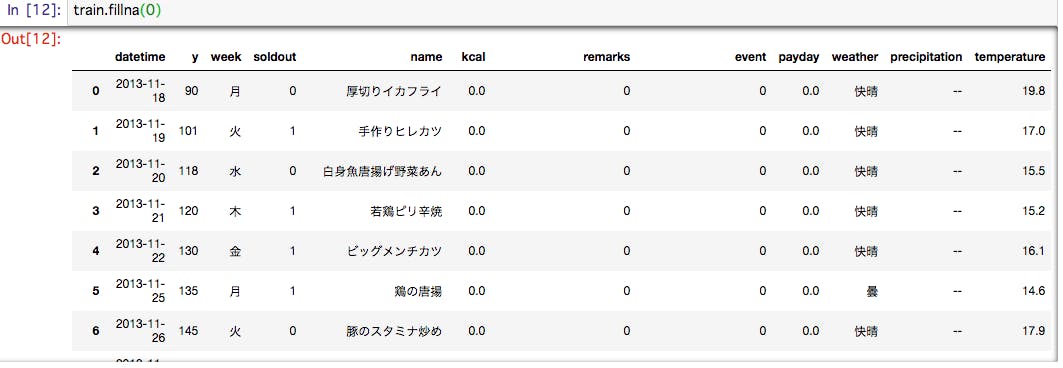
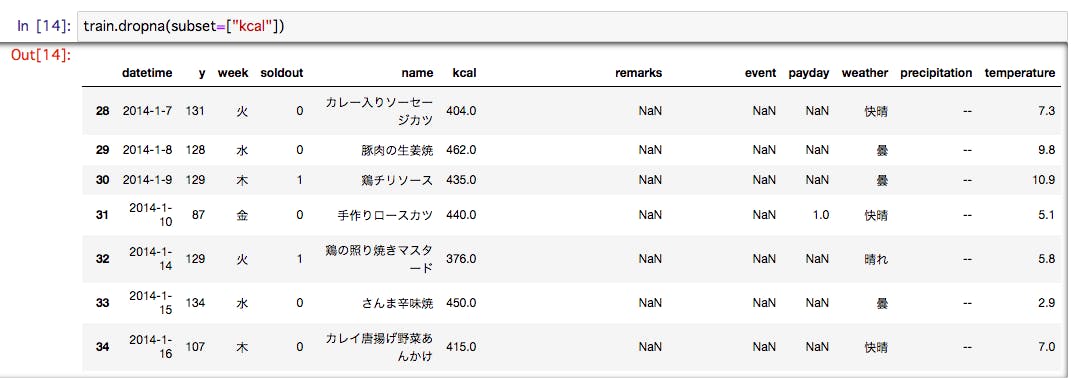
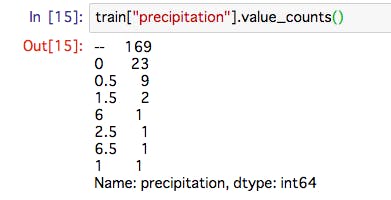
- 投稿日:2019-01-27T22:51:47+09:00
【matplotlib基本】動的グラフを書いてみる♬
音声アプリを作ろうとすると、グラフを動的に書きたくなった。そして、配置も工夫したいということでmatplotlibの使い方の基本を押さえることとした。
qiitaにも参考のような記事があり、ほぼまんまですが、動的というところが少し新しいかもです。【参考】
①matplotlibでのプロットの基本
②matplotlib基礎 | figureやaxesでのグラフのレイアウトやったこと
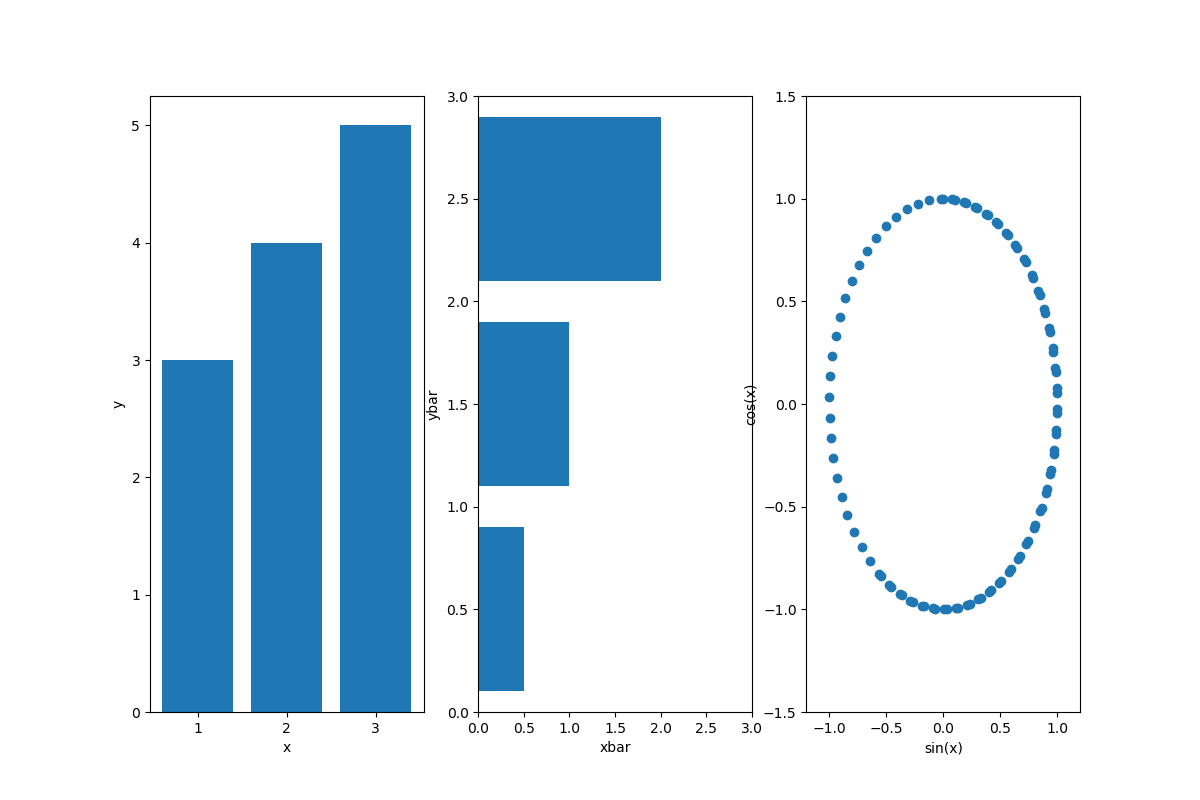
・グラフを書く
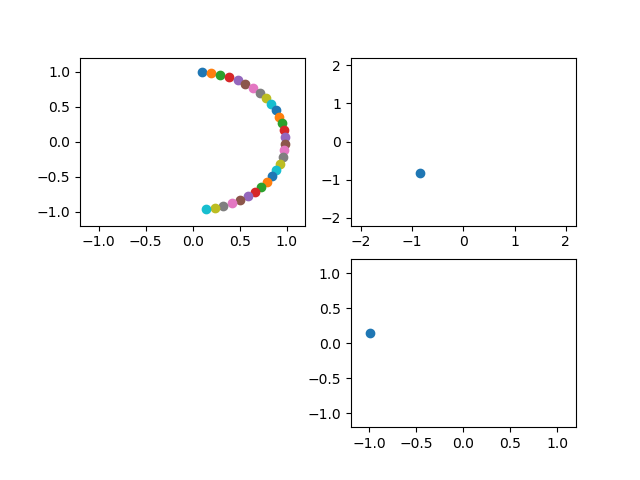
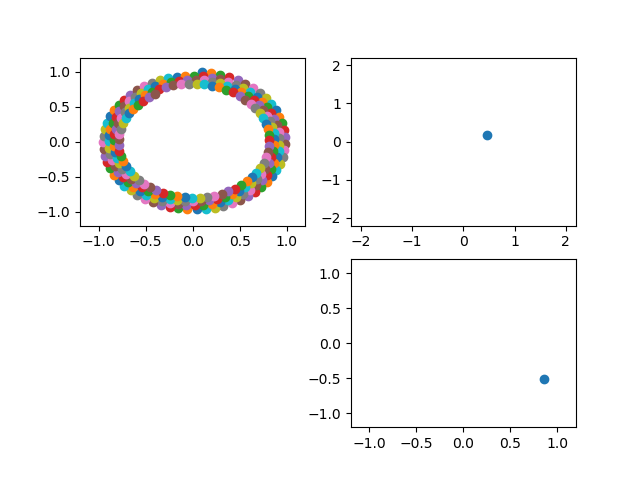
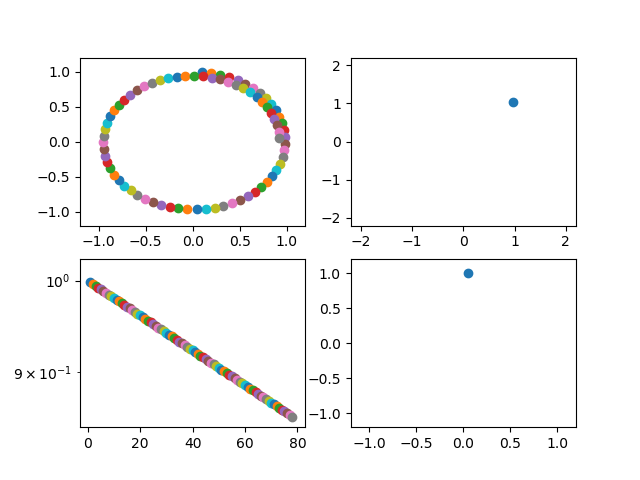
・動的グラフを書く・グラフを書く
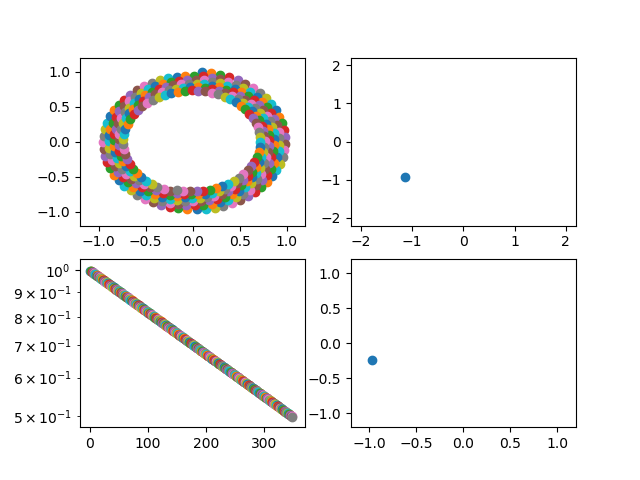
import matplotlib.pyplot as plt import numpy as np # データ生成 x = np.linspace(0, 10, 100) y1 = np.sin(x) y2 = np.cos(x) # プロット領域(Figure, Axes)の初期化 plt.figure(figsize=(12, 8)) fig1=plt.subplot(131) #配置は参考②のとおり fig2=plt.subplot(132) fig3=plt.subplot(133) # 棒グラフの作成 fig1.bar([1,2,3],[3,4,5]) fig1.set_xlabel("x") fig1.set_ylabel("y") fig2.barh([0.5,1.5,2.5],[0.5,1,2]) fig2.set_xlabel("xbar") fig2.set_ylabel("ybar") fig2.set_xlim(0,3) fig2.set_ylim(0,3) fig3.scatter(y1, y2) plt.xlabel("sin(x)") #pltのときはset_無しでplt.xlabelと記載 plt.ylabel("cos(x)") plt.xlim(-1.2,1.2) #pltのときはset無しでplt.xlimと記載 plt.ylim(-1.5,1.5) #fig3.set_xlabel("sin(x)") #もちろんこっちが正解だけど、上のようにも書ける #fig3.set_ylabel("cos(x)") plt.show()・動的グラフを書く
import matplotlib.pyplot as plt import numpy as np # プロット領域(Figure, Axes)の初期化 fig = plt.figure() ax1 = fig.add_subplot(221) ax2 = fig.add_subplot(222) ax3 = fig.add_subplot(224) ax1.axis([-1.2, 1.2, -1.2, 1.2]) # 棒グラフの作成 s = 1 while True: # ax1-3を削除する.ただし、ax1を残すと軌跡が見える #fig.delaxes(ax1) fig.delaxes(ax2) fig.delaxes(ax3) #plt.pause(0.01) #これ入れるとちかちかする #ax1 = fig.add_subplot(221) ax2 = fig.add_subplot(222) ax3 = fig.add_subplot(224) #ax1.axis([-1.2, 1.2, -1.2, 1.2]) ax2.axis([-2.2, 2.2, -2.2, 2.2]) ax3.axis([-1.2, 1.2, -1.2, 1.2]) y1 = np.sin(s/10)*np.exp(-s/1000) #減衰振動にしてみる y2 = np.cos(s/10)*np.exp(-s/1000) y3 = np.sin(s/10) #ゆっくりスムーズに動かしたいときは100とかで割る y4 = np.cos(s/10) ax1.scatter(y1, y2) ax2.scatter(y1+y4, y2+y3) #リサじゅーっぽいアプリにした ax3.scatter(y4, y3) plt.pause(0.001) #表示時間は短くすると動きは速い s += 1import matplotlib.pyplot as plt import numpy as np # プロット領域(Figure, Axes)の初期化 fig = plt.figure() ax1 = fig.add_subplot(221) ax2 = fig.add_subplot(222) ax3 = fig.add_subplot(224) ax4 = fig.add_subplot(223) #追記 ax1.axis([-1.2, 1.2, -1.2, 1.2]) # 棒グラフの作成 s = 1 while True: # ax1-3を削除する #fig.delaxes(ax1) fig.delaxes(ax2) fig.delaxes(ax3) #plt.pause(0.01) #これ入れるとちかちかする #ax1 = fig.add_subplot(221) ax2 = fig.add_subplot(222) ax3 = fig.add_subplot(224) #ax1.axis([-1.2, 1.2, -1.2, 1.2]) ax2.axis([-2.2, 2.2, -2.2, 2.2]) ax3.axis([-1.2, 1.2, -1.2, 1.2]) y1 = np.sin(s/10)*np.exp(-s/1000) y2 = np.cos(s/10)*np.exp(-s/1000) y3 = np.sin(s/10) y4 = np.cos(s/10) ax1.scatter(y1, y2) ax2.scatter(y1+y4, y2+y3) ax3.scatter(y4, y3) ax4.scatter(s,y1*y1+y2*y2) #追記 ax4.set_yscale("log")#y軸のscale追記 plt.pause(0.001) s += 1まとめ
・matplotlibの表記方法がゆらぎがあって迷うので、まとめておいた
・動的なグラフを作成してみた・ああ、すっきりした
- 投稿日:2019-01-27T22:07:23+09:00
python3 標準入力 まとめ
はじめに
python3 標準入力まとめです。
paizaの問題などでよく使いそうなものをまとめています。1行に要素1個
単語1個
入力aiueomain.pyhoge = input() print(type(hoge)) print(hoge)出力<class 'str'> aiueo整数1個
入力100main.pyhoge = int(input()) print(type(hoge)) print(hoge)出力<class 'int'> 1001行に要素n個
1行に複数要素ある場合。
要素間はスペース区切りを想定しています。単語n個を配列に格納
入力foo bar bazmain.pylist = [s for s in input().split()] print(type(list)) print(list)出力<class 'list'> ['foo', 'bar', 'baz']単語n個を各変数に格納
入力foo bar bazmain.pystr1, str2, str3 = [s for s in input().split()] print(type(str1)) print(str1) print(type(str2)) print(str2) print(type(str3)) print(str3)出力<class 'str'> foo <class 'str'> bar <class 'str'> fbaz整数n個を配列に格納
入力100 200 300main.pylist = [int(i) for i in input().split()] print(type(list)) print(list)出力<class 'list'> ['100', '200', '300']整数n個を各変数に格納
入力100 200 300main.pynum1, num2, num3 = [int(i) for i in input().split()] print(type(num1)) print(num1) print(type(num2)) print(num2) print(type(num3)) print(num3)出力<class 'int'> 100 <class 'int'> 200 <class 'int'> 300n行に要素m個
n行に整数m個
要素5つ, 3行の入力を想定しています。
入力5 3 10 11 12 13 14 20 21 22 23 24 30 31 32 33 34main.pyn, m = [int(i) for i in input().split()] table = [[int(i) for i in input().split()] for m in range(m)] print(type(n)) print(n) print(type(m)) print(m) print(type(table)) print(table)出力<class 'int'> 5 <class 'int'> 3 <class 'list'> [[10, 11, 12, 13, 14], [20, 21, 22, 23, 24], [30, 31, 32, 33, 34]]おわりに
しっかりしたエラー処理などはしていないため、使用の際は気をつけてください。。。
参考文献
- 投稿日:2019-01-27T21:23:09+09:00
Pythonを始めた時のメモ
はじめに
数年前にpythonを触り始めた時のメモをまとめた備忘録です。
※今思うとこの数年でたくさんのことを知ることができて嬉しいです。順不同に色々
Pythonを簡単に
pythonはインタープリタ言語で、コンパイルやリンクが必要出ないため、短時間でコーディングできる。対話的に(jupyter)利用することもでき、簡単な関数テストもできる。C言語との拡張も可能
引数
スクリプトと引数は、変数sys.argvとして渡される。引数がない時は、sys.argv[0]には起動時スクリプト名が渡される。-cコマンドの形式では、argv[0]は
'-c'となり、-mモジュールの時は、argv[0]は、モジュールのファイル名となる。環境変数設定
pythonを起動する際に、毎度同じコマンドを打つのが面倒なときに環境変数PYTHONSTARTUPに、起動時の実行コマンドを記載したファイルを指定することで、実行が可能。対話型のみで有効なのは注意。
複数の変数へ代入できる
>>> a = b = c = 123 >>> a 123 >>> b 123 >>> c 123 >>>複数の型の混合した計算
intはfloatに変換される
>>> 3*2 6 >>> 3*2.0 6.0 >>> 3.0*2.0 6.0 >>>複素数
虚数は「j」または「J」で表す
complex(実部,虚部)の関数でも作れる。>>> 3j*3j (-9+0j) >>> 3j*complex(0,3) (-9+0j) >>> 3.0j*complex(0,3.0) (-9+0j) >>>複素数は常に実部、虚部が浮動小数点数で表される。
.realと.imagで取り出すことができる。>>> a = 2+3J >>> a.real 2.0 >>> a.imag 3.0 >>>複素数は実数に変化する正しい方法が存在しないため、int()やfloat()での変換はできない。
大きさを求める場合には、abs()が利用できる。一応計算式も。>>> a = 2+3J >>> a.real 2.0 >>> a.imag 3.0 >>> float(a) Traceback (most recent call last): File "<stdin>", line 1, in <module> TypeError: can't convert complex to float >>> >>> abs(a) 3.605551275463989 >>> >>> import math >>> math.sqrt(a.real**2+a.imag**2) 3.605551275463989アンダースコア
最後に計算された式は、アンダースコアに代入されている。
>>> 3+2 5 >>> 3+_ 8文字列結合
当たり前の結合。注目すべきはシングルクオートであること。
>>> a = "hello" >>> b = "world" >>> a+b 'helloworld'文字列の中に、シングルクオートが含まれていると、最後がダブルクオートになる。
>>> c = "I've said " >>> c+a+b "I've said helloworld"print()を使うとクオートが無くなる。
>>> print(c+a+b) I've said helloworld並列で置くだけでも結合される
>>> "hello" "world" 'helloworld'あくまでリテラル同士であることが大事
>>> "hello".strip() "world" File "<stdin>", line 1 "hello".strip() "world" ^ SyntaxError: invalid syntaxこういう時は演算子使う
>>> "hello".strip()+"world" 'helloworld'スライスを作る
基本はこれ。
>>> a = "Helloworld" >>> len(a) 10 >>> a[0] 'H' >>> a[1] 'e' >>> a[5] 'w' >>> a[9] 'd'負の数だとこう。
>>> a[-1] 'd' >>> a[-1:] 'd' >>> a[-5:] 'world'注意が必要なのは「0」と「−0」。全く同じです。
>>> a[0] 'H' >>> >>> a[-0] 'H'スライスを作るときの考え方も簡単
+---+---+---+---+---+---+ | H | e | l | l | o | w | +---+---+---+---+---+---+ 0 1 2 3 4 5 6 -6 -5 -4 -3 -2 -1スライスは第一のデフォルトが0で、第二が文字列のサイズになっています。
不適切なものが入ると空文字列が返ってきます。>>> a[0:3] 'Hel' >>> a[3:] 'loworld' >>> a[:100] 'Helloworld' >>> a[5:1] '' >>> a[-1:-5] '' >>> a[10:] '' >>> a[10] Traceback (most recent call last): File "<stdin>", line 1, in <module> IndexError: string index out of rangeC言語とは異なり、文字列は変更不能です。新しい文字列を生成することで同じようにできます。
>>> a[:4]="goodmorning" Traceback (most recent call last): File "<stdin>", line 1, in <module> TypeError: 'str' object does not support item assignment >>> >>> "goodmorning"+a[5:] 'goodmorningworld'リストの操作
リストは[,]角括弧のカンマ区切りで、変更が可能です。型は同じでなくても問題ありません。
pop()やremove()やcopy()を使わない操作
>>> a=["hiro","yuki",33,[175,"80kg"]] >>> a ['hiro', 'yuki', 33, [175, '80kg']] >>> >>> a[0:2] ['hiro', 'yuki'] >>> a[0:2] = [] >>> a [33, [175, '80kg']] >>> a[0] = "hiro" >>> a ['hiro', [175, '80kg']] >>> a[1:1]="yuki" >>> a ['hiro', 'y', 'u', 'k', 'i', [175, '80kg']] >>> a[1]="yuki" >>> a ['hiro', 'yuki', 'u', 'k', 'i', [175, '80kg']] >>> a[:0]=a >>> a ['hiro', 'y', 'u', 'k', 'i', 'yuki', 'u', 'k', 'i', [175, '80kg'], 'hiro', 'y', 'u', 'k', 'i', 'yuki', 'u', 'k', 'i', [175, '80kg']]forやwhileの中でのリスト
反復対象のシーケンスを、ループの中で変更を加えるときには、リストのスライスコピーを作る必要がある。
以下では、無限ループがおきます。
>>> a = ["bbb","ccccc","ddddddd","eeeeeeeee"] >>> for x in a: ... if len(x)>6: ... a.insert(0,x) >>>スライスコピーすれば、元のリストは安全です。
>>> a = ["bbb","ccccc","ddddddd","eeeeeeeee"] >>> for x in a[:]: ... if len(x)>6: ... a.insert(0,x) >>>range()について
終端値がリストに入らないのは周知ですね。
他の記事も書きましたが、出力の仕方が大事です。>>> range(10) range(0, 10) >>> print(range(10)) range(0, 10) >>> list(range(10)) [0, 1, 2, 3, 4, 5, 6, 7, 8, 9] >>> list(range(0,10)) [0, 1, 2, 3, 4, 5, 6, 7, 8, 9] >>> list(range(0,10,2)) [0, 2, 4, 6, 8]関数の実行と参照について
関数が実行されると、関数内のローカルのシンボリックリンクに格納されます。
変数の参照は
・ローカルのシンボリックリンク
・グローバルのシンボリックリック
・ビルトインのシンボリックリンク
を調べるようになっています。そのため関数内では、グローバル変数は,グローバル文を使わない限りは、参照しかできないようになっています。
また関数のデフォルト値は、関数を定義した時点で定義を行なっているスコープで評価されます。
キーワード引数について
>>> def intro(name,age = 33,weight = 80,height = 175): ... print("my name is",name) ... print("my age is",age) ... print("my weight is",weight) ... print("my height is",height) ... >>> intro("hiro") my name is hiro my age is 33 my weight is 80 my height is 175 >>> intro(name = "hiro",age = 35) my name is hiro my age is 35 my weight is 80 my height is 175引数が足りないと以下のようになる
>>> intro() Traceback (most recent call last): File "<stdin>", line 1, in <module> TypeError: intro() missing 1 required positional argument: 'name' >>> intro(age = 33) Traceback (most recent call last): File "<stdin>", line 1, in <module> TypeError: intro() missing 1 required positional argument: 'name'引数の重複やキーワード引数の後にキーワードでないものを置いた場合、定義されていないキーワード。
要は
引数のリストでは、位置指定型引数を先に、キーワード引数を後に、コール時には仮引数を使う
必要がある>>> intro("hiro",name = "yuki") Traceback (most recent call last): File "<stdin>", line 1, in <module> TypeError: intro() got multiple values for argument 'name' >>> intro(name = "hiro",35) File "<stdin>", line 1 SyntaxError: positional argument follows keyword argument >>> intro(name = "hiro","yuki") File "<stdin>", line 1 SyntaxError: positional argument follows keyword argument >>> intro(area = "chiba") Traceback (most recent call last): File "<stdin>", line 1, in <module> TypeError: intro() got an unexpected keyword argument 'area'ドキュメンテーション文字列(docstirng)
・一行目にオブジェクトの目的を簡潔に書く
・大文字始まりで、ピリオド終わり
・複数行の場合は2行目は空行リストの操作
list.remove(x):xである最初のアイテムを削除する。該当がなければエラー
list.pop(i):指定された位置のアイテムをリストから削除し。消したアイテムを返す。位置が指定されない時は、最後のアイテムを削除する。
list.sort():インプレース(元のリストのコピーを取らずに)でソートします。reverse()は逆順。
del list[i]:pop()と似ているがスライスを使えるところが便利。値は返さない。シーケンスのアンパッキング
シーケンスと同等の長さの変数のリストがあればOK
>>> a = ("a","b","c") >>> a ('a', 'b', 'c') >>> x,y,z = a >>> x 'a'keyvalueペアになっているタプルからなるリストは辞書に簡単に変換できる。
>>> a [('A', 1), ('B', 2), ('C', 3)] >>> dict(a) {'A': 1, 'B': 2, 'C': 3}zip()を使えば、forループを使うときに、簡単にペアにできる
>>> a = ["a","b","c"] >>> b = ["x","y","z"] >>> for i,v in zip(a,b): ... print(i+"と"+v+"はセット") ... aとxはセット bとyはセット cとzはセットreversed()やsorted()を使うと簡単に順番を変えられる。
>>> for i,v in zip(a,reversed(b)): ... print(i+"と"+v+"はセット") ... aとzはセット bとyはセット cとxはセット条件についてmemo
inとnot inははシーエンスに存在するかどうかのチェック
isとnot isは完全に同一であるかどうか>>> a = ["a","b","c"] >>> a ['a', 'b', 'c'] >>> b = a >>> b ['a', 'b', 'c'] >>> a == b True >>> a is b True >>> c = a.copy() >>> c ['a', 'b', 'c'] >>> c == a True >>> c == b True >>> c is a False >>> c is b False比較演算子は連結できる
aがbより小さく、かつbがcに等しい
a < b == cブール演算子は、比較演算子よりも優先順位は低いことは、とても重要。
ブール演算子の中では、not>and>orの順位になります。以下では 「aでありbではないもの」もしくは「c」であるもの
a and not b or cオブジェクトの比較
>>> 0 == 0.0 True >>> 0 is 0.0 False一旦ここまで
メモはまだ1/5くらい、また更新します。
- 投稿日:2019-01-27T21:06:23+09:00
Djangoのurls.pyに書くpath()関数の引数の意味について調べた(不明点あり)
Djangoでアプリを作ろうとするときに、通ることを避けられない、
urls.py。
そして、その中で使うpath関数。チュートリアルではフンフンと流していましたが、実際のところ、どんなものを入力すれば良いのか、調べてみました。
(例)
myapp.urls.pyurlpatterns = [ path('admin/', admin.site.urls), path("bbs/", include("bbs.urls")), path("", RedirectView.as_view(url="/bbs/")) ]path関数の引数
各pathの第一引数("admin/"等)に対応するurlへのリクエストが来た場合に、第二引数に対応するviewsやを呼び出します。
pathは、公式ドキュメントによると、以下の引数を取ります。
https://docs.djangoproject.com/ja/2.1/ref/urls/第一引数は、"admin/"等のroute、
第二引数は、viewを取る、
ということです。viewを取る、というのは、どういうことかというと、
The view argument is a view function or the result of as_view() for class-based views. It can also be an django.urls.include().
つまり、引数viewは、以下のいずれかを取る、ということのようです。
① view関数
② class-based viewsのas_view()の出力
③ include()関数上の例でいうと、
include("bbs.urls")は③、
RedirectView.as_view(url="/bbs/")は②、
ということになります。
admin.site.urlsは、一見、どこにも当てはまりません・・・。
以下のように、URLPattern型のオブジェクトのリストが帰ります。
まあ、最初から入っている特別なオブジェクト、ということで良いのかな・・。>>python manage.py shell >>> admin.site.urls ([<URLPattern '' [name='index']>, <URLPattern 'login/' [name='login']>, <URLPattern 'logout/' [name='logout']>, <URLPattern 'password_change/' [name='password_change']>, <URLPattern 'password_change/done/' [name='password_change_done']>, <URLPattern 'jsi18n/' [name='jsi18n']>, <URLPattern 'r/<int:content_type_id>/<path:object_id>/' [name='view_on_site']>, <URLResolver <URLPattern list> (None:None) 'auth/group/'>, <URLResolver <URLPattern list> (None:None) 'auth/user/'>, <URLResolver <URLPattern list> (None:None) 'bbs/article/'>, <URLPattern '^(?P<app_label>auth|bbs)/$' [name='app_list']>], 'admin', 'admin')
- 投稿日:2019-01-27T21:05:35+09:00
画面に好きな文字を表示してみよう
前回:
次回:
目次:https://qiita.com/New_enpitsu_15/private/479c69897780cabd01f4Hello world
プログラミングを始める第一歩。
まずは、適当なファイル名.pyというファイルを作って
適当なテキストエディタで開いてみましょう。そしたら、
file.pyprint("Hello world") input()と書いて、保存。実行してみてください。(ファイルをダブルクリックすれば実行されます。)
表示されたら、あなたはもうパイソニアです。
これから解説サイトを片手にプログラムを楽しんでいきましょう。ナポリタンが食べたい
ひとまず、先ほどのファイルを表示してください。
file.pyprint("ナポリタンが食べたい") input()と書き換えて実行してみましょう。
ナポリタンが食べたいと画面に表示されたはずです。では、最後にあなたの好きな食べ物の名前を表示してみましょう。
できましたか?わからなかったらコメントで聞いてくださいね。解説
print()
()内の文字を表示します。
例えばprint("ナポリタン")だったら、"ナポリタン"と表示されるはず…
いいえ、ナポリタンと表示されます。なぜでしょう?それは、ダブルクオーテーション
"とシングルクオーテーション'は文字列を表す文字だからです。詳しくは次回説明するので、今は
""か''で囲わなければならないのだと思っておいてください。input()
input()という関数は私たち(ユーザー)からの入力を待つ関数です。
試しに、print(input("入力:")) input()というファイルを作り、実行してみてください。
黒い画面に入力した文字が、もう一度表示されましたね。私たち(ユーザー)からの入力がprintに文字列として渡されました。
ですが、最後の行の
input()は何でしょう?
これは、画面をとどめておくために書いているものです。では試しにprint("Hello world")だけで実行してみましょう。どうですか?
「実行できない」、「黒い画面が出てすぐ消えてしまう」という感想を持ったでしょう。
これは、print("Hello world")を実行したらすぐにプログラムが終了して画面が閉じてしまうからです。そのため、
input()でユーザーからの入力を待つことにより一旦処理を停止して、画面が閉じないようにしているのです。次回は…
文字列と数字について説明します。
- 投稿日:2019-01-27T20:58:45+09:00
Amazon Comprehendをpythonから実行
はじめに
AmazonComprehend(AWSの自然言語処理サービス)のAPIを、Python3から実行してみました。幾つかの記事を参考にさせて頂きましたが、一つにまとまっているものはなかったので、それらをまとめてみました。
環境
MacOS Sierra 10.12.6
python 3.7.0インストール
$ sudo pip install awscli次に、AWSアクセスキーを取得する。
参考サイト:
- インストール https://qiita.com/reflet/items/e4225435fe692663b705
- AWSアクセスキー https://aws.amazon.com/jp/developers/access-keys/
AWSアクセスキーは、リクエストを正常に AWS API に送信するために必要、とのことです。
AWSアクセスキーの取得
以下の記事を参照してください。
https://qiita.com/miwato/items/291c7a8c557908de5833プログラムの実行
以下の記事にサンプルコードや結果がまとめられています。
https://dev.classmethod.jp/cloud/comprehend-operations-using-python-boto3-ja/感想
Qiita初投稿、ということもあり、至らぬ点やアドバイスいただけると幸いです。
また参考にさせて頂いた記事の著者の皆様、ありがとうございました。
- 投稿日:2019-01-27T19:26:13+09:00
AWS SageMakerでモデルのデプロイから推論(バッチ変換ジョブ)までを行う
本記事でやること
- 前回の記事で行なったトレーニングジョブ結果から生成されたモデルのデプロイを行う。
- S3に置いてあるデータをまとめて推論するバッチ変換ジョブを実行する。
本記事でやらないことは以下の通りなので、他の記事を参照してください。
- 前回の記事で行なった独自アルゴリズムを使ったトレーニングジョブの実行方法
- SageMakerやS3などにアクセスするためのIAMロールの作成
対象読者
- とりあえずAWS SageMakerを動かしてみたい人
- Dockerに関して一通りの基礎知識がある人
使用言語
- Python 3.6.3
トレーニングジョブ結果から生成されたモデルのデプロイを行う
公式ドキュメントに記載がある通り、推論を行う際にアプリケーションがリクエストを送るエンドポイントは、モデルをデプロイした際に取得されます。なので、トレーニングジョブが完了された後にモデルのデプロイを必ず行う必要があります。
以下、コードからモデルのデプロイを行います。
class SagemakerClient: def __init__(self): self.client = Session(profile_name="hoge").client("sagemaker", region_name="ap-northeast-1") def create_model(self, model_data_url): model_params = { "ExecutionRoleArn": "arn:aws:iam::123:role/dev-sagemaker", "ModelName": "sample-model", # モデル名 "PrimaryContainer": { "Image": "123.dkr.ecr.ap-northeast-1.amazonaws.com/sagemaker-repo:latest", # ECRにプッシュしたイメージURL "ModelDataUrl": model_data_url # モデルデータが格納されているS3のパス } } self.client.create_model(**model_params) if __name__ == '__main__': model_data_url = ... SagemakerClient().create_model(model_data_url)上記コードでの
create_modelメソッドの引数(model_data_url)は、boto3のdescribe_training_jobから得られるのでこちらの公式ドキュメントをご参照ください。参考程度に
model_data_urlを取得するコードを以下に記載いたします。また、describe_training_jobの引数TrainingJobNameはトレーニングジョブを送信する際に返ってくるTransformJobArnから取得をしています。client = Session(profile_name="hoge").client("sagemaker", region_name="ap-northeast-1") model_data_url = client.describe_training_job(TrainingJobName=training_job_name)['ModelArtifacts']['S3ModelArtifacts']以下のように
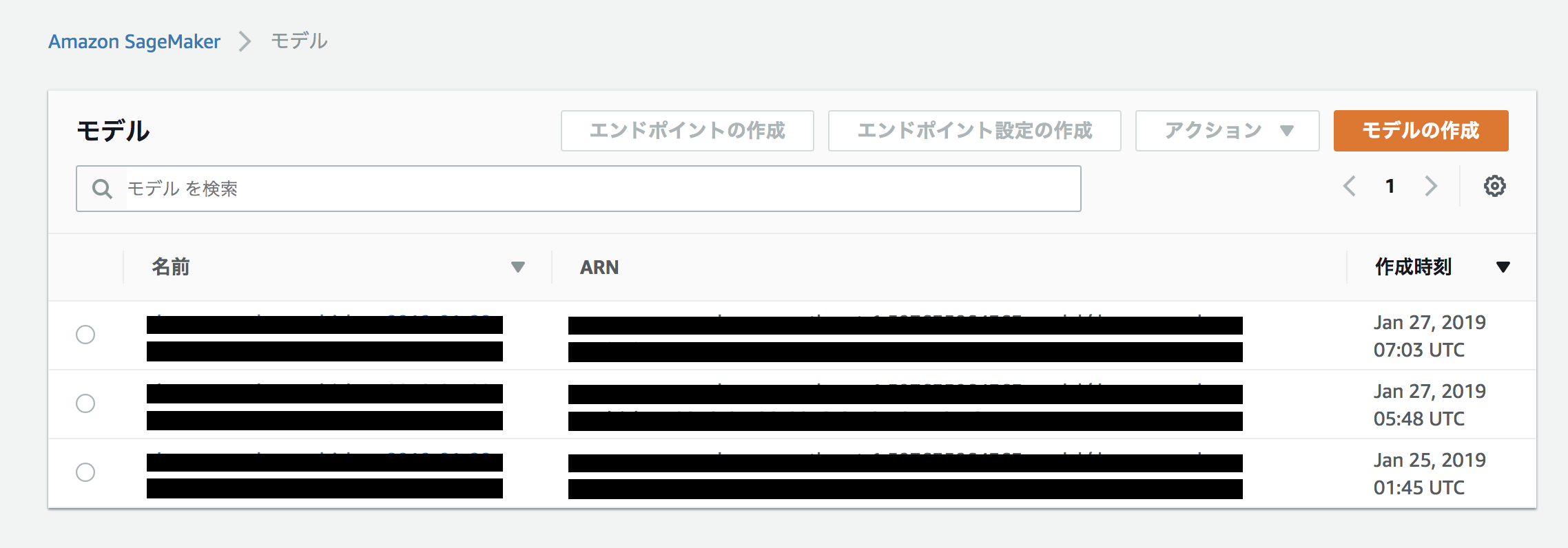
Amazon SageMaker > モデルにモデルが現れていれば無事完了です。S3に置いてあるデータをまとめて推論するバッチ変換ジョブを実行
今回、推論時に使用したデータや推論結果の出力先などは以下のようなフォルダ構成でS3に置きました。
bucket ├── input-data-prediction │ └── YYYY-MM-DD │ └── multiclass │ └── iris.csv ├── output-data-prediction │ └── YYYY-MM-DD │ └── multiclass │ ├── input-data-training │ └── YYYY-MM-DD │ └── multiclass │ └── iris.csv └── output-model └── YYYY-MM-DD └── multiclass └── model名(←ここからはSageMakerが出力する)以下のコードからバッチ変換ジョブを送信します。
ジョブを送信する際の引数であるModelNameは、デプロイしたモデル名を使用します。
デプロイしたモデル名は、boto3のlist_modelsメソッドで得られるのでこちらの公式ドキュメントをご参照ください。(モデル名に含まれている文字列とデプロイした時間からしか検索ができないみたいです)class SagemakerClient: def __init__(self): self.client = Session(profile_name="hoge").client("sagemaker", region_name="ap-northeast-1") def submit_transform_job(self): model_name = self.client.list_models( NameContains="base_model_name", # 各自デプロイしたモデル名に含まれている文字列 SortOrder='Descending', SortBy='CreationTime')["Models"][0]["ModelName"] transform_params = { "TransformJobName": "sample-transform_job_name", # バッチ変換ジョブ名 "ModelName": model_name, # デプロイしたモデル名 "MaxConcurrentTransforms": 2, "MaxPayloadInMB": 50, "BatchStrategy": "MultiRecord", "TransformOutput": { "S3OutputPath": "s3://bucket/output-data-prediction/YYYY-MM-DD/multiclass/" # 推論結果を格納するS3パス }, "TransformInput": { "DataSource": { "S3DataSource": { "S3DataType": "S3Prefix", "S3Uri": "s3://bucket/input-data-prediction/YYYY-MM-DD/multiclass/" # 推論を行うインプットデータが格納されているS3パス } }, "ContentType": "text/csv", "SplitType": "Line" }, "TransformResources": { "InstanceType": "ml.c4.xlarge", "InstanceCount": 1 } } self.client.create_transform_job(**transform_params) if __name__ == '__main__': SagemakerClient().submit_transform_job()以下のように
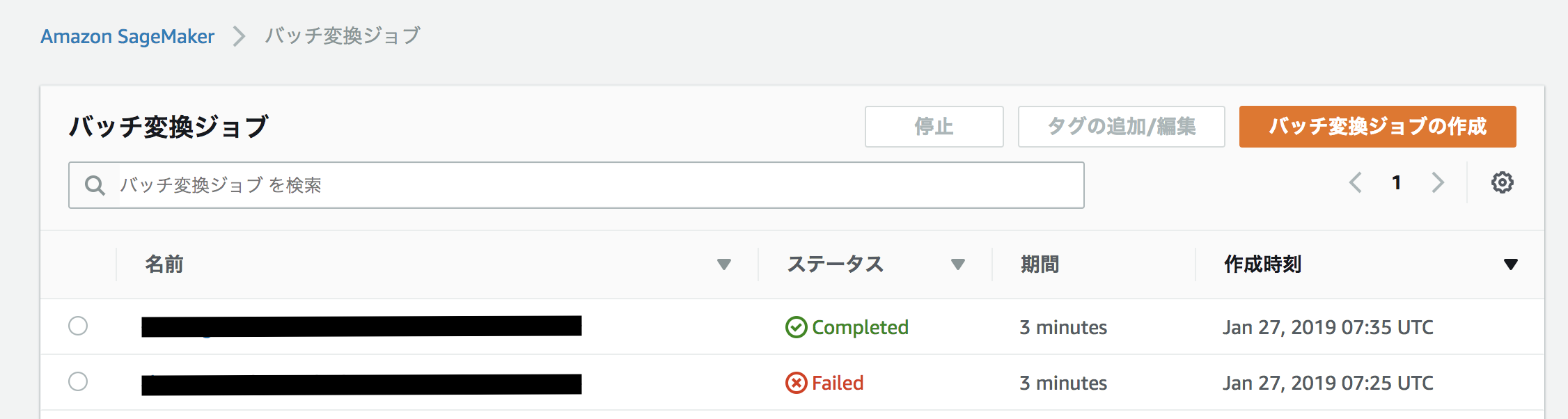
Amazon SageMaker > バッチ変換ジョブでステータスがCompletedになれば無事完了です。上記で指定したS3のパスに推論結果が格納されているはずです。終わりに
今回は、モデルをデプロイ~バッチ変換ジョブを送信するコードのみを記載しましたが、SageMakerのDockerコンテナ内にある
predictor.pyやserveは公式のgithubにあるサンプルをそのまま使用しています。
- 投稿日:2019-01-27T19:17:03+09:00
只今勉強中! 機械学習ライブラリがやっていること
・はじめに
Kerasを使ったディープラーニングを使いこなすにあたって必須知識のメモです。
・次元(ベクトルとテンソル)
・ベクトルデータ
1. 2次元テンソル
(samples, features)
例:生命表データセット
- ・データ数:10万件
- ・生命表(年齢、国、収入)
- ・テンソル:(100000, 3)
samples age country annual income 1 25 jp 680 2 34 usa 1200 3 53 china 7000 ... 42 usa 1500 100000 43 korea 650 2. 3次元テンソル
(samples, timesteps, features)
例:株価のデータセット(1分毎の最高値と最安値を360日分用意)
- ・データ数:360件(360日分)
- ・取引時間:390分
- ・データ(1分毎, 最高値、最安値)
- ・テンソル:(360, 3, 390)timesteps: 390
※取引時間/1分毎
samples max min 1 20000 19900 2 20250 19950 3 20170 19800 ... 21330 21100 360 23500 23300 3. 4次元テンソル
(samples, height, width, channels) または(samples, channels, height, width)
※TensorFlowは前者(チャネルラスト)を採用、Theanoは後者(チャネルファースト)を採用。例:画像のデータセット
- ・データ数:10000件
- ・データサイズ:{"height" : "300px", "width" : "250px"}
- ・チェンネル数:3 ※RGB
- ・テンソル:(10000, 300, 250, 3)3. 5次元テンソル
(samples, frames, height, width, channels) または(samples, frames, channels, height, width)
例:Youtubeの1分間動画(4FPS、144×256)のビデオクリップのサンプリング
- ・データ数:動画10本
- ・フレーム数:240※60 × 4
- ・チャンネル数:3
- - ・テンソル:(10, 240, 144, 256, 3)4. MNISTの概要
1. (train_images, train_labels), (test_images, test_labels) = mnist.load_data()
・訓練データと訓練用ラベル、テストデータをテスト用ラベルを取得
- 訓練データの中身はこんな感じ※サイズ28×28の画像データが60000件
(60000, 28, 28)
- 訓練用ラベルの中身
(2, 1, 3, 9, 7...)
データとラベルは対になっている。
samples labels 1 2 2 1 3 3 ... 8 60000 0 2. train_images = train_images.reshape(60000, 28 * 28))
・データを2次元テンソル形式に変換
3. train_images = train_images.astype('float32') / 255
・データを8ビット整数値から32ビットfloat型へ変換
4. network = models.Sequential()
ニューラルネットワークモデルを初期化
5. network.add(layers.Dense(512, activation='relu', input_shape=(28 * 28, )))
密結合ネットワークを実装
layers.Dense(出力数, 演算タイプ, 入力数) : 2次元テンソルで活用される密結合モデル
演算タイプ :入力テンソルから出力テンソルへ変換する際の演算タイプを指定
入力数 : テンソル数を指定6. network.add(layers.Dense(10, activation='softmax'))
密結合ネットワークを実装
※入力数は省略可7. network.compile(optimizer='rmsprop', loss='categorical_crossentropy', metrics=['accuracy'])
optimizer : 最適化関数
入力パラメータ(重み)の値を最適化していく
ニューラルネットワークモデルそのものが最適化されていくloss : 損失関数
目的値と予想値から損失率を割り出すための導関数
損失率=0に近づく値を探していくmetrics : 訓練とテストを監視するための指標
評価基準の定義8. network.fit(train_images, train_labels, epochs=5, batch_size=128)
128サンプルの小分けされたバッチを用いて訓練データの学習(イテレーション)を5回繰り返す。
※訓練データ全体に渡るイテレーションはエポックと呼ばれる。
イテレーションごとにネットワークはバッチごとの損失値から重みの勾配を計算し、重みを更新していく。
これを5エポック繰り返す。(※各エポックで469回、重みを更新、全体では2345回の値を更新)
[もう少し解説]
1. 60000件のデータを128件ごとに損失率をだし、最適化を行う
2. 上記1を5回繰り返す
- 投稿日:2019-01-27T18:32:41+09:00
Strawberry Fieldsで光量子計算をする(その4) ボゾンサンプリング
$$
\def\bra#1{\mathinner{\left\langle{#1}\right|}}
\def\ket#1{\mathinner{\left|{#1}\right\rangle}}
\def\braket#1#2{\mathinner{\left\langle{#1}\middle|#2\right\rangle}}
$$ボゾンサンプリングとは
例によって、主に公式文献のp.18に従って学んでいきます。
もう少し詳細な文献だとこれとか良さそうです。ボゾンサンプリングはパッシブな線形光学素子だけで構成可能な、比較的シンプルな量子計算機です。
ただしそれゆえにユニバーサルな量子計算機にはなり得ないです。
では何が嬉しいかと言うと、ボゾンサンプリングはPermanent関数の結果を吐き出します。
ただし現実的に有用かというと実はそうでもないようです。そこで今回は、このような流れで書いていこうと思います。
- Permanent関数とは
- ボゾンサンプリングの実装
- ボゾンサンプリングの解析的計算
- 実際のところ役に立つの?Permanent関数とは
Permanent関数は次のような数式で書けます。
$$
\mathrm{Perm(A)}= \sum_{\sigma \in S_n} \prod_{i=1}^{n} a_{i,\sigma (i)}
$$$A$は$a_{i,j}$を要素にもつ$n \times n$行列で、$S_n$はn個の数字で可能な順列の集合です。
例えば、
$S_3 = $ { $(1,2,3),(1,3,2),(2,1,3),(2,3,1),(3,1,2),(3,2,1)$ }
です。簡単に$A=2\times 2$行列の場合を考えると、
$\mathrm{Perm(A)}=a_{11} a_{22} + a_{12} a_{21}$
です。
$n=3$以降も計算するとわかるのですが、行列式と非常に似ています。
ただし行列式は各項の符号が偶置換or奇置換で変わりますが、Permanentは符号が変わりません。応用例として、n人でランダムにプレゼント交換する場合に、誰も自分が持ってきたプレゼントを引き当てないような組み合わせの数を求めることができるようです。(リンク先の"derangement problem")
Permanent関数は古典計算だと最も効率的なアルゴリズムでも $O(2^{n-1} n^2)$ の計算量が必要だそうで、nについて指数関数的な計算リソースが必要です。よってもしこれを量子計算で効率的に求められれば量子計算の優位性を示すことができます。
ボゾンサンプリングの実装
ここはほとんど公式文献のまま、Jupiter notebook 実装ですが、入力光子数状態をランダム生成できるようにコードを追加しています(コメントアウトしてますが)。
import numpy as np import strawberryfields as sf import random as rd from strawberryfields.ops import * from numpy.linalg import multi_dot from scipy.linalg import block_diag import math NodeNum = 4 SumOfPhoton = 3 # 入力光子数状態(FockList_ini)をランダムに生成 ''' FockList_ini = np.zeros(4, dtype=int) for i in range(SumOfPhoton): tmp = rd.randint(0, NodeNum-1) FockList_ini[tmp] += int(1) ''' # 1つのポートに2光子以上存在しない場合 ''' arr = np.arange(NodeNum) np.random.shuffle(arr) for i in range(SumOfPhoton): FockList_ini[arr[i]] += int(1) ''' FockList_ini = [1,1,0,1] eng, q = sf.Engine(4) with eng: # prepare the input fock states Fock(FockList_ini[0]) | q[0] Fock(FockList_ini[1]) | q[1] Fock(FockList_ini[2]) | q[2] # vacuum state (optional) Fock(FockList_ini[3]) | q[3] # rotation gates Rgate(0.5719) Rgate(-1.9782) Rgate(2.0603) Rgate(0.0644) # beamsplitter array BSgate(0.7804, 0.8578) | (q[0], q[1]) BSgate(0.06406, 0.5165) | (q[2], q[3]) BSgate(0.473, 0.1176) | (q[1], q[2]) BSgate(0.563, 0.1517) | (q[0], q[1]) BSgate(0.1323, 0.9946) | (q[2], q[3]) BSgate(0.311, 0.3231) | (q[1], q[2]) BSgate(0.4348, 0.0798) | (q[0], q[1]) BSgate(0.4368, 0.6157) | (q[2], q[3]) # end circuit # not performing measurement # run the engine state = eng.run('fock', cutoff_dim=8) # extract the joint Fock probabilities probs = state.all_fock_probs() # print the joint Fock state probabilities print(probs[1,1,0,1])出力結果は、出力側光子検出器で検出されたフォトン数が{1,1,0,1}となる確率を示しています。
当然、光子数は入出力間で保存します。0.17468916048563937RゲートとBSゲートの結合は1つの$4 \times 4$ユニタリゲート$U$と等価で、入力ポート$i$のフォトンが出力ポート$j$から出てくる確率は$U_{ij}$で表せます。
この結果がPermanent関数と結びつく直感的な説明はこれのp.5がわかりやすいです。例えば、入力ポート1,2に1つずつ存在したフォトンが、出力ポート3,4から1つずつ検出される確率は以下のように書けますね。
$$
U_{13} U_{24} + U_{14} U_{23}
$$
上の$2\times 2$行列のPermanent関数と見比べると、これは$4 \times 4$ユニタリゲート$U$を上手く整形してPermanent関数に渡してやれば出てきそうな気がしませんか?ボゾンサンプリングの解析的計算
出力光子数状態${n_{1},n_{2},…,n_{N}}$が測定される確率は以下の通りです。
$$
\braket{ n_{1},n_{2},…,n_{N}}{ \psi^{'}} ^{2} =
\frac{|\mathrm{Perm}(U_{st})|^{2}}{m_{1}!m_{2}!,…,m_{N}!n_{1}!n_{2}!,…,n_{N}!}
$$これを計算する実装例は以下の通りです。
元ネタとより詳細な説明はこちらになります。def transform(photonNumList): resList = [] for i in range(len(photonNumList)): for j in range(photonNumList[i]): resList.append(int(i)) return resList # Permanent関数を求める古典アルゴリズム def perm(M): n = M.shape[0] d = np.ones(n) j = 0 s = 1 f = np.arange(n) v = M.sum(axis=0) p = np.prod(v) while (j < n-1): v -= 2*d[j]*M[j] d[j] = -d[j] s = -s prod = np.prod(v) p += s*prod f[0] = 0 f[j] = f[j+1] f[j+1] = j+1 j = f[0] return p/2**(n-1) def Fact_Fock(FockList_ini, FockList_after): FockList_ini_Fact = [np.math.factorial(i) for i in FockList_ini] FockList_after_Fact = [np.math.factorial(i) for i in FockList_after] res = 1 for i in range(len(FockList_ini)): res *= FockList_ini_Fact[i] for i in range(len(FockList_after)): res *= FockList_after_Fact[i] return res # RゲートとBSゲートを結合し、1つのユニタリゲートで記述する ################### Uphase = np.diag([np.exp(0.5719*1j),np.exp(-1.9782*1j),np.exp(2.0603*1j),np.exp(0.0644*1j)]) BSargs = [ (0.7804, 0.8578), (0.06406, 0.5165), (0.473, 0.1176), (0.563, 0.1517), (0.1323, 0.9946), (0.311, 0.3231), (0.4348, 0.0798), (0.4368, 0.6157) ] t_r_amplitudes = [(np.cos(q), np.exp(p*1j)*np.sin(q)) for q,p in BSargs] BSunitaries = [np.array([[t, -np.conj(r)], [r, t]]) for t,r in t_r_amplitudes] # list of 2x2 numpy array UBS1 = block_diag(*BSunitaries[0:2]) UBS2 = block_diag([[1]], BSunitaries[2], [[1]]) UBS3 = block_diag(*BSunitaries[3:5]) UBS4 = block_diag([[1]], BSunitaries[5], [[1]]) UBS5 = block_diag(*BSunitaries[6:8]) U = multi_dot([UBS5, UBS4, UBS3, UBS2, UBS1, Uphase]) ################### # 出力側光子検出器の検出結果(FockList_after)をランダムに生成 ########### FockList_after = np.zeros(4, dtype=int) for i in range(SumOfPhoton): tmp = rd.randint(0, NodeNum-1) FockList_after[tmp] += int(1) ########### # 任意の値を使用する場合 FockList_after = [1,0,1,1] print("Input photon number states") print(list(FockList_ini)) print("Measured photon number states") print(list(FockList_after), '\n') # ボゾンサンプリングの出力を解析的に計算 div = Fact_Fock(FockList_ini, FockList_after) ini_vec = transform(FockList_ini) after_vec = transform(FockList_after) clasic_calc = np.abs(perm(U[:,ini_vec][after_vec]))**2 / div Boson_calc = probs[FockList_after[0], FockList_after[1], FockList_after[2], FockList_after[3]] print('Probability calculated with:') print('Classical algorithm',clasic_calc) print('Boson sampling',Boson_calc)大まかな流れとして、
- RゲートとBSゲートを結合し、1つのユニタリゲート$U$で記述する
- 光子が存在する入出力ポートに合わせて$U$を上式中$U_{st}$に整形し、Permanent関数を計算する。
- 解析的に得られた出力確率と、先ほど実装したボゾンサンプリング回路で得られた出力確率を比較する。
といったことを行っています。出力結果はこのようになります。
比較の結果で言うと、非常によく一致します。
Strawberry Fieldsが非常によくできていることのデモンストレーションですね。Input photon number states [1, 1, 0, 1] Measured photon number states [1, 0, 1, 1] Probability calculated with: Classical algorithm 0.046297023812036465 Boson sampling 0.046297023812036486このように特定の入出力状態とそれが得られる確率がわかれば、$\mathrm{Perm}(U_{st})$を計算できます。
実際のところ役に立つの?
ここまで上手くいっていますが、ボゾンサンプリングには実用上の問題点があるようです。
まず、現実にボゾンサンプリングを実行しても上記コードのように直接出力確率が得られることはなく、あくまで1つの出力結果が得られるだけです。
よって、ある入出力状態について確率を知るには十分な回数のサンプリングを繰り返す必要があります。ところが得られる出力光子数状態の組み合わせは光子数$n$に対してsuper exponentialです。
よって、サンプリングによって一定の精度を担保しようとすると結局指数関数的にリソースが増えかねないです。
また、そもそもどれだけサンプリングすればどれだけの精度を担保できるか自体が非常に難しい問題です。入力光子数状態の生成にも課題があります。
各ポートには光子がほぼ同時に入力される必要がありますが、光子生成源があるtime bin $\delta t$ の間に光子を吐く確率が$p$であるような性質を持っている場合、$\delta t$の間にn個の光子が生成される確率は $p^n$ となってしまいます。
よって試行回数は光子数$n$の指数関数でスケールされてしまいます。確率的でなく決定論的に単一光子を生成するのは技術的ハードルが高いようです。
あまり詳しくないですが。
ただし、入力光子数状態の問題は後に Gaussian Boson Sampling という手法が提案され回避可能になります。過去の研究者の苦難の跡が見えますね。色々と勉強になります。
次は流れ的には Gaussian Boson Sampling ですが、似ているので飛ばすかもしれません。
読んでいただきありがとうございました!参考にした文献
・公式ドキュメント
・https://en.wikipedia.org/wiki/Permanent_(mathematics)
・https://arxiv.org/abs/1406.6767
- 投稿日:2019-01-27T18:16:21+09:00
pip install poencv-pythonで入れたopenCVのバージョンを変更する
- 投稿日:2019-01-27T17:59:54+09:00
PyQt5チュートリアルのテトリスをMVC化
ZetCodeのPyQt5チュートリアルにあるテトリスのソースコードを拝見したのですが、モデルが分離されておらず、気になってしまったので MVC(Model-View-Controller) 化に挑戦しました。
- Model(データ) は表示に左右されない部分、PyQt5(GUI)は登場しない
- Modelは大きく Tetrimino と Field の2つで構成、FacadeパターンでModelに統合
- View(表示) と Controller(操作) で PyQt5を使用
- チュートリアルではwidgetを継承して使用しているが、名前衝突を嫌って継承しないように変更
改善点などありましたらコメントお願いします。
何か参考になることがあれば幸いです。tetrimino.pyclass Tetrimino: """ # python -m doctest -v tetrimino.py >>> len(tetriminos) 7 >>> print(tetrimino_names['Z'].rotate_left()) ## ## >>> print(tetrimino_names['S'].rotate_right()) ## ## >>> print(tetrimino_names['I'].rotate_left()) #### >>> print(tetrimino_names['T']) ### # >>> print(tetrimino_names['O']) ## ## >>> tetrimino_names['O'] is tetrimino_names['O'].rotate_right() True >>> print(tetrimino_names['L'].rotate_right().rotate_right()) # # ## >>> print(tetrimino_names['J'].rotate_left().rotate_left()) # # ## """ def __init__(self, name, *offsets, rotate=True): self.name = name self.offsets = offsets self.can_rotate = rotate def __iter__(self): return iter(self.offsets) def __str__(self): xs = range(self.min_x(), self.max_x() + 1) ys = range(self.min_y(), self.max_y() + 1) return '\n'.join(''.join(' #'[(x, y) in self.offsets] for x in xs) for y in ys) def min_x(self): return min(x for x, y in self) def max_x(self): return max(x for x, y in self) def min_y(self): return min(y for x, y in self) def max_y(self): return max(y for x, y in self) def move(x, y): return Tetrimino(self.name, *self.offsets, rotate=self.can_rotate) def rotate_unclockwise(self): if not self.can_rotate: return self return Tetrimino(self.name, *((y, -x) for x, y in self.offsets)) def rotate_clockwise(self): if not self.can_rotate: return self return Tetrimino(self.name, *((-y, x) for x, y in self.offsets)) tetriminos = ( # (+0, +0) is rotation center Tetrimino('Z', (+0, -1), (+0, +0), (-1, +0), (-1, +1)), Tetrimino('S', (+0, -1), (+0, +0), (+1, +0), (+1, +1)), Tetrimino('I', (+0, -1), (+0, +0), (+0, +1), (+0, +2)), Tetrimino('T', (-1, +0), (+0, +0), (+1, +0), (+0, +1)), Tetrimino('O', (+0, +0), (+1, +0), (+0, +1), (+1, +1), rotate=False), Tetrimino('L', (-1, -1), (+0, -1), (+0, +0), (+0, +1)), Tetrimino('J', (+1, -1), (+0, -1), (+0, +0), (+0, +1)), ) tetrimino_names = {tetrimino.name: tetrimino for tetrimino in tetriminos}field.pyclass TetrisField: """ # python -m doctest -v field.py >>> field = TetrisField(6, 4) >>> print(field) | | | | | | | | >>> field.put(((-1, -1), (0, -1), (-1, 0), (0, 0)), 1, 3) >>> print(field) | | | | |## | |## | >>> field.clear_complete_lines() 0 >>> field.can_put(((0, 0), (1, 0), (2, 0), (3, 0)), 2, 3) True >>> field.put(((0, 0), (1, 0), (2, 0), (3, 0)), 2, 3) >>> print(field) | | | | |## | |######| >>> field.clear_complete_lines() 1 >>> print(field) | | | | | | |## | """ WALL = (-1, 0) def __init__(self, width, height): self.width = width self.height = height self.tiles = [[None] * width for y in range(height)] def __str__(self): return '\n'.join('|' + ''.join('# '[tile is None] for tile in row) + '|' for row in self.tiles) def __iter__(self): return iter(self.tiles) def __setitem__(self, coodinates, tile): x, y = coodinates if 0 <= x < self.width and 0 <= y < self.height: self.tiles[y][x] = tile def __getitem__(self, y_or_xy): if isinstance(y_or_xy, int): y = y_or_xy return self.tiles[y] if 0 <= y < self.height else () x, y = y_or_xy if y < 0: return None elif 0 <= x < self.width and y < self.height: return self.tiles[y][x] else: return self.WALL def copy(self): field = TetrisField(self.width, self.height) for y, tiles in enumerate(self): for x, tile in enumerate(tiles): field[x, y] = tile return field def can_put(self, tetrimino, x, y): tetrimino = tetrimino or self.WALL return all(self[x + dx, y + dy] is None for dx, dy in tetrimino) def put(self, tetrimino, x, y): for dx, dy in tetrimino: self[x + dx, y + dy] = tetrimino def clear_complete_lines(self): for y, tiles in enumerate(self.tiles[:]): if all(tile is not None for tile in tiles): del self.tiles[y] clear_lines = self.height - len(self.tiles) if clear_lines > 0: self.tiles[:0] = [[None] * self.width for y in range(clear_lines)] return clear_linesmodel.pyimport random from tetrimino import tetriminos, tetrimino_names from field import TetrisField class TetrisModel: """ # python -m doctest -v model.py >>> model = TetrisModel(4, 6) >>> model.put(tetrimino_names['T'], 2, 0) True >>> model.drop_down() >>> print(model) | | | | | | | | | ###| | # | >>> model.put(tetrimino_names['I'], 0, 0) True >>> model.drop_down() >>> print(model) | | | | | | |# | |# | |# # | >>> model.score 1 """ SCORE = (0, 1, 2, 4, 8) # by clear lines def __init__(self, width, height): self.width = width self.height = height self.field = TetrisField(width, height) self.tetrimino = None self.x = 0 self.y = 0 self.score = 0 self.alive = True def __iter__(self): field = self.field.copy() self.tetrimino and field.put(self.tetrimino, self.x, self.y) return iter(field) def __str__(self): field = self.field.copy() self.tetrimino and field.put(self.tetrimino, self.x, self.y) return str(field) def is_alive(self): return self.alive def dead(self): self.alive = False def put_new_tetrimino(self): tetrimino = random.choice(tetriminos) x = self.field.width // 2 for y in range(tetrimino.min_y(), tetrimino.max_y() + 1): if self.field.can_put(tetrimino, x, -y): self.put(tetrimino, x, -y) return self.dead() def put(self, tetrimino, x, y): if self.field.can_put(tetrimino, x, y): self.tetrimino = tetrimino self.x = x self.y = y return True return False def replace(self, tetrimino): return self.put(tetrimino, self.x, self.y) def move(self, x, y): return self.tetrimino and self.put(self.tetrimino, x, y) def move_left(self): self.move(self.x - 1, self.y) def move_right(self): self.move(self.x + 1, self.y) def rotate_clockwise(self): self.tetrimino and self.replace(self.tetrimino.rotate_clockwise()) def rotate_unclockwise(self): self.tetrimino and self.replace(self.tetrimino.rotate_unclockwise()) def move_down(self): if self.tetrimino is None: return False if self.move(self.x, self.y + 1): return True self.fixed() return False def drop_down(self): while self.move_down(): pass def fixed(self): if self.tetrimino is None: return True self.field.put(self.tetrimino, self.x, self.y) self.tetrimino = None clear_lines = self.field.clear_complete_lines() self.score += self.SCORE[clear_lines] return False def step(self): if not self.is_alive(): return if self.tetrimino: self.move_down() else: self.put_new_tetrimino()qt5view.pyfrom PyQt5.QtCore import Qt from PyQt5.QtWidgets import QMainWindow, QFrame, QDesktopWidget from PyQt5.QtGui import QPainter, QColor TILE_COLORS = { 'Z': QColor(0xCC6666), 'S': QColor(0x66CC66), 'I': QColor(0x6666CC), 'T': QColor(0xCCCC66), 'O': QColor(0xCC66CC), 'L': QColor(0x66CCCC), 'J': QColor(0xDAAA00), } class ModelView: def __init__(self, parent, model): self.model = model self.widget = QFrame(parent) self.widget.setFocusPolicy(Qt.StrongFocus) self.widget.paintEvent = self.draw def draw(self, event): width = self.widget.contentsRect().width() // self.model.width height = self.widget.contentsRect().height() // self.model.height tiles = ((x, y, tile) for y, tiles in enumerate(self.model) for x, tile in enumerate(tiles) if tile is not None) for x, y, tile in tiles: color = TILE_COLORS[tile.name] self.drawTile(x * width, y * width, width, height, color) def drawTile(self, x, y, width, height, color): x2, y2 = x + width, y + height painter = QPainter(self.widget) painter.fillRect(x, y, width, height, color) painter.setPen(color.lighter()) painter.drawLine(x, y, x2, y) painter.drawLine(x, y, x, y2) painter.setPen(color.darker()) painter.drawLine(x + 1, y2 - 1, x2 - 1, y2 - 1) painter.drawLine(x2 - 1, y + 1, x2 - 1, y2 - 1) class TetrisView: def __init__(self, model, width, height): self.widget = QMainWindow() self.model_view = ModelView(self.widget, model) self.status = self.widget.statusBar() self.widget.setWindowTitle('Tetris') self.widget.setCentralWidget(self.model_view.widget) self.widget.resize(width, height) self.centering() def centering(self): screen = QDesktopWidget().screenGeometry() size = self.widget.geometry() self.widget.move((screen.width() - size.width()) // 2, (screen.height() - size.height()) // 2) def show(self): self.widget.show() def showStatus(self, message): self.status.showMessage(message) def update(self): self.model_view.widget.update() self.widget.update() def on_keyin(self, handler): # call handler(key) when key is pressed def hook(event): handler(event.key()) self.widget.keyPressEvent = hook def on_close(self, handler): def closeEvent(event): handler() self.widget.closeEvent = closeEventqt5tetris.pyimport sys from PyQt5.QtCore import Qt, QObject, pyqtSignal, QBasicTimer from PyQt5.QtWidgets import QApplication from model import TetrisModel from qt5view import TetrisView class Status(QObject): signal = pyqtSignal(str) def on(self, handler): self.signal[str].connect(handler) def set(self, message): # It will call handler(message) that set self.on(handler) self.signal.emit(message) class Timer(QObject): def __init__(self, handler): super().__init__() self.handler = handler self.timer = QBasicTimer() def start(self, interval): # It will call self.timerEvent(event) after each interval self.timer.start(interval, self) def stop(self): self.timer.stop() def timerEvent(self, event): if event.timerId() == self.timer.timerId(): self.handler() class Tetris: SPEED = 300 def __init__(self, app): self.app = app self.model = TetrisModel(width=10, height=20) self.view = TetrisView(self.model, width=180, height=380) self.status = Status() self.status.on(self.view.showStatus) self.key_map = self.make_key_map() self.view.on_keyin(self.keyin) self.view.on_close(self.quit) self.timer = Timer(self.step) self.is_started = False def make_key_map(self): return { Qt.Key_Q: self.quit, Qt.Key_P: self.pause, Qt.Key_Left: self.model.move_left, Qt.Key_Right: self.model.move_right, Qt.Key_Up: self.model.rotate_clockwise, Qt.Key_Down: self.model.rotate_unclockwise, Qt.Key_Space: self.model.drop_down, Qt.Key_D: self.model.move_down, } def keyin(self, key): if key not in self.key_map: return False self.key_map[key]() return True def start(self): if not self.is_started: self.is_started = True self.view.show() self.timer.start(self.SPEED) def step(self): self.model.step() if self.model.is_alive(): self.status.set(f"Score: {self.model.score}") else: self.status.set(f"Game Over (Score: {self.model.score})") self.view.update() def pause(self): if self.is_started: return self.is_paused = not self.is_paused if self.is_paused: self.timer.stop() self.status.set("Paused") else: self.timer.start(self.SPEED) self.status.set(f"Score: {self.model.score}") def quit(self): self.app.quit() def main(): app = QApplication(sys.argv) tetris = Tetris(app).start() sys.exit(app.exec_()) if __name__ == '__main__': main()
- 投稿日:2019-01-27T17:57:42+09:00
Pythonista3でブラウザで開いているURLのタイトルを取得する
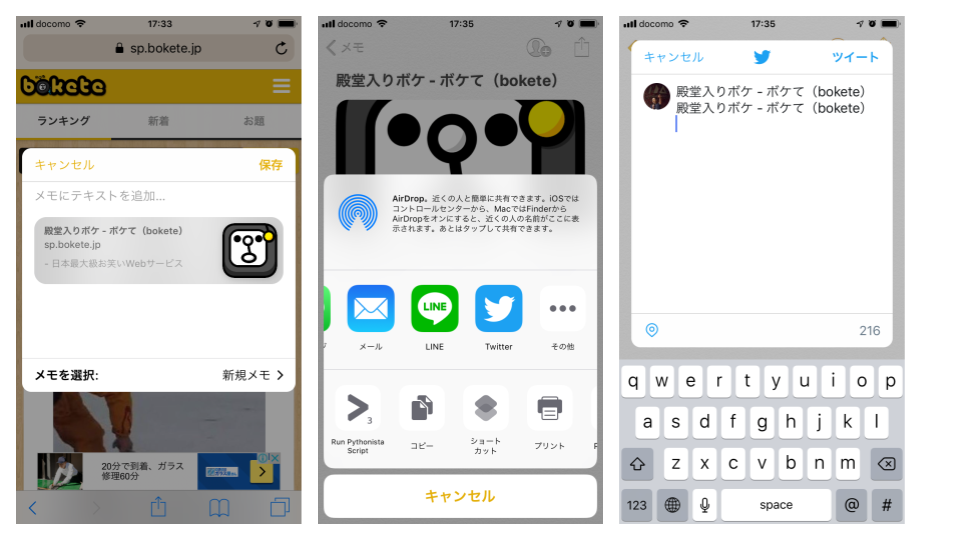
iPhoneで見ていたページを共有する方法は幾つかあるが、その場で共有するならともかく、一旦、どこかで保持してから共有するのは、どうも使いにくい。下の3枚の画像は①SafariからiOS標準のメモ帳で記録し、②メモ腸からTwitterで共有する、という流れだが③でURLの情報が失われている。
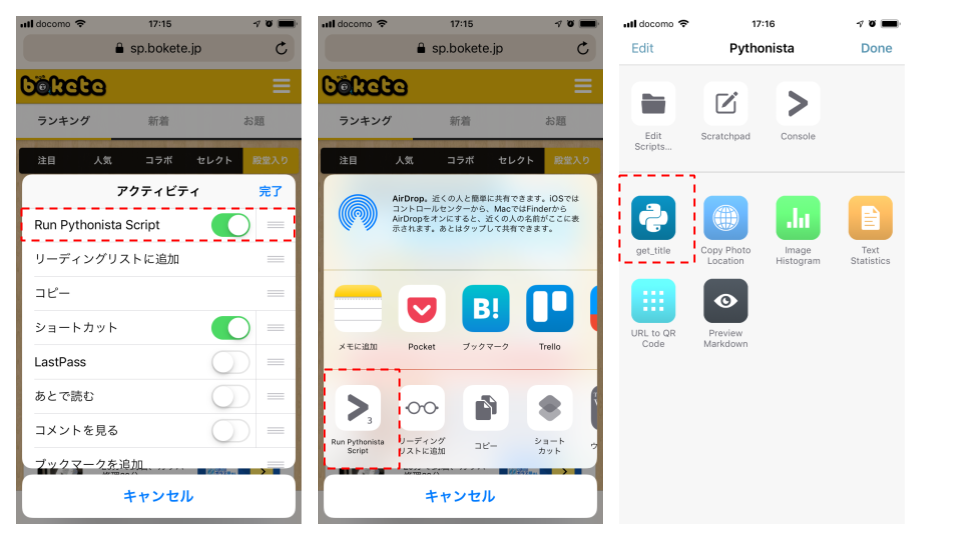
といって、URLだけ保存していると、QiitaのURLのように、あとでURLを見て何のページかわからない。そこでPythonista3でURLにタイトルを付与するというスクリプトを書いてみた。利用イメージとしては、下の3枚の画像のように①事前にPythonistaを有効にしておき、②Safariの共有で「Run Pythonista Script」を起動して、③URLを送るスクリプトを送るという流れである。
URLはappex.get_url()もしくはクリップボードから取得し、タイトルの取得にはBeautifulSoupを使っている。
import sys import appex import urllib.request import clipboard from bs4 import BeautifulSoup def main(): if appex.is_running_extension(): url = appex.get_url() else: url = clipboard.get() try: html = urllib.request.urlopen(url=url) soup = BeautifulSoup(html, 'html.parser') title = soup.title.string print(title+'\r\n'+url) clipboard.set(title) except: print(url) print(sys.exc_info()[0]) pass if __name__ == '__main__': main()後半に
print(title+'\r\n'+url)で書いているようにタイトル、URLの順で画面表示され、クリップボードにタイトルが格納される。単にタイトルを取得するだけでなく、あんなことやこんなことにも応用できそうだ。
- 投稿日:2019-01-27T17:38:47+09:00
dplyr使いのためのpandas dfply window関数編
はじめに
pandasデータフレームをRのdplyr同様に操作可能にするdfplyライブラリについてまとめるシリーズです。
dfplyについてはこちらをご参照ください。
dplyr使いのためのpandas dfplyすごい編Window関数?
SQLではおなじみですが、主に集計や分析に使われる関数ですね。
実行結果がgroup_byしたときのように集約されるわけではなく、入力データに対しての実行結果が戻されます。そのためmutateと一緒に使うことが多いと思います。
dplyrでのwindow関数は、matsuou1氏がまとめているこちらも参考にして頂けるとわかりやすいです。事前準備、例データ
今回も、みんな大好きtitanicを使用します。
Python:事前準備、例データimport pandas as pd import numpy as np from dfply import * #dfply読み込み import seaborn as sns titanic = sns.load_dataset('titanic') #titanic読み込みランキング
選択列の値に対して順位番号をつけていく関数です。
dfplyでは、以下の関数が実装されているようです。
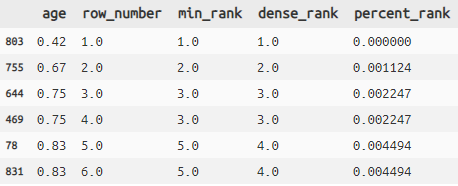
関数 説明 row_number 選択列の値に対して順番付け(デフォルトは昇順) min_rank 選択列の値に対して順番付け(同値は同番号、その場合次番号は飛ぶ) dense_rank 選択列の値に対して順番つけ(同値は同番号、その場合次番号は飛ばない) percent_rank min_rankのスケールを0~1に変換したもの Python:ランキングtitanic >> arrange(X.age) >> select(X.age) >> mutate( row_number=row_number(X.age, ascending=True), min_rank=min_rank(X.age, ascending=True), dense_rank=dense_rank(X.age, ascending=True), percent_rank=percent_rank(X.age, ascending=True)) >> head(6)
- 各順位番号はnull(None, NaN)を対象には含みません。
- ascending = Trueは省略可オフセット
選択列の前後のレコードの値を取得できます。
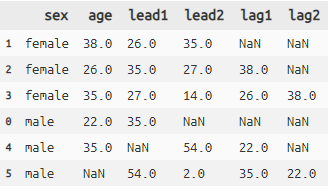
関数 説明 lead 選択列の値に対して前レコードの値をとる lag 選択列の値に対して後レコードの値をとる Python:オフセット# lag lead titanic >> group_by(X.sex) >> mutate( lead1=lead(X.age, i=1), lead2=lead(X.age, i=2), lag1=lag(X.age, i=1), lag2=lag(X.age, i=2)) >> \ head(3) >> select(X.sex, X.age, X.lead1, X.lead2, X.lag1, X.lag2)- group_byを使うとそのグループ内でのオフセットも可能です(dplyrと一緒です)
- i = は何レコード分ずらすかの指定です(省略可)
- dplyrではnull値を埋める数値も指定できましたが、dfplyではできないようです累積
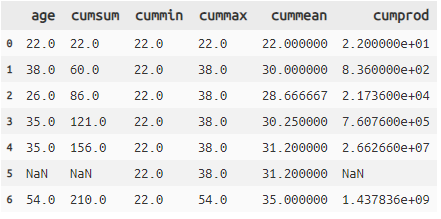
選択した列に対して1レコードずつ累積で関数を処理していきます。例で見た方が理解が早いですね。
累積1:cumsum, cummin, cummax, cummean, cumprod
関数 説明 cumsum 対象レコードまでの累積和(足し算) cummin 対象レコードまでの最小値 cummax 対象レコードまでの最大値 cummean 対象レコードまでの平均値 cumprod 対象レコードまでの積 Python:累積1titanic >> select(X.age) >> mutate( cumsum=cumsum(X.age), cummin=cummin(X.age), cummax=cummax(X.age), cummean=cummean(X.age), cumprod=cumprod(X.age)) >> head(7)
- 例えばcummaxはageの6レコード目に54がでてきて累積値で一番高い数値になったため、54が返されています累積2:cumany, cumall
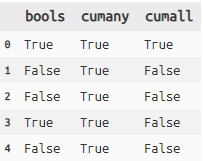
cumany,cumallは少々ややこしいです(使用機会も少ないと思いますが)。
選択列がTrue or Falseのbool型のときに判定を行う関数です。これも例を見た方が理解が早いですね。Python:累積2#bool型のデータを作成 test = pd.DataFrame({'bools':[True,False,False,True,False]}) #cumany, cumall test >> mutate(cumany=cumany(X.bools), cumall=cumall(X.bools))
- cumanyは選択列にTrueが表れた時点で以降すべてTrueと判定します
- cumallは選択列にFalseが表れた時点で以降すべてをFalseと判定します
その他
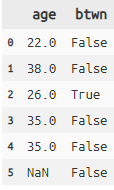
between
選択した列の値が、指定した値の範囲内にあるかないかを判定しbool型を返します。
Python:between#ageが25~30の範囲内にあるかどうか判定 titanic >> mutate(age_btwn=between(X.age, 25, 30, inclusive=True)) >> \ select(X.age, X.age_btwn) >> head(6)まとめ
window関数もdplyr同様しっかり再現されてますね。
dfplyやっぱりすごいです。
- 投稿日:2019-01-27T17:24:51+09:00
Python手遊び(wget)
この記事、何?
pythonを使って、Windowsでhttpsからファイルを取得するという話。
ちょっと前の記事でWindowsでwget代わりにPowerShellでInvoke-WebRequestしてた。
ただ、https相手だとうまくいかなかった。
それをトラブルシュートするよりpythonしたほうが楽っぽかったので頼ってみた、という話。どういう人向け?
まあ・・・自分あて。
クローニングとかもいろいろ潤沢な時代に「httpsあてでもファイル取れるわ~」って言われても、ね?
でもQiitaの記事を取れるバッチファイルも付けたのでよければどうぞ。参考にした記事
結局は他力本願なんですけどね。
Alice1017さん、ありがとうございます。■Pythonでバイナリファイルを保存する
https://qiita.com/Alice1017/items/34befe8168cd771f535fやったこと
Pythonスクリプト1つ、呼出用のバッチが一つ。
#17-01.py #Qiitaの投稿ID(?)をもらって、Markdownをファイル化 import sys import shutil import requests args = sys.argv id = args[1] URL = 'https://qiita.com/siinai/items/' + id + '.md' print(URL) filepath = id + '.md' print(filepath) res = requests.get(URL, stream = True) with open(filepath, 'wb') as fp: shutil.copyfileobj(res.raw, fp)rem 呼び出し用のバッチファイル rem python 17-01.py [qiita-id] python 17-01.py 4deb8529a2224a238b91 python 17-01.py 7b4196f4271448093a1f python 17-01.py 457977b61b6c4cb15ac4 ...感想
まあ・・・普通。
Pythonってこんなに簡単なのね・・・
いいわぁ♪
- 投稿日:2019-01-27T16:07:56+09:00
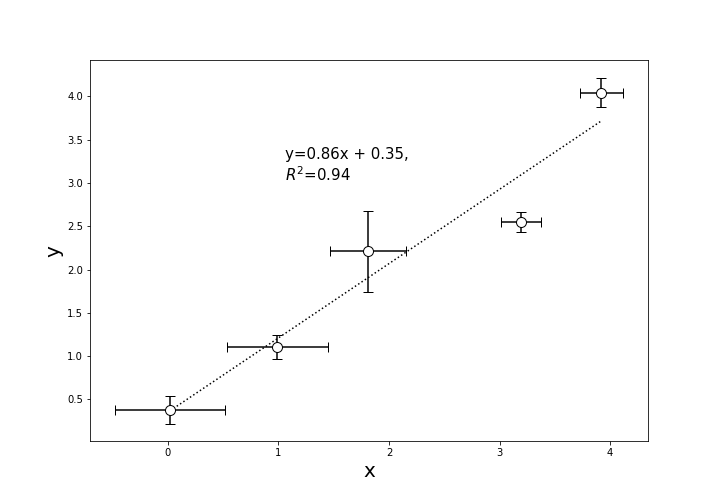
matplotlib エラーバー付きのグラフを描く
概要
グラフにエラーバーをつけたいことは多々ありますよね。
今回はグラフにエラーバーをつけて、検量線を描くことを目標とします。データの用意
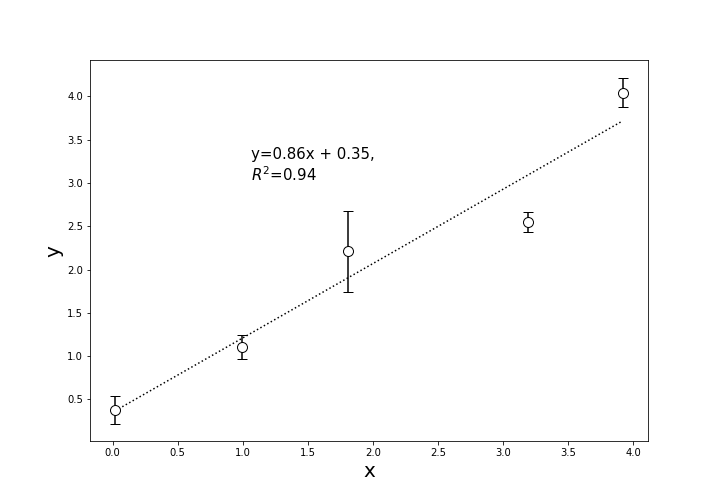
from matplotlib import pyplot as plt import random import numpy as np from sklearn.metrics import r2_score ## テストデータの作成 x = [i+random.uniform(-0.2, 0.2) for i in range(5)] # 変数を初期化 y = [i+random.uniform(-0.5, 0.5) for i in range(5)] x_err = [random.uniform(-0.5, 0.5) for i in range(5)] # 誤差範囲を乱数で生成 y_err = [random.uniform(-0.5, 0.5) for i in range(5)]y軸方向にerror barを設定
もっとも多く使うのはy軸方向にerror barを設定するケースだと思います。
put.errorbarのyerrに誤差の入った配列を設定します。以下によく使う引数をまとめました。
引数 意味 x x軸の値(配列) y y軸の値(配列) xerr x軸の誤差の値(配列) yerr y軸の誤差の値(配列) fmt マーカーの表示, 'o', 'v'など 詳しくはこちら markersize マーカーの大きさ color マーカーの色 markeredgecolor マーカーの縁の色 ecolor エラーバーの色 capsize エラーバーの横線の長さ (matplotlib.pyplot.errorbarにはもっと詳しくあります。)
また、検量線は
np.polyfitで一次近似して、scikit_learnのr2_scoreで決定係数を出します。# y軸方向にのみerrorbarを表示 plt.figure(figsize=(10,7)) plt.errorbar(x, y, yerr = y_err, capsize=5, fmt='o', markersize=10, ecolor='black', markeredgecolor = "black", color='w') coef=np.polyfit(x, y, 1) appr = np.poly1d(coef)(x) plt.plot(x, appr, color = 'black', linestyle=':') y_pred = [coef[0]*i+coef[1] for i in x] r2 = r2_score(y, y_pred) plt.text(max(x)/3.7, max(y)*6/8, 'y={:.2f}x + {:.2f}, \n$R^2$={:.2f}'.format(coef[0], coef[1], r2), fontsize=15) plt.xlabel('x', fontsize=20) plt.ylabel('y', fontsize=20) # plt.savefig('y_error_bar.png')x, y軸両方にerror barを設定
x軸方向にもerror barをつけたいこともあるでしょう。
その場合は、xerr引数に誤差配列を渡せばできます。# x軸方向にもerrorbarを表示 plt.figure(figsize=(10,7)) plt.errorbar(x, y, yerr = y_err, xerr = x_err, capsize=5, fmt='o', markersize=10, ecolor='black', markeredgecolor = "black", color='w') coef=np.polyfit(x, y, 1) appr = np.poly1d(coef)(x) plt.plot(x, appr, color = 'black', linestyle=':') y_pred = [coef[0]*i+coef[1] for i in x] r2 = r2_score(y, y_pred) plt.text(max(x)/3.7, max(y)*6/8, 'y={:.2f}x + {:.2f}, \n$R^2$={:.2f}'.format(coef[0], coef[1], r2), fontsize=15) plt.xlabel('x', fontsize=20) plt.ylabel('y', fontsize=20) # plt.savefig('xy_error_bar.png')リソース
以下のrepositoryに実行コードがあります。
https://github.com/YutoOhno/ErrorBar
- 投稿日:2019-01-27T15:59:23+09:00
【Qiita記事無作為試用①】Google Colaboratoryで簡単な顔認識に挑戦
・膨大な素晴らしきプログラムの集積地であるQiitaの中から、無作為に試してみて、惜しげもなく我が物顔を炸裂する記事第一弾。
・興味のあるものは何の躊躇も恥もなく無心で模倣してきた「自己解決反対運動」の信者のため、思う存分発揮していこう。
・初回として、顔認識処理が簡単に扱える記事を発見したので、これを試してみよう。概要
- Google Colaboratoryという、Jupyter Notebook環境を構築不要ですぐに試せるサービスを使って、指定画像を顔認識処理にかけて、返してくれる。
※Jupyter Notebookとは、実行記録型のデータ分析ツールである。主にPython実行環境として使用されていたが、現在は様々な言語に対応している。結果
使用環境・技術
- 1. Google Colaboratory
- 2. Python
- 3. OpenCV(解析ライブラリ)
手順
使用画像の用意
- 今回顔認識処理に用いる好きな画像を用意する。(※公開等なければ、著作権はあまり気にしない。)
- 画像決定すれば、名前をつけて保存。その際の保存名はメモしておく。
※今回の保存名は「test.jpg」で、使用画像は下記。
解析ライブラリの設定
- 画像の顔認識処理のために、解析ライブラリであるOpenCVの公式GitHubから下記のファイルを自分のパソコンにダウンロードする。
「ファイル名」・・・haarcascade_frontalface_default.xml
「取得リンク」・・・ここGoogle Colaboratoryの設定
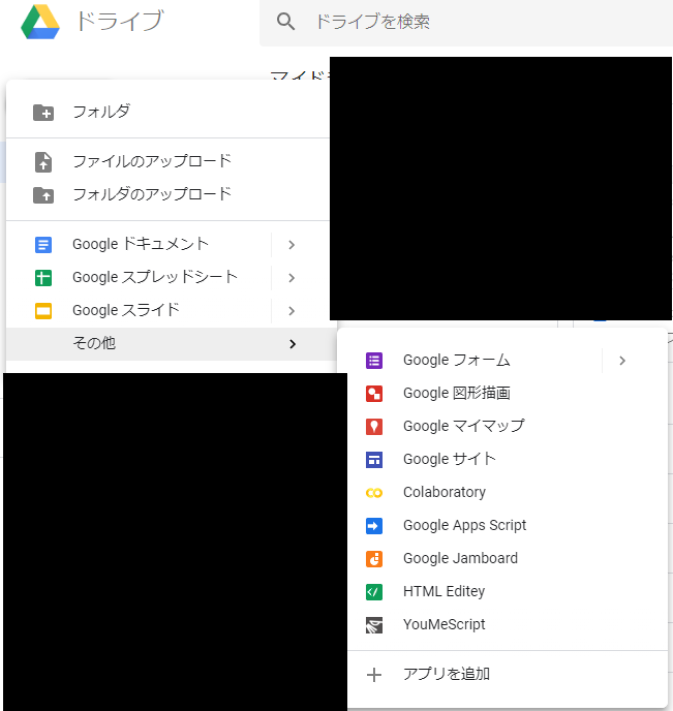
- GoogleドライブからColaboratoryプロジェクト作成。
※Googleアカウントを持っていない人は、作成する。
- Googleドライブを起動
- 「新規」→ 「その他」→「アプリを追加」の順にクリック。
- アプリ検索欄に「colab」と入力して検索。
- 検索結果に「Colaboratory」が表示されるので、そこの「接続」ボタンをクリック
- 改めて「新規」→ 「その他」の順にクリックしていくと、「Colaboratory」が表示されているので、クリック。
- 起動したプロジェクトに名前をつける
※プロジェクト名が反映されるわけではないが、自分がわかる名前をつける。
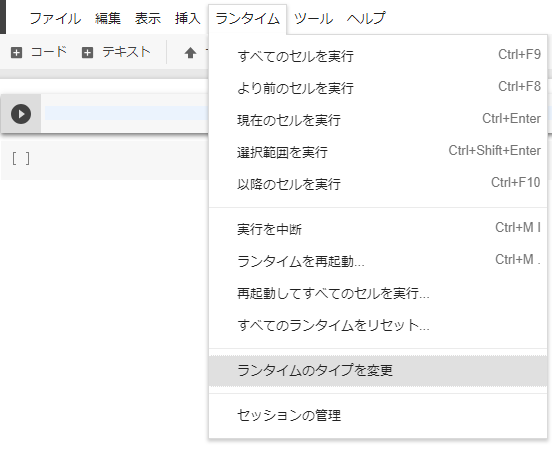
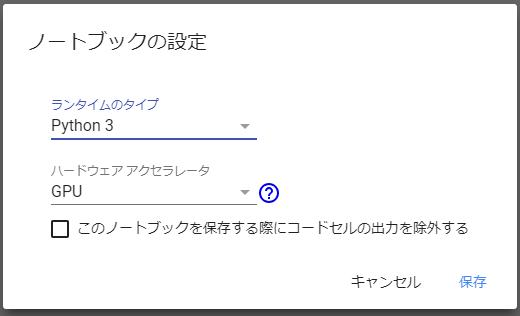
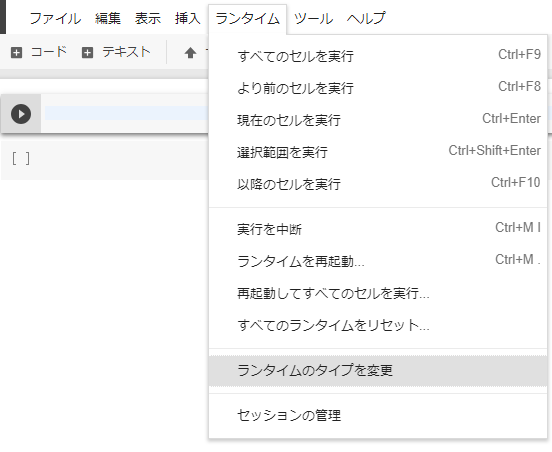
- GPU割り当てのため、プロジェクト上部メニューの「ランタイム」から「ランタイムのタイプを変更」をクリック。
ノートブック設定後、プロジェクトに下記のコードを入力後、左の三角ボタンで実行する。
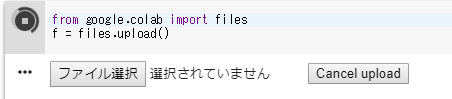
from google.colab import files f = files.upload()
ファイル選択ボタンが表示されるので、先程保存した下記の2つのファイルをアップロードする。
※アップロードは1つずつ行う。
①使用画像ファイル・・・今回は「test.jpg」
②解析ライブラリファイル・・・「haarcascade_frontalface_default.xml 」アップロード後、下記のコードを入力して、実行する。
import cv2 from matplotlib import pyplot as plt # 画像読み込み img=cv2.imread("./test.jpg") # 画像のRGB形式への変換 img = cv2.cvtColor(img, cv2.COLOR_BGR2RGB) # 解析ライブラリの使用 cascade = cv2.CascadeClassifier('./haarcascade_frontalface_default.xml') # 検出処理 gray = cv2.cvtColor(img, cv2.COLOR_BGR2GRAY) face = cascade.detectMultiScale(gray, scaleFactor=1.1, minNeighbors=3, minSize=(30, 30)) # 検出領域のデザイン処理 for (x, y, w, h) in face: cv2.rectangle(img, (x, y), (x + w, y + h), (200,0,0), 3) # 標準軸の非表示処理 plt.grid(False) plt.tick_params(labelbottom=False, labelleft=False, labelright=False, labeltop=False) plt.imshow(img)※好きな画像を使用している場合は、「test.jpg」の部分を自分用に変える。
まとめ
- 初回は顔認識処理ということで、「賢人の方々の知の結晶のおかげで、こんなにも恩恵を受けているのだな」と、水分の跡も見られないまぶたをぬぐいながら、記事を書く。
- とはいえこの模倣学習は、GoogleやQiitaや作成者等の多くの方々の偉大な功績によるものなので、毎回の礼拝作業は欠かさない。
- 「今後も、自己満足促進のための素晴らしきプログラム探しに精進しよう」と、堂々他人依存決断。
参考
- https://qiita.com/FrozenVoice/items/d1dc5f3d780f5097bfab
→こちらの記事を参考にしました。大変お世話になりました。
- 投稿日:2019-01-27T15:51:54+09:00
Self-Attentionを利用したテキスト分類
Self-Attentionを利用したテキスト分類
TL;DR
テキスト分類問題を対象に、LSTMのみの場合とSelf-Attentionを利用する場合で精度にどのような差がでるのかを比較しました。
結果、テキスト分類問題においても、Self-Attentionを利用することで、LSTMのみを利用するよりも高い精度を得られることが確認できました。Self-Attentionの実装としてはkeras-self-attentionを利用しました。
ベンチマーク用データ
京都大学情報学研究科--NTTコミュニケーション科学基礎研究所 共同研究ユニットが提供するブログの記事に関するデータセットを利用しました。 このデータセットでは、ブログの記事に対して以下の4つの分類がされています。
- グルメ
- 携帯電話
- 京都
- スポーツ
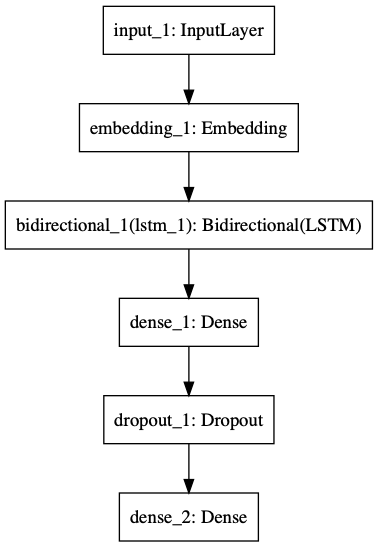
LSTMのみ利用した場合のモデルと結果
text-vectorianを利用してベクトル表現に変換したテキスト入力し、LSTMにより学習、
分類先となる4ラベルの何れに該当するかを推論するモデルです。モデル
クラシフィケーションレポート
F1値は0.76でした。
precision recall f1-score support 京都 0.71 0.75 0.73 137 携帯電話 0.80 0.81 0.80 145 スポーツ 0.68 0.72 0.70 47 グルメ 0.84 0.72 0.78 90 micro avg 0.76 0.76 0.76 419 macro avg 0.76 0.75 0.75 419 weighted avg 0.77 0.76 0.76 419Self-Attentionを利用した場合のモデルと結果
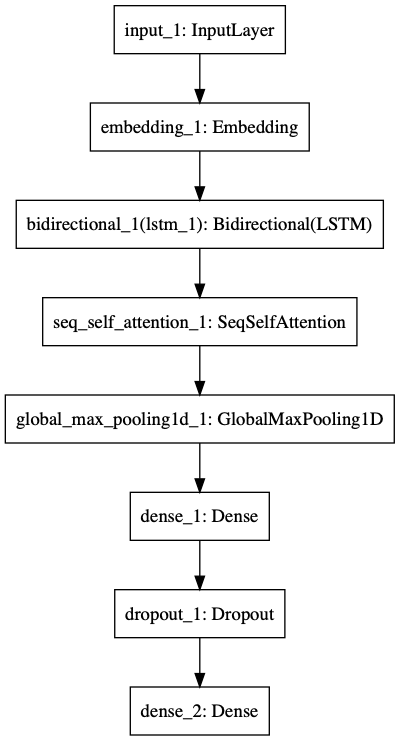
LSTMのみを利用した場合のモデルにSelf-Attentionの層を追加したものです。
Self-Attentionの出力は入力と同じ(sample, time, dim)の3階テンソルであるため、GlobalMaxPooling1DによりShapeを変換しています。モデル
クラシフィケーションレポート
F1値が0.79でした。
precision recall f1-score support 京都 0.71 0.84 0.77 137 携帯電話 0.88 0.78 0.83 145 スポーツ 0.66 0.74 0.70 47 グルメ 0.88 0.74 0.81 90 micro avg 0.79 0.79 0.79 419 macro avg 0.78 0.78 0.78 419 weighted avg 0.80 0.79 0.79 419総括
LSTMのみでは0.76であったF1値がSelf-Attentionを追加することで3%増加し0.79に向上しました。
それぞれ複数回試しても±1-2%程度の誤差範囲でしたので、Self-Attentionを追加することは有意であると考えられます。
AttentionはNMT(Neural Machine Translation)のようなシーケンスからシーケンスを推論する問題(seq2seq)への適用が注目されますが、
単純な分類問題にも入出力の形を変えずにそのまま適用出来るため、とりあえず使って見るのは悪く無さそうです。参考文献
- 投稿日:2019-01-27T15:51:12+09:00
化合物の記述子:RDKitを計算してみた
この記事、何?
というわけでRDKitを。
ちゃちゃっと計算さしてくれるページないかと思ってたんだけど、どれも微妙にそのままでは動かなかったのでつぎはぎして作ってみた。
まあ、動けばいいというレベルなのであとで書き直すと思う。どういう人向け?
いつも通り自分向け。
まあ、似たようなこと考えている方、pythonスクリプト一つで、SDFファイルを
csvにしてくれると助かるという方向け。試した環境は?
Windows 8 Pro (x64)
rdkit 2017.09.2.0やりたいこと
RDKitの計算。
最終的にはRDBに突っ込んでからだけど、まずはcsv化ということで。やったこと
まずはここを参考に動かしてみた。
ただ、そのままじゃ動かなかった。
(変数の記述が足りてない?)■Rdkitを使って、SDFファイルを分解
https://qiita.com/beginnerhuman/items/cb1ce16ce7f9cdd667a1なので、大先生のサイトから記載をちょっと流用。(すみません、お世話になります!)
■Support Vector Machine
https://funatsu-lab.github.io/open-course-ware/machine-learning/support-vector-machine/で、何とか動いた。
#16-02.py import rdkit import pandas as pd from rdkit import Chem from rdkit.Chem import Descriptors from rdkit.ML.Descriptors import MoleculeDescriptors suppl = Chem.SDMolSupplier('00000001_00025000_mini.sdf') mols = [x for x in suppl if x is not None] descs = [desc_name[0] for desc_name in Descriptors._descList] #化合物の数 print(len(mols)) #記述子の数 print(len(descs)) desc_calc = MoleculeDescriptors.MolecularDescriptorCalculator(descs) descriptors = pd.DataFrame([desc_calc.CalcDescriptors(mol) for mol in mols]) descriptors.columns = descs smiles = [Chem.MolToSmiles(mol) for mol in mols] descriptors.index = smiles #descriptors.to_csv('00000001_00025000_mini.csv') y_name = '_Name' y = pd.DataFrame([mol.GetProp(y_name) for mol in mols]) y.index = smiles y.columns = [y_name] dataset = pd.concat([y, descriptors], axis=1) dataset.to_csv('00000001_00025000_mini.csv')いろいろと課題が。。。
Pandas
全然わからない・・・
とてもじゃないけど転用して上にのせたようなコード、自力で作れる気がしない。。。
勉強しましょうね、はい。記述子の数
RDKitって、200個くらい記述子あるんじゃなかったっけ?
なんか、途中で115個って出るんですが・・・
他に作ったWin10やCentOSだと同じコードで200個演算されるし、それぞれの環境で、conda list > list.txtでとったテキストを比較しても主だった差がないようにしか見えないんだけど・・・
とりあえず、rdkitインストールし直してみようかな?
・・・やってみた。
悪化した。from rdkit import Chemすら通らなくなった・・・orz
まあ、こんな時の仮想環境ということで。
conda remve -n py36 --all
さよなら~
あっ、しまった。残しておいて、あとから環境問題のトラブルシュート用に取っておいてもよかったたかも。。。化合物ファイル
別記事のこれで集めたファイル2Dと3Dがあるうちの、2D版だったみたい。
■化合物を300万個目指していっぱい集めてみた
https://qiita.com/siinai/items/7b4196f4271448093a1f「記述子が足らない?あっ、3Dじゃないからか~」と勝手に納得したおかげで気づいた。
おかげで情報量が多いほうを再入手。
PowerShellでwgetするんでこんな感じで。Invoke-WebRequest -Uri ftp://ftp.ncbi.nlm.nih.gov/pubchem/Compound_3D/01_conf_per_cmpd/SDF/00000001_00025000.sdf.gz -OutFile C:\shared\20190127\00000001_00025000.sdf.gz Invoke-WebRequest -Uri ftp://ftp.ncbi.nlm.nih.gov/pubchem/Compound_3D/01_conf_per_cmpd/SDF/00025001_00050000.sdf.gz -OutFile C:\shared\20190127\00025001_00050000.sdf.gz Invoke-WebRequest -Uri ftp://ftp.ncbi.nlm.nih.gov/pubchem/Compound_3D/01_conf_per_cmpd/SDF/00050001_00075000.sdf.gz -OutFile C:\shared\20190127\00050001_00075000.sdf.gz Invoke-WebRequest -Uri ftp://ftp.ncbi.nlm.nih.gov/pubchem/Compound_3D/01_conf_per_cmpd/SDF/00075001_00100000.sdf.gz -OutFile C:\shared\20190127\00075001_00100000.sdf.gz Invoke-WebRequest -Uri ftp://ftp.ncbi.nlm.nih.gov/pubchem/Compound_3D/01_conf_per_cmpd/SDF/00100001_00125000.sdf.gz -OutFile C:\shared\20190127\00100001_00125000.sdf.gz Invoke-WebRequest -Uri ftp://ftp.ncbi.nlm.nih.gov/pubchem/Compound_3D/01_conf_per_cmpd/SDF/00125001_00150000.sdf.gz -OutFile C:\shared\20190127\00125001_00150000.sdf.gz Invoke-WebRequest -Uri ftp://ftp.ncbi.nlm.nih.gov/pubchem/Compound_3D/01_conf_per_cmpd/SDF/00150001_00175000.sdf.gz -OutFile C:\shared\20190127\00150001_00175000.sdf.gz Invoke-WebRequest -Uri ftp://ftp.ncbi.nlm.nih.gov/pubchem/Compound_3D/01_conf_per_cmpd/SDF/00175001_00200000.sdf.gz -OutFile C:\shared\20190127\00175001_00200000.sdf.gz ...あっ、for文書けよって話ですよね、分かっちゃいるんですが、つい。。。
ま、気が向いたらそのうち。感想
まあまあ面白かった。
こうやって小さなレンガを積み上げていくといいかも。
- 投稿日:2019-01-27T15:23:12+09:00
Python機械学習プログラミング カーネル主成分分析
非線形問題を解くためには、
低次元の空間に射影し、線形分離する必要がある。カーネル主成分分析は、
一旦高次元空間に変換した後に
低次元空間に射影することによって、
非線形の関数を線形に変換する。ソースコードを以下に示す。
from scipy.spatial.distance imoirt pdist, squreform from scipy import exp from scopy.linalg import eigh import numpy as np def rbf_kernel_pca(X, gamma, n_components): """RBFカーネルPCAの実装 パラメータ ------------ X: {Numpy ndarray}, shape = [n_samples, n_features] gamma: float RBFカーネルのチューニングパラメータ n_components: int 返される主成分の個数 戻り値 -------------- X_pc: {Numpy ndarray}, shape = [n_samples, k_features] 射影されたデータセット """ # M×N次元のデータセットでペアごとのユークリッド距離の2乗を計算 sq_dists = pdist(X, 'sqeuclidean') # ペアごとの距離を正方行列に変換 mat_sq_dists = squreform(sq_dists) # 対称カーネル行列を計算 K = exp(-gamma * mat_sq_dists) # カーネル行列を中心化 N = K.shape[0] one_n = np.ones((N, N)) / N K = K - one_n.dot(K) - K.dot(one_n) + one_n.dot(K).dot(one_n) # 中心化されたカーネル行列から固有対を取得 # scipy.linalg.eighはそれらを昇順で返す eigvals, eigvecs = eigh(K) eigvals, eigvecs = eigvals[::-1], eigvecs[:, ::-1] # 上位K個の固有ベクトル(射影されたサンプル)を収集 X_pc = np.column_stack((eigvecs[:, i] for i in range(n_components))) return X_pc
- 投稿日:2019-01-27T15:05:56+09:00
pandas group_byとpct_changeの併用時bug (0.23.N)
bugの内容
概要
pandasのpct_change関数をgroup byと共に使用する際に、group毎にpct_changeを区切って実行してくれないという事象が発生していた。(pandas 0.24.0で修正済み。)
何をしたかったのか
以下に参考用のテーブルを用意した。
このデータのValueのpct_changeをして、昨対比(MoM)を見たいという場面を想定。
Company Group Date Value A X 2015-01 1 A X 2015-02 2 A X 2015-03 1.5 A XX 2015-01 1 A XX 2015-02 1.5 A XX 2015-03 0.75 A XX 2015-04 1 B Y 2015-01 1 B Y 2015-02 1.5 B Y 2015-03 2 B Y 2015-04 3 B YY 2015-01 2 B YY 2015-02 2.5 B YY 2015-03 3 Company, Group, Dateでgroup byをして、Valueのpct_changeを実施する。
df['Pct_change'] = df.sort_values('Date').groupby(['Company', 'Group']).Value.pct_change()欲しかった結果
Company Group Date Value Pct_change A X 2015-01 1 NaN A X 2015-02 2 1 A X 2015-03 1.5 -0.25 A XX 2015-01 1 NaN A XX 2015-02 1.5 0.5 A XX 2015-03 0.75 -0.5 A XX 2015-04 1 0.33 B Y 2015-01 1 NaN B Y 2015-02 1.5 0.5 B Y 2015-03 2 0.33 B Y 2015-04 3 0.5 B YY 2015-01 2 NaN B YY 2015-02 2.5 0.25 B YY 2015-03 3 0.2 実際に吐き出された結果
Company Group Date Value Pct_change A X 2015-01 1 NaN A X 2015-02 2 1 A X 2015-03 1.5 -0.25 A XX 2015-01 1 -0.33 A XX 2015-02 1.5 0.5 A XX 2015-03 0.75 -0.5 A XX 2015-04 1 0.33 B Y 2015-01 1 0 B Y 2015-02 1.5 0.5 B Y 2015-03 2 0.33 B Y 2015-04 3 0.5 B YY 2015-01 2 -0.33 B YY 2015-02 2.5 0.25 B YY 2015-03 3 0.2 要は、group_byの最初のrow(ex.4行目)のPct_changeカラムに-0.33と入ってしまう。
ここはgroupが違うので、NaNであるべきなのに。解決方法
概要
pandasのversionを0.24.0にupしましょう。
sample
anaconda userはterminalを立ち上げて、以下のコマンドを打ちましょう。
conda install pandas=0.24.0参考
https://pandas.pydata.org/pandas-docs/stable/reference/api/pandas.DataFrame.pct_change.html
https://stackoverflow.com/questions/40273251/pandas-groupby-with-pct-change
https://github.com/pandas-dev/pandas/issues/21200
- 投稿日:2019-01-27T14:22:15+09:00
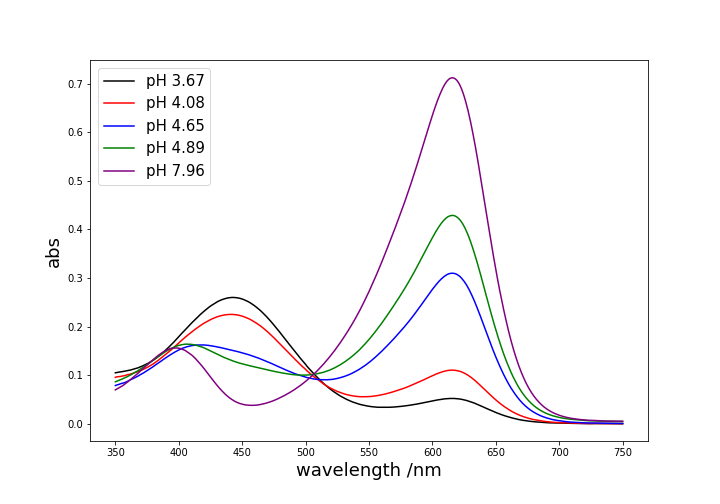
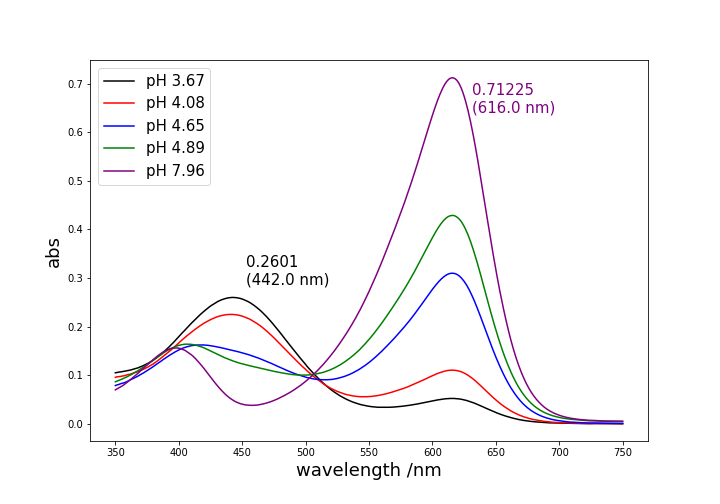
matplotlibでUV-Visスペクトルを描く
概要
matplotlibを使って、UV-Visスペクトルを描画する手法を紹介します!
今回はpHによって、吸収スペクトルが変化するケースを例にとります。
グラフの重ね合わせ、凡例の表示、ピークラベルの表示を実装しています。データの形式
以下の例で使用する
Uv-Vis.csv(こちらからダウンロード)は'wavelength'列に波長を持ち、その他の列は異なるpHごとのabs(吸光度)が入っています。wavelength,1,2,3,4,5 750.0,0.00018,0.00033,0.0007,0.00468,0.00536 749.0,0.00018999999999999998,0.00033,0.00073,0.00468,0.005379999999999999 748.0,0.00021,0.00029,0.00074,0.00468,0.005379999999999999 ...シンプルに重ね合わせる
まずは、複数のグラフを重ね合わせ、それに対応する凡例を表示させます。
from matplotlib import pyplot as plt import numpy as np # シンプルに重ね合わせる data = np.loadtxt("UV-Vis.csv", delimiter=",", skiprows=1) lines = [] colors = ['black', "red", "blue", "green", "purple"] pHs = ['3.67', '4.08', '4.65', '4.89', '7.96'] plt.figure(figsize=(10,7)) for i in range(1, 6): lines.append(plt.plot(data[:, 0], data[:, i], color=colors[i-1])) plt.xlabel("wavelength /nm", fontsize=18) plt.ylabel("abs", fontsize=18) plt.legend([i[0] for i in lines], ['pH {}'.format(i) for i in pHs], fontsize=15, loc='upper left') # plt.savefig("UV-Vis.png")必要に応じて保存こんな感じの出力。概ね良いですが、少し物足りない感じがしますね。
ピーク位置にラベルを付けましょう!ピーク位置にラベルをつける
plt.textでラベルを付けます。
ラベルの位置については自分でいい感じに調整してください。# ピークを位置表示 data = np.loadtxt("UV-Vis.csv", delimiter=",", skiprows=1) lines = [] colors = ['black', "red", "blue", "green", "purple"] pHs = ['3.67', '4.08', '4.65', '4.89', '7.96'] plt.figure(figsize=(10,7)) for i in range(1, 6): lines.append(plt.plot(data[:, 0], data[:, i], color=colors[i-1])) # pH 7.96の600nm付近のピーク peak1_abs = data[:, 5].max() peak1_wav = data[:, 0][data[:, 5].argmax()] plt.text(peak1_wav*1.025, peak1_abs*0.9, '{}\n({} nm)'.format(peak1_abs, peak1_wav), color='purple', fontsize=15) # 位置は手動で微調整... # pH 3.67の450nm付近のピーク peak2_abs = data[:, 1].max() peak2_wav = data[:, 0][data[:, 1].argmax()] plt.text(peak2_wav*1.025, peak2_abs*1.1, '{}\n({} nm)'.format(peak2_abs, peak2_wav), color='black', fontsize=15) # 位置は手動で微調整... plt.xlabel("wavelength /nm", fontsize=18) plt.ylabel("abs", fontsize=18) plt.legend([i[0] for i in lines], ['pH {}'.format(i) for i in pHs], fontsize=15, loc='upper left') # plt.savefig("UV-Vis_label.png")ラベルもつきいい感じになりました!
リソース
以下のrepositoryで同じことができます。
https://github.com/YutoOhno/UV-vis/tree/master
- 投稿日:2019-01-27T14:18:18+09:00
GoogleMapsAPIで施設名、地名から住所を取得する方法 Python編
ググってもヒットしなかったため備忘録として
ジオコードから住所のみ参照する方法
必要なもの
- GoogleMap Api key
- googlemapsモジュール1
- python
例として東京タワーのジオコードをまず
gmaps.goecodeで取得します。
日本語で返してもらうため引数にlanguage='jaを指定します。import googlemaps gmaps = googlemaps.Client(key='xxxxxxxxx') results = gmaps.geocode('東京タワー', language='ja') print(results) print(type(results)) ''' 結果 [{'address_components': [{'long_name': '8', 'short_name': '8', 'types': ['premise']}, {'long_name': '2', 'short_name': '2', 'types': ['political', 'sublocality', 'sublocality_level_4']}, {'long_name': '4丁目', 'short_name': '4丁目', 'types': ['political', 'sublocality', 'sublocality_level_3']}, {'long_name': '芝公園', 'short_name': '芝公園', 'types': ['political', 'sublocality', 'sublocality_level_2']}, {'long_name': '港区', 'short_name': '港区', 'types': ['locality', 'political']}, {'long_name': '東京都', 'short_name': '東京都', 'types': ['administrative_area_level_1', 'political']}, {'long_name': '日本', 'short_name': 'JP', 'types': ['country', 'political']}, {'long_name': '105-0011', 'short_name': '105-0011', 'types': ['postal_code']}], 'formatted_address': '日本、〒105-0011 東京都港区芝公園4丁目2−8', 'geometry': {'location': {'lat': 35.6585805, 'lng': 139.7454329}, 'location_type': 'ROOFTOP', 'viewport': {'northeast': {'lat': 35.6599294802915, 'lng': 139.7467818802915}, 'southwest': {'lat': 35.6572315197085, 'lng': 139.7440839197085}}}, 'place_id': 'ChIJCewJkL2LGGAR3Qmk0vCTGkg', 'plus_code': {'compound_code': 'MP5W+C5 日本、東京', 'global_code': '8Q7XMP5W+C5'}, 'types': ['establishment', 'point_of_interest', 'premise']}] <class 'list'> '''listの中にdictが収まっている形ですね。
このなかのdictformatted_addressに目的の値が格納されているようです。results = gmaps.geocode('東京タワー', language='ja') add = [d.get('formatted_address') for d in results] # リストが排出されるので参照先を取得 print(add[0]) ''' 結果 日本、〒105-0011 東京都港区芝公園4丁目2−8 '''おお!ちゃんと取得できました。
わざわざforで回さなくともリストで格納された辞書ならgetで取得できるので楽です。(dict['key']でもいけます)
日本、の部分はこの場合必要ないので削除します。print(add[0].strip('日本、')) ''' 〒105-0011 東京都港区芝公園4丁目2−8 '''この住所が本当に東京タワーの住所なのか東京タワーのHPで調べてみましょう。
本社 東京都港区芝公園4丁目2-8 合ってましたね!東京タワー以外でもいろいろ試したのですが、ほとんど正解でした。(検索できない場合はエラーが排出されます。)
おまけ
jsonでもできました。(初めこっちでやってました。)
import requests url = 'https://maps.googleapis.com/maps/api/place/textsearch/json' q = {'query': '東京タワー', 'language': 'ja', 'key': 'xxxxxxxxxxxxxxxxxxxx'} s = requests.Session() r = s.get(url, params=q) json_o = r.json() print(json_o['results'][0]['formatted_address']) ''' 日本、〒105-0011 東京都港区芝公園4丁目2−8 '''
pipでもcondaでも入れられますが、conda持ってるならcondaで入れたほうが無難です。 ↩
- 投稿日:2019-01-27T14:11:45+09:00
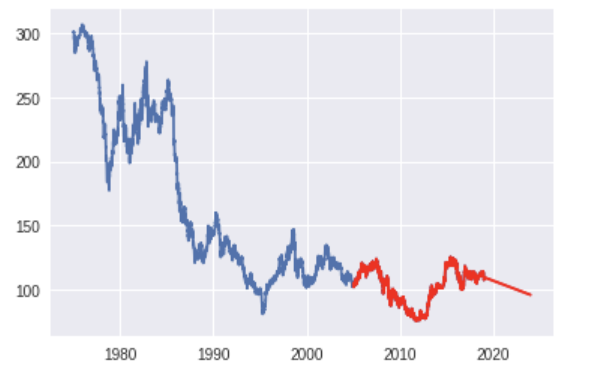
データ分析で株価予測をしてみた
時系列分析を学習したので、そのアウトプットとして
Quandlのデータを用いて株価を予測してみましたGoogleColaboratoryを使ってみました
目次
・1.データの読み込み
・2.データの整理
・3.データの可視化
・4.パラメーターの決定
・5.モデルの構築
・6.データとの予測とその可視化
・7.感想1.データの読み込み
Quandlのインストール
GoogleColaboratoryのライブラリにQuandlが入っていないためpipを用いてインストールします
!pip install quandl日経平均株価の取得
取得し、最古と最新のデータを5件ずつと、データをグラフで表示します。
最古と最新のデータから欠落があることが確認できます。%matplotlib inline import matplotlib.pyplot as plt import quandl import pandas as pd quandl.ApiConfig.api_key = "" yen_data = quandl.get('BOE/XUDLJYD') # 日経平均株価 print(yen_data.head()) print(yen_data.tail()) plt.xlabel("date") plt.ylabel("yen") plt.plot(yen_data, c="b") plt.show()2.データの整理
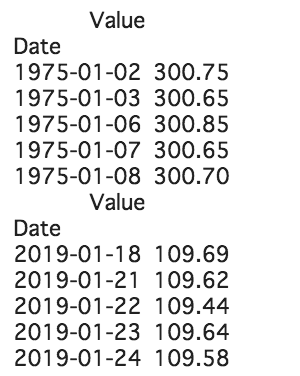
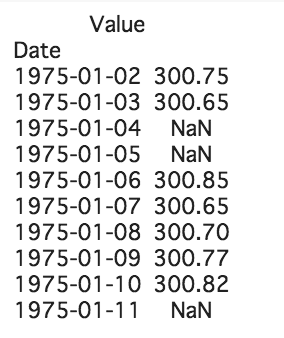
インデックスの欠落を補う
補うためにasfreqを用います。
asfreqは、現在のインデックスで同じ値を保持しながら異なる頻度に変更することができます。
このままでは値にNaN(欠損値)が見られます。yen_data = yen_data.asfreq('D') print(yen_data.head(10))欠損値を補う
fillnaを用い欠損値を前の値で置き換えます
import warnings import itertools import pandas as pd import numpy as np import statsmodels.api as sm import matplotlib.pyplot as plt %matplotlib inline index = pd.date_range("1975-01-02","2019-01-24", freq="D") yen_data.index=index yen_data = yen_data['Value'].fillna(method='ffill') print(yen_data.head()) print(yen_data.tail())3.データの可視化
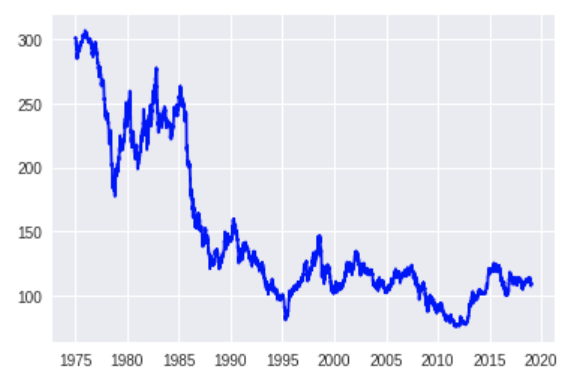
日経平均株価のデータをグラフで表します
plt.plot(yen_data, c="b") plt.show()4.パラメーターの決定
p(自己相関度), q(移動平均)を決める
自動推定関数を用いてARMA(p,q)の次数を求める
aic_min_order':(4,2)より(p,q)のベストな組み合わせが(4,2)とわかるyen_data_diff = yen_data - yen_data.shift() yen_data_diff = yen_data_diff.dropna() yen_data_diff.plot() import warnings warnings.filterwarnings('ignore') # 計算警告を非表示 # 自動ARMAパラメータ推定関数 res_selection = sm.tsa.arma_order_select_ic(yen_data_diff, ic='aic', trend='nc') res_selection5.モデルの構築
def selectparameter(DATA,s): p = d = q = range(0, 2) pdq = list(itertools.product(p, d, q)) seasonal_pdq = [(x[0], x[1], x[2], s) for x in list(itertools.product(p, d, q))] parameters = [] BICs = np.array([]) for param in pdq: for param_seasonal in seasonal_pdq: try: mod = sm.tsa.statespace.SARIMAX(DATA, order=param, seasonal_order=param_seasonal) results = mod.fit() parameters.append([param, param_seasonal, results.bic]) BICs = np.append(BICs,results.bic) except: continue return parameters[np.argmin(BICs)] # 予測 selectparameter(yen_data, 12) SARIMA_yen_data = sm.tsa.statespace.SARIMAX(yen_data, order=(4, 1, 2), seasonal_order=(0, 1, 1, 12), enforce_stationarity = False, enforce_invertibility = False).fit() pred = SARIMA_yen_data.predict("2005-01-01", "2023-12-31", freq="D")6.データとの予測ともとの時系列データの可視化
plt.plot(pred, c="r") plt.show()7.感想
今回、インデックスの欠落を補うところが大変でした。
補うためにasfreqを用いたら簡単に補えることを知らず、reindexなどを用いて補おうと苦戦していました。
何時間もかけても解決できなかったところが適切な関数を用いると一瞬で解決でき、
問題を解決したい時に適した関数が分かっている事の大切さを実感しました。
今後も学んだことはアウトプットし、力をつけていきたいです。参考記事
pandasで時系列データをリサンプリングするresample, asfreq
Pythonで時系列分析の練習(9)SARIMAモデルで未来予測
- 投稿日:2019-01-27T13:55:21+09:00
Cross-validation: KFold と StratifiledKFold の違い
目的
本ページの目的は交差検証におけるランダム性の違いを確認する。そのために、以下の内容で話を進める。
- 説明を簡単にするために iris データを利用
- Kfold 検定のランダム性ありとランダム性なしを比較
- Kfold 検定とStratifiledKFoldを比較する背景
以前までは機械学習技術を利用して作成されたモデルはブラックボックスとされてきた。しかし、近年では機械学習を利用して作成されたモデルはいくつかの方法で説明できるようになりつつある。その中の一つとして、学習データを振り分ける際のランダム性によってモデルが如何に妥当であるか、という議論もなされるようになってきた。
学術的な背景 (間違ってたらご指摘願います。学者以外はスキップ)
- Kohavi さんの論文 (被引用数 9000 オーバー)では ten-fold-Stratified-cross-validation がモデル選択にベストな方法であると述べている(ただ手法が古いけど)。データや現在の手法では異なるかもしれないが、利用する価値はありそうである。
- 恐らく、Kohavi さんの論文によると、Weiss, S. M. (1991)がStratified-cross-validationを初めに比較したように読める。
本ページを読み終えて理解すること
- 交差検証による分類対象(クラス)の振り分けを均等にするためには、StratifiledKFoldを利用する必要がある。
データの読み込み
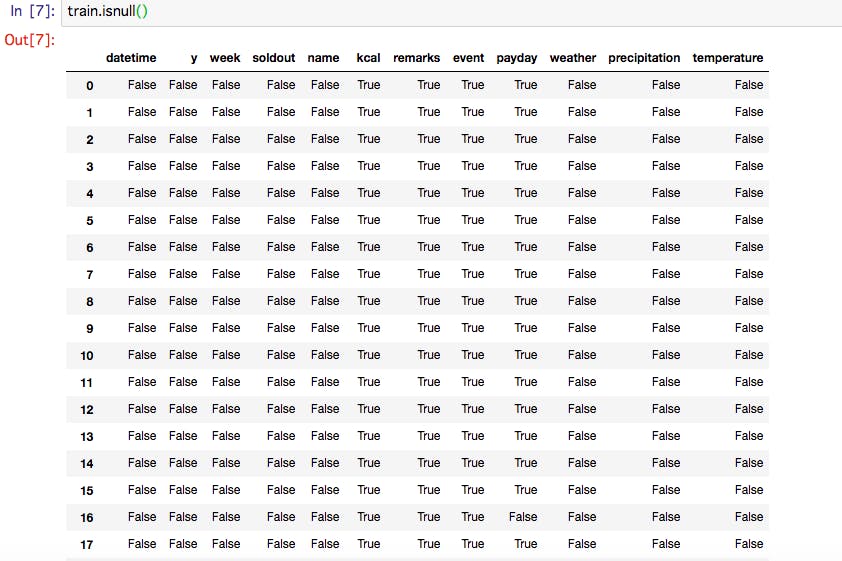
データ読み込みと変換の趣旨
- sklearn では様々なパッケージを利用する際に pandas が便利である。
- numpy 形式の iris データを pandas 形式に変換する。
- 重要変数
- data_setは機械学習の用語である特徴量(もしくは特徴変数) を表す
- target_setは機械学習の用語であるクラス (分類対象, setosa などはクラスラベル)を表す
- all_dataは data_set と target_set を結合させたもの
from sklearn.datasets import load_iris import seaborn as sns #データ読み込み iris = load_iris() #データの確認 pd.set_option('display.max_rows', 5) display(pd.DataFrame(iris.data, columns = iris.feature_names)) #データタイプの確認 print(type(iris.data), type(iris.target)) # pandas のデータフレーム に変換 (特徴量 data_set = pd.DataFrame(iris.data, columns=iris.feature_names) # クラスラベルを pandas の seriesに変換 target_set = pd.Series(iris.target) for i, val in enumerate(iris.target_names): target_set = target_set.replace(i,val) display(target_set) # これまでのデータを一つの変数にまとめる all_data = data_set.copy() all_data["target"] = target_set.copy() all_data.describe() # それぞれのクラスラベルをx軸にとり、各クラスラベルのデータを表示する sns.pairplot(data = all_data,hue="target") # 分割の仕方のメモ # X_train, X_test, y_train, y_test = train_test_split(iris.data,
sepal length (cm) sepal width (cm) petal length (cm) petal width (cm) 0 5.1 3.5 1.4 0.2 1 4.9 3.0 1.4 0.2 ... ... ... ... ... 148 6.2 3.4 5.4 2.3 149 5.9 3.0 5.1 1.8 150 rows × 4 columns
<class 'numpy.ndarray'> <class 'numpy.ndarray'> 0 setosa 1 setosa ... 148 virginica 149 virginica Length: 150, dtype: object交差検証
- 交差検証はクロスバリデーション(Cross-validation)とも呼ばれる。
- 交差検証には様々な種類がある。
- 最も代表的な K-分割交差検証 (k-fold cross-validation) と StratifiedKFold の違いを確認する。
Kfold shuffle=True
- プログラムの実行結果、クラスラベルの種類別個数の実行例は以下の通りである。
- クラスラベルの種類別個数(array(['setosa', 'versicolor', 'virginica'], dtype=object), array([8, 5, 2]))
- クラスラベルの種類別個数(array(['versicolor', 'virginica'], dtype=object), array([ 5, 10]))
- 以上の内容から、KFold ではクラスラベルの種類別個数が一致していない。
from sklearn.model_selection import StratifiedKFold, cross_validate, KFold# データを 10 分割させる。 # 分割する際には、分割させる前のデータからシャッフルする。 k = KFold(n_splits=10, shuffle=True, random_state=0) for train_index, test_index in k.split(data_set, target_set): print("分割されたデータセットの大きさ:{}".format(target_set[test_index].shape)) print("分割される以前のデータセットのどこを抽出したのか?:{}".format(test_index)) print("クラスラベル:{}".format(target_set[test_index])) print("クラスラベルの種類別個数{}".format( np.unique(target_set[test_index],return_counts=True)))分割されたデータセットの大きさ:(15,) 分割される以前のデータセットのどこを抽出したのか?:[ 7 33 40 51 54 62 63 71 73 76 86 100 107 114 134] クラスラベル:7 setosa 33 setosa ... 114 virginica 134 virginica Length: 15, dtype: object クラスラベルの種類別個数(array(['setosa', 'versicolor', 'virginica'], dtype=object), array([3, 8, 4])) 分割されたデータセットの大きさ:(15,) 分割される以前のデータセットのどこを抽出したのか?:[ 8 16 22 24 26 37 44 45 66 78 90 93 97 121 126] クラスラベル:8 setosa 16 setosa ... 121 virginica 126 virginica Length: 15, dtype: object クラスラベルの種類別個数(array(['setosa', 'versicolor', 'virginica'], dtype=object), array([8, 5, 2])) 分割されたデータセットの大きさ:(15,) 分割される以前のデータセットのどこを抽出したのか?:[ 2 10 18 27 43 59 61 83 84 92 112 127 132 137 141] クラスラベル:2 setosa 10 setosa ... 137 virginica 141 virginica Length: 15, dtype: object クラスラベルの種類別個数(array(['setosa', 'versicolor', 'virginica'], dtype=object), array([5, 5, 5])) 分割されたデータセットの大きさ:(15,) 分割される以前のデータセットのどこを抽出したのか?:[ 50 56 60 69 80 106 108 116 119 123 133 135 144 146 147] クラスラベル:50 versicolor 56 versicolor ... 146 virginica 147 virginica Length: 15, dtype: object クラスラベルの種類別個数(array(['versicolor', 'virginica'], dtype=object), array([ 5, 10])) 分割されたデータセットの大きさ:(15,) 分割される以前のデータセットのどこを抽出したのか?:[ 13 15 20 30 48 52 64 85 89 91 94 95 101 111 125] クラスラベル:13 setosa 15 setosa ... 111 virginica 125 virginica Length: 15, dtype: object クラスラベルの種類別個数(array(['setosa', 'versicolor', 'virginica'], dtype=object), array([5, 7, 3])) 分割されたデータセットの大きさ:(15,) 分割される以前のデータセットのどこを抽出したのか?:[ 3 6 11 12 46 68 96 98 102 104 109 110 120 128 149] クラスラベル:3 setosa 6 setosa ... 128 virginica 149 virginica Length: 15, dtype: object クラスラベルの種類別個数(array(['setosa', 'versicolor', 'virginica'], dtype=object), array([5, 3, 7])) 分割されたデータセットの大きさ:(15,) 分割される以前のデータセットのどこを抽出したのか?:[ 1 4 5 17 38 41 42 53 105 113 124 129 139 143 148] クラスラベル:1 setosa 4 setosa ... 143 virginica 148 virginica Length: 15, dtype: object クラスラベルの種類別個数(array(['setosa', 'versicolor', 'virginica'], dtype=object), array([7, 1, 7])) 分割されたデータセットの大きさ:(15,) 分割される以前のデータセットのどこを抽出したのか?:[ 0 23 28 31 32 34 35 55 57 65 74 75 118 131 138] クラスラベル:0 setosa 23 setosa ... 131 virginica 138 virginica Length: 15, dtype: object クラスラベルの種類別個数(array(['setosa', 'versicolor', 'virginica'], dtype=object), array([7, 5, 3])) 分割されたデータセットの大きさ:(15,) 分割される以前のデータセットのどこを抽出したのか?:[ 14 19 25 29 49 72 77 79 82 99 115 122 130 136 145] クラスラベル:14 setosa 19 setosa ... 136 virginica 145 virginica Length: 15, dtype: object クラスラベルの種類別個数(array(['setosa', 'versicolor', 'virginica'], dtype=object), array([5, 5, 5])) 分割されたデータセットの大きさ:(15,) 分割される以前のデータセットのどこを抽出したのか?:[ 9 21 36 39 47 58 67 70 81 87 88 103 117 140 142] クラスラベル:9 setosa 21 setosa ... 140 virginica 142 virginica Length: 15, dtype: object クラスラベルの種類別個数(array(['setosa', 'versicolor', 'virginica'], dtype=object), array([5, 6, 4]))Kfold shuffle=False
KFold の 引数 shuffle = False を利用すると、下記のようにシャッフルされない。
- 実行結果は例えば次の通りである。
- 分割される以前のデータセットのどこを抽出したのか?:[ 0 1 2 3 4 5 6 7 8 9 10 11 12 13 14]
- 分割される以前のデータセットのどこを抽出したのか?:[15 16 17 18 19 20 21 22 23 24 25 26 27 28 29]
- 以上よりデータの並び順で交差検証用のデータを作成していることがわかる。このまま利用すると学習時に大きな問題を抱えそう。
そのため、shuffle は 基本的に Trueが推奨されると考えられる。k = KFold(n_splits=10, shuffle=False, random_state=0) for train_index, test_index in k.split(data_set, target_set): print("分割されたデータセットの大きさ:{}".format(target_set[test_index].shape)) print("分割される以前のデータセットのどこを抽出したのか?:{}".format(test_index)) print("クラスラベル:{}".format(target_set[test_index])) print("クラスラベルの種類別個数{}".format( np.unique(target_set[test_index],return_counts=True)))分割されたデータセットの大きさ:(15,) 分割される以前のデータセットのどこを抽出したのか?:[ 0 1 2 3 4 5 6 7 8 9 10 11 12 13 14] クラスラベル:0 setosa 1 setosa ... 13 setosa 14 setosa Length: 15, dtype: object クラスラベルの種類別個数(array(['setosa'], dtype=object), array([15])) 分割されたデータセットの大きさ:(15,) 分割される以前のデータセットのどこを抽出したのか?:[15 16 17 18 19 20 21 22 23 24 25 26 27 28 29] クラスラベル:15 setosa 16 setosa ... 28 setosa 29 setosa Length: 15, dtype: object クラスラベルの種類別個数(array(['setosa'], dtype=object), array([15])) 分割されたデータセットの大きさ:(15,) 分割される以前のデータセットのどこを抽出したのか?:[30 31 32 33 34 35 36 37 38 39 40 41 42 43 44] クラスラベル:30 setosa 31 setosa ... 43 setosa 44 setosa Length: 15, dtype: object クラスラベルの種類別個数(array(['setosa'], dtype=object), array([15])) 分割されたデータセットの大きさ:(15,) 分割される以前のデータセットのどこを抽出したのか?:[45 46 47 48 49 50 51 52 53 54 55 56 57 58 59] クラスラベル:45 setosa 46 setosa ... 58 versicolor 59 versicolor Length: 15, dtype: object クラスラベルの種類別個数(array(['setosa', 'versicolor'], dtype=object), array([ 5, 10])) 分割されたデータセットの大きさ:(15,) 分割される以前のデータセットのどこを抽出したのか?:[60 61 62 63 64 65 66 67 68 69 70 71 72 73 74] クラスラベル:60 versicolor 61 versicolor ... 73 versicolor 74 versicolor Length: 15, dtype: object クラスラベルの種類別個数(array(['versicolor'], dtype=object), array([15])) 分割されたデータセットの大きさ:(15,) 分割される以前のデータセットのどこを抽出したのか?:[75 76 77 78 79 80 81 82 83 84 85 86 87 88 89] クラスラベル:75 versicolor 76 versicolor ... 88 versicolor 89 versicolor Length: 15, dtype: object クラスラベルの種類別個数(array(['versicolor'], dtype=object), array([15])) 分割されたデータセットの大きさ:(15,) 分割される以前のデータセットのどこを抽出したのか?:[ 90 91 92 93 94 95 96 97 98 99 100 101 102 103 104] クラスラベル:90 versicolor 91 versicolor ... 103 virginica 104 virginica Length: 15, dtype: object クラスラベルの種類別個数(array(['versicolor', 'virginica'], dtype=object), array([10, 5])) 分割されたデータセットの大きさ:(15,) 分割される以前のデータセットのどこを抽出したのか?:[105 106 107 108 109 110 111 112 113 114 115 116 117 118 119] クラスラベル:105 virginica 106 virginica ... 118 virginica 119 virginica Length: 15, dtype: object クラスラベルの種類別個数(array(['virginica'], dtype=object), array([15])) 分割されたデータセットの大きさ:(15,) 分割される以前のデータセットのどこを抽出したのか?:[120 121 122 123 124 125 126 127 128 129 130 131 132 133 134] クラスラベル:120 virginica 121 virginica ... 133 virginica 134 virginica Length: 15, dtype: object クラスラベルの種類別個数(array(['virginica'], dtype=object), array([15])) 分割されたデータセットの大きさ:(15,) 分割される以前のデータセットのどこを抽出したのか?:[135 136 137 138 139 140 141 142 143 144 145 146 147 148 149] クラスラベル:135 virginica 136 virginica ... 148 virginica 149 virginica Length: 15, dtype: object クラスラベルの種類別個数(array(['virginica'], dtype=object), array([15]))StratifiedKFold の検証
StratifiledKFold 関数は Kfold 関数とクラスラベルの扱いが異なる。
StratifiledKFold 関数は なるべくクラスラベルが均等に割り振られるように交差検証を行う。
引用: StratifiedKFold
- 実行結果は例えば次のようなものが挙げられる。
- クラスラベルの種類別個数(array(['setosa', 'versicolor', 'virginica'], dtype=object), array([5, 5, 5]))
以上からクラスラベルが均等に分けられた上で交差検証用のデータが作成されていることがわかる。
skf = StratifiedKFold(n_splits=10, shuffle=True, random_state=0) for train_index, test_index in skf.split(iris.data, iris.target): print("分割されたデータセットの大きさ:{}".format(target_set[test_index].shape)) print("分割される以前のデータセットのどこを抽出したのか?:{}".format(test_index)) print("クラスラベル:{}".format(target_set[test_index])) print("クラスラベルの種類別個数{}".format( np.unique(target_set[test_index],return_counts=True)))分割されたデータセットの大きさ:(15,) 分割される以前のデータセットのどこを抽出したのか?:[ 2 10 11 28 41 52 60 61 78 91 102 110 111 128 141] クラスラベル:2 setosa 10 setosa ... 128 virginica 141 virginica Length: 15, dtype: object クラスラベルの種類別個数(array(['setosa', 'versicolor', 'virginica'], dtype=object), array([5, 5, 5])) 分割されたデータセットの大きさ:(15,) 分割される以前のデータセットのどこを抽出したのか?:[ 4 22 27 31 38 54 72 77 81 88 104 122 127 131 138] クラスラベル:4 setosa 22 setosa ... 131 virginica 138 virginica Length: 15, dtype: object クラスラベルの種類別個数(array(['setosa', 'versicolor', 'virginica'], dtype=object), array([5, 5, 5])) 分割されたデータセットの大きさ:(15,) 分割される以前のデータセットのどこを抽出したのか?:[ 18 26 33 34 35 68 76 83 84 85 118 126 133 134 135] クラスラベル:18 setosa 26 setosa ... 134 virginica 135 virginica Length: 15, dtype: object クラスラベルの種類別個数(array(['setosa', 'versicolor', 'virginica'], dtype=object), array([5, 5, 5])) 分割されたデータセットの大きさ:(15,) 分割される以前のデータセットのどこを抽出したのか?:[ 7 14 29 45 48 57 64 79 95 98 107 114 129 145 148] クラスラベル:7 setosa 14 setosa ... 145 virginica 148 virginica Length: 15, dtype: object クラスラベルの種類別個数(array(['setosa', 'versicolor', 'virginica'], dtype=object), array([5, 5, 5])) 分割されたデータセットの大きさ:(15,) 分割される以前のデータセットのどこを抽出したのか?:[ 15 16 30 32 42 65 66 80 82 92 115 116 130 132 142] クラスラベル:15 setosa 16 setosa ... 132 virginica 142 virginica Length: 15, dtype: object クラスラベルの種類別個数(array(['setosa', 'versicolor', 'virginica'], dtype=object), array([5, 5, 5])) 分割されたデータセットの大きさ:(15,) 分割される以前のデータセットのどこを抽出したのか?:[ 8 13 20 25 43 58 63 70 75 93 108 113 120 125 143] クラスラベル:8 setosa 13 setosa ... 125 virginica 143 virginica Length: 15, dtype: object クラスラベルの種類別個数(array(['setosa', 'versicolor', 'virginica'], dtype=object), array([5, 5, 5])) 分割されたデータセットの大きさ:(15,) 分割される以前のデータセットのどこを抽出したのか?:[ 1 5 17 40 49 51 55 67 90 99 101 105 117 140 149] クラスラベル:1 setosa 5 setosa ... 140 virginica 149 virginica Length: 15, dtype: object クラスラベルの種類別個数(array(['setosa', 'versicolor', 'virginica'], dtype=object), array([5, 5, 5])) 分割されたデータセットの大きさ:(15,) 分割される以前のデータセットのどこを抽出したのか?:[ 6 12 23 24 37 56 62 73 74 87 106 112 123 124 137] クラスラベル:6 setosa 12 setosa ... 124 virginica 137 virginica Length: 15, dtype: object クラスラベルの種類別個数(array(['setosa', 'versicolor', 'virginica'], dtype=object), array([5, 5, 5])) 分割されたデータセットの大きさ:(15,) 分割される以前のデータセットのどこを抽出したのか?:[ 9 19 21 36 39 59 69 71 86 89 109 119 121 136 139] クラスラベル:9 setosa 19 setosa ... 136 virginica 139 virginica Length: 15, dtype: object クラスラベルの種類別個数(array(['setosa', 'versicolor', 'virginica'], dtype=object), array([5, 5, 5])) 分割されたデータセットの大きさ:(15,) 分割される以前のデータセットのどこを抽出したのか?:[ 0 3 44 46 47 50 53 94 96 97 100 103 144 146 147] クラスラベル:0 setosa 3 setosa ... 146 virginica 147 virginica Length: 15, dtype: object クラスラベルの種類別個数(array(['setosa', 'versicolor', 'virginica'], dtype=object), array([5, 5, 5]))参考文献
- 投稿日:2019-01-27T13:17:39+09:00
相関関係を確認
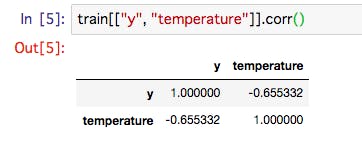
相関関係とは
相関関係とは、Aという事象とBという事象の間、双方向の動きに関係があること。
例えば、気温が上がると弁当の売り上げ数もあがる関係があった場合、正の相関がある。
逆に、気温が上がると弁当の売り上げが下がる関係があった場合、負の相関があると言う。なお、相関関係と因果関係は異なる為、注意が必要。
この関係の度合は相関係数と呼ばれる数値で表されます
(相関関係の度合い(強さ))を表すtrain[["y", "temperature"]].corr()** -1 ~ 1 の間。**
- -1 に近いほど、負の関数。
- 1 に近いほど、正の関数。
相関関係は欠損値を測定できないため、自動的に欠損値の行は削除される。
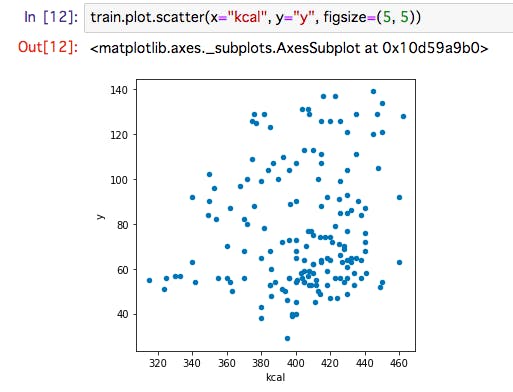
train[["y", "kcal"]].corr()相関関係の散布図を描画。
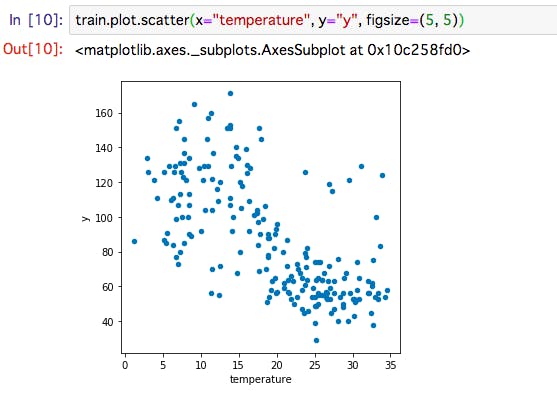
- 相関関係は散布図を書いて見るとわかりやすい。
train.plot.scatter(x="temperature", y="y", figsize=(5, 5))temperatureが高いほど、yの値は減っているということが視覚的にわかる。
その他 一例
- 関係性は見られず、満遍なくバラバラ。
- つまり、これらの相関関係は無いに等しい
数値だけではなく、散布図を用いて確認することを強く推奨。
もし、1つでも外れ値があった場合、数値だけでは発見できず、想定外の動きをすることが。
- 投稿日:2019-01-27T09:50:24+09:00
Python内のデフォルトパスを通す方法(Windows, Linux)
やりたいこと
自分のpyファイルを
sysなどのモジュールのようにimportして使いたい。次のような書き方を、
import sys sys.path例えば自分が作った
mypy.pyでもやりたい。import mypy mypy.func()つまり、Pythonの中でパスを通したい。
2019年の今、色んなページにこの方法(全部または一部)が書かれていますが、ちょっとハマってしまったのでQiitaに書きます。
venvで仮想環境を作るほどでもないときに使うのだと思います。方法
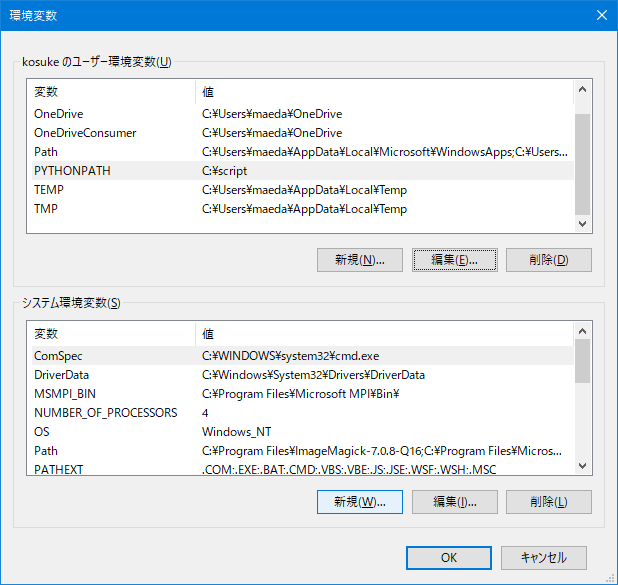
PYTHONPATHというPythonが使用する環境変数があります。
PYTHONPATHの値に、自分のpyファイルが置いてあるフォルダのパスを入力すればOKです。
PYTHONPATHの公式の説明が読みたい方は、こちらへ。https://docs.python.jp/3/using/cmdline.html#envvar-PYTHONPATH
Windowsで
PYTHONPATHを新規作成する例えば、自分のpyファイル
mypy.pyがC:\scriptにあるとします。フォルダ構成はこんな感じだとして、
C: \script \mypy.py \mypy2.py \sub-script subpy.pyエクスプローラーを開いて、
左ナビゲーションバーのPCを右クリック > プロパティ > システムの詳細設定 > 環境変数 > 新規
にて環境変数
PYTHONPATHを入力し、値にフォルダのパスを入力します。複数のフォルダを指定したい場合は、
C:\script;C:\script2のように;つなぎです。そして、もし、SpyderやAnaconda Promptを起動していたら、再起動してください(Windowsの再起動は不要です)。
パスが通せたか確認する
その後、
import sys sys.pathを実行すれば、
C:\\scriptがパスに追加されていることが確認できます(\は自動で\\になります)。In[11]: sys.path Out[11]: ['C:\\Users\\maech', 'C:\\script', ... <-- 追加されている自分のpyファイルをimportする
これで、
C:\script\*.pyのすべてのpyファイルがimportできます。# C:\script\mypy.pyをインポート import mypy # mypy.pyに定義したfunc()を使う a = mypy.func() # mypy.pyに定義したクラスmypyのfunc()を使う a = mypy.mypy.func() # この場合は from mypy import mypy とすると a = mypy.func() で呼び出せる # C:\script\mypy2.pyをインポート import mypy2 # C:\script\sub-script\subpy.pyをインポート import sub-script.subpyLinuxでやりたい人
例えば、
~/.bashrcにPYTHONPATHと値を追加してください。複数パスの指定は:つなぎです。詳しくは省略。
参考
PYTHONPATH — 1. コマンドラインと環境 — Python 3.6.5 ドキュメント https://docs.python.jp/3/using/cmdline.html#envvar-PYTHONPATH
Pythonでimportの対象ディレクトリのパスを確認・追加(sys.pathなど) | note.nkmk.me https://note.nkmk.me/python-import-module-search-path/
python - Setting PYTHONPATH on Windows under Anaconda without elevated privileges - Stack Overflow https://stackoverflow.com/questions/43099096/setting-pythonpath-on-windows-under-anaconda-without-elevated-privileges
- 投稿日:2019-01-27T01:53:31+09:00
Machine Learning 欠損値を確認
欠損値とは?
- 何らかの理由で値が入っていない状態のこと。
- データ分析において欠損がある場合の対応はとても重要
train.isnull()これだとわかりづらいので...
カラムにNullが1つでもある場合、Trueを返す。
train.isnull().any()各カラムにいくつNullが存在するか
train.isnull().sum()欠損値の取り扱い方
基本的には二択
- 別のなんらかの値を代入(補間)する。
欠損値を含む行を削除する。
まずは、欠損値を0で補完
train.fillna(0) // nullに0を代入
- 欠損値を削除
train.dropna(subset=["kcal"])kcalカラムが存在する行のみ表示された。
欠損の見落としを確認
カラム特有の表現の仕方(null以外)で、欠損値を表現しているケース
- 指定カラムの行のそれぞれの値がいくつあるか
train["precipitation"].value_counts()