- 投稿日:2020-09-13T23:56:39+09:00
【Python】"copy()"したのにコピーされていない?深いコピーに関する思い込みと失敗
投稿日:2020/9/13
はじめに
この記事では参照の代入、浅いコピー、深いコピーについて書いています。すでに複数の記事がありますが、この記事では私が間違いに気づいた時の状況や、その他調べている中で新しく知った内容を含めています。
私は浅いコピーと深いコピーを理解できておらず、参照の代入=浅いコピー、
.copy()=深いコピーであると思っていました。しかし、今回の失敗で調べてみて、代入には3種類あることを知りました。3つの代入
参照の代入
a = [1,2] b = a b[0] = 100 print(a) # [100, 2] print(b) # [100, 2]こうすると、
bを書き換えるとaも書き換わりますよね。これが参照の代入1です。aとbは同じ物(オブジェクト)を指しているため、片方を書き換えるともう片方も書き換わったように見えます。
id()でオブジェクトIDを確認してみましょう。print(id(a)) # 2639401210440 print(id(b)) # 2639401210440 print(a is b) # Trueidが同じですね。
aとbが同じであることがわかります。浅いコピー
では、
bをaとは別のオブジェクトとして扱いたい場合はどうすれば良いでしょうか。私は普段.copy()を使っています。a = [1,2] b = a.copy() # 浅いコピー b[0] = 100 print(a, id(a)) # [1, 2] 1566893363784 print(b, id(b)) # [100, 2] 1566893364296 print(a is b) # Falseちゃんと
aとbが分かれていますね。これが浅いコピーです。浅いコピーは他にも方法があります。リストの浅いコピー# リストの浅いコピー import copy a = [1,2] b = a.copy() # .copy()を使う例 b = copy.copy(a) # copyモジュールを使う例 b = a[:] # スライスを使う例 b = [x for x in a] # リスト内包表記を使う例 b = list(a) # list()を使う例辞書の浅いコピー# 辞書の浅いコピー import copy a = {"hoge":1, "piyo":2} b = a.copy() # .copy()を使う例 b = copy.copy(a) # copyモジュールを使う例 b = dict(a.items()) # items()で取り出したものを辞書にする例深いコピー?
では、深いコピーをやってみましょう。
import copy a = [1,2] b = copy.deepcopy(a) # 深いコピー b[0] = 100 print(a, id(a)) # [1, 2] 2401980169416 print(b, id(b)) # [100, 2] 2401977616520 print(a is b) # False結果は浅いコピーと変わらないですね。
"copy()"したのにコピーされていない
では、次の例はどうでしょうか。
a = [[1,2], [3,4]] # 変更 b = a.copy() b[0][0] = 100 print(a) # [[100, 2], [3, 4]] print(b) # [[100, 2], [3, 4]]一行目の
aを二次元リストにしました。
コピーを取ったはずなのに、aも書き換わっていますね。先程の例との違いはなんでしょうか。ミュータブルオブジェクト
Pythonにはミュータブル(変更可能)オブジェクトとイミュータブル(変更不可能)オブジェクトがあります。分類すると,
ミュータブル:list, dict, numpy.ndarray2など
イミュータブル:int, str, tupleなどという感じです3 4。上の例ではリスト(list)の中にリストを入れています。つまりミュータブルオブジェクトの中にミュータブルオブジェクトを入れました。では、同じオブジェクトかどうかを確認しましょう。
print(a is b, id(a), id(b)) # False 2460506990792 2460504457096 print(a[0] is b[0], id(a[0]), id(b[0])) # True 2460503003720 2460503003720外側のリスト
abは異なりますが、その中のリストa[0]b[0]は同じです。つまり、b[0]を書き換えるとa[0]も書き換わります。よってこの挙動は、オブジェクトの中にミュータブルオブジェクトが入っているのに浅いコピーを使ったことが原因です5。そして、このようなときに用いるのが深いコピーです。
解決策 - 深いコピー
1.深いコピーを使う
深いコピーを使います。
import copy a = [[1,2], [3,4]] b = copy.deepcopy(a) b[0][0] = 100 print(a) # [[1, 2], [3, 4]] print(b) # [[100, 2], [3, 4]]同じオブジェクトかどうかも見てみましょう。
print(a is b, id(a), id(b)) # False 2197556646152 2197556610760 print(a[0] is b[0], id(a[0]), id(b[0])) # False 2197556617864 2197556805320 print(a[0][1] is b[0][1], id(a[0][1]), id(b[0][1])) # True 140736164557088 140736164557088あれ、最後は同じですね。
b[0][1]はイミュータブルオブジェクトであるintであり、再代入時に自動的に別オブジェクトが作られるので問題ありません6。それを除けば、ミュータブルオブジェクトはidが異なるので、コピーされたことがわかります。
2. numpy.ndarrayを使う
今回の内容とは少しずれているということと、余計難しくなるような内容なので、下に持っていきました。「解決策その2 numpy.ndarrayにする」の項を見てください。
私が間違いに気づいたときのコード
私が間違いに気づいた時とほぼ同じコードを掲載します。私は以下のようなデータを作成しました。
import numpy as np a = {"data":[ {"name": "img_0.jpg", "size":"100x200", "img": np.zeros((100,200))}, {"name": "img_1.jpg", "size":"100x100", "img": np.zeros((100,100))}, {"name": "img_2.jpg", "size":"150x100", "img": np.zeros((150,100))}], "total_size": 5000 }このように、辞書の中にリストを、その中に辞書、さらに画像(ndarray)といった感じで、ミュータブルオブジェクトを入れ子にしたデータを作成しました。そして、この中から
imgだけを省いた別の辞書をjson書き出し用に作成しました。その後、元の辞書から
imgを取り出そうとするとKeyErrorが発生しました。コピーしたはずなのにどうしてだろう、としばらく悩んで、辞書の中のオブジェクトの参照が同じである可能性に気づきました。# 問題を起こしたコード data = a["data"].copy() # ここが間違い for i in range(len(data)): del data[i]["img"] # 辞書からimgを削除 b = {"data":data, "total_size":a["total_size"]} # 新しい辞書 img_0 = a["data"][0]["img"] # aを触っていないのにKeyError # KeyError: 'img'解決方法としては
data = copy.deepcopy(a["data"])のように深いコピーに変更するのが一番簡単ですが、この場合、後で消す画像をわざわざコピーすることになり、メモリや実行速度に影響が出る可能性があります。ゆえに、元データから不要なデータを消すのではなく、必要なデータを取り出す形で書くのが良いと思います。
# 必要なデータを取り出す形で書きなおしたコード data = [] for d in a["data"]: new_dict = {} for k in d.keys(): if(k=="img"): # imgだけ含めない continue new_dict[k] = d[k] # コピーではないので注意 data.append(new_dict) b = {"data":data, "total_size":a["total_size"]} # 新しい辞書 img_0 = a["data"][0]["img"] # 動作する私は、コピーしたデータをjson形式で書き出すために使ったので、上のコードで問題ないですが、もしコピー後のデータを書き換えるのであればdeepcopyを使わなければなりません(ミュータブルオブジェクトを含む場合)。
浅いコピーと深いコピー
以上の例からも分かる通り、
浅いコピー:対象のオブジェクトのみ
深いコピー:対象のオブジェクト+対象のオブジェクトに含まれるミュータブルオブジェクト全てがコピーされます。詳しくは、Pythonのドキュメント(copy) をご覧ください。一度目を通しておくと良いと思います。
実行速度検証
テキストを含む辞書
aを作成し、浅いコピーと深いコピーの実行速度テストを行いました。import copy import time import numpy as np def test1(a): start = time.time() # b = a # b = a.copy() # b = copy.copy(a) b = copy.deepcopy(a) process_time = time.time()-start return process_time a = {i:"hogehoge"*100 for i in range(10000)} res = [] for i in range(100): res.append(test1(a)) print(np.average(res)*1000, np.min(res)*1000, np.max(res)*1000)結果
処理 平均(ms) 最小(ms) 最大(ms) b=a 0.0 0.0 0.0 a.copy() 0.240 0.0 1.00 copy.copy(a) 0.230 0.0 1.00 copy.deepcopy(a) 118 78.0 414 適当な検証なのであまりあてになりませんが、浅いコピーと深いコピーの差が大きいことは分かります。よって書き換えしないデータは浅いコピーを使うなど、使用するデータや使用方法によって使い分けたほうがよさそうです。
その他検証等
自作クラスのコピー
import copy class Hoge: def __init__(self): self.a = [1,2,3] self.b = 3 hoge = Hoge() # hoge_copy = hoge.copy() # copyメソッドがないのでエラー hoge_copy = copy.copy(hoge) # 浅いコピー hoge_copy.a[1] = 10000 hoge_copy.b = 100 print(hoge.a) # [1, 10000, 3](書き換わっている) print(hoge.b) # 3 (書き換わっていない)自作クラスの場合も、メンバ変数がミュータブルオブジェクトなら浅いコピーだけでは不十分です。
タプルのコピー
タプルと言っても、タプルの中にミュータブルオブジェクトを入れた場合です。
import copy a = ([1,2],[3,4]) b = copy.copy(a) # 浅いコピー print(a) # ([1, 2], [3, 4]) b[0][0] = 100 # これが実行できる print(a) # ([100, 2], [3, 4])(書き換わっている) b[0] = [100,2] # TypeErrorで書き換え不可タプルはイミュータブルなので値の書き換えができませんが、タプルに含まれるミュータブルオブジェクトの中は書き換えができてしまいます。この場合も同様に浅いコピーでは中のオブジェクトはコピーされません。
リストの
.copy()についてリストのコピー
b = a.copy()について、どのような処理になっているのか気になったので、Pythonのソースコードを見てみました。cpytnon/Objects/listobject.c 812行目 (2020/9/11現在のmasterブランチより引用)
ソースのリンク(位置は変わっているかもしれません)/*[clinic input] list.copy Return a shallow copy of the list. [clinic start generated code]*/ static PyObject * list_copy_impl(PyListObject *self) /*[clinic end generated code: output=ec6b72d6209d418e input=6453ab159e84771f]*/ { return list_slice(self, 0, Py_SIZE(self)); }コメントにもあるように、
Return a shallow copy of the list.
と、浅いコピー(shallow copy)であることが書かれています。その下の実装も
list_sliceと書いてあるので、b = a[0:len(a)]のようにスライスしているだけだと思われます。解決策その2-numpy.ndarrayにする
今回の話とは少しずれますが、多次元配列を扱うのであればリストではなくNumPyのndarrayを使う方法もあります。ただし、注意が必要です。
import numpy as np import copy a = [[1,2],[3,4]] a = np.array(a) # ndarrayに変換 b = copy.copy(a) # b = a.copy() #これでも可 b[0][0] = 100 print(a) # [[1 2] # [3 4]] print(b) # [[100 2] # [ 3 4]]このように、
copy.copy()を使うか、.copy()を使えば問題ありませんが、スライスを使うとリストと同じく、元の配列が書き換わってしまいます。これは、NumPyのcopyとviewの違いによるものです。参考:https://deepage.net/features/numpy-copyview.html (NumPyのコピー(copy)とビュー(view)を分かりやすく解説)
# スライスを使った場合 import numpy as np a = [[1,2], [3,4]] a = np.array(a) b = a[:] # スライス(= viewを作成) # b = a[:,:] # これでも同じ # b = a.view() # これと同じ b[0][0] = 100 print(a) # [[100 2] # [ 3 4]] print(b) # [[100 2] # [ 3 4]]また、この場合は
isによる比較結果がリストと同じようにはなりません。import numpy as np def check(a, b): print(id(a[0]), id(b[0])) print(a[0] is b[0], id(a[0])==id(b[0])) # リストをスライスした場合 a = [[1,2],[3,4]] b = a[:] check(a,b) # 1778721130184 1778721130184 # True True # ndarrayをスライス(viewを作成)した場合 a = np.array([[1,2],[3,4]]) b = a[:] check(a,b) # 1778722507712 1778722507712 # False True最後の行を見ればわかるように、idは同じですが,isによる比較結果がFalseになっています。そのため、
is演算子でオブジェクトの同一性を確認して、Falseであったとしても書き換わってしまうことがあるので注意しなければなりません。
is演算子について調べるとidが同じかどうかを返すと書かれていますが7 8 9、この場合そうはなっていません。numpyが特殊なのでしょうか。よくわかりません。実行環境はPython3.7.4 & numpy1.16.5+mklです。
おわりに
私はこれまで
.copy()で困ったことがなかったので、コピーについて全く気にしたことがありませんでした。これまでに書いたコードの中に、思いがけず書き換えを行っているデータがあるかもしれないと思うと非常に恐ろしいです。Pythonにおけるミュータブルオブジェクトの問題には関数のデフォルト引数の話もあります。こちらも知らないと気づかずに意図しないデータを生成することになるので、ご存知ない方は確認してみてください。
http://amacbee.hatenablog.com/entry/2016/12/07/004510 (Pythonの値渡しと参照渡し)
https://qiita.com/yuku_t/items/fd517a4c3d13f6f3de40 (引数のデフォルト値はimmutableなものにする)# デフォルト引数にミュータブルオブジェクトを指定しないほうが良い def hoge(data=[1,2]): # 悪い例 def hoge(data=None): # 良い例1 def hoge(data=(1,2)): # 良い例2# こういうことも起きる a = [[0,1]] * 3 print(a) # [[0, 1], [0, 1], [0, 1]] a[0][0] = 3 print(a) # [[3, 1], [3, 1], [3, 1]] (全部書き換わっている)参考
[1] https://qiita.com/Kaz_K/items/a3d619b9e670e689b6db (Pythonのcopyとdeepcopyについて)
[2] https://www.python.ambitious-engineer.com/archives/661 (copyモジュール 浅いコピーと深いコピー)
[3] https://snowtree-injune.com/2019/09/16/shallow-copy/ ( Python♪次は理屈で覚えよう「参照渡し」「浅いコピー」「深いコピー」)
[4] https://docs.python.org/ja/3/library/copy.html (copy --- 浅いコピーおよび深いコピー操作)
参照の代入と書きましたが、Pythonにおいてインターネット上でそのような言い方は見つかりませんでした。「参照渡し」は関数の引数で用いられる用語であり、他に良さそうな言い方が見つからなかったので、参照の代入としました。 ↩
ndarrayはイミュータブルにすることも可能らしい(https://note.nkmk.me/python-numpy-ndarray-immutable-read-only/ ) ↩
https://hibiki-press.tech/python/data_type/1763 (主な組み込み型のミュータブル、イミュータブル、イテラブル) ↩
https://gammasoft.jp/blog/python-built-in-types/ (Pythonの組み込みデータ型の分類表(ミュータブル等)) ↩
Pythonのドキュメント には、「複合オブジェクト(リストやクラスインスタンスのような他のオブジェクトを含むオブジェクト)」と書いてあります。よって正確にいうと、複合オブジェクトを浅いコピーしたのが原因です。別項で書きましたが、イミュータブルであるタプルの中にリストを入れても同じことが発生します。 ↩
https://atsuoishimoto.hatenablog.com/entry/20110414/1302750443 (is演算子のふしぎ)Pythonはメモリ削減のためにイミュータブルオブジェクトは使いまわすらしい。 ↩
https://www.python-izm.com/tips/difference_eq_is/ (==とisの違い) ↩
https://qiita.com/i13602/items/6d8914e019c13e858c72 (pythonにおける「==」と「is」の違いについて) ↩
https://docs.python.org/ja/3/reference/expressions.html#is (6.10.3. 同一性の比較)公式リファレンスにも「オブジェクトの同一性はid()関数を使って判定されます。」と書いてある。 ↩
- 投稿日:2020-09-13T23:44:09+09:00
2020年にSelenium (+Python)でスクレイピングしてハマったところ
iframe
症状:「実行画面では表示されているボタンなのにElementが見つからないと言われる!」
→原因と解決策:iframeはいちいち切り替えないとelementが見つけられない
driver.switch_to.frame(driver.find_elements_by_tag_name("iframe")[0]) driver.find_element_by_css_selector("#hogefuga").click() driver.switch_to.default_content()Popupによる干渉
症状:ボタンのelementを取得できているのに押せない
エラー文面:Element <a class="hoge" href="/fuga">...</a> is not clickable at point (748, 591). Other element would receive the click: <div class="piyo">...</div>→原因と解決策:一旦Bodyなどをクリックしておく
driver.find_element_by_tag_name("body").click()
- 投稿日:2020-09-13T23:08:34+09:00
AtCoder Beginner Contest 178 参戦記
AtCoder Beginner Contest 178 参戦記
ABC178A - Not
1分半で突破. xor を使っちゃったけど、タイトルは Not だった(笑).
x = int(input()) print(x ^ 1)ABC178B - Product Max
2分で突破. 端同士のどれかが最大のはずだ.
a, b, c, d = map(int, input().split()) print(max(a * c, a * d, b * c, b * d))ABC178C - Ubiquity
5分で突破. すべての組み合わせの数から、0を含まない組み合わせの数と9を含まない組み合わせの数を引いて、それだと0と9を両方含まない組み合わせの数がダブって引かれているので足し戻したものが答え.
m = 1000000007 N = int(input()) print((pow(10, N, m) - pow(9, N, m) * 2 + pow(8, N, m)) % m)ABC178D - Redistribution
29分で突破. 数列の長さが n の時に S - 3×n を n 個に分配する場合分けの数ってどうするんだって唸っていたら、仕切り棒を入れると思うんだと ABC132D - Blue and Red Balls を思い出して解けた. n種類のものから重複を許してr個選ぶ場合の数はnHr=n+r−1Cr通り.
def make_factorial_table(n): result = [0] * (n + 1) result[0] = 1 for i in range(1, n + 1): result[i] = result[i - 1] * i % m return result def mcomb(n, k): if n == 0 and k == 0: return 1 if n < k or k < 0: return 0 return fac[n] * pow(fac[n - k], m - 2, m) * pow(fac[k], m - 2, m) % m m = 1000000007 S = int(input()) fac = make_factorial_table(S + 10) result = 0 for i in range(1, S // 3 + 1): result += mcomb(S - i * 3 + i - 1, i - 1) result %= m print(result)ABC178E - Dist Max
突破できず. この問題がこんなに解かれているということはと、確信を持って「マンハッタン距離最大」でググったらやっぱりでてきましたね orz.
- 投稿日:2020-09-13T23:02:01+09:00
Keras 多クラス分類 Iris
環境
- Tensorflow 2.3.0
- Google Colaboratory
イントロダクション
- (注意) 重きは、多クラス分類の内容ではなく、tf.kerasを使用した事への内容です
- Tensorflow kerasにより多層パーセプトロン(MLP)を作成
- 簡単なtf.kerasの使い方とモデルの保存、保存したモデルの読込を行う
- kaggleをやっている際、必要にかられ、kerasコードの読み方を今一度確認しました
データセット
- scikit-learnに付属のアヤメ品種データ(Iris plants dataset)を用いて検証
- Irisデータセットには(setosa, versicolor, virginica)の3種の情報が格納されている為、多クラス分類に使える
ソースコード全体
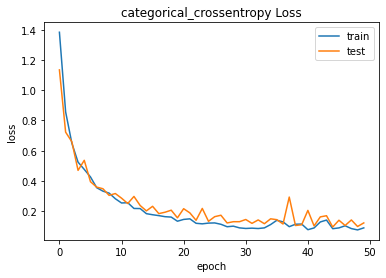
import numpy as np from sklearn.datasets import load_iris from sklearn.model_selection import train_test_split from tensorflow.keras.utils import to_categorical import tensorflow as tf import matplotlib.pyplot as plt # irisデータセット読込 iris = load_iris() # データセットを訓練データ, テストデータに分割 data_X = iris.data data_y = to_categorical(iris.target) # one-hotエンコーディング train_X, test_X, train_y, test_y = train_test_split(data_X, data_y, test_size=0.3, random_state=0) # モデルの構築 model = tf.keras.models.Sequential([ tf.keras.layers.Input(4), tf.keras.layers.Dense(100, activation='relu'), tf.keras.layers.Dense(3, activation='softmax') ]) # モデルのコンパイル model.compile(optimizer=tf.keras.optimizers.Adam(learning_rate=0.01), loss='categorical_crossentropy', metrics=['accuracy']) # モデルの学習 result = model.fit(train_X, train_y, batch_size=32, epochs=50, validation_data=(test_X, test_y), verbose=1) # Accuracyのプロット plt.figure() plt.title('Accuracy') plt.xlabel('epoch') plt.ylabel('accuracy') plt.plot(result.history['accuracy'], label='train') plt.plot(result.history['val_accuracy'], label='test') plt.legend() # Lossのプロット plt.figure() plt.title('categorical_crossentropy Loss') plt.xlabel('epoch') plt.ylabel('loss') plt.plot(result.history['loss'], label='train') plt.plot(result.history['val_loss'], label='test') plt.legend() plt.show() # model.evaluateを使用し、学習を終えたモデルの誤差と精度を呼び出し train_score = model.evaluate(train_X, train_y) test_score = model.evaluate(test_X, test_y) print('Train loss:', train_score[0]) print('Train accuracy:', train_score[1]) print('Test loss:', test_score[0]) print('Test accuracy:', test_score[1]) # 結果の確認(predict) pred_train = model.predict(train_X) pred_test = model.predict(test_X) pred_train = np.argmax(pred_train, axis=1) pred_test = np.argmax(pred_test, axis=1) print(pred_train) print(np.argmax(train_y, axis=1)) print(pred_test) print(np.argmax(test_y, axis=1))データロード
- scikit-learnを使いirisデータのロードを行う
from sklearn.datasets import load_iris # irisデータセット読込 iris = load_iris()# サイズを確認 (iris.data.shape), (iris.target.shape) # ((150, 4), (150,)) print(iris.feature_names) # ラベルの確認 print(iris.data) # 説明変数 print(iris.target_names) # ラベルの確認 print(iris.target) # 目的変数 # データセットのタイプを確認 type(iris.data) type(iris.target) # numpy.ndarray
- 説明変数
['sepal length (cm)', 'sepal width (cm)', 'petal length (cm)', 'petal width (cm)'][[5.1 3.5 1.4 0.2] [4.9 3. 1.4 0.2] [4.7 3.2 1.3 0.2] [4.6 3.1 1.5 0.2] [5. 3.6 1.4 0.2]...
- 目的変数
['setosa' 'versicolor' 'virginica']array([0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2])データ処理
- irisの目的変数をニューラルネットワークで扱いやすいone-hotエンコーディングへ変換
from tensorflow.keras.utils import to_categorical # one-hotエンコーディング data_y = to_categorical(iris.target) # 0 => [1, 0, 0] # 1 => [0, 1, 0] # 2 => [0, 0, 1]
- 以下の様にも記載可能
# 例: クラスベクトル(整数)をバイナリクラス行列に変換 to_categorical(iris.target, num_classes=None, dtype='float32')
詳細は以下参照
https://www.tensorflow.org/api_docs/python/tf/keras/utils/to_categorical続いてデータセットを訓練データ, テストデータへ分割
# データセットを訓練データ, テストデータへ分割 from sklearn.model_selection import train_test_split data_X = iris.data data_y = to_categorical(iris.target) # one-hotエンコーディング train_X, test_X, train_y, test_y = train_test_split(data_X, data_y, test_size=0.3, random_state=0)
- test_size=0.3
- 学習データ7割, テストデータ3割へ分割
- random_state ランダムシードを設定
ネットワークアーキテクチャ
- 入力層 4変数
- 中間層 100変数
- 出力層 3変数
- 活性化関数 relu
- 損失関数 categorical_crossentropy
- 最適化関数Adam
import tensorflow as tf # モデルの構築 model = tf.keras.models.Sequential([ tf.keras.layers.Input(4), tf.keras.layers.Dense(100, activation='relu'), tf.keras.layers.Dense(3, activation='softmax') ]) # モデルのコンパイル model.compile(optimizer=tf.keras.optimizers.Adam(learning_rate=0.01), loss='categorical_crossentropy', metrics=['accuracy']) # モデルの学習 result = model.fit(train_X, train_y, batch_size=32, epochs=50, validation_data=(test_X, test_y), verbose=1)Epoch 1/50 4/4 [==============================] - 0s 36ms/step - loss: 1.0097 - accuracy: 0.5810 - val_loss: 0.7336 - val_accuracy: 0.6222 Epoch 2/50 4/4 [==============================] - 0s 6ms/step - loss: 0.7082 - accuracy: 0.7048 - val_loss: 0.6553 - val_accuracy: 0.6000 Epoch 3/50 4/4 [==============================] - 0s 5ms/step - loss: 0.5494 - accuracy: 0.7905 - val_loss: 0.4738 - val_accuracy: 0.9111
- model.fitのverbose=0を設定する事により途中結果の出力をしない
モデル評価
# model.evaluateを使用し、学習を終えたモデルの誤差と精度を呼び出し train_score = model.evaluate(train_X, train_y) test_score = model.evaluate(test_X, test_y) print('Train loss:', train_score[0]) print('Train accuracy:', train_score[1]) print('Test loss:', test_score[0]) print('Test accuracy:', test_score[1])4/4 [==============================] - 0s 2ms/step - loss: 0.0649 - accuracy: 0.9714 2/2 [==============================] - 0s 2ms/step - loss: 0.1223 - accuracy: 0.9778 Train loss: 0.06492960453033447 Train accuracy: 0.9714285731315613 Test loss: 0.12225695699453354 Test accuracy: 0.9777777791023254# 結果の確認(predict) pred_train = model.predict(train_X) pred_test = model.predict(test_X) pred_train = np.argmax(pred_train, axis=1) pred_test = np.argmax(pred_test, axis=1) print(pred_train) print(np.argmax(train_y, axis=1)) print(pred_test) print(np.argmax(test_y, axis=1))[1 2 2 2 2 1 2 1 1 2 1 2 2 1 2 1 0 2 1 1 1 1 2 0 0 2 1 0 0 1 0 2 1 0 1 2 1 0 2 2 2 2 0 0 2 2 0 2 0 2 2 0 0 1 0 0 0 1 2 2 0 0 0 1 1 0 0 1 0 2 1 2 1 0 2 0 2 0 0 2 0 2 1 1 1 2 2 1 1 0 1 2 2 0 1 1 1 1 0 0 0 2 1 2 0] [1 2 2 2 2 1 2 1 1 2 2 2 2 1 2 1 0 2 1 1 1 1 2 0 0 2 1 0 0 1 0 2 1 0 1 2 1 0 2 2 2 2 0 0 2 2 0 2 0 2 2 0 0 2 0 0 0 1 2 2 0 0 0 1 1 0 0 1 0 2 1 2 1 0 2 0 2 0 0 2 0 2 1 1 1 2 2 1 1 0 1 2 2 0 1 1 1 1 0 0 0 2 1 2 0] [2 1 0 2 0 2 0 1 1 1 2 1 1 1 1 0 1 1 0 0 2 1 0 0 2 0 0 1 1 0 2 1 0 2 2 1 0 2 1 1 2 0 2 0 0] [2 1 0 2 0 2 0 1 1 1 2 1 1 1 1 0 1 1 0 0 2 1 0 0 2 0 0 1 1 0 2 1 0 2 2 1 0 1 1 1 2 0 2 0 0]モデルの保存
# jsonファイルでモデルを保存 # 重みをhdf5で保存 config = model.to_json() with open('model.json','w') as file: file.write(config) model.save_weights('weights.hdf5')モデルの読込
with open('model.json','r') as file: model_json = file.read() model = tf.keras.models.model_from_json(model_json) model.load_weights('weights.hdf5') model.compile(optimizer=tf.keras.optimizers.Adam(learning_rate=0.01), loss='categorical_crossentropy', metrics=['accuracy']) train_score = model.evaluate(train_X, train_y) test_score = model.evaluate(test_X, test_y) print('Train loss:', train_score[0]) print('Train accuracy:', train_score[1]) print('Test loss:', test_score[0]) print('Test accuracy:', test_score[1])4/4 [==============================] - 0s 2ms/step - loss: 0.0649 - accuracy: 0.9714 2/2 [==============================] - 0s 2ms/step - loss: 0.1223 - accuracy: 0.9778 Train loss: 0.06492960453033447 Train accuracy: 0.9714285731315613 Test loss: 0.12225695699453354 Test accuracy: 0.9777777791023254
- tf.keras.models.model_from_jsonについての詳細は以下参照
https://www.tensorflow.org/api_docs/python/tf/keras/models/model_from_json
参考文献
- https://snova301.hatenablog.com/entry/2018/10/29/175028
- https://aidiary.hatenablog.com/entry/20161108/1478609028
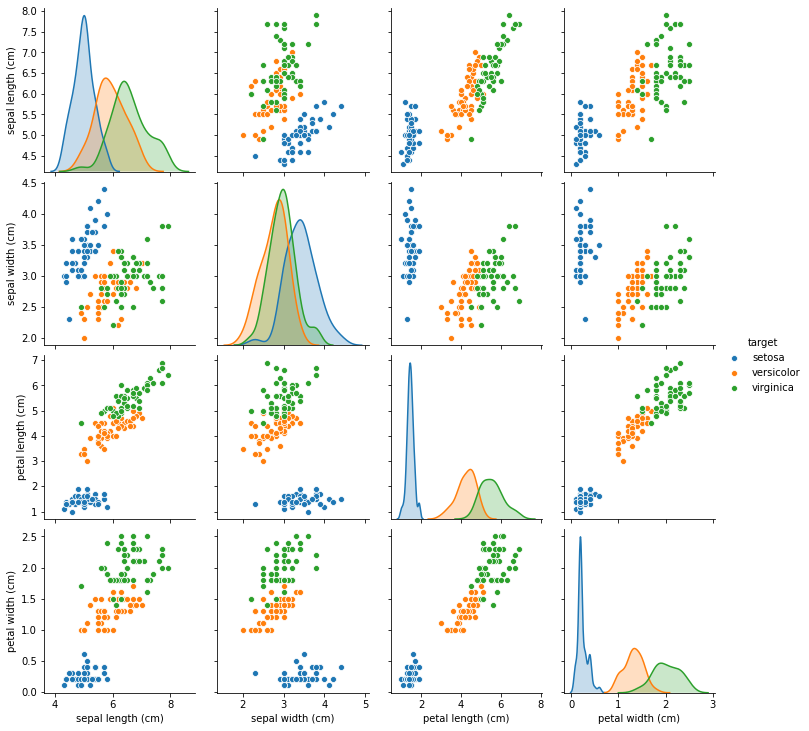
おわり (データの中身を把握するには)
- pandasを使ってみる
- seabornを使ってみる
import pandas as pd import seaborn as sns df = pd.DataFrame(iris.data, columns=iris.feature_names) df['target'] = iris.target df.loc[df['target'] == 0, 'target'] = "setosa" df.loc[df['target'] == 1, 'target'] = "versicolor" df.loc[df['target'] == 2, 'target'] = "virginica" df.head(2)
sepal length (cm) sepal width (cm) petal length (cm) petal width (cm) target 5.1 3.5 1.4 0.2 setosa 4.9 3.0 1.4 0.2 setosa sns.pairplot(df, hue="target")
- 投稿日:2020-09-13T22:58:20+09:00
RustでPythonライクなrangeを使う
はじめに
RustでPythonライクなrangeを使う方法を紹介します。
以下のように使うことができます。
// for i in range(5): // print(i) for i in range!(5) { println!("{}", i); } // for i in range(1, 5): // print(i) for i in range!(1, 5) { println!("{}", i); } // for i in range(1, 5, 2): // print(i) for i in range!(1, 5, 2) { println!("{}", i); } // for i in range(5, 1, -1): // print(i) for i in range!(5, 1, -1) { println!("{}", i); }やり方
以下のマクロでPythonライクなrangeを実装できます。
macro_rules! range { ($stop:expr) => { 0..$stop }; ($start:expr, $stop:expr) => { $start..$stop }; ($start:expr, $stop:expr, -$step:expr) => { ($stop + 1..$start + 1).rev().step_by($step) }; ($start:expr, $stop:expr, $step:expr) => { ($start..$stop).step_by($step) }; }基本的には、Pythonのrange関数と同じイテレータを返します。
できないこと
負のステップを指定するときに、
step_by()はusizeしか受け取れないため、rev()でイテレータを逆順にする必要があります。
そこでパターンマッチで-の有無を判定して、rev()するかを決めています。
なので変数に負の数が入っていてステップとして与えると、パターンマッチで負の数と判定することができないため、コンパイルエラーとなります。// コンパイルエラー let x = -1; for i in range!(5, 1, x) { println!("{}", i); }ここらへんをうまく実装できる方法があれば、教えていただきたいです。
最後に
Rustのマクロは自由度が高いので、とても楽しいです。
- 投稿日:2020-09-13T22:31:54+09:00
ゼロから作るDeep Learning❷で素人がつまずいたことメモ:5章
はじめに
ふと思い立って勉強を始めた「ゼロから作るDeep Learning❷ーー自然言語処理編」の5章で私がつまずいたことのメモです。
実行環境はmacOS Catalina + Anaconda 2019.10、Pythonのバージョンは3.7.4です。詳細はこのメモの1章をご参照ください。
5章 リカレントニューラルネットワーク(RNN)
この章は、リカレントニューラルネットワークの説明です。
5.1 確率と言語モデル
言語モデルの説明と、CBOWを言語モデルに使おうとした時の問題点が解説されています。式5.8が近似になっているのは、CBOWだと単語の並びを無視してしまうからということかと思います。
word2vecは単語の並びを無視してしまうので、分散表現に使うならこの章で学ぶRNNの方が良さそうに思えるのですが、RNNが先に生まれ、語彙数増加と質の向上のために後からword2vecが提案されたとのことで、実際には逆の流れだったというのが興味深いです。
5.2 RNNとは
RNNの解説です。活性化関数としてtanh関数(双曲線正接関数)がでてきますが、なぜかこの本では解説がないので、詳細は「tanh」でググりましょう。
あと少し気になったのは、ミニバッチ学習でデータを終端まで使った時に先頭に戻す対応です。これだとコーパスの末尾と先頭がつながってしまいます。ただ、そもそもこの本ではPTBコーパスを「ひとつの大きな時系列データ」として扱っていて、文の区切りすら意識していません(P.87の中央のサソリマーク部分参照)。そのため、末尾と先頭がつながってしまうことを気にしても意味がないレベルなのかも知れません。
5.3 RNNの実装
実装に当たっては、図5-19と図5-20でバイアス $b$ の後のRepeatノードが省略されているので少し注意が必要です。順伝播はブロードキャストが行われるので図の通りに実装できますが、逆伝播で $db$ を求める際は意識して加算する必要があります。ちょうどこの部分のQAがteratailにもありました(teratail : RNNの逆伝播でdbをaxis=0でsumする理由に関して)。
あと、今回出てきたtanh関数が解説なしで実装されていますが、順伝播は本のコードのように
numpy.tanh()で計算できます。逆伝播はdt = dh_next * (1 - h_next ** 2)の部分がtanhの微分になりますが、これについては本の終わりにある「付録A sigmoid関数とtanh関数の微分」に詳しい解説があります。また、P.205で「...(3点ドット)」の話がでてきますが、これはP.34で出てきた「3点リーダー」と同じです。このメモの1章にも書きましたが、3点ドットだと上書きになると覚えるよりも、ndarrayのスライスとビューの関係を理解するのがオススメです。
5.4 時系列データを扱うレイヤの実装
コードの解説が省略されていますが、シンプルなので見れば理解できました。
Time Embeddingレイヤー(common/time_layers.pyの
TimeEmbeddingクラス)は単純に $T$ 個のEmbeddingレイヤーをループで処理しているだけです。Time Affineレイヤー(common/time_layers.pyの
TimeAffineクラス)では、$T$ 回ループする代わりにバッチサイズ $N$ が $T$ 倍になる形に変形して一気に計算し、結果を元の形に変形することで効率化しています。Time Softmax With Loss レイヤー(common/time_layers.pyの
TimeSoftmaxWithLossクラス)は本の解説通りなのですが、ignore_labelを使ったマスクが実装されているのが気になりました。正解ラベルが -1の場合は損失も勾配も0にして $L$ を求める際の分母の $T$ からも除外しているのですが、正解ラベルを-1にするような処理は今の所なかったかと思います。この後の章で使うのかも知れないので、とりあえず放置しておきます。5.5 RNNLMの学習と評価
残念ながら今回の実装ではPTBデータセット全体を使うと良い結果が出ないとのことなので、前章で遊んだ青空文庫の分かち書き済みテキストでの学習もやめておきました。次章で改良するとのことなので、そこで試してみようと思います。
余談ですが、
SimpleRnnlm.__init__()のrn = np.random.randnというコードを見て、今さらながらPythonは関数を簡単に変数に入れて使えるので便利だなと思いました。C言語だと変数に関数を入れる(関数のエントリポイントを変数に入れる)のに*とか()とかたくさんついて複雑で、それを使うのもややこしくて、現役時代はホント苦手でした5.6 まとめ
なんとか時系列データが取り扱えるようになってきました。
この章は以上です。誤りなどありましたら、ご指摘いただけますとうれしいです。
- 投稿日:2020-09-13T21:53:20+09:00
RとPythonのDocker作業環境の構築2: 日本語対応
はじめに
先日に以下の記事を投稿し,RとPythonが使える環境を構築するDockerfileを紹介しました。
こちらの記事のDockerfileでは,以下の問題がありました。
- 日本語の対応ができていない
- 文字コードが異なっており,他OSのテキスト解析の結果を再現できない
そこで,localeを変更することで,上記問題を解決するDockerfileに修正しました。
FROM ubuntu:18.04 # set timezone RUN apt-get update \ && apt-get install tzdata \ && ln -sf /usr/share/zoneinfo/Asia/Tokyo /etc/localtime RUN date # install packages RUN ["/bin/bash", "-c", "\ apt-get update \ && apt-get install -y \ vim \ build-essential \ git curl llvm sqlite3 libssl-dev libbz2-dev \ libreadline-dev libsqlite3-dev libncurses5-dev \ libncursesw5-dev python-tk python3-tk tk-dev aria2 \ lsb-release locales\ "] RUN locale-gen ja_JP.UTF-8 ENV LANG ja_JP.UTF-8 ENV LANGUAGE ja_JP:ja ENV LC_ALL ja_JP.UTF-8 RUN ["/bin/bash", "-c", "apt-get install -y software-properties-common"] RUN apt-add-repository ppa:ansible/ansible -y # install r RUN apt-key adv --keyserver keyserver.ubuntu.com --recv-keys E298A3A825C0D65DFD57CBB651716619E084DAB9 #RUN add-apt-repository 'deb https://cran.rstudio.com/bin/linux/ubuntu $(lsb_release -cs)-cran35/' RUN add-apt-repository 'deb https://cran.rstudio.com/bin/linux/ubuntu bionic-cran35/' RUN ["/bin/bash", "-c", "\ apt-get update \ && apt-get install -y r-base \ "] RUN Rscript --version CMD ["/bin/bash", "-c"]差分は以下の通りです。
- python3.8 python3-pip のインストールをやめた
- インストールするパッケージにlocalesを追加した
- locale-genから始まるブロックで,日本語に設定した
以上です。
コンテナ上でpyenvを構築するためのスクリプトを作成中で,これができたらとりあえずPCを変えても再現できる環境が作れるのではないかと思っています。
スクリプトができたらまた記事書きます。
- 投稿日:2020-09-13T21:43:38+09:00
画像縮小(LEDマトリクスパネル 16×32)
ppmファイルを縮小して、RGBマトリクスパネル16×32に表示する画像を作ります。
1.ライブラリ
pip install Pillow
resize.pyencoding:UTF-8 #-*- codinf:utf-8 -*- from PIL import Image img = Image.open("{変換前jpgファイル}") width, height = img.size img = img.resize((int(img.width / (img.height / 16 )), 16)) img.save("{変換後ppmファイル}")2.実行
python resize.py
- 投稿日:2020-09-13T21:40:24+09:00
【定義】フレームワークとは何か
はじめに
本記事の主題であるフレームワーク定義の部分は記事の後半部にありますので、前半部は読み飛ばしていただいても問題ありません。
フレームワークを調べるに至るまでの変遷
venvとは何かを調べていたところ、「PyCharmでVenvをGitで共有するときにすること」というサイトを教えてもらったのですが、冒頭の導入部分にこんなことが書いてありました。
今回友達とpythonのDjangoで何かサービスを開発しようということになり、環境はVirtualEnv環境(以降venv)を使おうという話になりました!
venvとは、Djangoを使うときに使えるのか…
いやまず、Djangoってなんだ?
Wikipediaにはこう書かれていました。Django(ジャンゴ)は、Pythonで実装されたWebアプリケーションフレームワーク。
あれ、ウェブアプリケーションフレームワークってなんか聞いたことあるぞ。なんだっけ…
FlaskをWikipediaで調べてみるとこう書かれていました。Flask(フラスク)は、プログラミング言語Python用の、軽量なウェブアプリケーションフレームワークである。標準で提供する機能を最小限に保っているため、自身を「マイクロフレームワーク」と呼んでいる。
そうだ!Flaskもウェブアプリケーションフレームワークなんだ!
flaskってどんな使い方をするんだっけ…
例えば…main.pyfrom flask import Flask app = Flask(__name__, static_folder='.', static_url_path='') @app.route('/') def index(): return app.send_static_file('index.html') app.run(port=8000, debug=True)あれ?つまり、ウェブアプリケーションフレームワークというのはライブラリのことなのか…?
ウェブアプリケーションフレームワーク
じゃあ、ウェブアプリケーションフレームワークって何なんだろう。例によってWikipediaを開きました。
Web アプリケーションフレームワーク(ウェブアプリケーションフレームワーク、英: Web Application Framework)は、動的な ウェブサイト、Webアプリケーション、Webサービスの開発をサポートするために設計されたアプリケーションフレームワークである。
アプリケーションフレームワーク…?
とりあえず続きを読む。フレームワークの目的は、Web開発で用いられる共通した作業に伴う労力を軽減することである。たとえば、多数のフレームワークがデータベースへのアクセスのためのライブラリや、テンプレートエンジン(→Webテンプレート)、セッション管理を提供し、コードの再利用を促進させるものもある。
やっぱりライブラリでもあるんだけど、それだけじゃないみたいだ。
アプリケーションフレームワーク(or フレームワーク)
とりあえずアプリケーションフレームワークとは何か、Wikipediaを読む。
アプリケーションフレームワーク (英: application framework) とは、プログラミングにおいて、アプリケーションソフトウェアの標準構造を実装するのに使われるライブラリ(サブルーチンやクラスなど)の集まりである。単にフレームワークとも呼ぶ。
やっぱりライブラリのことなんだ!
じゃあ、なんでフレームワークなんて言い方をするんだろう…?「ライブラリ」と「フレームワーク」は何が違うのか [本題]
以上がフレームワークとは何かを調べるに至った経緯です。フレームワークとは何かという質問に答えるならば、上記の通り、「誰かが書いたコードの集まり、すなわちライブラリみたいなものと認識しておけば十分」だと思われます。
しかし、このように言葉が分けられているということは、そこに違いがあると考えるのが普通ですよね。こちらの記事によりますと、実は、ライブラリとフレームワークにおける技術的な違いは「制御の反転の有無」という用語に集約されます。これは主導権が「我々」にあるのか、「クラスやサブルーチンの集まり」にあるのかということです。
すなわち、ライブラリを使うときには、ライブラリの機能を使うタイミングは我々が決定するのに対し、フレームワークを使うときには、その機能を使うタイミングをフレームワーク自身が決定するのです。つまり、フレームワークを使う際には、プログラムの制御をするのが使っている人間ではなくそのプログラム自身になってしまうという「制御の反転」ということが起こってくるのです。
ここでもう一度ウェブアプリケーションフレームワークのWikipediaの記事を読んでみると、
フレームワークの目的は、Web開発で用いられる共通した作業に伴う労力を軽減することである。たとえば、多数のフレームワークがデータベースへのアクセスのためのライブラリや、テンプレートエンジン(→Webテンプレート)、セッション管理を提供し、コードの再利用を促進させるものもある。
とありますね。つまり、ライブラリの提供に加えて、テンプレートエンジン(テンプレートエンジンはテンプレートと呼ばれる雛形と、あるデータモデルで表現される入力データを合成し、成果ドキュメントを出力するソフトウェアまたはソフトウェアコンポーネント(Wikipediaより))やセッション管理の提供がなされるとところがよりフレームワーク-likeな部分だと思います。
より詳しい具体例についてはこちらの記事を参照してください。
たとえ話
最後に、引用になりますが、ライブラリとフレームワークの違いのわかりやすいたとえです。
私はよくWeb開発における概念のメタファーとして、家を使います。
例えばライブラリはIKEAに行くようなものです。あなたはすでに自分の家を持っていますが、家具が少し足りないとします。ですが、テーブルを一から作る気にはなれません。そんな時、IKEAに行けば数ある選択肢の中から気に入ったものを持ち帰ることができます。それを選ぶのはあくまでも、あなたです。
一方、フレームワークはモデルハウスを建築するようなものです。いくつかのテンプレートの中から、限られたアーキテクチャとデザインを選ぶことができます。あなたが基本的に選べるのは請負業者とテンプレートのみ。あとはその業者がピンポイントであなたが介入できるところを教えてくれます。
- 投稿日:2020-09-13T21:28:35+09:00
WinAutomationから自分環境のPythonを使う
概要
入力処理の自動化をする場合、前処理としてPythonのプロセスを組み込みたいことがあるかもしれません。
WinAutomationにはPythonアクションがありますがIronPythonというもので、Pandas等ライブラリの使用は難しいようです。
そこで自身が使いたいライブラリをインストールした環境のPythonをWinautomationから実行する方法を説明したいと思います。お題
次ようなnull(空欄)が含まれる1万行のExcelデータを0(ゼロ)で埋めて他システムに入力
0埋めする部分をpython、入力部分はWinautomationで行います(仮想としてExcel to Excel)前提条件
- Windows10 64bit
- WinAutomation 9.2.1
- Python 3.8.5
Python環境構築についてはディストリビューション使用等、様々なお作法があるかもしれませんが、今回は公式版で説明していきます。インストール済みの環境がある場合、複数環境になると正常動作出来なくなる場合がありますので自己責任でお願いいたします。
環境構築
Pythonのインストール
https://www.python.org/
から3.8.5をダウンロード、パスは通さずインストール。
同時にインストールされるランチャーアプリのPy.exeからスクリプトやコマンドの実行を行う。インストール状況とランチャーの動作をバージョン情報を調べて確認
C:\Users\username>py -V Python 3.8.5初期インストールされているライブラリの確認
最新版でないライブラリの確認C:\Users\username>py -m pip list Package Version --------------- ------- pip 20.2.1 setuptools 49.6.0 C:\Users\username>py -m pip list --outdate最新版でなければ両方ともアップデート
C:\Users\user>py -m pip install -U ライブラリ名py -m pip listで再確認
データ処理に必要なライブラリをインストール
今回はデータ処理にpandas、エクセルファイルの読み書きにopenpyxlとxlrdを使用
numpyや他必要ライブラリはpandsインストール時に自動でインストールされるC:\Users\user>py -m pip install pandas C:\Users\user>py -m pip install openpyxl C:\Users\user>py -m pip install xlrd C:\Users\user>py -m pip list Package Version --------------- ------- et-xmlfile 1.0.1 jdcal 1.4.1 numpy 1.19.2 openpyxl 3.0.5 pandas 1.1.2 pip 20.2.3 python-dateutil 2.8.1 pytz 2020.1 setuptools 50.3.0 six 1.15.0 xlrd 1.2.0同様に使いたいライブラリがあれば追加
なおコードエディターにはVisual Studio CodeをしているWinautomationのプロセス
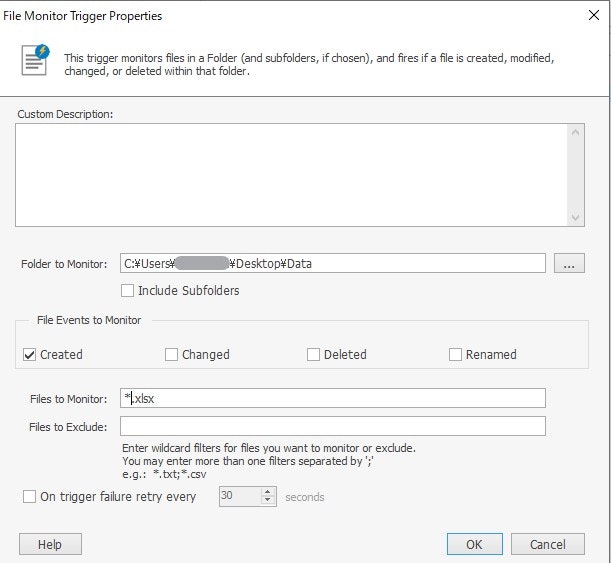
監視フォルダの設定
WinAutomation ConsoleからTriggersでFile Monitor Triggerを設定する
OKしたあと起動するプロセス名を紐づけFile Monitor TriggerのCreatedが起動することで
以下の変数が自動的に値を持つ
Process Designerには表示されない変数なので覚えておくと便利
変数名 内容 %FileTriggerFileName% 拡張子まで含めたファイル名 %FileTriggerFilePath% ファイルの完全なパス及びファイルのすべてのプロパティを保持 %FileTriggerFilePath.NameWithoutExtension% 拡張子を含まないファイル名 プロセスの作成
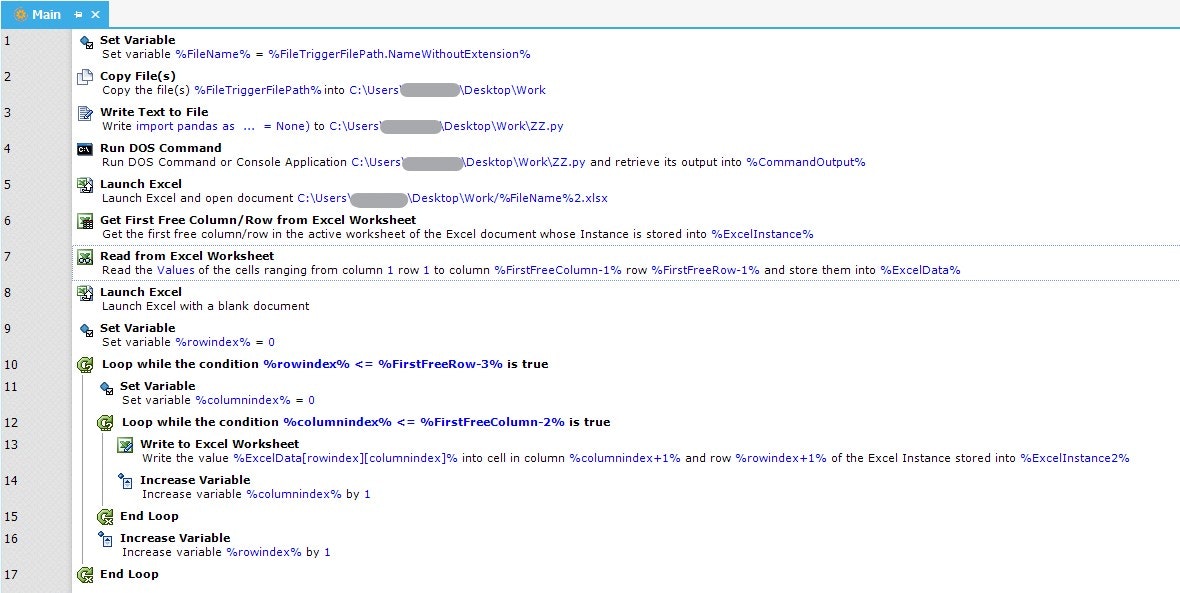
各アクションの解説
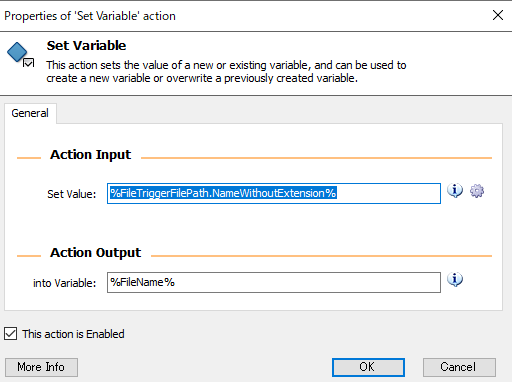
1.Set Variable
File Monitor Triggeから渡されるファイル名の変数が長すぎるのでわかりやすくしただけ
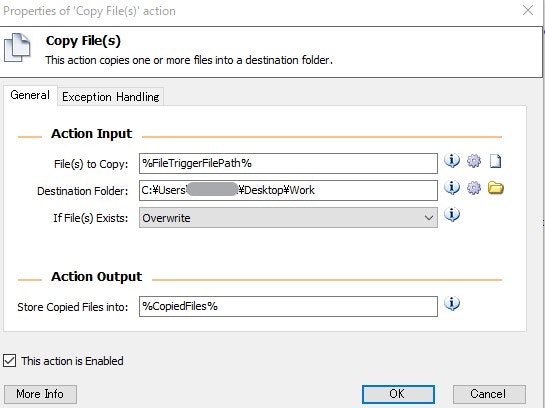
2.Copy File(s)
Dataフォルダに検知したファイルを作業(Work)フォルダにコピー
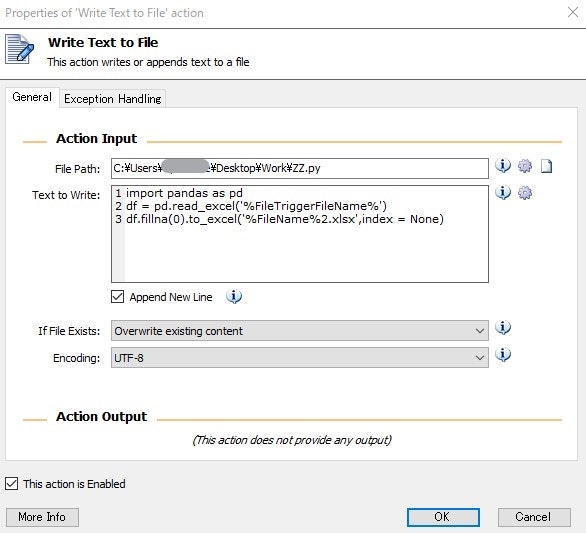
3.Write Text to File
Python Script(.py)の作成
投入するデータファイルが固定されたファイル名であればpython scriptも毎回生成する必要はない
この場合はファイル名に日付が入っているなど変動する場合を想定しているため、毎回ファイル名をTriggerの変数より取得して書き込むようにしている
ZZ.pyという名前を付けてUTF-8で保存
Python ScriptはPandasに読み込んでゼロフィル、ファイル名2.xlsxで保存するだけのショボい内容
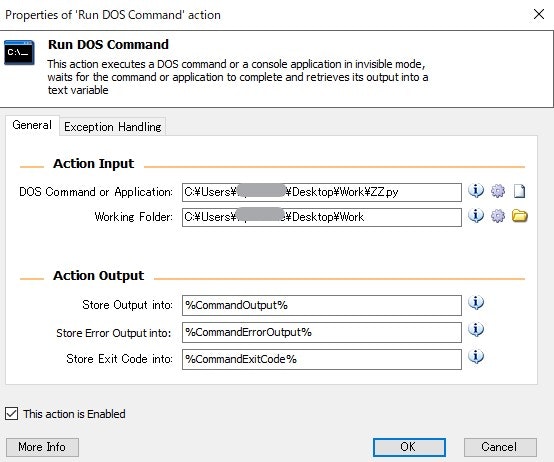
トリガーから作成されると変数部分にファイル名が入るimport pandas as pd df = pd.read_excel('%FileTriggerFileName%') df.fillna(0).to_excel('%FileName%2.xlsx',index = None)4.Run DOS Command
3で作ったZZ.pyファイルを実行
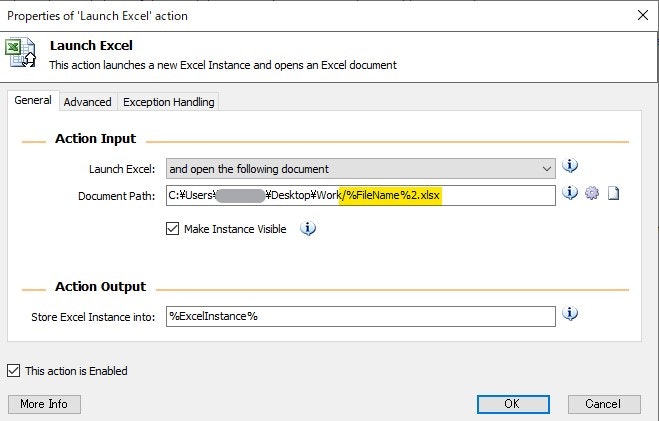
5.Launch Excel
Pythonで生成されたエクセルファイルを開くのだが、変数化したファイル名のパス表記が、変数の手前だけ¥ではなく/とする
はまりポイントなので注意したい
またMake Instance Visibleのチェックを外した場合、画面に表示せず実行できるが、その場合は必ずプロセスの終わりにClose Excelを入れてインスタンスを閉じてあげないと裏で開いたままになるので注意6~17のプロセスについて
これら他システムへの入力の代わりにブランクのExcelに1セルずつデータ部分転記しているだけなので省略する
一括コピーをしないのは他システムで入力するにあたり、トランザクションが発生することを想定しているため
まとめ
ランチャーのpy.exeのおかげで.py Scriptが簡単に実行できます。
WinAutomationのプロセスにPandasなどの便利なライブラリが使えた上で組み込み可能。実をいうとゼロ埋めをするだけならWinAutomationからExcelの置換を使って行うことができます。(一番簡単)
しかし複雑な前処理が必要な場合はExcelの制御は難しいように思います。
WinAutomation単体でも転記する際に空欄ならゼロにするプロセスにすれば同様のことが行えます。
ただしデータを前処理した場合より遅くなります。
筆者のボロマシンCorei7 3770 8Gでは1万行*7列を別Excelに1セルづつ転記した場合前処理ありだと5分50秒
前処理なしでWinAutomationで空欄ならゼロ処理させながら転記した場合は6分30秒でした。WinAutomationの情報はまだ少ないので寂しいなぁ。
- 投稿日:2020-09-13T20:08:48+09:00
Django templates.html を使ってみる
form.pyは、正直全然わかっていないし初めて使います。
ネットで複数記事読みましたが、全然頭に入らず…(笑)でも、とりあえず書いてすすめてみようと思います。
form.pyfrom django import forms from .models import Staff class CreateUserForm(forms.ModelForm): #modelformを継承する class meta(): model = Staff fields = ("name ", "password", "roll", "nyushabi", "taishabi", "hyoujijyun", "jikyu", "delete ")これで、htmlに連携されるってことみたいです。
つづいて、Viewsを作成します。
views.pyfrom django.shortcuts import render from . import CreateUserForm def form_name_view(request): form = forms.UserForm() if request.method == 'POST': form = forms.CreateUserForm(request.POST) if form.is_valid(): form.save() return render(request, 'templates/staff.create.html', {'CreateUserForm': form})if request.method=="POST"
は、初回表示ではないリクエストの時にってことなので、何か入力した後に登録ボタンを押下した時に動作する命令になります。Postでなければ、テンプレートを呼び込んで、FomsからCreateUserForm を呼び込みするイメージです。
本当にこれでいいのかわかっていませんが(笑)次は、テンプレートの作成。
まずは、base.htmlをbootstarpからコピーして一部、改修します。base.html<!doctype html> <html lang="ja"> <head> <!-- Required meta tags --> <meta charset="utf-8"> <meta name="viewport" content="width=device-width, initial-scale=1, shrink-to-fit=no"> <!-- Bootstrap CSS --> <link rel="stylesheet" href="https://stackpath.bootstrapcdn.com/bootstrap/4.5.0/css/bootstrap.min.css" integrity="sha384-9aIt2nRpC12Uk9gS9baDl411NQApFmC26EwAOH8WgZl5MYYxFfc+NcPb1dKGj7Sk" crossorigin="anonymous"> <title> {% block title %} {% endblock title %} </title> </head> <body> {% blcok header %} {% endblock header %} {% block content %} {% endblock content %} <!-- Optional JavaScript --> <!-- jQuery first, then Popper.js, then Bootstrap JS --> <script src="https://code.jquery.com/jquery-3.5.1.slim.min.js" integrity="sha384-DfXdz2htPH0lsSSs5nCTpuj/zy4C+OGpamoFVy38MVBnE+IbbVYUew+OrCXaRkfj" crossorigin="anonymous"></script> <script src="https://cdn.jsdelivr.net/npm/popper.js@1.16.0/dist/umd/popper.min.js" integrity="sha384-Q6E9RHvbIyZFJoft+2mJbHaEWldlvI9IOYy5n3zV9zzTtmI3UksdQRVvoxMfooAo" crossorigin="anonymous"></script> <script src="https://stackpath.bootstrapcdn.com/bootstrap/4.5.0/js/bootstrap.min.js" integrity="sha384-OgVRvuATP1z7JjHLkuOU7Xw704+h835Lr+6QL9UvYjZE3Ipu6Tp75j7Bh/kR0JKI" crossorigin="anonymous"></script> </body> </html>これで、ベースとなる部分の作成完了。
登録用のHTMLは、
staff.create.py{% extends 'staff/base.html' %} {% block content %} {{ form.as_p }} <input type="submit" class="btn btn-primary" value="Submit"> {% block content %}これで、ベースから読み込みしてcontentに差し込みされて表示がされるはず。
最後に、urls.pyを修正
staff.url.pyfrom django.urls import path, include from .views import createstaff urlpatterns = [ path('create/', createstaff, name = "createstaff"), ]これで、サーバーを起動させて一度表示できるか挑戦してみます。
viewsの5行目にある、forms が見つからないってエラーですね。
staff.viewsfrom django.shortcuts import render from .form import CreateUserForm def createstaff(request): form = CreateUserForm.UserForm() if request.method == 'POST': form = forms.CreateUserForm(request.POST) if form.is_valid(): form.save() return render(request, 'templates/staff.create.html', {'CreateUserForm': form})これでリトライしてみる。
まだダメですね。
この後、少しいじって挑戦しましたがエラーが解決できなかったので、formを使うのをやめてclassで作成をしてみるように切替てすすめていこうと思います
- 投稿日:2020-09-13T19:57:23+09:00
tqdmでタスクバー表示
- 投稿日:2020-09-13T19:56:52+09:00
boto3でno attributeのエラーがでたときはバージョンを確認しよう
boto3のエラー
boto3のput_function_event_invoke_configを使用したところ、
import botocore import boto3 client = boto3.client('lambda') response = client.put_function_event_invoke_config( FunctionName=FunctionName, MaximumRetryAttempts=0, )以下のエラーが発生
AttributeError: 'Lambda' object has no attribute 'put_function_event_invoke_config'翻訳すると
AttributeError: 'Lambda' オブジェクトには 'put_function_event_invoke_config' 属性がありません。
とのこと。
lambdaに以下を追記して、boto3とbotocoreのバージョンを確認しました。
print('botocore vertion is {0}'.format(botocore.__version__)) print('boto3 vertion is {0}'.format(boto3.__version__))これが結果
botocore vertion is 1.12.253 boto3 vertion is 1.14.57現在のバージョンを以下で確認したところ・・
Releases · boto/boto3 · GitHub
Releases · boto/botocore · GitHubbotocoreが半年以上古いようです。
botocoreを最新化する
以下の記事の通りで最新化できます。
【AWS】Lambdaでのbotoの使い方と最新botoの使用方法
ServerlessFrameworkの場合は、
serverless-python-requirementsのプラグインを入れてrequirement.txtにboto3とbotocoreを追記することで、最新化できます。以下の記事が参考になります。
【Tips】Serverless Frameworkで最新のboto3をインストールしたLambda Layerをデプロイする解決
botocoreを最新化することで、エラーが解消しました。
参考
- 投稿日:2020-09-13T19:43:48+09:00
一次元と二次元の頂点検出処理
一次元の頂点検出
[-1, 1]で畳み込み処理を行うと上昇している箇所は0を超えた値となり下降している箇所は0未満となる、この事を利用して上昇フラグ、下降フラグを作成、そのフラグが重なりあった箇所を頂点とする。
import math import matplotlib import matplotlib.pyplot as plt import numpy as np import PIL.Image import random import scipy.ndimage # 一次元の頂点検出 x = np.array([0, 0, 1, 0, 0]) print("x", x, "対象配列") conv1 = np.convolve(x, [1, -1], mode="full") print("conv1", conv1, "[-1, 1]のフィルタにより上昇箇所、下降箇所の目印を作る") flag1 = (conv1 > 0).astype(int) print("flag1", flag1, "上昇箇所のフラグ") flag2 = (conv1 <= 0).astype(int) print("flag2", flag2, "下降箇所のフラグ") flag1 = flag1[:-1] print("flag1", flag1, "上昇フラグの末尾を1つ削って長さをあわせる") flag2 = flag2[1:] print("flag2", flag2, "下降フラグの先頭を1つ削って頂点箇所と合わせ、長さも揃える") flag3 = flag1 & flag2 print("flag3", flag3, "上昇フラグと下降フラグを AND した結果が頂点箇所となる")実行結果
x [0 0 1 0 0] 対象配列 conv1 [ 0 0 1 -1 0 0] [-1, 1]のフィルタにより上昇箇所、下降箇所の目印を作る flag1 [0 0 1 0 0 0] 上昇箇所のフラグ flag2 [1 1 0 1 1 1] 下降箇所のフラグ flag1 [0 0 1 0 0] 上昇フラグの末尾を1つ削って長さをあわせる flag2 [1 0 1 1 1] 下降フラグの先頭を1つ削って頂点箇所と合わせ、長さも揃える flag3 [0 0 1 0 0] 上昇フラグと下降フラグを AND した結果が頂点箇所となるサンプルの作成
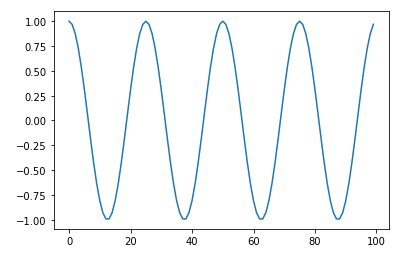
cycle = 4 data = np.zeros(100) cycleWidth = len(data) / cycle unit = math.pi / cycleWidth * 2 for i in range(cycle): for j in range(int(cycleWidth)): data[i * int(cycleWidth) + j] = math.cos(unit * float(j)) plt.plot(data) plt.show()サンプルに対して頂点検出の実行
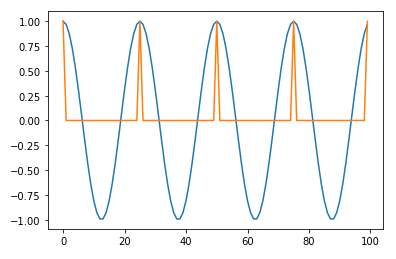
# 一次元の頂点検出を関数化しておく def detectPeak1D(x): conv1 = np.convolve(x, [1, -1], mode="full") flag1 = (conv1 > 0).astype(int) flag2 = (conv1 <= 0).astype(int) flag1 = flag1[:-1] flag2 = flag2[1:] flag3 = flag1 & flag2 return flag3 peaks = detectPeak1D(data) plt.plot(data) plt.plot(peaks) plt.show()水平箇所については立ち上がりが頂点として検出される
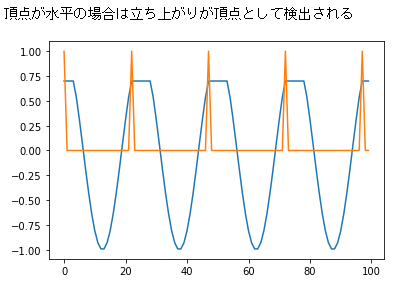
data[data > 0.7] = 0.7 peaks = detectPeak1D(data) print("頂点が水平の場合は立ち上がりが頂点として検出される") plt.plot(data) plt.plot(peaks) plt.show()二次元の頂点検出
一次元の頂点検出を「全ての行に対して行ったフラグ(二次元配列)」と「全ての列に対して行ったフラグ(二次元配列)」の2つをANDした結果を2次元データの頂点とする
# 2次元の頂点検出 x = np.array([ [0, 0, 1, 0, 0], [0, 2, 3, 2, 0], [1, 3, 5, 3, 1], [0, 2, 3, 2, 0], [0, 0, 1, 0, 0], ]) print(x, "対象データ") # すべての行で頂点検出を実行 peaks1 = [] for ix in x: peak = detectPeak1D(ix) peaks1.append(peak) peaks1 = np.array(peaks1) print(peaks1, "横方向の頂点検出フラグ") # すべての列で頂点検出を実行 peaks2 = [] for ix in x.transpose(): peak = detectPeak1D(ix) peaks2.append(peak) peaks2 = np.array(peaks2).transpose() # transposeにより検出を実行したのでもとに戻す print(peaks2, "縦方向の頂点検出フラグ") peaks3 = (peaks1 & peaks2).astype(int) print(peaks3, "行、列の検出フラグをANDして残ったフラグが2次元の頂点フラグとなる")実行結果
[[0 0 1 0 0] [0 2 3 2 0] [1 3 5 3 1] [0 2 3 2 0] [0 0 1 0 0]] 対象データ [[0 0 1 0 0] [0 0 1 0 0] [0 0 1 0 0] [0 0 1 0 0] [0 0 1 0 0]] 横方向の頂点検出フラグ [[0 0 0 0 0] [0 0 0 0 0] [1 1 1 1 1] [0 0 0 0 0] [0 0 0 0 0]] 縦方向の頂点検出フラグ [[0 0 0 0 0] [0 0 0 0 0] [0 0 1 0 0] [0 0 0 0 0] [0 0 0 0 0]] 行、列の検出フラグをANDして残ったフラグが2次元の頂点フラグとなる# 二次元の頂点検出を関数化しておく def detectPeak2D(x): peaks1 = [] for ix in x: peak = detectPeak1D(ix) peaks1.append(peak) peaks1 = np.array(peaks1) peaks2 = [] for ix in x.transpose(): peak = detectPeak1D(ix) peaks2.append(peak) peaks2 = np.array(peaks2).transpose() flag = (peaks1 & peaks2).astype(int) return flag二次元データサンプルの作成
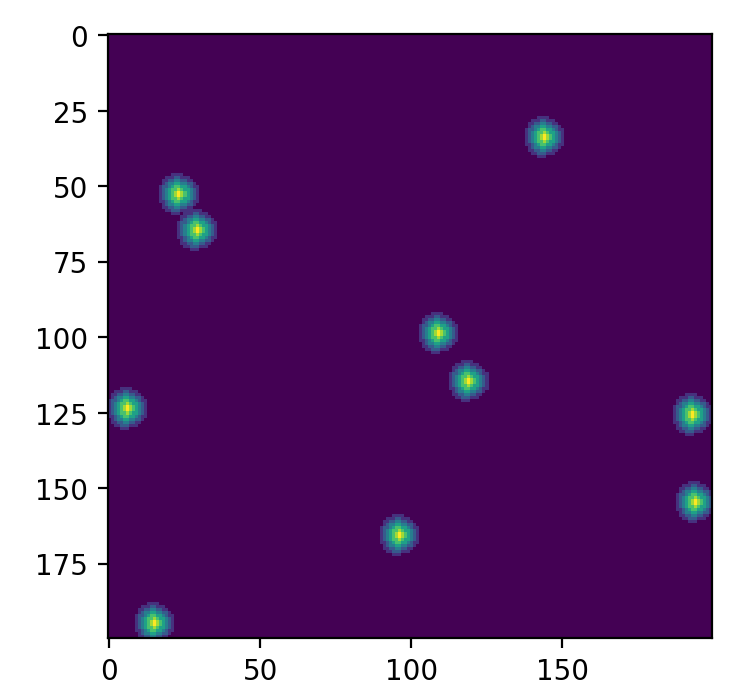
# 二次元頂点検出の試験用データを作成する、一次元の頂点のあるデータを回転させて作る random.seed(1) data2d = np.zeros((200, 200)) pattern = np.array([1, 2, 3, 4, 5, 6, 7, 6, 5, 4, 3, 2, 1]) for i in range(10): ox, oy = random.randrange(200), random.randrange(200) for j in range(50): rad = (math.pi / 50) * j for ix, v in enumerate(pattern): pos = ix - len(pattern) / 2 x = ox + math.cos(rad) * pos y = oy + math.sin(rad) * pos if x < 0: continue if x >= 200: continue if y < 0: continue if y >= 200: continue data2d[int(x)][int(y)] = v plt.figure(figsize=(10,4),dpi=200) plt.imshow(data2d) plt.show()二次元サンプル画像
二次元頂点検出の実行
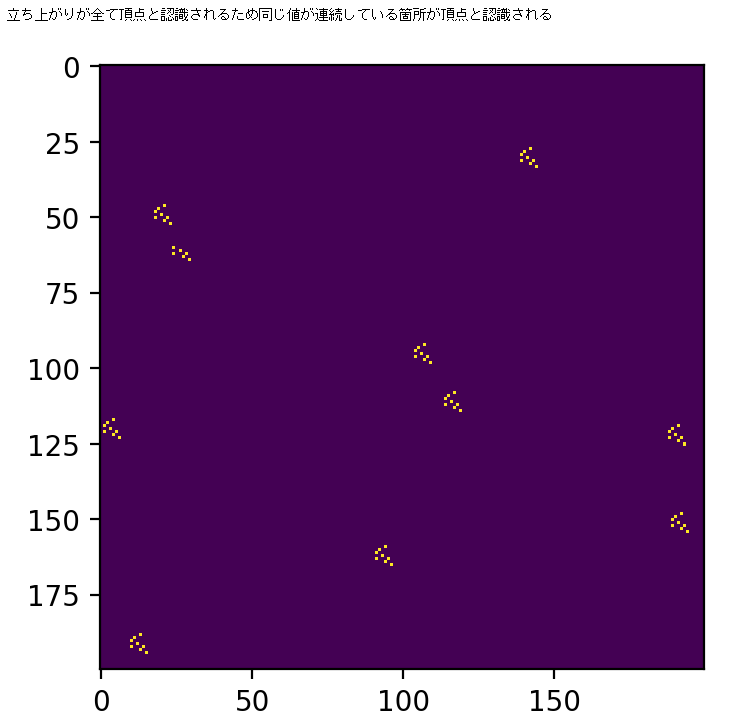
peaks = detectPeak2D(data2d) print("立ち上がりが全て頂点と認識されるため同じ値が連続している箇所が頂点と認識される") plt.figure(figsize=(10,4),dpi=200) plt.imshow(peaks) plt.show()平滑化処理を施してから二次元頂点検出の実行
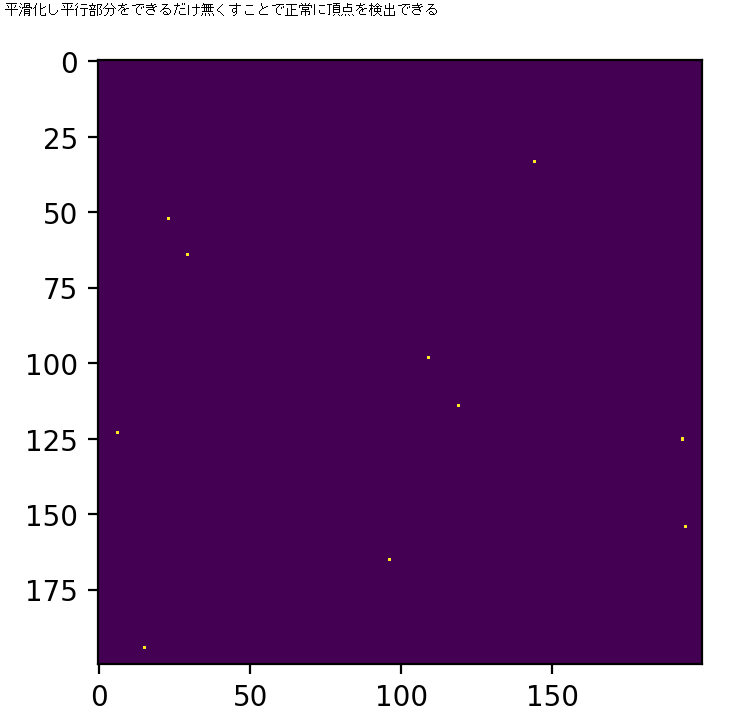
data2dGaussian = scipy.ndimage.gaussian_filter(data2d, sigma=1) peaks = detectPeak2D(data2dGaussian) print("平滑化し平行部分をできるだけ無くすことで正常に頂点を検出できる") plt.figure(figsize=(10,4),dpi=200) plt.imshow(peaks) plt.show()注意等
前処理も加工処理もなくキレイに山なりの形をしているデータを想定した処理です、基本的には平滑化してからでないと想定通りの動作をしない事が多いと思います、そのまま使うのではなく移動平均でもガウシアンフィルタでもなんでも良いので平滑化処理を行ってから使用します。
以上です。
- 投稿日:2020-09-13T19:02:38+09:00
Google Colaboratoryで書籍「Pythonで儲かるAIをつくる」の実習コードを動かす方法
はじめに
書籍「Pythonで儲かるAIをつくる」の著者です。
Amazonリンク
https://www.amazon.co.jp/dp/4296106961/この本では、書籍サポートサイトに実習コードをすべて公開しています。
https://github.com/makaishi2/profitable_ai_book_info/blob/master/README.mdこれらのコードは導入なしに利用できるクラウド環境Google Colaboratoryで動かすことを前提にしています。
書籍購入を検討中の方が、実習コードを試しに動かすことの参考となるよう、Google Colaboratoryでこの本の実習コードを動かすための手順を、当記事で説明します。事前準備
必要な事前準備は、gmailアカウントの取得と、Chromeブラウザの導入の2つです。
gmail アカウント
gmailアカウント取得に関しては、いろいろなところでガイドが出ているので、説明を省略します。
Chrome導入
Colaboratoryを動かす場合の推奨ブラウザは、Google Chromeです。
まだ、自分のパソコンに導入していない場合は、以下のリンクから、ダウンロード・導入を済ませて下さい。https://www.google.co.jp/chrome/
Chromeでgmailにログイン
上の2つの準備が終わったら、Chromeを立ち上げて、gmailにログインした状態にします。これで、すべての準備は完了しました。
Colaboratory起動
Colaboratory立ち上げ
新しいタブを開いて、以下のURLを入力して下さい。
https://colab.research.google.com/
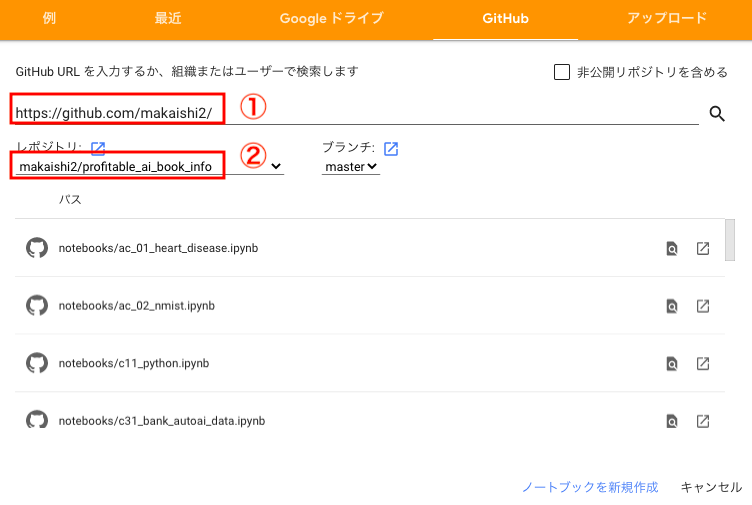
下のような画面が出てくるので、Githubのタブを選択します。
下の画面が出てきたら
① 一番上の欄にhttps://github.com/makaishi2と入力し、Enterキーを押します。② その下の欄がdropdownに変化するので、クリックして
profitable_ai_book_infoを選択します。下の画面になるので、読み込みたいファイルを選択します。
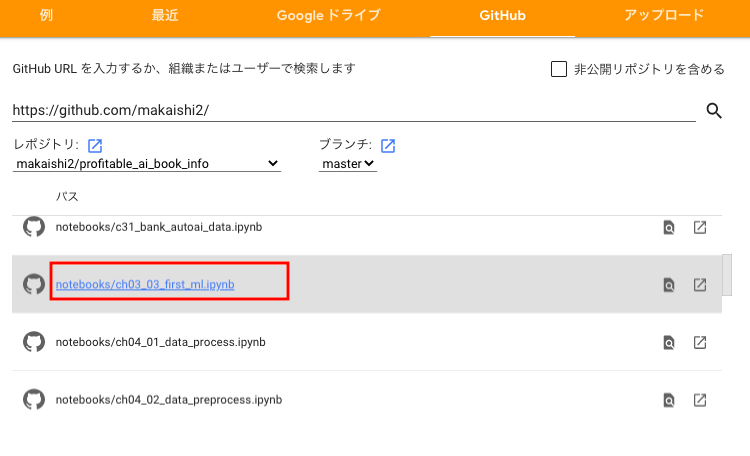
ファイル名と、中身の対応については、次のリンクを参考にして下さい。
https://github.com/makaishi2/profitable_ai_book_info/blob/master/refs/notebooks.md例えば、3章の実習を選択したい場合、下の図のように
ch03_03_first_ml.ipynbを選びます。下の図のようになっていれば、Notebookの読み込みに成功しています。
最低限の使い方として、この状態で、Shift + Enterをひたすら繰り返すと、セルと呼ばれる処理の単位を一つずつ順に実行することが可能です。下のワーニングが出る場合は、右の「このまま実行」を選んで下さい。
最後までうまく実行できると、こんな画面になるはずです。
- 投稿日:2020-09-13T18:49:23+09:00
【Python】Slackbotでパスワードを生成する
こんにちは、みやびのです。
今回はSlackbot+Pythonでパスワードを実装する方法について説明します。
具体的には以下の2つについて説明します。・Pythonでパスワードを生成する方法
・Slackbot+Pythonでパスワードを生成するPythonで作るSlackbotの基本については「SlackbotをPythonで作成しよう」をお読みください。
Pythonでパスワード生成する方法
Pythonのrandomライブラリを活用すれば比較的簡単にパスワード生成処理を作成できます。
関数実装例引数1:パスワードの桁数(デフォルト8) 引数2:文字列の種類(デフォルト:大文字・小文字・数字) リターン値:パスワード引数1に指定した桁数分の文字列をリターン。
さらに引数2によって「小文字と数字」、「大文字と小文字」、「大文字だけ」のように文字列を細かく指定できるようにします。import random def make_password(digit=8, word_type=None): words = '' password = '' uppercase = 'ABCDEFGHIJKLMNOPQRSTUVWXYZ' lowercase = 'abcdefghijklmnopqrstuvwxyz' number = '0123456789' # 引数がない場合は全て指定 if word_type is None: word_type = ['uppercase', 'lowercase', 'number'] # 使用する文字の判定 if 'uppercase' in word_type: words = words + uppercase if 'lowercase' in word_type: words = words + lowercase if 'number' in word_type: words = words + number # パスワードを生成ループ for i in range(0, digit): # 配列で返ってくるので0番目の値を取得 password = password + random.sample(words, 1)[0] return passwordパスワードの実装方法の詳細については「Pythonでパスワード生成処理を実装する」をお読みください。
Slackbot+Pythonでパスワードを生成する
上記処理をSlackbotの応答に埋め込みます。
Slackbotの応答仕様
引数1:パスワードの桁数
引数2:パスワードの文字の種類文字の種類は以下の形で指定します。
数字:数字のみ 大英数字:大文字の英字と数字 小英数字:小文字の英字と数字 英字:小文字と大文字の英字 大英字:大文字の英字のみ 小英字:小文字の英字のみ それ以外:英数字全てパスワードをダイレクトメッセージに送信
message.send()でチャンネルに応答してしまうと他の人にもパスワードが見えてしまうためセキュリティ上あまりよろしくありません。
よってダイレクトメッセージに応答します。ダイレクトメッセージへの応答はslackerライブラリを使用すれば実現できます。
ダイレクトメッセージを使う場合はチャンネル名の代わりにユーザIDの指定が必要です。
ユーザIDはmessageに格納されています。
```pyユーザーID取得
user_id = message.user['id']
ダイレクトメッセージでパスワードを送信
slack = Slacker(slackbot_settings.API_TOKEN)
slack.chat.post_message(user_id, password, as_user=True)
```実装例は以下の通り。
import random from slackbot.bot import respond_to from slacker import Slacker import slackbot_settings def make_password(digit=8, word_type=None): words = '' password = '' uppercase = 'ABCDEFGHIJKLMNOPQRSTUVWXYZ' lowercase = 'abcdefghijklmnopqrstuvwxyz' number = '0123456789' # 引数がない場合は全て指定 if word_type is None: word_type = ['uppercase', 'lowercase', 'number'] # 使用する文字の判定 if 'uppercase' in word_type: words = words + uppercase if 'lowercase' in word_type: words = words + lowercase if 'number' in word_type: words = words + number # パスワードを生成ループ for i in range(0, digit): # 配列で返ってくるので0番目の値を取得 password = password + random.sample(words, 1)[0] return password @respond_to('^パスワード生成s(.*)s(.*)$') def response_pass(message, digit, param_type): # ユーザーID取得 user_id = message.user['id'] # パターンの切り分け if param_type == '数字': word_type = ['number'] elif param_type == '大英数字': word_type = ['lowercase', 'number'] elif param_type == '小英数字': word_type = ['lowercase', 'number'] elif param_type == '英字': word_type = ['lowercase', 'uppercase'] elif param_type == '大英字': word_type = ['uppercase'] elif param_type == '小英字': word_type = ['lowercase'] else: word_type = ['uppercase', 'lowercase', 'number'] # パスワード生成 password = make_password(int(digit), word_type) # ダイレクトメッセージでパスワードを送信 slack = Slacker(slackbot_settings.API_TOKEN) slack.chat.post_message(user_id, password, as_user=True) message.send('パスワードをダイレクトメッセージに送信しました!')パスワードはダイレクトメッセージの方に送信されています。
終わりに
今回紹介したように比較的簡単にSlackbotをパスワード生成ツールにすることができます。
ちょっとしたパスワードを作りたい場合にあると便利ですね。パスワード生成サイトなどもありますが、自由にカスタマイズできるのが人の作った生成サイトにはないメリットです。
今回は英数字1パターンの表示のみでしたが複数パターンの表示をしたり、記号も使えるようにしたりなどいろいろカスタマイズしてみてください。Slackbotの活用方法はブログでもまとめています。
Slackbotの作り方マニュアル〜Python編〜
- 投稿日:2020-09-13T18:40:32+09:00
Pythonの数値計算ライブラリ「Numpy」の配列(ndarray)操作一覧
Pythonの数値計算ライブラリ「Numpy」が提供するN次元配列オブジェクト(以下、ndarray)の操作方法を整理する。
Pythonはデータ型のサポートがない為に計算が低速であり、大量のデータを使って計算する際にはndarrayを使う事が標準的です。前提
import numpy as npA. 配列生成
ndarrayを生成する。
【
array(object, dtype=None, copy=True, order='K', subok=False, ndmin=0)】
- リストからndarrayを作成する。
- object:元となるリスト(又はそのように扱う事ができるもの)
- dtype:要素のデータ型。未指定の場合は要素に基づいて自動選択。(参考:NumPyのデータ型dtype一覧とastypeによる変換(キャスト) | note.nkmk.me)
y = np.array([1,2,3]) X = np.array([[1,2,3], [4,5,6]], dtype=np.int64)【
zeros(shape, dtype=float, order='C')】
- 各要素が0で埋められたndarrayを生成する。
- shape:各次元の要素数(整数又は整数のタプル)
y = np.zeros(3) # array([0., 0., 0.]) X = np.zeros((2,3)) # array([[0., 0., 0.], # [0., 0., 0.]])【
ones(shape, dtype=None, order='C')】
- 各要素が1で埋めれらたndarrayを生成する。
y = np.ones(3) # array([1., 1., 1.]) X = np.ones((2,3)) # array([[1., 1., 1.], # [1., 1., 1.]])【
empty(shape, dtype=None, order='C')】
- 各要素が初期化されていないndarrayを生成する。
y = np.empty(3) # array([0.39215686, 0.70980392, 0.80392157]) X = np.empty((2,3)) # array([[2.6885677e-316, 0.0000000e+000, 0.0000000e+000], # [0.0000000e+000, 0.0000000e+000, 0.0000000e+000]])【
identity(n, dtype=None)】
- 単位行列となるndarrayを生成する。
X = np.identity(3) # array([[1., 0., 0.], # [0., 1., 0.], # [0., 0., 1.]])【
arange([start,] stop[, step,], dtype=None)】
- 等差数列となるndarrayを生成する。
- start:等差数列の開始値
- stop:等差数列の終了の基準値(指定した値以上の値は数列に含まれない)
- step:差
y = np.arange(5) # array([0, 1, 2, 3, 4]) y = np.arange(2,5) # array([2, 3, 4]) y = np.arange(5,2,-1) # array([5, 4, 3])【
linspace(start, stop, num=50, endpoint=True, retstep=False, dtype=None)】
- 指定範囲を分割した等差数列となるndarrayを生成する。
- start:等差数列の開始値
- stop:等差数列の終了値
- step:分割値(配列の要素数)
y = np.linspace(2,10,3) # array([ 2., 6., 10.]) y = np.linspace(2,10,5) # array([ 2., 4., 6., 8., 10.])【
unique(ar, return_index=False, return_inverse=False, return_counts=False, axis=None)】
- ソートした上で重複を削除したndarrayを生成する。
y = np.unique(['C', 'E', 'C', 'A', 'B', 'E', 'D', 'C']) # array(['A', 'B', 'C', 'D', 'E'], dtype='<U1')B. 配列要素取得
ndarrayの要素は、Pythonでのリスト要素の取得と同じ方法で取得できる。
ただし、多次元配列の場合は指定方法が変わるので注意する。X = [[1,2,3],[4,5,6]] print(X[1][2]) # 6 X_numpy = np.array(X) print(X_numpy[1,2]) # 6又、通常の取得方法とは別に次の方法でも取得できる。
1. インデックスの配列で取得する。
X = np.array([[1,2,3],[4,5,6]]) print(X[[0,1,-1]]) # [[1 2 3], # [4 5 6], # [4 5 6]] print(X[[0,1,-1], 0]) # [1, 4, 4] print(X[[0,1,-1], ::-1]) # [[3 2 1], # [6 5 4], # [6 5 4]] print(X[[0,1,-1],[1,0,-1]]) # [2, 4, 6] """ ※この指定は、次と同じ結果になっている。 np.array([X[0,1], X[1,0], X[-1,-1]]) """2. 条件式で取得する。
X = np.array([[1,2,3],[4,5,6]]) print(X > 4) # [[False, False, False], # [False, True, True]] print(np.where(X > 4)) # (array([1, 1]), array([1, 2])) print(X[X > 4]) # [5, 6] print(X[np.where(X > 4)]) # [5, 6]C. 配列情報の取得
ndarrayの情報を取得する。
【
shape】
- 各次元の要素数を取得する。
X = np.array([[1,2,3], [4,5,6]]) X.shape # (2, 3)【
dtype】
- データ型を取得する。
X = np.array([[1,2,3], [4,5,6]]) X.dtype # dtype('int64')【
ndim】
- 次元数を取得する。
X = np.array([[1,2,3], [4,5,6]]) X.ndim # 2【
size】
- 要素数を取得する。
X = np.array([[1,2,3], [4,5,6]]) X.size # 6D. 配列操作
ndarrayを操作する。
【
<ndarray>.flatten(order='C')】
- 一次元配列にする。
X = np.array([[1,2,3], [4,5,6]]) print(X.flatten()) # [1, 2, 3, 4, 5, 6]【
<ndarray>.reshape(shape, order='C')】
- 配列の形状(次元数および各次元の要素数)を変える。
X = np.array([1,2,3,4,5,6]) print(X.reshape((3,2))) # [[1, 2], # [3, 4], # [5, 6]]【
sort(a, axis=-1, kind='quicksort', order=None)】
- 並び替える。(対象をコピーし、コピーを並べ替えて返す)
- axis:ソートする次元
X1 = np.array([[5,4,6], [3,1,2]]) X2 = np.sort(X1) print(X1) # [[5 4 6] # [3 1 2]] print(X2) # [[4 5 6] # [1 2 3]] X3 = np.sort(X1, axis=0) print(X3) # [[3 1 2] # [5 4 6]]【
<ndarray>.sort(axis=-1, kind='quicksort', order=None)】
- 並び替える。(対象を並び替える)
- axis:ソートする次元
X = np.array([[5,4,6], [3,1,2]]) X.sort() print(X) # [4 5 6] # [1 2 3]] X.sort(axis=0) print(X) # [[3 1 2] # [5 4 6]]【
<ndarray>.argsort(axis=-1, kind='quicksort', order=None)】
- 並び替えた時のインデックスを取得する。
X = np.array([[5,4,6], [3,1,2]]) idxs = X.argsort() print(idxs) # [[1 0 2] # [1 2 0]] idxs = X.argsort(axis=0) print(idxs) # [[1 1 1] # [0 0 0]]【
<ndarray>.astype(dtype, order='K', casting='unsafe', subok=True, copy=True)】
- ndarrayのデータ型を変換する。
X = np.array([[1,2,3], [4,5,6]]) print(X.dtype) # int64 print(X) # [[1 2 3] # [4 5 6]] X2 = X.astype(np.float64) print(X2.dtype) # float64 print(X2) # [[1. 2. 3.] # [4. 5. 6.]]E. 行列操作
ndarrayで行列操作を行う。
【
<ndarray>.T】
- 転置
X = np.array([[1,2,3], [4,5,6]]) print(X.T) # [[1, 4], # [2, 5], # [3, 6]]【
numpy.dot】【<ndarray>.dot】
- 内積
X1 = np.array([[1, 2, 3], [4, 5, 6]]) X2 = np.array([[10, 20], [100, 200], [1000, 2000]]) print(np.dot(X1, X2)) # [[ 3210 6420] # [ 6540 13080]] print(X1.dot(X2)) # [[ 3210 6420] # [ 6540 13080]]【
numpy.trace】【<ndarray>.trace】
- 対角成分の合計
X = np.array([[1, 2, 3], [4, 5, 6], [7, 8, 9]]) print(np.trace(X)) # 15 print(X.trace()) # 15【
numpy.linalg.det】
- 行列式
X1 = np.array([[3., 2.], [5., 4.]]) print(np.linalg.det(X1)) # 2.0000000000000013【
numpy.linalg.eig】
- 固有値・固有ベクトル
X = np.array([[2, 0], [0, 0.25]]) print(np.linalg.eig(X)) # (array([2. , 0.25]), array([[1., 0.], [0., 1.]]))【
numpy.linalg.svd】
- 特異値分解
X = np.array([[2, 0], [0, 0.25], [0, 1]]) print(np.linalg.svd(X)) # (array([[ 1., 0. , 0. ], # [ 0., -0.24253563, -0.9701425 ], # [ 0., -0.9701425 , 0.24253563]]), # array([2., 1.03077641]), # array([[ 1., 0.], # [-0., -1.]]))【
numpy.linalg.inv】
- 逆行列
X = np.array([[1, 1], [1, -1]]) print(np.linalg.inv(X)) # [[ 0.5 0.5] # [ 0.5 -0.5]]【
numpy.linalg.pinv】
- ムーア・ペンローズの擬似逆行列
X = np.array([[1, 1], [1, -1], [1, 0]]) print(np.linalg.pinv(X)) # [[ 0.33333333 0.33333333 0.33333333] # [ 0.5 -0.5 0. ]]【
numpy.linalg.qr】
- QR分解
X = np.array([[1, 2], [3, 4], [5, 6]]) print(np.linalg.qr(X)) # (array([[-0.16903085, 0.89708523], # [-0.50709255, 0.27602622], # [-0.84515425, -0.34503278]]), # array([[-5.91607978, -7.43735744], # [ 0. , 0.82807867]]))F. 算術演算
二つのndarray同士で、算術演算をする。
【
add】
- 足し算(加算)
a = np.array([[1, 2, 3], [4, 5, 6]]) print(np.add(a, 10)) # [[11 12 13] # [14 15 16]] print(a + 10) # [[11 12 13] # [14 15 16]] print(np.add(a, [10, 20, 30])) # [[11 22 33] # [14 25 36]] print(a + [10, 20, 30]) # [[11 22 33] # [14 25 36]] print(np.add(a, np.array([[10, 20, 30], [40, 50, 60]]))) # [[11 22 33] # [44 55 66]] print(a + np.array([[10, 20, 30], [40, 50, 60]])) # [[11 22 33] # [44 55 66]]【
subtract】
- 引き算(減算)
a = np.array([[11, 22, 33], [44, 55, 66]]) print(np.subtract(a, 10)) # [[ 1 12 23] # [34 45 56]] print(a - 10) # [[ 1 12 23] # [34 45 56]] print(np.subtract(a, [10, 20, 30])) # [[ 1 2 3] # [34 35 36]] print(a - [10, 20, 30]) # [[ 1 2 3] # [34 35 36]] print(np.subtract(a, np.array([[10, 20, 30], [40, 50, 60]]))) # [[1 2 3] # [4 5 6]] print(a - np.array([[10, 20, 30], [40, 50, 60]])) # [[1 2 3] # [4 5 6]]【
multiply】
- 掛け算(乗算)
a = np.array([[1, 2, 3], [4, 5, 6]]) print(np.multiply(a, 10)) # [[10 20 30] # [40 50 60]] print(a * 10) # [[10 20 30] # [40 50 60]] print(np.multiply(a, [10, 20, 30])) # [[ 10 40 90] # [ 40 100 180]] print(a * [10, 20, 30]) # [[ 10 40 90] # [ 40 100 180]] print(np.multiply(a, np.array([[10, 20, 30], [40, 50, 60]]))) # [[ 10 40 90] # [160 250 360]] print(a * np.array([[10, 20, 30], [40, 50, 60]])) # [[ 10 40 90] # [160 250 360]]【
divide】
- 割り算(除算)
a = np.array([[10, 20, 30], [40, 50, 60]]) print(np.divide(a, 10)) # [[1. 2. 3.] # [4. 5. 6.]] print(a / 10) # [[1. 2. 3.] # [4. 5. 6.]] print(np.divide(a, [10, 20, 30])) # [[1. 1. 1. ] # [4. 2.5 2. ]] print(a / [10, 20, 30]) # [[1. 1. 1. ] # [4. 2.5 2. ]] print(np.divide(a, np.array([[10, 20, 30], [40, 50, 60]]))) # [[1. 1. 1.] # [1. 1. 1.]] print(a / np.array([[10, 20, 30], [40, 50, 60]])) # [[1. 1. 1.] # [1. 1. 1.]]【
floor_divide】
- 割り算の商
a = np.array([[22, 33, 44], [45, 67, 89]]) print(np.floor_divide(a, 2)) # [[11 16 22] # [22 33 44]] print(a // 2) # [[11 16 22] # [22 33 44]] print(np.floor_divide(a, [2, 3, 4])) # [[11 11 11] # [22 22 22]] print(a // [2, 3, 4]) # [[11 11 11] # [22 22 22]] print(np.floor_divide(a, np.array([[2, 3, 4], [4, 6, 8]]))) # [[11 11 11] # [11 11 11]] print(a // np.array([[[2, 3, 4], [4, 6, 8]]])) # [[11 11 11] # [11 11 11]]【
mod】
- 割り算の余り
a = np.array([[22, 33, 44], [45, 67, 89]]) print(np.mod(a, 2)) # [[0 1 0] # [1 1 1]] print(a % 2) # [[0 1 0] # [1 1 1]] print(np.mod(a, [2, 3, 4])) # [[0 0 0] # [1 1 1]] print(a % [2, 3, 4]) # [[0 0 0] # [1 1 1]] print(np.mod(a, np.array([[2, 3, 4], [4, 6, 8]]))) # [[0 0 0] # [1 1 1]] print(a % np.array([[[2, 3, 4], [4, 6, 8]]])) # [[0 0 0] # [1 1 1]]【
power】
- 累乗
a = np.array([[1, 2, 3], [4, 5, 6]]) print(np.power(a, 2)) # [[ 1 4 9] # [16 25 36]] print(a ** 2) # [[ 1 4 9] # [16 25 36]] print(np.power(a, [2, 3, 4])) # [[ 1 8 81] # [ 16 125 1296]] print(a ** [2, 3, 4]) # [[ 1 8 81] # [ 16 125 1296]] print(np.power(a, np.array([[2, 3, 4], [1/2, 1/3, 1/4]]))) # [[ 1. 8. 81. ] # [ 2. 1.70997595 1.56508458]] print(a ** np.array([[2, 3, 4], [1/2, 1/3, 1/4]])) # [[ 1. 8. 81. ] # [ 2. 1.70997595 1.56508458]]G. 比較演算
二つのndarrayを比較する。
【
greater】
- より大きい
a1 = np.array([10, 10, 10, 10, 10]) a2 = np.array([9, 9.9, 10, 10.1, 11]) print(np.greater(a1, a2)) # [ True True False False False] print(a1 > a2) # [ True True False False False]【
greater_equal】
- 以上
a1 = np.array([10, 10, 10, 10, 10]) a2 = np.array([9, 9.9, 10, 10.1, 11]) print(np.greater_equal(a1, a2)) # [ True True True False False] print(a1 >= a2) # [ True True True False False]【
less】
- より小さい
a1 = np.array([10, 10, 10, 10, 10]) a2 = np.array([9, 9.9, 10, 10.1, 11]) print(np.less(a1, a2)) # [False False False True True] print(a1 < a2) # [False False False True True]【
less_equal】
- 以下
a1 = np.array([10, 10, 10, 10, 10]) a2 = np.array([9, 9.9, 10, 10.1, 11]) print(np.less_equal(a1, a2)) # [False False True True True] print(a1 <= a2) # [False False True True True]【
equal】
- 等しい
a1 = np.array([10, 10, 10, 10, 10]) a2 = np.array([9, 9.9, 10, 10.1, 11]) print(np.equal(a1, a2)) # [False False True False False] print(a1 == a2) # [False False True False False]【
not_equal】
- 等しくない
a1 = np.array([10, 10, 10, 10, 10]) a2 = np.array([9, 9.9, 10, 10.1, 11]) print(np.not_equal(a1, a2)) # [ True True False True True] print(a1 != a2) # [ True True False True True]H. 論理演算
二つのndarrayを論理演算する。
【
logical_and】
- AND
a1 = np.array([True, False, True, False]) a2 = np.array([True, False, False, True]) print(np.logical_and(a1, a2)) # [ True False False False]【
logical_or】
- OR
a1 = np.array([True, False, True, False]) a2 = np.array([True, False, False, True]) print(np.logical_or(a1, a2)) # [ True False True True]【
logical_xor】
- XOR
a1 = np.array([True, False, True, False]) a2 = np.array([True, False, False, True]) print(np.logical_xor(a1, a2)) # [False False True True]【
logical_not】
- NOT
a = np.array([True, False, True, False]) print(np.logical_not(a)) # [False True False True]【
<ndarray>.any()】
- いずれかの要素がTrue
a1 = np.array([0, 1, 0, 0]) print(a1.any()) # True a2 = np.array([1, 1, 1, 1]) print(a2.any()) # True a3 = np.array([0, 0, 0, 0]) print(a3.any()) # False【
<ndarray>.all()】
- すべての要素がTrue
a1 = np.array([0, 1, 0, 0]) print(a1.all()) # False a2 = np.array([1, 1, 1, 1]) print(a2.all()) # True a3 = np.array([0, 0, 0, 0]) print(a3.all()) # FalseI. 集合演算
ndarrayでの集合演算をする。
※集合演算自体はPythonのsetでも行える。参考:Pythonの集合演算【
union1d(ar1, ar2)】
- 和集合(OR)
X = np.unique([1,2,3,4,5,6]) Y = np.unique([4,5,6,7,8,9]) Z = np.union1d(X,Y) print(Z) # [1 2 3 4 5 6 7 8 9]【
intersect1d(ar1, ar2)】
- 積集合(AND)
X = np.unique([1,2,3,4,5,6]) Y = np.unique([4,5,6,7,8,9]) Z = np.intersect1d(X,Y) print(Z) # [4 5 6]【
setxor1d(ar1, ar2)】
- 対称差(XOR)
X = np.unique([1,2,3,4,5,6]) Y = np.unique([4,5,6,7,8,9]) Z = np.setxor1d(X,Y) print(Z) # [1 2 3 7 8 9]【
setdiff1d(ar1, ar2)】
- 差集合
X = np.unique([1,2,3,4,5,6]) Y = np.unique([4,5,6,7,8,9]) Z = np.setdiff1d(X,Y) print(Z) # [1 2 3]【
in1d(ar1, ar2)】
- 前者の集合の各要素が後者に含まれるかの判定
X = np.unique([1,2,3,4,5]) Y = np.unique([1,2,3]) Z = np.in1d(X,Y) print(Z) # [ True True True False False]J. 最大値・最小値・符号抽出
ndarrayの最大値・最小値・符号を抽出する。
【
maximum】
- 最大値(2つのndarrayを比較し、大きい方を取得)
- 片方の要素がNaNの場合は、NaNを取得。
a1 = np.array([4, 2, np.nan]) a2 = np.array([3, 6, 5]) print(np.maximum(a1, a2)) # [ 4. 6. nan]【
fmax】
- 最大値(2つのndarrayを比較し、大きい方を取得)
- 片方の要素がNaNの場合は、もう片方を取得。
a1 = np.array([4, 2, np.nan]) a2 = np.array([3, 6, 5]) print(np.fmax(a1, a2)) # [4. 6. 5.]【
minimum】
- 最小値(2つのndarrayを比較し、小さい方を取得)
- 片方の要素がNaNの場合は、NaNを取得。
a1 = np.array([4, 2, np.nan]) a2 = np.array([3, 6, 5]) print(np.minimum(a1, a2)) # [ 3. 2. nan]【
fmin】
- 最小値(2つのndarrayを比較し、小さい方を取得)
- 片方の要素がNaNの場合は、もう片方を取得。
a1 = np.array([4, 2, np.nan]) a2 = np.array([3, 6, 5]) print(np.fmin(a1, a2)) # [3. 2. 5.]【
copysign】
- 1つ目のndarrayに、2つ目のndarrayで対応する要素の符号を当てる。
a1 = np.array([1, 2, 3, 4, 5]) a2 = np.array([0, -1, 1, 2, -2]) print(np.copysign(a1, a2)) # [ 1. -2. 3. 4. -5.]K. 切り上げ・切り捨て・四捨五入
ndarrayの各値で、小数点以下の値を処理する。
【
ceil(ndarray)】
- 切り上げ
y = np.array([-5.6, -5.5, -5.4, 0, 5.4, 5.5, 5.6]) z = np.ceil(y) print(z) # [-5. -5. -5. 0. 6. 6. 6.]【
floor(ndarray)】
- 切り捨て
y = np.array([-5.6, -5.5, -5.4, 0, 5.4, 5.5, 5.6]) z = np.floor(y) print(z) # [-6. -6. -6. 0. 5. 5. 5.]【
rint(ndarray)】
- 四捨五入
y = np.array([-5.6, -5.5, -5.4, 0, 5.4, 5.5, 5.6]) z = np.rint(y) print(z) # [-6. -6. -5. 0. 5. 6. 6.]【
modf(ndarray)】
- 整数部と小数部の分割
y = np.array([-3.6, -2.5, -1.4, 0, 1.4, 2.5, 3.6]) z = np.modf(y) print(z) # (array([-0.6, -0.5, -0.4, 0. , 0.4, 0.5, 0.6]), array([-3., -2., -1., 0., 1., 2., 3.]))L. 二乗・平方根・絶対値
ndarrayの各値で、二乗・平方根・絶対値を算出する。
【
square(ndarray)】
- 二乗
y = np.arange(1,6) z = np.square(y) print(z) # [ 1 4 9 16 25] z = y ** 2 print(z) # [ 1 4 9 16 25]【
sqrt(ndarray)】
- 平方根
y = np.array([1,4,9,16,25]) z = np.sqrt(y) print(z) # [1. 2. 3. 4. 5.] z = y ** 0.5 print(z) # [1. 2. 3. 4. 5.]【
abs(ndarray)】
- 絶対値
y = np.array([-1,-2,3,4,5j]) z = np.abs(y) print(z) # [1, 2, 3, 4, 5]【
fabs(ndarray)】
- 絶対値(虚数・複素数不可だが、absより高速)
y = np.array([-1,-2,3,4,-5]) z = np.fabs(y) print(z) # [1, 2, 3, 4, 5]M. 指数・対数関数
ndarrayの各値で、指数・対数を算出する。
【
exp(ndarray)】
- 指数
y = np.arange(1,6) z = np.exp(y) print(z) # [ 2.71828183 7.3890561 20.08553692 54.59815003 148.4131591 ] print(np.e) # 2.718281828459045【
log(ndarray)】
- 自然対数
y = np.e ** np.array([1, 2, 3, 4, 5]) z = np.log(y) print(z) # [1. 2. 3. 4. 5.]【
log10(ndarray)】
- 常用対数
y = 10 ** np.array([1, 2, 3, 4, 5]) z = np.log10(y) print(z) # [1. 2. 3. 4. 5.]【
log2(ndarray)】
- 二進対数
y = 2 ** np.array([1, 2, 3, 4, 5]) z = np.log2(y) print(z) # [1. 2. 3. 4. 5.]N. 三角関数
ndarrayの各値に、三角関数を適用する。
【
sin(ndarray)】
- 正弦
y = np.linspace(-1, 1, 5) * np.pi print(np.sin(y)) # [-1.2246468e-16 -1.0000000e+00 0.0000000e+00 1.0000000e+00 1.2246468e-16]【
cos(ndarray)】
- 余弦
y = np.linspace(-1, 1, 5) * np.pi print(np.cos(y)) # [-1.000000e+00 6.123234e-17 1.000000e+00 6.123234e-17 -1.000000e+00]【
tan(ndarray)】
- 正接
y = np.linspace(-1, 1, 5) * np.pi print(np.tan(y)) # [ 1.22464680e-16 -1.63312394e+16 0.00000000e+00 1.63312394e+16 -1.22464680e-16]O. 双曲線関数
ndarrayの各値に、双曲線関数を適用する。
【
sinh(ndarray)】
- 双曲線正弦
y = np.array([-np.inf, -2, -1, 0, 1, 2, np.inf]) print(np.sinh(y)) # [-inf -3.62686041 -1.17520119 0. 1.17520119 3.62686041 inf]【
cosh(ndarray)】
- 双曲線余弦
y = np.array([-np.inf, -2, -1, 0, 1, 2, np.inf]) print(np.cosh(y)) # [inf 3.76219569 1.54308063 1. 1.54308063 3.76219569 inf]【
tanh(ndarray)】
- 双曲線正接
y = np.array([-np.inf, -2, -1, 0, 1, 2, np.inf]) print(np.tanh(y)) # [-1. -0.96402758 -0.76159416 0. 0.76159416 0.96402758 1.]P. 逆三角関数
ndarrayの各値に、逆三角関数を適用する。
【
arcsin(ndarray)】
- 逆正弦
y = np.linspace(-1, 1, 5) print(np.arcsin(y)) # [-1.57079633 -0.52359878 0. 0.52359878 1.57079633]【
arccos(ndarray)】
- 逆余弦
y = np.linspace(-1, 1, 5) print(np.arccos(y)) # [3.14159265 2.0943951 1.57079633 1.04719755 0.]【
arctan(ndarray)】
- 逆正接
y = np.linspace(-1, 1, 5) print(np.arctan(y)) # [-0.78539816 -0.46364761 0. 0.46364761 0.78539816]Q. 逆双曲線関数
ndarrayの各値に、逆双曲線関数を適用する。
【
arcsinh(ndarray)】
- 逆双曲線正弦
y = np.array([-np.inf, -3.62686041, -1.17520119, 0., 1.17520119, 3.62686041, np.inf]) print(np.arcsinh(y)) # [-inf -2. -1. 0. 1. 2. inf]【
arccosh(ndarray)】
- 逆双曲線余弦
y = np.array([1., 1.54308063, 3.76219569, np.inf]) print(np.arccosh(y)) # [ 0. 1. 2. inf]【
arctanh(ndarray)】
- 逆双曲線正接
y = np.array([-1., -0.96402758, -0.76159416, 0., 0.76159416, 0.96402758, 1.]) print(np.arctanh(y)) # [-inf -2. -1.00000001 0. 1.00000001 2. inf]R. 乱数生成
Numpyで乱数生成する。
【
numpy.random.rand(d0, d1, ..., dn)】
- 一様分布
- 指定した要素数/次元数の乱数配列を生成する。(乱数の範囲:0以上1未満)
print(np.random.rand()) # 0.5504876218756463 print(np.random.rand(2)) # [0.70029403 0.48969378] print(np.random.rand(2, 2)) # [[0.98181874 0.47001957] # [0.79277853 0.12536121]]【
numpy.random.randint(low, high=None, size=None, dtype='l')】
- 一様分布
- 指定範囲の整数での乱数を生成する。
- low:最小値(以上。乱数の範囲に含まれる)
- high:最大値(未満。乱数の範囲に含まれない)
- size:要素数/次元数
- dtype:データ型
print(np.random.randint(1,4)) # 2 print(np.random.randint(1,4, 2)) # [2 2] print(np.random.randint(1,4, (2, 2))) # [[3 2] # [3 1]]【
numpy.random.uniform(low=0.0, high=1.0, size=None)】
- 一様分布
- 指定範囲の乱数を生成する。
- low:最小値(以上。乱数の範囲に含まれる)
- high:最大値(未満。乱数の範囲に含まれない)
- size:要素数/次元数
print(np.random.uniform(1,4)) # 3.5515484791542056 print(np.random.uniform(1,4, 2)) # [1.51270014 3.02435494] print(np.random.uniform(1,4, (2, 2))) # [[3.47188029 1.17177563] # [3.87198389 3.91458379]]【
numpy.random.randn(d0, d1, ..., dn)】
- 正規分布
- 指定した要素数/次元数の乱数配列を生成する。(平均:0, 標準偏差:1)
print(np.random.randn()) # -0.5775065521096695 print(np.random.randn(2)) # [-1.50501689 1.46743032] print(np.random.randn(2, 2)) # [[ 1.16357112 -0.24601519] # [ 2.07269576 -0.39272309]]【
numpy.random.normal(loc=0.0, scale=1.0, size=None)】
- 正規分布
- 指定の平均・標準偏差の乱数を生成する。
- loc:平均
- scale:標準偏差
- size:要素数/次元数
print(np.random.normal(50, 10)) # 63.47995333571061 print(np.random.normal(50, 10, (2, 2))) # [[56.02364177 55.25423891] # [45.44840171 29.8303964 ]] print(np.random.normal((50, 0), (10, 1), (5, 2))) # [[45.48754234 -0.74792452] # [60.84696747 1.01209036] # [42.38844146 -0.10453915] # [39.77544056 1.22264549] # [41.60250782 1.64150462]]【
numpy.random.binomial(n, p, size=None)】
- 二項分布
- 指定の試行数・確率の乱数を生成する。
- n :試行数
- p:確率
- size:要素数/次元数
print(np.random.binomial(100, 0.5)) # 53 print(np.random.binomial(100, 0.5, (2, 2))) # [[53 53] # [48 50]] print(np.random.binomial((100, 1000), (0.3, 0.7), (5, 2))) # [[ 33 699] # [ 30 660] # [ 34 698] # [ 26 688] # [ 25 683]]【
numpy.random.beta(a, b, size=None)】
- ベータ分布
print(np.random.beta(3, 5)) # 0.09838262724430759 print(np.random.beta(8, 2, (2, 2))) # [[0.92800788 0.86391443] # [0.67249524 0.97299346]] print(np.random.beta((3, 8), (5, 2), (5, 2))) # [[0.11825463 0.74320634] # [0.24992266 0.79029969] # [0.13345269 0.57807883] # [0.32374525 0.92666103] # [0.64669681 0.84867388]]【
numpy.random.chisquare(df, size=None)
- カイ二乗分布
print(np.random.chisquare(1)) # 0.05074259859574817 print(np.random.chisquare(5, (2, 2))) # [[ 6.15617206 5.54859677] # [ 2.60704305 10.35079434]] print(np.random.chisquare((1, 5), (5, 2))) # [[2.3942405 6.43803251] # [1.97544231 2.73456762] # [0.63389405 7.81526263] # [0.05035459 7.8224598 ] # [1.01597309 1.46098368]]【
numpy.random.gamma(shape, scale=1.0, size=None)】
- ガンマ分布
- shape:形状パラメータ k
- scale:尺度パラメータ θ
print(np.random.gamma(1, 2)) # 0.48471788864900295 print(np.random.gamma(9, 1, (2, 2))) # [[10.71101589 16.68686166] # [ 5.22150652 5.87160223]] print(np.random.gamma((1, 9), (2, 1), (5, 2))) # [[ 3.4102224 6.31938602] # [ 0.03882968 7.71108072] # [ 2.62781738 10.70853193] # [ 5.07929584 5.83489052] # [ 1.50577929 11.21572879]]S. 基本統計
ndarrayの値から基本統計量を算出する。
【
max】
- 最大値
a = np.array([10, 20, 30, 40, 50]) print(np.max(a)) # 50【
argmax】
- 最大値のインデックス
a = np.array([10, 20, 30, 40, 50]) print(np.argmax(a)) # 4【
min】
- 最小値
a = np.array([10, 20, 30, 40, 50]) print(np.min(a)) # 10【
argmin】
- 最小値のインデックス
a = np.array([10, 20, 30, 40, 50]) print(np.argmin(a)) # 0【
sum】
- 合計
a = np.array([10, 20, 30, 40, 50]) print(np.sum(a)) # 150【
mean】
- 平均値
a = np.array([10, 20, 30, 40, 50]) print(np.mean(a)) # 30.0【
var】
- 分散
a = np.array([10, 20, 30, 40, 50]) print(np.var(a)) # 200.0【
std】
- 標準偏差
a = np.array([10, 20, 30, 40, 50]) print(np.std(a)) # 14.142135623730951【
cumsum】
- 累積和
a = np.array([10, 20, 30, 40, 50]) print(np.cumsum(a)) # [ 10 30 60 100 150]【
cumsum】
- 累積積
a = np.array([10, 20, 30, 40, 50]) print(np.cumprod(a)) # [ 10 200 6000 240000 12000000]T. 検証処理
値の検証処理を行う。
【
isnan(ndarray)】
- NaN(Not a number)か否かの判定
y = np.array([np.nan, np.inf, 0, 1]) print(np.isnan(y)) # [ True False False False]【
isfinite(ndarray)】
- 有限(無限でもなくNaNでもない)か否かの判定
y = np.array([np.nan, np.inf, -np.inf, 0, 1]) print(np.isfinite(y)) # [False False False True True]【
isfin(ndarray)】
- 無限(有限でもなくNaNでもない)か否かの判定
y = np.array([np.nan, np.inf, -np.inf, 0, 1]) print(np.isinf(y)) # [False True True False False]【
sign(ndarray)】
- 符号の判定
y = np.array([-np.inf, -5, 0, 5, np.inf]) print(np.sign(y)) # [-1. -1. 0. 1. 1.]備考
本記事はブログ「雑用エンジニアの技術ノート」からの移行記事です。先のブログは削除予定です。
- 投稿日:2020-09-13T18:35:36+09:00
[CovsirPhy] COVID-19データ解析用Pythonパッケージ: Parameter estimation
Introduction
COVID-19のデータ(PCR陽性者数など)のデータを簡単にダウンロードして解析できるPythonパッケージ CovsirPhyを作成しています。
紹介記事:
今回はParameter estimation(SIR-F modelなどのパラメータ推定)のご紹介です。
英語版のドキュメントはCovsirPhy: COVID-19 analysis with phase-dependent SIRs, Kaggle: COVID-19 data with SIR modelをご参照ください。
1. 実行環境
CovsirPhyは下記方法でインストールできます!Python 3.7以上, もしくはGoogle Colaboratoryをご使用ください。
- 安定版:
pip install covsirphy --upgrade- 開発版:
pip install "git+https://github.com/lisphilar/covid19-sir.git#egg=covsirphy"import covsirphy as cs cs.__version__ # '2.8.2'
実行環境 OS Windows Subsystem for Linux / Ubuntu Python version 3.8.5 2. 準備
本記事の表及びグラフは2020/9/12時点のデータを使用して作成しました。実データをCOVID-19 Data Hub1よりダウンロードするコード2はこちら:
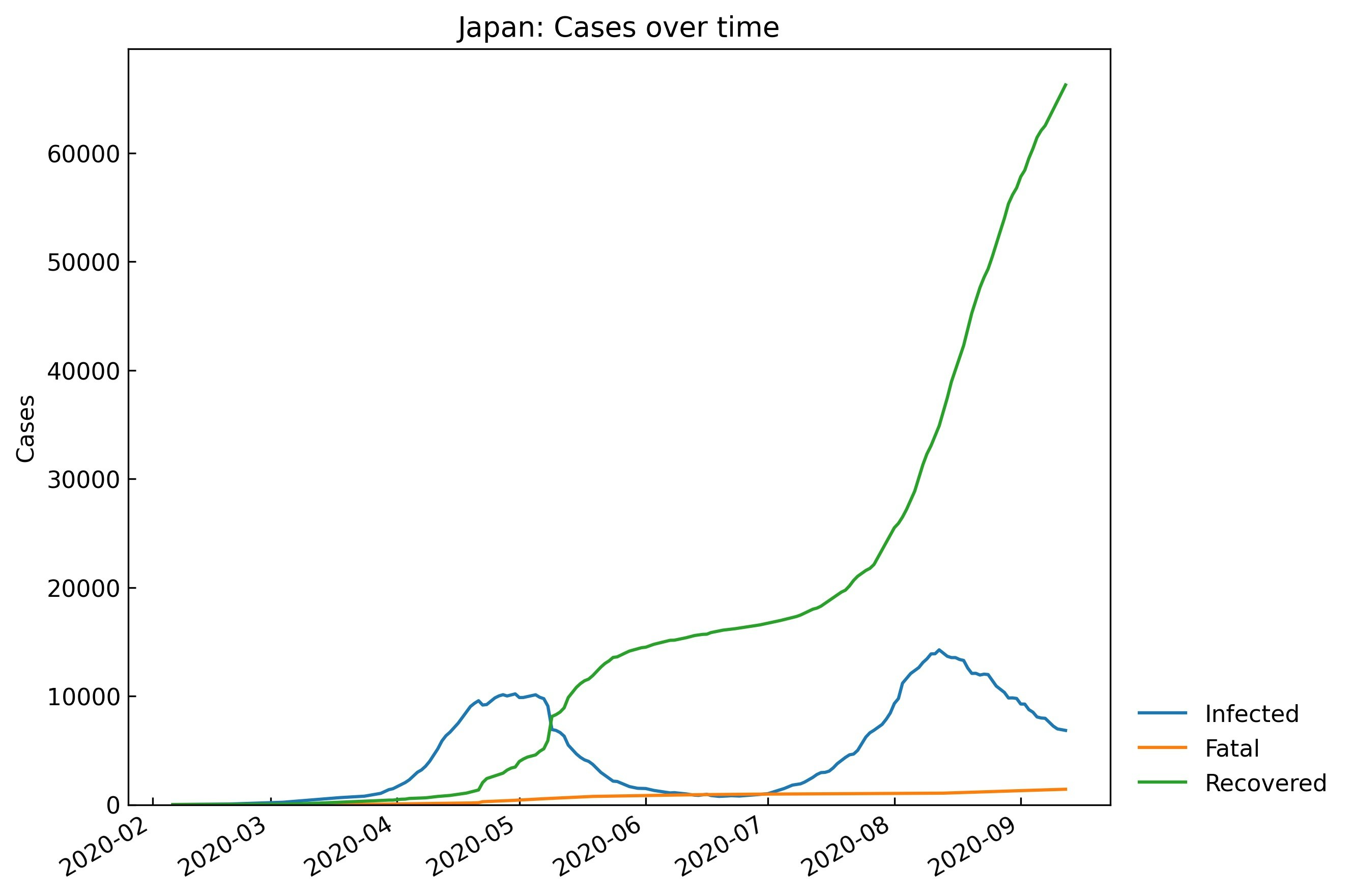
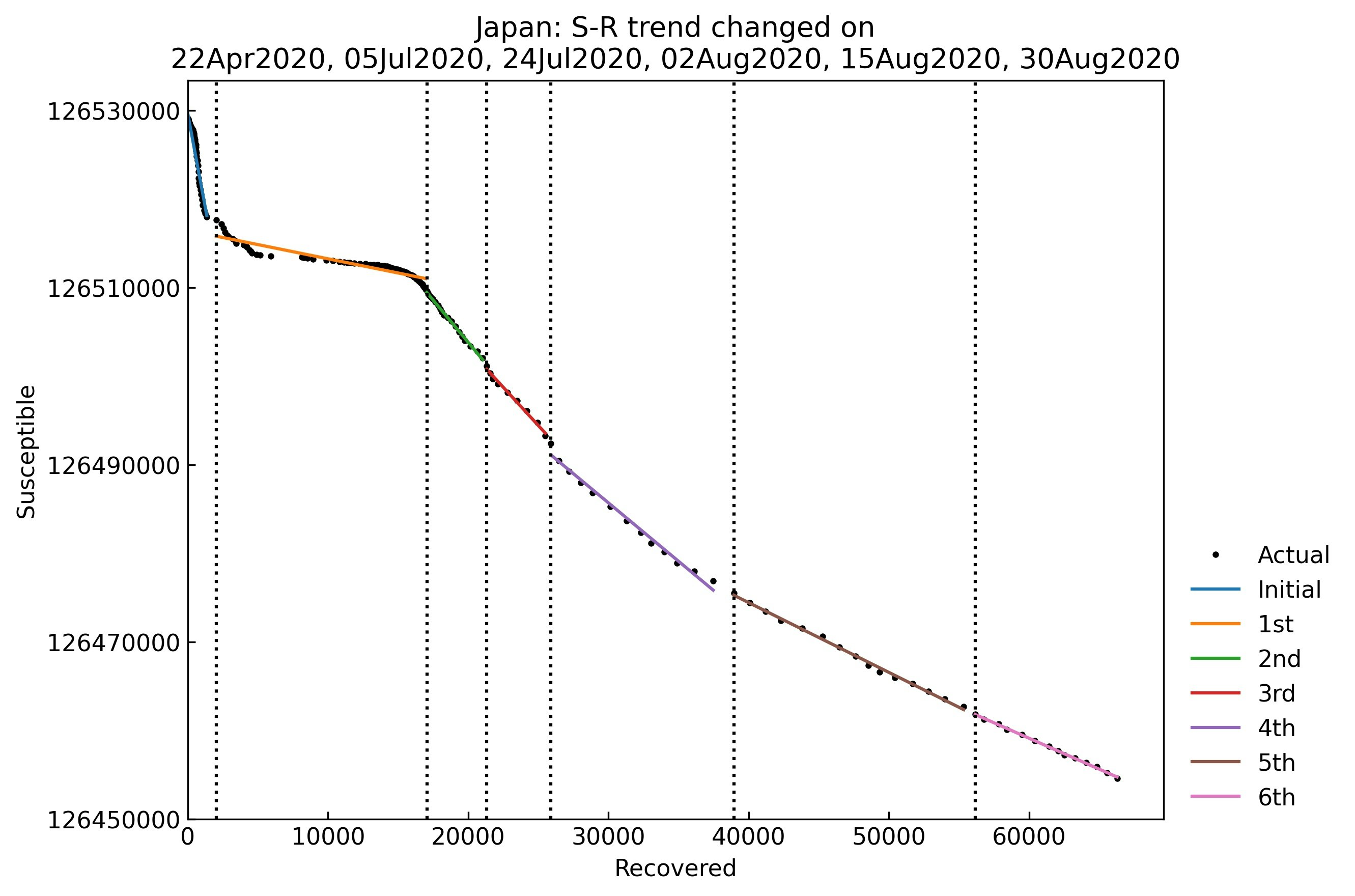
data_loader = cs.DataLoader("input") jhu_data = data_loader.jhu() population_data = data_loader.population()また、下記コードにより実データの確認とS-R trend analysis(感染拡大状況のトレンドの分析法)3を実行してください。
# (Optional) 厚生労働省データの取得 japan_data = data_loader.japan() jhu_data.replace(japan_data) print(japan_data.citation) # 解析用クラスのインスタンス生成 snl = cs.Scenario(jhu_data, population_data, country="Japan") # 実データの確認 snl.records(filename=None) # S-R trend analysis snl.trend(filename=None) # Phase設定の確認 snl.summary()Phase設定:
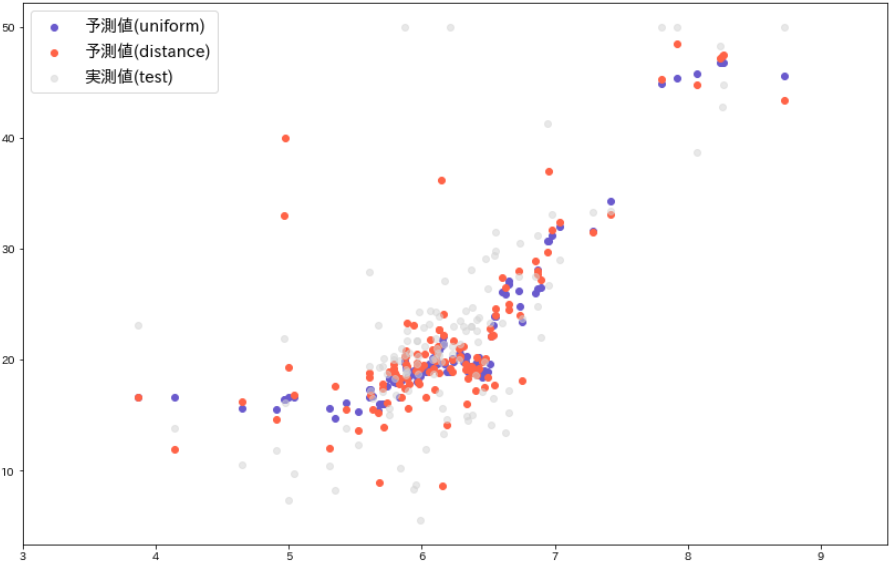
Type Start End Population 0th Past 06Feb2020 21Apr2020 126529100 1st Past 22Apr2020 04Jul2020 126529100 2nd Past 05Jul2020 23Jul2020 126529100 3rd Past 24Jul2020 01Aug2020 126529100 4th Past 02Aug2020 14Aug2020 126529100 5th Past 15Aug2020 29Aug2020 126529100 6th Past 30Aug2020 12Sep2020 126529100 3. 実行例
S-R trend analysisにより、"Phase"(パラメータが一定となる期間)に分割することができました。本記事では、各"Phase"のデータ(例えば0th phaseであれば2020/2/6 - 2020/4/21のデータ)を用いてパラメータの値を推定します。
推定のしくみについては別記事を作成する予定です。
optunaパッケージを使ってパラメータの値を提案、scipy.integrate.solve_ivpによって数値解を計算、実データとの誤差の少ないパラメータセットを選び出しています。結果の見方は後述しますが、下記2行で実行・結果一覧の取得ができます。今回はSIR-F model4を使用しました。
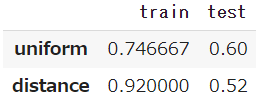
# Parameter estimation with SIR-F model snl.estimate(cs.SIRF) # パラメータ一覧の取得 snl.summary()# 標準出力の例(CPU数などによって処理時間は異なる) # 詳細後述:最新のPhase = 6th phaseにてtauを含めた推定を行った後、tauを固定して0-5thのパラメータ推定 <SIR-F model: parameter estimation> Running optimization with 8 CPUs... 6th phase (30Aug2020 - 12Sep2020): finished 704 trials in 0 min 25 sec 5th phase (15Aug2020 - 29Aug2020): finished 965 trials in 1 min 0 sec 3rd phase (24Jul2020 - 01Aug2020): finished 965 trials in 1 min 0 sec 1st phase (22Apr2020 - 04Jul2020): finished 913 trials in 1 min 0 sec 4th phase (02Aug2020 - 14Aug2020): finished 969 trials in 1 min 0 sec 0th phase (06Feb2020 - 21Apr2020): finished 853 trials in 1 min 0 sec 2nd phase (05Jul2020 - 23Jul2020): finished 964 trials in 1 min 0 sec Completed optimization. Total: 1 min 27 sec
Type Start End Population ODE Rt theta kappa rho sigma tau 1/alpha2 [day] 1/gamma [day] alpha1 [-] 1/beta [day] RMSLE Trials Runtime 0th Past 06Feb2020 21Apr2020 126529100 SIR-F 5.54 0.0258495 0.0002422 0.0322916 0.00543343 480 1376 61 0.026 10 1.17429 804 1 min 0 sec 1st Past 22Apr2020 04Jul2020 126529100 SIR-F 0.41 0.0730748 0.000267108 0.0118168 0.0264994 480 1247 12 0.073 28 1.11459 861 1 min 0 sec 2nd Past 05Jul2020 23Jul2020 126529100 SIR-F 2.01 0.000344333 7.92419e-05 0.0467789 0.023201 480 4206 14 0 7 0.0331522 910 1 min 0 sec 3rd Past 24Jul2020 01Aug2020 126529100 SIR-F 1.75 0.00169155 4.05087e-05 0.0459332 0.0260965 480 8228 12 0.002 7 0.0201773 923 1 min 0 sec 4th Past 02Aug2020 14Aug2020 126529100 SIR-F 1.46 0.000634554 0.000116581 0.0325815 0.0221345 480 2859 15 0.001 10 0.0751473 909 1 min 0 sec 5th Past 15Aug2020 29Aug2020 126529100 SIR-F 0.8 0.00244294 9.30884e-05 0.0272693 0.0337857 480 3580 9 0.002 12 0.0420563 907 1 min 0 sec 6th Past 30Aug2020 12Sep2020 126529100 SIR-F 0.69 5.36037e-05 0.000467824 0.0219379 0.0312686 480 712 10 0 15 0.0132161 724 0 min 25 sec 4. パラメータ推定値
横に長いので順番に見ていきましょう。まずはパラメータの推定値です。
# cs.SIRF.PARAMETERS: SIR-F modelのパラメータ名リスト cols = ["Start", "End", "ODE", "tau", *cs.SIRF.PARAMETERS] snl.summary(columns=cols)
Start End ODE tau theta kappa rho sigma 0th 06Feb2020 21Apr2020 SIR-F 480 0.0258495 0.0002422 0.0322916 0.00543343 1st 22Apr2020 04Jul2020 SIR-F 480 0.0730748 0.000267108 0.0118168 0.0264994 2nd 05Jul2020 23Jul2020 SIR-F 480 0.000344333 7.92419e-05 0.0467789 0.023201 3rd 24Jul2020 01Aug2020 SIR-F 480 0.00169155 4.05087e-05 0.0459332 0.0260965 4th 02Aug2020 14Aug2020 SIR-F 480 0.000634554 0.000116581 0.0325815 0.0221345 5th 15Aug2020 29Aug2020 SIR-F 480 0.000644851 0.000383424 0.0274104 0.0337156 6th 30Aug2020 12Sep2020 SIR-F 480 5.36037e-05 0.000467824 0.0219379 0.0312686 SIR-F model4:
\begin{align*} \mathrm{S} \overset{\beta I}{\longrightarrow} \mathrm{S}^\ast \overset{\alpha_1}{\longrightarrow}\ & \mathrm{F} \\ \mathrm{S}^\ast \overset{1 - \alpha_1}{\longrightarrow}\ & \mathrm{I} \overset{\gamma}{\longrightarrow} \mathrm{R} \\ & \mathrm{I} \overset{\alpha_2}{\longrightarrow} \mathrm{F} \\ \end{align*}$\alpha_2$, $\beta$, $\gamma$は「時間」を次元として有しています。この「時間」は連立常微分方程式$f'(T)=x(T)$を離散化した方程式$f(T+\Delta T) - f(T) = x(T) \Delta T$の$\Delta T$に依存しています。1日おきにデータを取得している以上$\Delta T$は1日(=1440 min)未満となりますが、具体的な値はわかりません。国や地域によって異なる可能性もあり、異なる国の有次元パラメータ$\alpha_2$, $\beta$, $\gamma$の比較を行うことは困難です。
そこで$\Delta T$に相当する変数$\tau$を設定し、有次元のパラメータを無次元化しました。
\begin{align*} (S, I, R, F) = & N \times (x, y, z, w) \\ (T, \alpha_1, \alpha_2, \beta, \gamma) = & (\tau t, \theta, \tau^{-1}\kappa, \tau^{-1}\rho, \tau^{-1}\sigma) \\ 1 \leq \tau & \leq 1440 \\ \end{align*}このとき4、
\begin{align*} & 0 \leq (x, y, z, w, \theta, \kappa, \rho, \sigma) \leq 1 \\ \end{align*}同一国内では$\tau$の値が一定となるように、最新のPhaseデータを用いて$\tau$と無次元パラメータを推定した後、他のPhaseのパラメータ推定時には同じ値を$\tau$として使用するようにしています。今回は表の通り480 minとなりました。計算を単純化するため1日=1440 minの約数を使用するようにプログラミングしています。
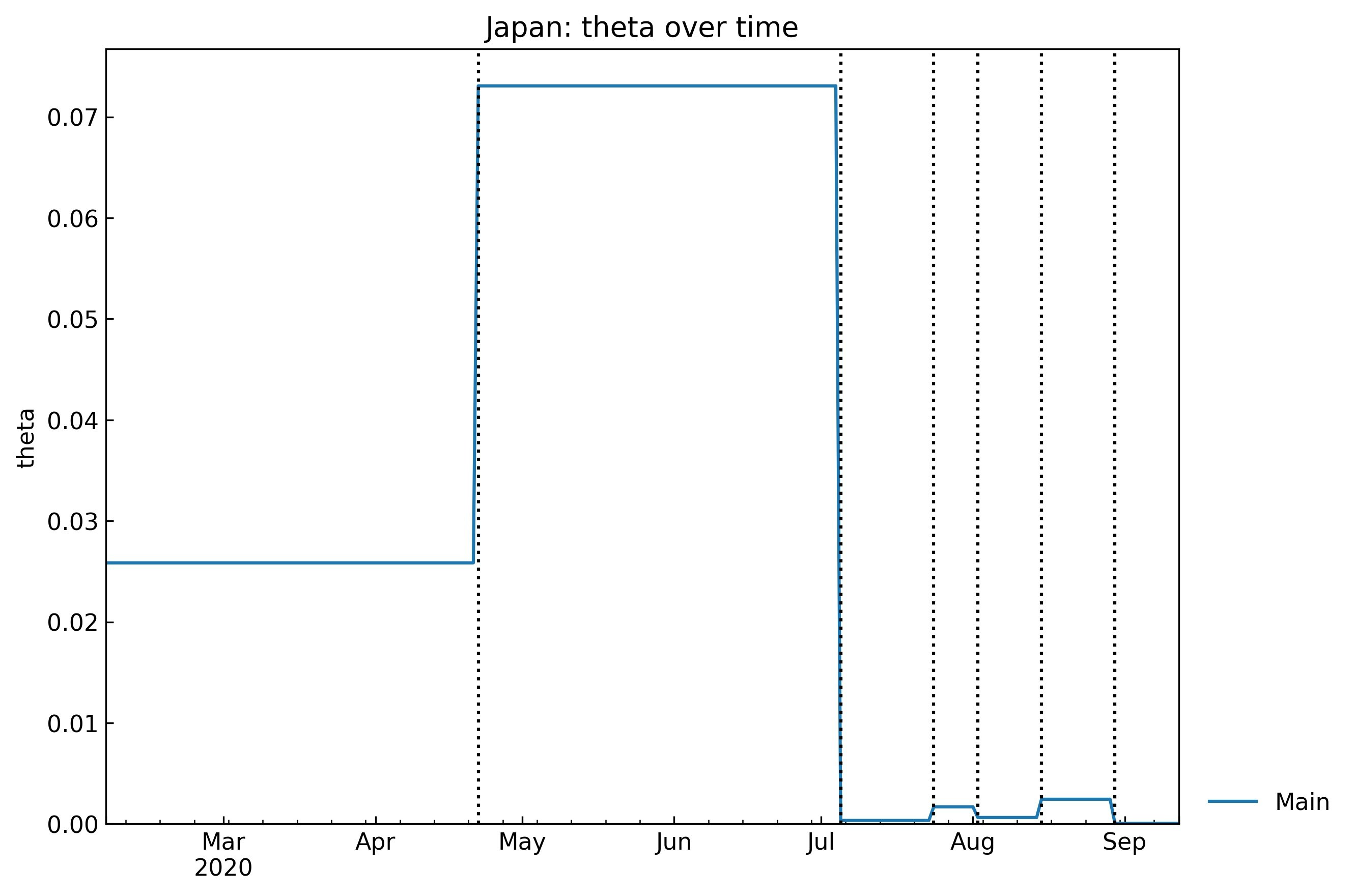
では、各無次元パラメータの推移をグラフ化してみましょう。
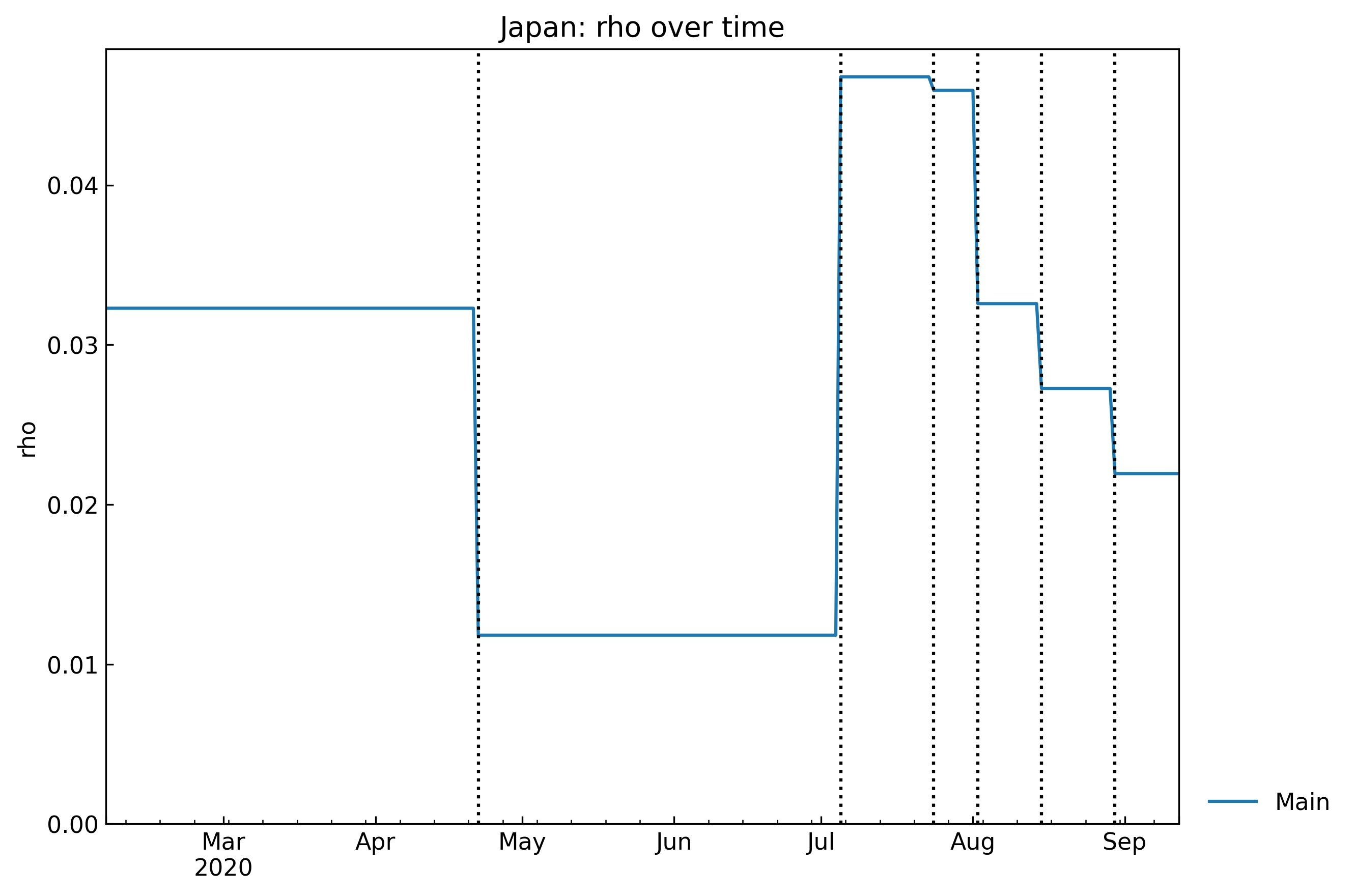
\begin{align*} & \frac{\mathrm{d}x}{\mathrm{d}t}= - \rho x y \\ & \frac{\mathrm{d}y}{\mathrm{d}t}= \rho (1-\theta) x y - (\sigma + \kappa) y \\ & \frac{\mathrm{d}z}{\mathrm{d}t}= \sigma y \\ & \frac{\mathrm{d}w}{\mathrm{d}t}= \rho \theta x y + \kappa y \\ \end{align*}rhoの推移
Susceptible(感受性保持者)がInfected(感染者)と接触したとき、感染する確率 $\rho$の推移:
snl.history(target="rho", filename=None)緊急事態宣言(区域限定2020/4/7, 全国2020/4/16, 全国で解除2020/5/25)の効果が4月後半に現れ、7月初旬まで維持されていた、と解釈しています。その後はね上がり、少しずつ低下してきています。詳細については議論が必要ですが、3密回避などの対策の効果が直接的に反映されるパラメータとなっています。
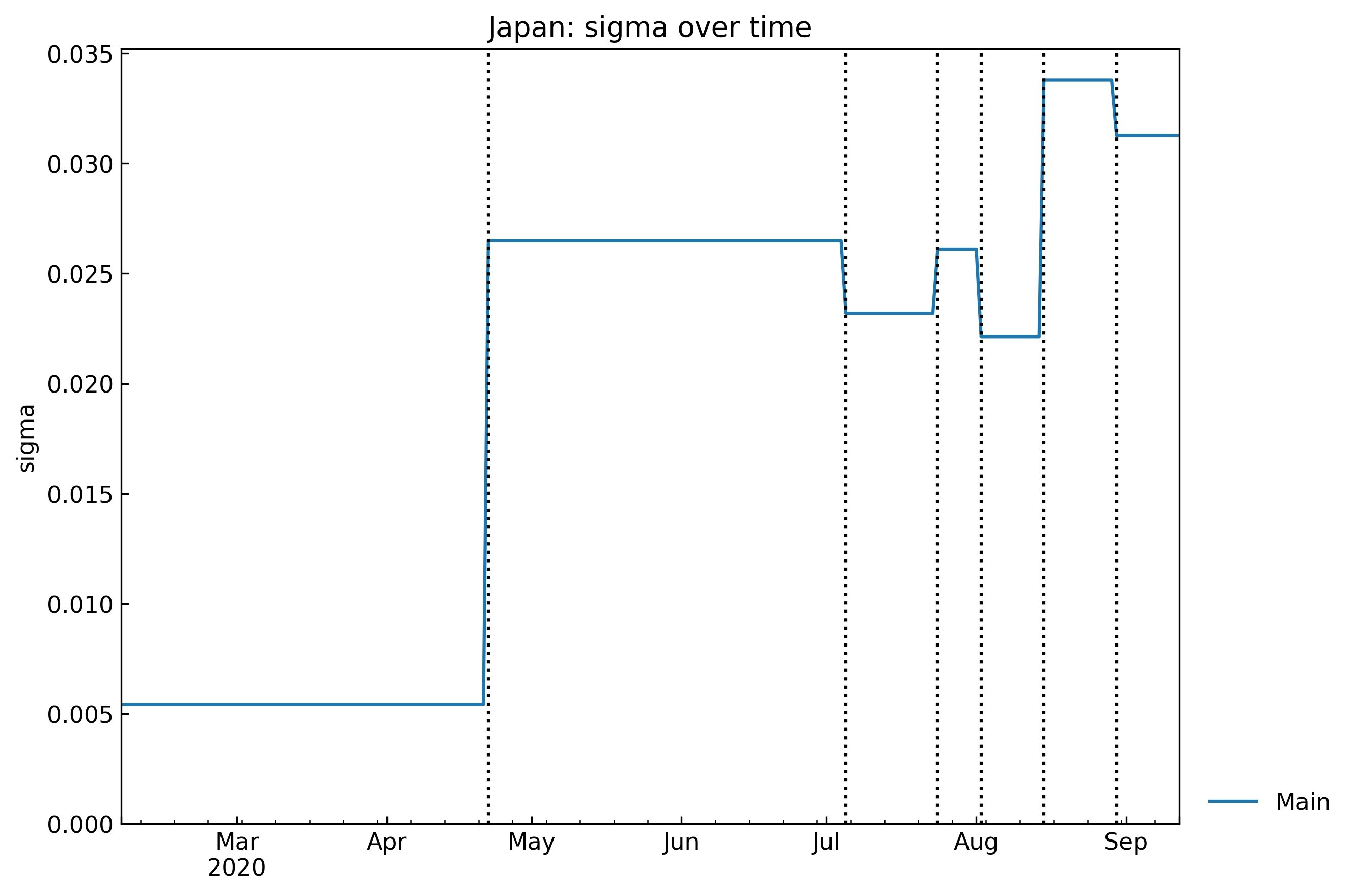
sigmaの推移
InfectedからRecovered(回復者)に移行する確率 $\sigma$の推移:
snl.history(target="sigma", filename=None)4月後半に大きく上昇した後、上昇/下降を繰り返しながら上昇傾向にあります。医療提供体制、新薬の開発/供給状況が反映されるパラメータです。
kappaの推移
感染者の死亡率$\kappa$の推移:
snl.history(target="kappa", filename=None)大きく変動しているように見えますが、値の絶対値が小さいため、ある程度一定に抑えられていると考えてよいのではないでしょうか(他国との比較による検証が必要)。医療体制を整え、新薬の十分な供給により$\kappa$を限りなく0に近づけていくことが必要です。
thetaの推移
確定診断を受けた感染者のうち、確定診断の時点で亡くなっていた感染者の割合$\theta$の推移:
snl.history(target="theta", filename=None)感染初期においては医療提供体制自体が極めて逼迫していたため解釈は難しいですが、検査が遅れて適切な治療を行えなかった場合などに値が上昇すると考えられます。
5. 有次元パラメータの推移
有次元のパラメータについても表に含まれています。解釈しやすくするため、もともと無次元の$\alpha_1$を除き、逆数にして単位を[day]にしています。
cols = ["Start", "End", "ODE", "tau", *cs.SIRF.DAY_PARAMETERS] fh.write(snl.summary(columns=cols).to_markdown())
Start End ODE tau alpha1 [-] 1/alpha2 [day] 1/beta [day] 1/gamma [day] 0th 06Feb2020 21Apr2020 SIR-F 480 0.026 1376 10 61 1st 22Apr2020 04Jul2020 SIR-F 480 0.073 1247 28 12 2nd 05Jul2020 23Jul2020 SIR-F 480 0 4206 7 14 3rd 24Jul2020 01Aug2020 SIR-F 480 0.002 8228 7 12 4th 02Aug2020 14Aug2020 SIR-F 480 0.001 2859 10 15 5th 15Aug2020 29Aug2020 SIR-F 480 0.002 3580 12 9 6th 30Aug2020 12Sep2020 SIR-F 480 0 712 15 10 有次元パラメータは無次元パラメータと同じ経過を示すため省略しますが、グラフは下記コードで取得できます。
# betaの場合 -> グラフ省略 snl.history(target="1/beta [day]", filename=None)6. 実効産生産数の推移
SIR-F model4の基本/実効産生産数Rtは次の通り定義しています。
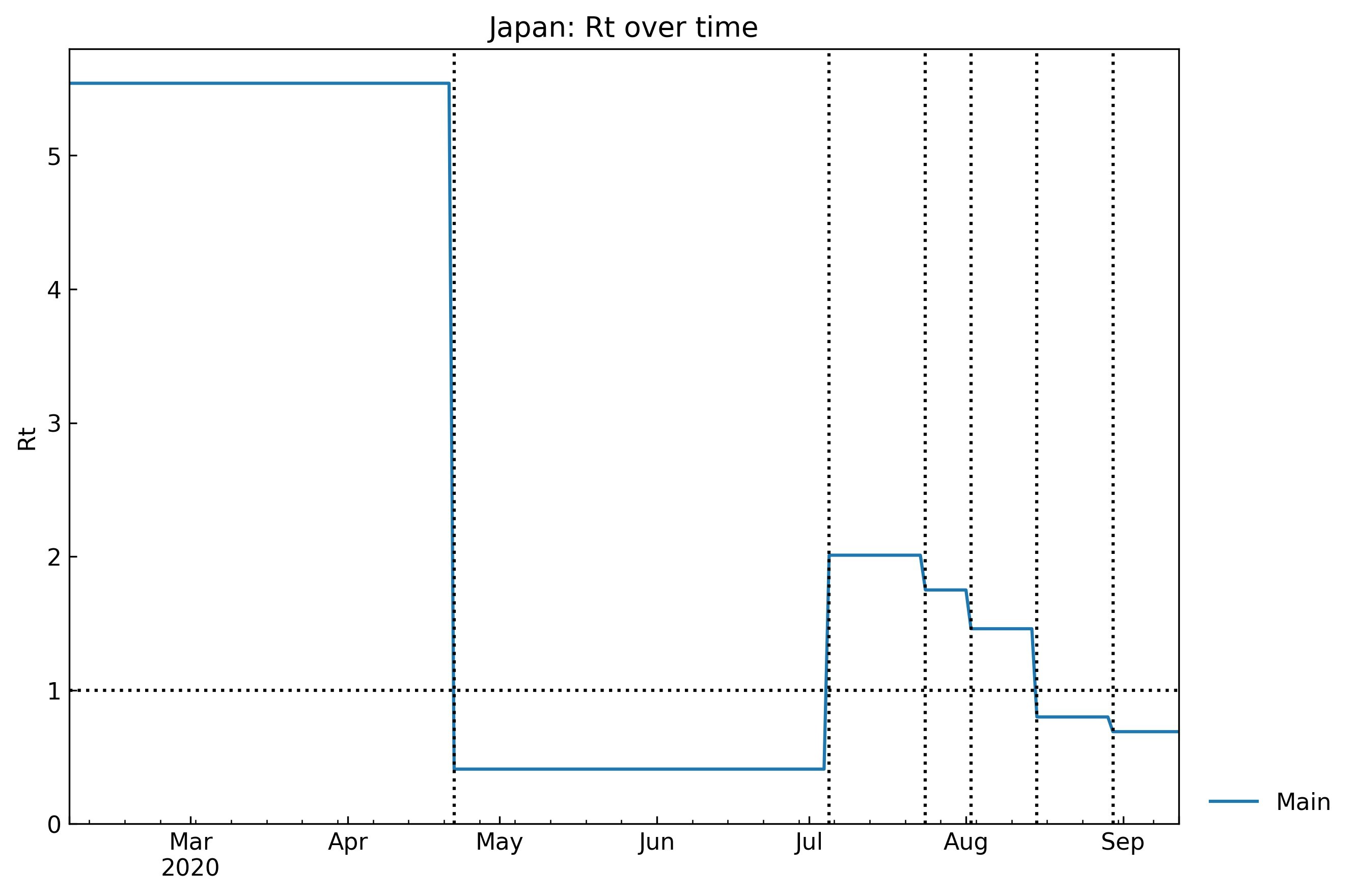
\begin{align*} R_t = \rho (1 - \theta) (\sigma + \kappa)^{-1} = \beta (1 - \alpha_1) (\gamma + \alpha_2)^{-1} \end{align*}推移:
# 一覧 cols = ["Start", "End", "ODE", "tau", "Rt"] snl.summary(columns=cols) # グラフ snl.history(target="Rt", filename="rt.jpg")
Start End ODE Rt 0th 06Feb2020 21Apr2020 SIR-F 5.54 1st 22Apr2020 04Jul2020 SIR-F 0.41 2nd 05Jul2020 23Jul2020 SIR-F 2.01 3rd 24Jul2020 01Aug2020 SIR-F 1.75 4th 02Aug2020 14Aug2020 SIR-F 1.46 5th 15Aug2020 29Aug2020 SIR-F 0.8 6th 30Aug2020 12Sep2020 SIR-F 0.69 $Rt > 1$が感染拡大の1つの目安となるため、水平線$Rt=1$を表示させました。
7. パラメータの正確性
パラメータの正確性を測るRMSLE score, パラメータを推定するために
optunaパッケージが提案したパラメータセットの数、実行時間についても表に含まれています。cols = ["Start", "End", "RMSLE", "Trials", "Runtime"] snl.summary(columns=cols)
Start End RMSLE Trials Runtime 0th 06Feb2020 21Apr2020 1.17429 690 1 min 1 sec 1st 22Apr2020 04Jul2020 1.11459 764 1 min 0 sec 2nd 05Jul2020 23Jul2020 0.0331522 810 1 min 1 sec 3rd 24Jul2020 01Aug2020 0.0201773 816 1 min 1 sec 4th 02Aug2020 14Aug2020 0.0751473 808 1 min 0 sec 5th 15Aug2020 29Aug2020 0.0420563 804 1 min 0 sec 6th 30Aug2020 12Sep2020 0.0132161 658 0 min 25 sec RMSLE score (Root Mean Squared Log Error)5の定義式は次の通りです。0に近いほど実データをよく反映していると言えます。省略しますが、推定方法の検証自体は理論データ(SIR-F modelの式とパラメータセットの例から理論的に作成される患者数データ)を使って行っております。
\begin{align*} & \sqrt{\cfrac{1}{n}\sum_{i=1}^{n}(log_{10}(A_{i} + 1) - log_{10}(P_{i} + 1))^2} \end{align*}$A$は実データ, $P$は予測値を示しています。$i=1, 2, 3, 4(=n)$のとき、$A_i$及び$P_i$はそれぞれ$S, I, R, F$の実データ/予測値です。
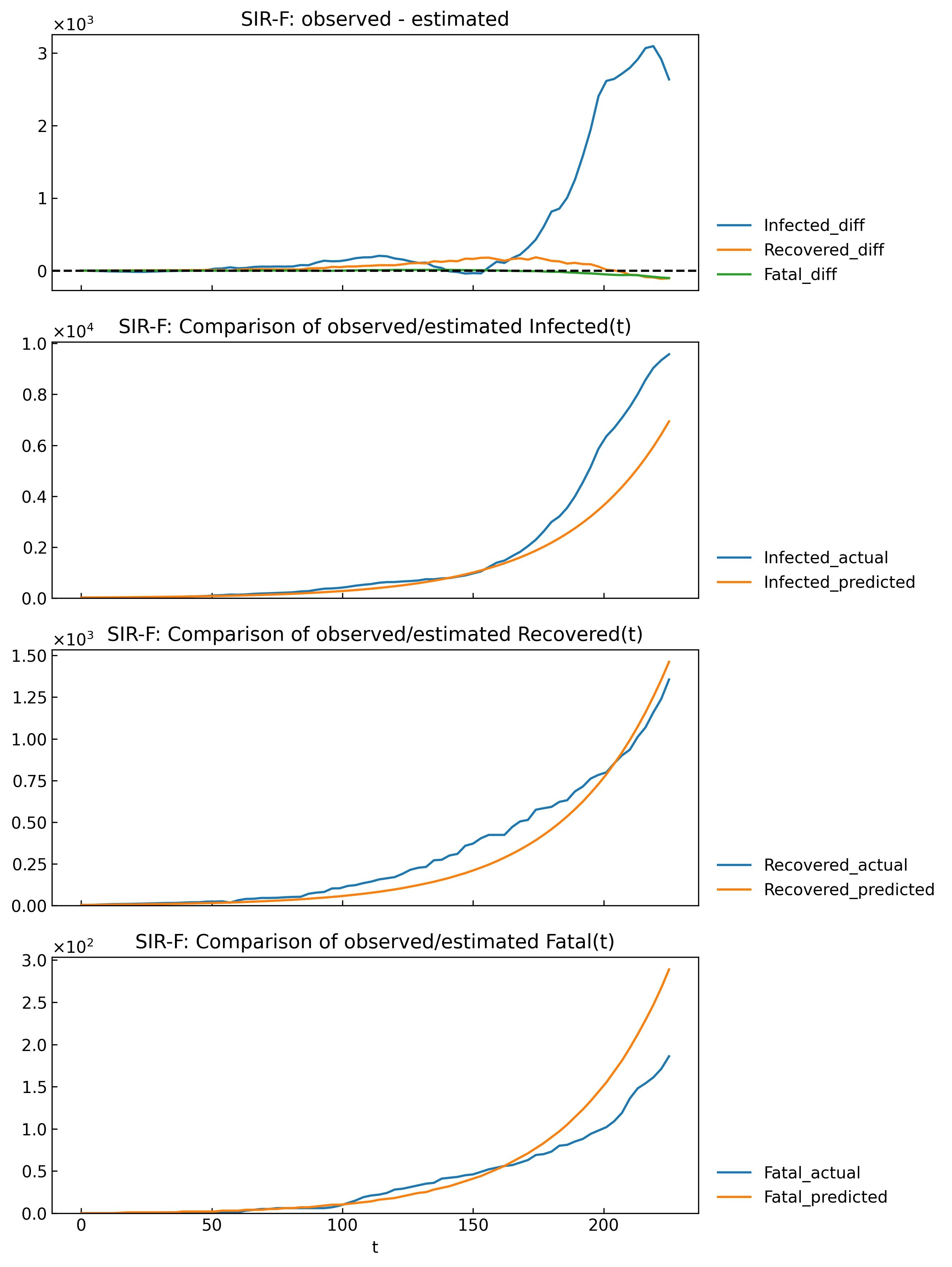
値だけではイメージがつきにくいためグラフ化してみました。まずはRMSLE値が最も大きい0th phaseについて。1番上のグラフは実データと予測値の差、2, 3, 4番目は変数ごとに実データと予測値の両方を表示しています。
snl.estimate_accuracy(phase="0th", filename=None)誤差がある程度生じています。
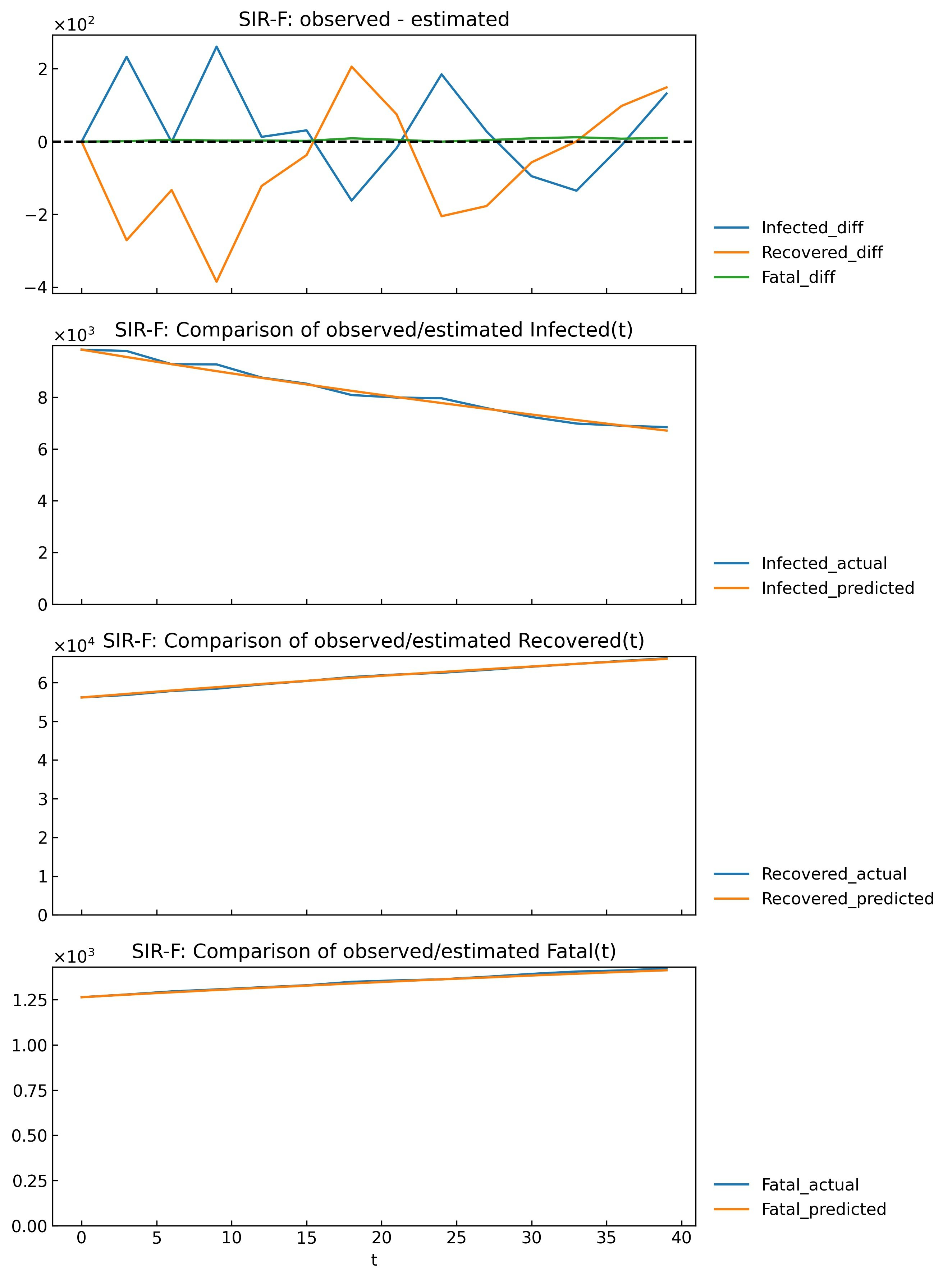
Scenario.separate()などを使って0th phaseを分割したほうが良いようです(別記事を作成予定)。一方でRMSLE scoreが最も小さい6th phaseでは実データと予測値がよく重なっています。
snl.estimate_accuracy(phase="6th", filename=None)8. あとがき
今回は各Phaseのパラメータを推定する方法についてご説明しました。流行開始から半年以上が経過し、パラメータの検証、今後のパラメータの予測、シナリオ分析が重要となる段階に来ていると思います。データサイエンスのテーマとしてCOVID-19の注目度が下がってきているようですが、データが蓄積されてきた分、ダッシュボードの作成に注力していた初期の頃より深い分析ができるようになってきました。
今回もお疲れさまでした!
Guidotti, E., Ardia, D., (2020), “COVID-19 Data Hub”, Journal of Open Source Software 5(51):2376, doi: 10.21105/joss.02376. ↩
Please refer to What’s the Difference Between RMSE and RMSLE? ↩
- 投稿日:2020-09-13T17:08:13+09:00
Djangoを使ったWEBアプリケーションの開発【Django起動編】
前提・環境
MacbookPro2017 13inch macOS Catalina 10.15.6
Python 3.6
Djanngo version 3.1.1 ←インストール手順書き忘れてました。そのうち書くかも。プロジェクトの作成とファイル構成の確認
今回は、djangoフォルダを作成し、その中にプロジェクトを作成していく。
コマンドプロンプトで下記を入力する。$ mkdir django $ cd django「django」フォルダに移動したら、下記を入力するとプロジェクトが作成される。
今回プロジェクト名は「project1」とした。$ django-admin startproject project1実行後、作成されたproject1フォルダ内に移動し、ファイルを確認する。
$ cd project1 $ ls manage.py project1さらに、project1フォルダ内のproject1フォルダに移動し、ファイルを確認する。
$ cd project1 $ ls __init__.py asgi.py settings.py urls.py wsgi.py以上のファイルが作成されていることを確認したら、一つ上のフォルダに戻ってDjangoサーバーの起動し動作を確認する。
$ cd .. $ python3.6 manage.py runserver Watching for file changes with StatReloader Performing system checks... System check identified no issues (0 silenced). You have 18 unapplied migration(s). Your project may not work properly until you apply the migrations for app(s): admin, auth, contenttypes, sessions. Run 'python manage.py migrate' to apply them. September 13, 2020 - 07:31:30 Django version 3.1.1, using settings 'project1.settings' Starting development server at http://127.0.0.1:8000/ Quit the server with CONTROL-C.サーバーが起動できたらブラウザに「http://127.0.0.1:8000/」を入力する。
ロケットの画像と「The install worked successfully! Congratulations!」の文字が表示されていれば成功です。
動作が確認できたら、コマンドプロンプトでControl + Cでサーバーを停止させることができます。
- 投稿日:2020-09-13T16:55:02+09:00
pythonから Amazon Athenaへクエリ実行(名前付きプロファイル使用)
はじめに
windows command lineにて、AWS CLIでAthenaに接続させることがあるが、
どうせなら、
クエリ結果を取得してグラフ描画まで出来ればなと思い、pythonで行うと思ったのだけれども
名前付きプロファイルの部分で行き詰まった個所があったので、整理マニュアル記載の方法(名前付きprofile未使用)
windows command lineでの方法
aws athena start-query-execution --query-string "select * from table_name" --result-configuration "s3://path/to/query/bucket/"AWSドキュメント:
https://docs.aws.amazon.com/cli/latest/reference/athena/start-query-execution.htmlpythonでの方法
athena = boto3.client('athena') # クエリ実行 exec_run = athena.start_query_execution( QueryString="select * from table_name", QueryExecutionContext={'Database': 'database_name'}, ResultConfiguration={'OutputLocation': 's3://path/to/query/bucket/'})AWSドキュメント:
https://boto3.amazonaws.com/v1/documentation/api/latest/reference/services/athena.html今回行いたいこと
名前付きプロファイルの情報を用いて、クエリ実行を行いたい。
名前付きプロファイルの設定方法が
「windows command line」と「python」で異なることに気が付かず無駄に時間を使ってしまった。名前付きプロファイルの設定方法は
windows command line:awsコマンドのパラメータとして設定
python :boto3.sessionクラスを使ってプロファイル情報を設定windows command lineでの方法(名前付きprofile使用)
aws athena start-query-execution --query-string "select * from table_name" --result-configuration "s3://path/to/query/bucket/" --profile "NRP"AWSドキュメント:
https://docs.aws.amazon.com/cli/latest/reference/athena/start-query-execution.html
https://docs.aws.amazon.com/cli/latest/reference/athena/start-query-execution.htmlpythonでの方法(名前付きprofile使用)
session = boto3.Session(profile_name='NRP') athena = session.client('athena') # 処理実行 exec_run = athena.start_query_execution( QueryString="select * from table_name", QueryExecutionContext={'Database': 'database_name'}, ResultConfiguration={'OutputLocation': 's3://path/to/query/bucket/'})AWSドキュメント:
https://boto3.amazonaws.com/v1/documentation/api/latest/reference/services/athena.html
https://boto3.amazonaws.com/v1/documentation/api/latest/guide/session.html
S3の接続についての記事:
boto3 で デフォルトprofile以外を使う最後に
boto3.sessionにたどり着くのが大変だった。エラー文ベースで調べてたのも良くなかった。
マニュアルにもしっかり書いてあったし
たどり着いてしまえば、いろいろとわかってくるもんだなと再認識。ただ、今度は取得結果が全件なかったりと課題はたくさん
以上。
参考情報
各箇所に記載
- 投稿日:2020-09-13T16:43:37+09:00
Python開発環境の整理
Python開発環境について、以前はローカル環境にインストールしたAnacondaからJupyter Notebookを使っていて、最近はAmazon SageMakerからJupyter Notebookを使っているのですが、あらためてJupyter Notebookをはじめとするディストリビューションの開発元やインストール方法などを調査・整理してみました。なお、機能については、いずれにおいても概要レベルの記載としています。
環境
Windows10
対象とするディストリビューション
①Anaconda
②Python(IDLE)
③Jupyter Notebook
④Jupyter Lab①Anaconda
開発元はAnaconda社。
Anacondaはデータサイエンス・機械学習・科学技術計算などを目的とした開発プラットフォームです。下のAnaconda Navigatorのとおり、Anacondaをインストールすれば、Jupyter NotebookやJupyter Labも使えるため、Anacondaで環境構築するのが手っ取り早く楽かもしれません。
インストール手順は様々なサイトに載っているので省略しますが、1.Anacondaダウンロード・インストール・設定などを参考にしていただければと思います。
②Python(IDLE)
開発元はPython Software Foundation(PSF)。Paython公式版。
上記公式サイトからPythonをインストールすると、IDLE(Integrated DeveLopment Environment)というPython用の統合開発環境が使えるようになります。下にあるような、インタラクティブシェルにより対話的にプログラミングを行います。こちらがPython開発環境の最もシンプルな構成かと思います。
インストール手順は様々なサイトに載っているので省略しますが、windows10にpythonをインストールなどを参考にしていただければと思います。
③Jupyter Notebook
開発元はProject Jupyter。
Jupyter Notebookはブラウザ上で動作する開発環境で、ノートブックと呼ばれるファイルにプログラムを記述し、実行結果を確認しながら開発を進めるものです。
インストールは、Pythonインストール後に付属のpipというパッケージ管理ツールを使って、PyPI(Python Package Index)というレポジトリサービスからパッケージを取得することで行います。
具体的には、コマンドプロンプトに
pip install notebookと入力して実行することでインストールできます。Jupyter Notebookの起動は、コマンドプロンプトに
jupyter notebookと入力して実行します。④Jupyter Lab
開発元はJupyter Notebbokと同様で、Project Jupyter。
Jupyter LabはJupyter Notebookの後継としてProject Jupyterにより開発されたもので、Jupyter Notebookよりも多機能かつ操作性が向上された開発環境です。
インストールは、Pythonインストール後に付属のpipというパッケージ管理ツールを使って、PyPI(Python Package Index)というレポジトリサービスからパッケージを取得することで行います。
具体的には、コマンドプロンプトに
pip install jupyterlabと入力して実行することでインストールできます。jupyter Labの起動は、コマンドプロンプトに
jupyter labと入力して実行します。まとめ
まとめとしては、手っ取り早く丸ごとインストールするのであればAnaconda、個別に必要なものだけインストールするのであれば、Pyrhon or (Python + Jupyter notebook) or (Python + Jupyter lab)のいずれかになると思います。
Pythonのディストリビューションについては他にもPyCharmなど色々あると思いますが、今回は上記について説明させていただきました。
参考
- 投稿日:2020-09-13T16:12:32+09:00
遺伝的アルゴリズムを勉強する(1)-基本的なOneMax問題
遺伝的アルゴリズムの勉強
platypus、deapを動かすのに必要な遺伝的アルゴリズムの知識を、日経ソフトウェアの2019年3月号の特集記事をもとに勉強しています。
遺伝的アルゴリズムとは
遺伝アルゴリズムとは「選択(淘汰)」「交叉」「突然変異」と言った遺伝的進化を模したアルゴリズムです。
また遺伝的アルゴリズムは、乱数を多く利用するため、乱択アルゴリズムでもある。解ける問題は次の2つを満たすこと。
・入力パラメーターに対する数値化(評価)ができる問題
・数値が最大化、最小化となる入力を探す問題遺伝子:形質を表現するための構成要素
染色体:複数の遺伝子が集まったもの
個体:一つの染色体によって表現される形質(個体=染色体と言う場合もある。)個体が環境にどれだけ適しているかの評価値が「適応度」
この適応度を高めるために3つの操作を行う。
選択(淘汰):適応度が高いものを残す(適応度が低いものを淘汰する)
交叉:2つの親から遺伝子を引き継いだ子を生成する
突然変異:個体の遺伝子を一定確率で変化させる。交叉が遺伝的アルゴリズムの特徴である。
これらの操作が一回すべて終了すると世代が1つ進む。
OneMax問題とは
OneMax問題とは、[1,0,0,0,1,0,1,1]のように1と0からなるリストの和が最大になるようなリストを探し出す問題です。
遺伝子:0または1
染色体:[1,0,0,0,1,0,1,1]
適応度:リストの和(この場合は、[1,0,0,0,1,0,1,1]=4)プログラムは、次の要素でできている。
パラメータ設定
関数(適応度を計算、集団を適応度順にソート、選択:適応度の高い個体を残す、交叉、突然変異)
メイン処理(初期集団を生成、選択、交叉、突然変異)パラメーターの設定
パラメーターとして与えるのは、5つ。遺伝子の長さ、個体数、世代数、突然変異の確率、選択割合
import random import copy # パラメータ LIST_SIZE = 10 # 0/1のリスト長(遺伝子長) POPULATION_SIZE = 10 # 集団の個体数 GENERATION = 25 # 世代数 MUTATE = 0.1 # 突然変異の確率 SELECT_RATE = 0.5 # 選択割合この例では、
GENERATION = 25なので、25世代で終了します。一般的には、適応度が変化しなくなったら終了するなどにします。LIST_SIZEを大きくした場合は、POPULATION_SIZEGENERATIONを大きくしないと最適解に到達しない可能性がある。突然変異の確率、選択割合などのチューニングが重要だが、今回は適当に決めている。関数
# 適応度を計算する def calc_fitness(individual):#individualは染色体 return sum(individual) # 染色体リスト要素の合計 # 集団を適応度順にソートする def sort_fitness(population): # 適応度と個体をタプルにしてリストへ格納する fp = [] for individual in population: fitness = calc_fitness(individual)#適応度を計算してfitnessに入れる。 fp.append((fitness, individual)) #fp に適応度と染色体を順に入れる fp.sort(reverse=True) # 適用度でソート(降順) sorted_population = [] # ソートした個体を格納する集団 for fitness, individual in fp: # 個体を取り出し、集団に格納する sorted_population.append(individual) return sorted_population #この中に、適用度が高い順にソートされた適用度と染色体が保存されている。 # 選択 # 適応度の高い個体を残す def selection(population): sorted_population = sort_fitness(population)#sort_populationにソートされた適用度と染色体が入っている。 #残す個体数。パラメーター設定で0.5なので個体数10×0.5で5個残る n = int(POPULATION_SIZE * SELECT_RATE) return sorted_population[0:n]#最初から5個が残る。0,1,2,3,4 #交叉 #まず、ind1をコピーして新しい個体indを作る。その後、ランダムに決めたr1~r2の範囲をind2の遺伝子に置き換える def crossover(ind1, ind2): r1 = random.randint(0, LIST_SIZE - 1)#r1の位置をランダムに決める r2 = random.randint(r1 + 1, LIST_SIZE)#r1以降でランダムにr2の位置を決める。 ind = copy.deepcopy(ind1)#ind1のコピーを作る ind[r1:r2] = ind2[r1:r2]#染色体ind2からランダムにきめたr1からr2の範囲を取り出し、ind1のコピーへ置き換える、。 return ind #突然変異:設定した突然変異確率に従って突然変異させる def mutation(ind1): ind2 = copy.deepcopy(ind1)#コピーでind2を作る。 for i in range(LIST_SIZE): if random.random() < MUTATE:#ランダムに数値を作り、突然変異率以下なら突然変異がおきる ind2[i] = random.randint(0, 1)#突然変異のおきたところに0か1を入れる return ind2メイン処理(初期集団を生成、選択、交叉、突然変異)
#初期集団を生成 population = [] # 集団 for i in range(POPULATION_SIZE): individual = [] # 染色体 for j in range(LIST_SIZE): # 0/1を乱数で追加 individual.append(random.randint(0, 1)) population.append(individual) for generation in range(GENERATION): print(str(generation + 1) + u"世代") #選択 population = selection(population) # 交叉と突然変異を繰り返す。交叉と突然変異で増やす個体数 n = POPULATION_SIZE - len(population) for i in range(n): # 集団から2個体をランダムに選び、 # 交叉した個体を生成する r1 = random.randint(0, len(population) - 1) r2 = random.randint(0, len(population) - 1) #交叉 individual = \ crossover(population[r1], population[r2]) #突然変異 individual = mutation(individual) #集団に追加する population.append(individual) for individual in population: print(individual)これでOne-Max問題が解けるプログラムです。
結果です。
1世代
[1, 1, 1, 0, 1, 1, 0, 0, 1, 1]
[1, 0, 1, 1, 0, 0, 1, 1, 1, 0]
[1, 0, 1, 0, 1, 1, 0, 1, 1, 0]
[1, 0, 1, 1, 1, 0, 0, 0, 0, 1]
[0, 0, 1, 1, 0, 1, 1, 0, 1, 0]
[1, 1, 1, 0, 1, 1, 0, 0, 1, 0]
[0, 1, 1, 0, 1, 1, 0, 0, 1, 0]
[0, 1, 1, 0, 1, 1, 1, 0, 1, 0]
[1, 0, 1, 1, 0, 1, 0, 0, 1, 0]
[1, 0, 1, 1, 1, 1, 0, 1, 1, 0]第14世代で、最適解が見つかりました。
14世代
[1, 1, 1, 1, 1, 1, 1, 1, 1, 1]
[1, 1, 1, 1, 1, 1, 0, 1, 1, 1]
[1, 1, 1, 1, 1, 1, 0, 1, 1, 1]
[1, 1, 1, 1, 1, 1, 0, 1, 1, 1]
[1, 1, 1, 1, 1, 1, 0, 1, 1, 1]
[1, 1, 1, 1, 1, 1, 0, 1, 1, 1]
[1, 1, 1, 1, 1, 0, 0, 1, 1, 1]
[1, 1, 1, 1, 1, 1, 1, 1, 1, 1]
[1, 1, 1, 1, 1, 0, 0, 1, 1, 1]
[1, 1, 1, 1, 1, 1, 0, 1, 1, 1]第20世代で、6つまで最適解になりました。
20世代
[1, 1, 1, 1, 1, 1, 1, 1, 1, 1]
[1, 1, 1, 1, 1, 1, 1, 1, 1, 1]
[1, 1, 1, 1, 1, 1, 1, 1, 1, 1]
[1, 1, 1, 1, 1, 1, 1, 1, 1, 1]
[1, 1, 1, 1, 1, 1, 1, 1, 1, 1]
[1, 1, 1, 1, 1, 1, 1, 1, 1, 1]
[1, 1, 1, 1, 1, 1, 1, 0, 1, 1]
[1, 1, 1, 1, 1, 1, 1, 1, 1, 1]
[1, 1, 1, 1, 1, 1, 1, 1, 1, 0]
[1, 1, 1, 0, 1, 1, 1, 1, 1, 1]第25世代ですべて最適解になりました。
25世代
[1, 1, 1, 1, 1, 1, 1, 1, 1, 1]
[1, 1, 1, 1, 1, 1, 1, 1, 1, 1]
[1, 1, 1, 1, 1, 1, 1, 1, 1, 1]
[1, 1, 1, 1, 1, 1, 1, 1, 1, 1]
[1, 1, 1, 1, 1, 1, 1, 1, 1, 1]
[1, 1, 1, 1, 1, 1, 1, 1, 1, 1]
[1, 1, 1, 1, 1, 1, 1, 1, 1, 1]
[1, 1, 1, 1, 1, 1, 1, 1, 1, 1]
[1, 1, 1, 1, 1, 1, 1, 1, 1, 1]
[1, 1, 1, 1, 1, 1, 1, 1, 1, 1]これが基本のOneMax問題の解き方です。
次回は、巡回セールスマン問題を解こうと思います。
- 投稿日:2020-09-13T15:45:18+09:00
2. Pythonで綴る多変量解析 8-3. k近傍法 [交差検証]
- k近傍法における2種類の重み関数uniformとdistanceの違いについて、前回は視覚的にとらえました。
- 各点を距離の逆数で重みづけするdistanceは過学習を引き起こしやすく、全点を等しく重みづけするuniformの方がモデルとしての汎可性に優れていました。
- 改めて交差検証(クロスバリデーション)を行ない、具体的な数値として確認します。
import numpy as np import pandas as pd # sklearn系のライブラリ類 from sklearn import datasets # データセット from sklearn.model_selection import train_test_split # データ分割 from sklearn.neighbors import KNeighborsClassifier # 分類モデル from sklearn.neighbors import KNeighborsRegressor # 回帰モデル # matplotlib系のライブラリ類 import matplotlib.pyplot as plt from matplotlib.colors import ListedColormap !pip install japanize-matplotlib # 日本語表示対応モジュール import japanize_matplotlib回帰モデル ―ボストン住宅価格―
⑴ データ作成
- sklearnに付属のデータセット「boston(ボストン住宅価格)」から、目的変数「住宅価格」と、説明変数としては「一戸当たり平均室数」のみを使うこととします。
- sklearnのデータ分割ユーティリティを利用して、データを訓練用とテスト用に分割します。
# データセットを取得 boston = datasets.load_boston() # 説明変数・目的変数を抽出 X = boston.data[:, 5].reshape(len(boston.data), 1) y = (boston.target).reshape(len(boston.target), 1) # 訓練・テストにデータ分割 X_train, X_test, y_train, y_test = train_test_split(X, y, random_state = 0)⑵ 交差検証
- 重み関数uniform、distance間で訓練データとテストデータの正解率を比較します。
# kパラメータ n_neighbors = 14 # 正解率を格納する変数 score = [] for w in ['uniform', 'distance']: # モデル生成 model = KNeighborsRegressor(n_neighbors, weights=w) model = model.fit(X_train, y_train) # 訓練データの正解率 r_train = model.score(X_train, y_train) score.append(r_train) # テストデータの正解率 r_test = model.score(X_test, y_test) score.append(r_test) # データフレームに表現 score = np.array(score) pd.DataFrame(score.reshape(2,2), columns = ['train', 'test'], index = ['uniform', 'distance'])
- まずuniformですが、訓練データでは61.0%、テストデータでは54.0%となっています。
- これに対してdistanceは、訓練データでは96.2%と非常に高く、テストデータになると著しく低下して40%を下回っています。
⑶ 可視化
- uniform、distance それぞれのモデルを生成し、同じテストデータを渡して予測を行います。
# kパラメータ n_neighbors = 14 # インスタンス生成 model_u = KNeighborsRegressor(n_neighbors, weights='uniform') model_d = KNeighborsRegressor(n_neighbors, weights='distance') # モデル生成 model_u = model_u.fit(X_train, y_train) model_d = model_d.fit(X_train, y_train) # 予測 y_u = model_u.predict(X_test) y_d = model_d.predict(X_test)
- これら2つの予測値$\hat{y}$と、実測値y_testを散布図に表します。
plt.figure(figsize=(14,6)) # 散布図 plt.scatter(X_test, y_u, color='slateblue', lw=1, label='予測値(uniform)') plt.scatter(X_test, y_d, color='tomato', lw=1, label='予測値(distance)') plt.scatter(X_test, y_test, color='lightgrey', label='実測値(test)') plt.legend(fontsize=15) plt.xlim(3, 9.5) plt.show()
- 灰色●が実測値y_test、青●がuniform、赤●がdistanceによる予測値$\hat{y}$です。
- uniformの予測値に対してdistanceの方はばらついており、大きく外れた値も目につきます。
分類モデル ―アイリスのがく片の長さ・幅―
- sklearnに付属のデータセット「アイリス」を用いますが、問題を単純にしたいので、目的変数である「種類」を2つだけ(versicolour=1, virginica=2)に絞って2値分類とします。
- この2種類は、所与の2変数では個体が入り混じって境界を分けにくい。それを承知の上で uniform と distance にどのような違いが出るかを観察します。
⑴ データ作成
# データセットを取得 iris = datasets.load_iris() # 説明変数・目的変数のみ抽出 X = iris.data[:, :2] y = iris.target y = y.reshape(-1, 1) # 形状変換 # 2値のみ抽出後、各変数を設定 data = np.hstack([X, y]) # X, yを結合 data = data[data[:, 2] != 0] # 2値のみ抽出 X = data[:, :2] y = data[:, -1] # データ分割 X_train, X_test, y_train, y_test = train_test_split(X, y, stratify = y, random_state = 0)⑵ 交差検証
- kパラメータ(kの個数)は前回を踏襲して15個とします。
# kパラメータ n_neighbors = 15 # 正解率を格納する変数 score = [] for i, w in enumerate(['uniform', 'distance']): # モデル生成 model = KNeighborsClassifier(n_neighbors, weights=w) model = model.fit(X_train, y_train) # 訓練データ r_train = model.score(X_train, y_train) score.append(r_train) # テストデータ r_test = model.score(X_test, y_test) score.append(r_test) # データフレームに表現 score = np.array(score) pd.DataFrame(score.reshape(2,2), columns = ['train', 'test'], index = ['uniform', 'distance'])
- uniformは、訓練データでは74.7%、テストデータでも60.0%となっています。
- これに対して distance は、訓練データでは92.0%という高率で、それがテストデータでは4割減の52.0%となっています。
⑶ 可視化
- 境界を予測させるためにモデルに渡すメッシュ間隔は、やはり前回と同じく0.02とします。
# kパラメータ n_neighbors = 15 # メッシュ間隔 h = 0.02 # マッピングのためのカラーマップを生成 cmap_surface = ListedColormap(['mistyrose', 'lightcyan']) cmap_dot = ListedColormap(['tomato', 'slateblue'])
- 重み関数をそれぞれuniform、distanceとする2つのモデルに、同じテストデータを渡して境界を生成し、そこへ実測値であるテストデータをプロットしてみます。
plt.figure(figsize=(18,6)) for j, w in enumerate(['uniform', 'distance']): # モデルを生成 model = KNeighborsClassifier(n_neighbors, weights = w) model = model.fit(X_train, y_train) # テストデータをセット X, y = X_test, y_test # x,y軸の最小値・最大値を取得 x_min, x_max = X[:, 0].min() - 1, X[:, 0].max() + 1 y_min, y_max = X[:, 1].min() - 1, X[:, 1].max() + 1 # 指定のメッシュ間隔で格子列を生成 xx, yy = np.meshgrid(np.arange(x_min, x_max, h), np.arange(y_min, y_max, h)) # 格子列をモデルに渡して予測 z = np.c_[xx.ravel(), yy.ravel()] # 一次元に平坦化してから結合 Z = model.predict(z) # 予測 Z = Z.reshape(xx.shape) # 形状変換 # 描画 plt.subplot(1, 2, j + 1) plt.pcolormesh(xx, yy, Z, cmap=cmap_surface) # カラープロット plt.scatter(X[:, 0], X[:, 1], c=y, cmap=cmap_dot, s=30) plt.xlim(xx.min(), xx.max()) plt.ylim(yy.min(), yy.max()) plt.xlabel('がく片の長さ', fontsize=12) plt.ylabel('がく片の幅', fontsize=12) plt.title("'%s'" % (w), fontsize=18) plt.show()
- distanceモデルは、訓練データにおける過学習のために境界は複雑となり、随所に飛び地も見られますが、それらはテストデータには適合せず意味をなしません。
- 位置関係は恒常的(いつも一定)であるとして、その間の距離は偶発的(たまたまそうなった)とみなす、というのが uniform の考え方でしょう。
- そうした偶発性をも情報として取り込み、データの特徴を色濃く反映する distance は、汎可性を求められるモデル生成には不向きといえそうです。
- 投稿日:2020-09-13T15:32:22+09:00
にしやま、Kaggleはじめるってよ。前編。タイタニックデータセットのコンペをお試し。
学生時代、気が狂ったようにデータサイエンス系の本を読み漁り、
『俺はコンピューターサイエンス界の王様になる!』くらいに意気込んでたのに、いざ社会人になってみると、一ミリもコンピューターサイエンス関連の研究・開発をしてない・・・
これが幸せな選択だったのか、俺にはわからないけど、仕事で出来ないのなら、
趣味でやるしか無いだろう、という発想にようやく至れた。ということで、Kaggleで俺のやりたいデータサイエンスを速攻とらいしてみよう。
事始めは下の記事見りゃなんとかなる
とりあえず始めたいって人は、下の記事をぱぱっと見ながらルールを覚えれば良いと思う。
- ぱぱっと始めたい人向け:Kaggle事始め
- しっかり参加したい人向け:Kaggleに登録したら次にやること ~ これだけやれば十分闘える!Titanicの先へ行く入門 10 Kernel ~
自分は甘い蜜を一刻も早く吸いたいので、はじめ方やルールに関する説明はしない。笑
みんな大好きタイタニックデータセットからスタート。
そうは言ってもいきなりCompetition参加すんのはハードル高いし、面倒だよなぁー。
まずは仕組みを理解したいなーっと思っていたら、あるじゃないですか、見慣れたタイタニックデータセット、、、笑Kaggle初心者が、まずお試しでできるように解説されてるコンペティションで、
期限なし、賞金なしのコンペティションみたいですね。僕みたいな人間ならば、事始めとして、ここからスタートしましょう。
(?に参照しといた記事もどっちもここから開始してますね)今回、この記事では、用意されてる記事とかテンプレートとか使わず、
1から仕組みを理解して、進めてみましょうか!トップダウン進めよう。まずNotebook作成。
ごにょごにょルールとか前提条件とか読むのめんどくさいから、まずコード書きます。笑
(本当はこういうの、ルールと前提条件を正確に理解することが何より重要だったりするけど、自分はまず触りたいねん。)ここのページの Notebooks ⇒ Your Work からこのコンペの Notebook が作成できるっぽい。
とりあえず適当にOKとかCreateとか押しておけばOK。違ったら後で直す。笑こんな感じに画面が現れる。いや、すごすぎ。ブラウザでプログラミングってすごい。
後は書いてRunするだけでしょ、これ多分。凄いなぁー。
今回はお試しだから適当にやるか。女性が生き残る作戦で予測だ!
今回は事始め記事なので、予測モデルをごりくそに組むことはしない。とりあえず、予測ができるモデルをクソ適当に組んで、
Submitすることを目標にしてる。(Submitの方法をそもそも知りたいので!笑)なんで今回は、女性は全員助かって、男性は助からない予測モデルを作ります!!!
実際にこんな感じで適当にコード自体は書いてみました。絶対アホみたいな書き方してるので、
賢い書き方があれば教えてください。配列操作とか、データ操作とかまだ苦手です。。。一応、メイン処理な感じのコードはいかに書いときます。(別に処理してないんだけどね、、、笑)
# 女性は必ず助かる部分のデータ操作部分 sub = pd.DataFrame(pd.read_csv('../input/titanic/test.csv', usecols=['PassengerId', 'Sex'])) sub["Gender"] = sub["Sex"].map({"female": 0, "male": 1}).astype(int) sub["Survived"] = 0 for index, gender in enumerate(sub["Gender"]): if gender == 0: sub["Survived"][index] = 1 else: sub["Survived"][index] = 0 sub = sub.drop(["Sex", "Gender"], axis=1) sub.head()詳しい前処理とかは、さっき乗っけたノートを参照してね。これで下コードみたいにサブミットファイルを作ろう。
sub.to_csv('submission.csv', index=False)はい、これで修了。予測結果ファイルは出来たね。
最後の最後、サブミットして予測精度を確認しよう!!!
最後は流れるように処理を淡々とこなすだけ、画面右上のSave Versionをクリックしましょう。
そうすると以下のような画面が出るので、バージョン名を変えたい場合は変えて、
Save & Run ALL(Commit)をクリックしよう。(まだ、サブミットできるバージョンじゃないよ!という場合には単純に Quick SaveでOK。)そうすると自分のページが以下のように更新されてます。
今回の場合、76.5%の精度で予測できましたー!という結果らしいです。きっと女性を優先的に助けたんだろうなという仮設を若干いだきましたね、
データサイエンスはここから仮設の幅を広げて精度をあげるのが面白いんです!!!最後に結果ファイルをサブミットするために右のOutputタブを選択して、
サブミットしましょう!!終わりです!!おわりの言葉と次回予告
今回は、Kaggleまじで初めたばっかの初心者が適当に、Kaggleのサブミッションをして、ランキングに参加した記事でした!
やり方自体はクソ簡単、ブラウザでプログラミングできるから余計な環境問題とかは考えないでよし、
純粋にデータサイエンスのみで勝負!ってのがKaggleの醍醐味って事がよくわかりました。今回は事始めなので、とりあえず女性は生き残る戦術で予測モデルとは呼べないモデルを生成して、
予測してみたら76.5%の予測精度でした、意外と高くてびっくり。。。多分まだまだ機能的な部分は使いこなせては無いんだろうけど、これから徐々にできるようにしていきたい所存。
次回は若干データサイエンスに入り込み、重回帰予測でやっていこうと思う!さらばじゃ!!!
- 投稿日:2020-09-13T15:18:22+09:00
Numpyの配列情報取得
ndarrayの情報を取得する。
import numpy as np【
shape】
- 各次元の要素数を取得する。
X = np.array([[1,2,3], [4,5,6]]) X.shape # (2, 3)【
dtype】
- データ型を取得する。
X = np.array([[1,2,3], [4,5,6]]) X.dtype # dtype('int64')【
ndim】
- 次元数を取得する。
X = np.array([[1,2,3], [4,5,6]]) X.ndim # 2【
size】
- 要素数を取得する。
X = np.array([[1,2,3], [4,5,6]]) X.size # 6
- 投稿日:2020-09-13T15:15:46+09:00
Natural Language : BERT Part2 - Unsupervised pretraining ALBERT
目標
Microsoft Cognitive Toolkit (CNTK) を用いた BERT の続きです。
Part2 では、Part1 で準備した日本語 Wikipedia を用いて BERT の事前学習を行います。
NVIDIA GPU CUDA がインストールされていることを前提としています。導入
Natural Language : BERT Part1 - Japanese Wikipedia Corpus では、日本語 Wikipedia を用いて事前学習コーパスを準備しました。
Part2 では、教師なし事前学習モデルを作成して訓練します。
BERT
Bidirectional Encoder Representations from Transformers (BERT) [1] は Transformer [2] の Encoder 部分だけを使用します。Transformer については Natural Language : Machine Translation Part2 - Neural Machine Translation Transformer で紹介しています。
また今回は BERT を軽量化した ALBERT [3] のベースモデルを実装するとともに、Pre-Layer Normalization Transformer [4] で構成しました。層構造の詳細を下図に示します。
BERT の Multi-Head Attention では Self-Attention を使用しており、これが双方向学習を可能にしています。
訓練における諸設定
各パラメータの初期値は分散 0.02 の正規分布に設定しました。
損失関数は、Masked LM におけるマスクされた単語の予測には Cross Entropy Error、Sentence Prediction における識別にはBinary Cross Entropy を使用します。
最適化アルゴリズムには Adam [5] を採用しました。Adam のハイパーパラメータ $β_1$ は 0.9、$β_2$ は CNTK のデフォルト値に設定しました。
学習率は、Cyclical Learning Rate (CLR) [6] を使って、最大学習率は 1e-4、ベース学習率は 1e-8、ステップサイズはエポック数の 10倍、方策は triangular2 に設定しました。
モデルの訓練はミニバッチ学習によって 3,000,000 Iteration を実行しました。
実装
実行環境
ハードウェア
・CPU Intel(R) Core(TM) i7-5820K 3.30GHz
・GPU NVIDIA Quadro RTX 6000 24GBソフトウェア
・Windows 10 Pro 1909
・CUDA 10.0
・cuDNN 7.6
・Python 3.6.6
・cntk-gpu 2.7
・cntkx 0.1.33
・MeCab 0.996
・numpy 1.17.3
・pandas 0.25.0実行するプログラム
訓練用のプログラムは GitHub で公開しています。
bert_pretraining.py解説
今回の実装の要となる内容について補足します。
Masked LM and Sentence Prediction
BERT の事前学習では下図のように、入力文は特別な単語 [CLS] で始まり、2つの文で構成されます。各文の末尾には特別な単語 [SEP] が挿入されます。そして、Masked LM と Sentence Prediction と呼ばれる 2種類の学習を行います。
Masked LM
Masked LM では、入力文の 15% を特別な単語 [MASK] で置き換えたものを入力文として、元の入力文の同じ位置の単語を正解データとします。
マスクされた単語の予測には以下の処理を用いて正解単語を予測します。このときパラメータの更新にはマスクされた位置のみの勾配を使用します。
bert_masked_lmdef bert_masked_lm(encode): """ Masked Language Model """ h = Dense(num_hidden, activation=Cx.gelu_fast, init=C.normal(0.02))(encode) h = LayerNormalization()(h) return Dense(num_word, activation=None, init=C.normal(0.02))(h)しかしながら、[MASK] は BERT の事前学習においてのみ使われる特別な単語であるため、ファインチューニング時に言語モデルの不自然さを招く一因になります。そこで、BERT では以下の戦略でその不自然さを軽減します。
・80% の確率で [MASK] に置き換える。
・10% の確率でランダムな単語に置き換える。
・10% の確率で置き換えずにそのままにしておく。これは実際の単語表現に近づける意味を持ちます。Next Sentence Prediction
Next Sentence Prediction では、入力文に含まれる 2つの文が繋がっているかどうかの 2値分類問題を解くことで文脈理解の獲得を目指します。
この分類には入力文の [CLS] の位置にある隠れ層(pooler)を特徴量として取り出して、全結合とシグモイド関数を適用します。
bert_sentence_predictiondef bert_sentence_prediction(pooler): """ Sentence Prediction """ h = C.sequence.unpack(pooler, padding_value=0, no_mask_output=True)[0, :] return Dense(1, activation=C.sigmoid, init=C.normal(0.02))(h)訓練データ中の 50% は連続した 2つ文として、残る 50% はランダムに文を繋げて不連続な負例とします。
A Lite BERT
A Lite BERT (ALBERT) は BERT の問題点であるモデルの軽量化と文脈理解の改良をしています。
Factorized embedding parameterization
因子分解により埋め込み層のパラメータ数を削減します。
単語数を $V$、隠れ層の次元を $H$、より低次元の埋め込み次元を $E$ とすると、これによりパラメータ数を $V \times H$ から、$V \times E + E \times H$ に削減することができます。
実際に単語数を $E=32,000$、隠れ層の次元を $H=768$、より低次元の埋め込み次元を $E=128$ とすると、
V \times H = 24,576,000 \\ V \times E + E \times H = 4,096,000 + 98,304 = 4,194,304となり、埋め込み層においてパラメータ数を約 83% 削減できることが分かります。
Cross-layer parameter sharing
ALBERT では、Transformer Encoder の Self-Attention の各ヘッドと Position-wise Feedfoward Network のパラメータを 12層すべてにおいて共有します。
これによりパラメータ数を大幅に削減することができます。
Sentence-Order Prediction
Next Sentence Prediction は文脈を理解するための問題としては簡単な問題であるため、RoBERTa [7] などでその有用性が疑問視されています。
そこで、ALBERT では Next Sentence Prediction の代わりに Sentence-Order Prediction で文脈理解の訓練を行います。
実装は非常に簡単で、ランダムに文を繋げていた負例としていたものを、2つの文を入れ替えたものを負例として準備するだけです。
GELU
Gaussian Error Linear Units (GELU) [8] は Dropout [9] と Zoneout [10]、そして ReLU を融合した活性化関数として提案されており、微分可能で滑らかな関数かつ入力を確率的に正則化する効果が期待できます。GELU は下図のようになります。
GELU は以下の式で表されます。
GELU(x) = x \Phi(x)ここで、$\Phi$ は正規分布の累積確率密度関数を表しており、入力 $x$ が Batch Normalization や Layer Normalization により平均 0、分散 1 に近づいていることを想定して、標準正規分布の累積確率密度を使用します。
\Phi(x) = \frac{1}{2} \left( 1 + erf \left( \frac{x - \mu}{\sqrt{\sigma^2}} \right) \right) \\ erf(x) = \frac{2}{\sqrt{\pi}} \int^x_0 e^{-u^2} duここで、$erf$ は誤差関数を表しています。実装では上の式を近似した以下の式を使用します。
GELU(x) \approx 0.5x \left( 1 + \tanh \left[ \sqrt{\frac{2}{\pi}}(x + 0.044715x^3) \right] \right)しかし上の近似式では計算に時間がかかるので、今回の実装では上の式をさらに近似した以下の式を使用しました。
GELU(x) \approx x\sigma(1.702x)ここで、$\sigma$ はシグモイド関数を表しています。
結果
Training loss
訓練時の損失関数のログを可視化したものが下図です。横軸は繰り返し回数、縦軸は損失関数の値を表しています。
Masked LM の予測
訓練した ALBERT に文章の穴埋め問題を解かせてみました。ここで、answer は元の文章、masked は元に文章の一部を [MASK] に置き換えた文章、そして albert が [MASK] の位置にある単語を予測したものです。
answer : 人類は知性を正しく用いて進化しなければならない。 masked : 人類は[MASK]を正しく用いて進化しなければならない。 albert : 人類は生物を正しく用いて進化しなければならない。Self-Attention の可視化
下図は Encoder の 11, 12層目の Self-Attention の各ヘッドにおける Attention マップを可視化したものを示しており、カラーマップは hot で表示しています。
Encoder 11
Encoder 12
BERT fine-tuning
BERT の本来の動機は事前学習モデルの転移学習です。そこで、今回事前学習したモデルを用いて Natural Language : Doc2Vec Part1 - livedoor NEWS Corpus で使用した livedoor NEWS Corpus の文書分類タスクで転移学習してみました。
テキストデータの前処理と形態素解析 MeCab と NEologd 辞書を用いて名詞・動詞・形容詞のみを抽出し、ストップワードの除去を実行してから、SentencePiece モデルで単語を id に変換しました。
今回は Pooler の出力に 9分類の全結合に加えて 5 Epoch の訓練を行いました。
Natural Language : Doc2Vec Part2 - Document Classification と同様に検証データで性能評価してみると以下のような結果になりました。Doc2Vec の 10 Epoch で 90% と比較すると低い性能になりました。
Accuracy 75.56%時間の都合上、ALBERT の事前学習が実質 1 Epoch しか訓練ができなかったこと、問題に対してモデルが複雑すぎること、などが性能低下の要因だと考えられます。
参考
Natural Language : Doc2Vec Part1 - livedoor NEWS Corpus
Natural Language : Doc2Vec Part2 - Document Classification
Natural Language : BERT Part1 - Japanese Wikipedia Corpus
- Jacob Devlin, Ming-Wei Chang, Kenton Lee, Kristina Toutanova. "BERT: Pre-training of Deep Bidirectional Transformers for Language Understanding", arXiv preprint arXiv:1810.04805, 2018.
- Ashish Vaswani, et. al. "Attention Is All You Need", Advances in neural information processing systems. 2017. p. 5998-6008.
- Zhenzhong Lan, Mingda Chen, Sebastian Goodman, Kevin Gimpel, Piyush Sharma, and Radu Soricut. "ALBERT: A Lite BERT for self-supervised learning of language representations", arXiv preprint arXiv:1909.11942 (2019).
- Ruibin Xiong, Yunchang Yang, Di He, Kai Zheng, Shuxin Zheng, Chen Xing, Huishuai Zhang, Yanyan Lan, Liwei Wang, and Tie-Yan Liu . "On Layer Normalization in the Transformer Architecture", arXiv preprint arXiv:2002.04745 (2020).
- Diederik P. Kingma and Jimmy Lei Ba. "Adam: A method for stochastic optimization", arXiv preprint arXiv:1412.6980 (2014).
- Leslie N. Smith. "Cyclical Learning Rates for Training Neural Networks", 2017 IEEE Winter Conference on Applications of Computer Vision. 2017, p. 464-472.
- Yinhan Liu, Myle Ott, Naman Goyal, Jingfei Du, Mandar Joshi, Danqi Chen, Omer Levy, Mike Lewis, Luke Zettlemoyer, and Veselin Stoyanov. "RoBERTa: A Robustly Optimized BERT Pretraining Approach." arXiv preprint arXiv:1907.11692 (2019).
- Dan Hendrycks and Kevin Gimpel. "Gaussian Error Linear Units(GELUs)", arXiv preprint arXiv:1606.08415, (2016).
- Nitish Srivastava, Geoffrey Hinton, Alex Krizhevshky, Ilya Sutskever, and Ruslan Salakhutdinov. "Dropout: A Simple Way to Prevent Neural Networks from Overfitting", The Journal of Machine Learning Research 15.1 (2014) p. 1929-1958.
- David Krueger, Tegan Maharaj, János Kramár, Mohammad Pezeshki, Nicolas Ballas, Nan Rosemary Ke, Anirudh Goyal, Yoshua Bengio, Aaron Courville, and Christopher Pal. "Zoneout: Regularizing RNNs by Randomly Preserving Hidden Activations", arXiv preprint arXiv:1606.01305 (2016).
- 投稿日:2020-09-13T15:10:16+09:00
Numpyの配列要素取得
ndarrayの要素は、Pythonでのリスト要素の取得と同じ方法で取得できる。
ただし、多次元配列の場合は指定方法が変わるので注意する。
import numpy as npX = [[1,2,3],[4,5,6]] print(X[1][2]) # 6 X_numpy = np.array(X) print(X_numpy[1,2]) # 6又、通常の取得方法とは別に次の方法でも取得できる。
1. インデックスの配列で取得する。
import numpy as npX = np.array([[1,2,3],[4,5,6]]) print(X[[0,1,-1]]) # [[1 2 3], # [4 5 6], # [4 5 6]] print(X[[0,1,-1], 0]) # [1, 4, 4] print(X[[0,1,-1], ::-1]) # [[3 2 1], # [6 5 4], # [6 5 4]] print(X[[0,1,-1],[1,0,-1]]) # [2, 4, 6] """ ※この指定は、次と同じ結果になっている。 np.array([X[0,1], X[1,0], X[-1,-1]]) """2. 条件式で取得する。
import numpy as npX = np.array([[1,2,3],[4,5,6]]) print(X > 4) # [[False, False, False], # [False, True, True]] print(np.where(X > 4)) # (array([1, 1]), array([1, 2])) print(X[X > 4]) # [5, 6] print(X[np.where(X > 4)]) # [5, 6]
- 投稿日:2020-09-13T14:59:59+09:00
AttributeError: 'NoneType' object has no attribute 'loader'
始めに
今回はタイトルにあるようなエラーが起きたときの対処法を僕の実体験を元に書いていきます。
エラー内容
Error processing line 3 of /home/cjones/.local/lib/python3.6/site-packages/googleapis_common_protos-1.5.8-py3.6-nspkg.pth: Traceback (most recent call last): File "/usr/lib/python3.6/site.py", line 174, in addpackage exec(line) File "<string>", line 1, in <module> File "<frozen importlib._bootstrap>", line 568, in module_from_spec AttributeError: 'NoneType' object has no attribute 'loader' Remainder of file ignored対処法
自分のエラー文のから、この記事のエラー内容の一番上と同じようなpathを見つける。
(最後から2番目くらいに「site-packages」とあるはず。)
そのパスに該当するところにターミナルで移動します。# /home/cjones/.local/lib/python3.6/site-packages/ #このように「site-packages」までを入力してください $ cd 見つけたパス移動が完了したら、次のようにターミナルに打ち込んでください。
# googleapis_common_protos-1.5.8-py3.6-nspkg.pth # site-packages以降の部分を打ち込みます。 $ vi site-packages以降の部分そうするとファイルが開くので、「i」を押して編集モードにします。
import sys, types, os;has_mfs = sys.version_info > (3, 5);p ...<rest of file>この部分を探し、次のように改行を入れてください。
import sys, types, os; has_mfs = sys.version_info > (3, 5);p ...<rest of file>※「i」を打たないと編集できないので注意してください。
それが終わったら「esc」を押してください。
最後に「:wq」を押すと保存+終了ができるので完了です。
最後に
今後も自分が体験したエラーについての記事も上げていきます。
参考サイト
AttributeError: 'NoneType' object has no attribute 'loader'
- 投稿日:2020-09-13T14:56:58+09:00
Natural Language : BERT Part1 - Japanese Wikipedia Corpus
目標
Microsoft Cognitive Toolkit (CNTK) を用いた BERT についてまとめました。
Part1 では、BERT のための準備を行います。
以下の順で紹介します。
- 日本語Wikipedia のダウンロードとテキストデータの抽出
- テキストデータの前処理と Sentence Piece モデルの作成
- 事前学習コーパスの作成
導入
日本語Wikipedia のダウンロード
今回は日本語コーパスとして、日本語Wikipedia を使用します。
上記のリンクから jawiki-latest-pages-articles-multistream.xml.bz2 をダウンロードします。次に、wikiextractor を用いてマークアップ言語を取り除きます。
$ python ./wikiextractor-master/WikiExtractor.py ./jawiki/jawiki-latest-pages-articles-multistream.xml.bz2 -o ./jawiki -b 500M今回のディレクトリの構成は以下のようにしました。
BERT
|―jawiki
jawiki-latest-pages-articles-multistream.xml.bz2
|―wikiextractor-master
WikiExtractor.py
...
bert_corpus.py
Doc2Vec
NMTT
STSA
Word2Vecテキストデータの前処理と Sentence Piece モデルの作成
これまでに実装してきた前処理に加えて、鍵括弧と句読点の表記の正規化や仮名漢字間のスペースの削除といった前処理を実行しました。
単語の分割に関しては、sentencepiece [1] を用いたサブワードモデルを作成します。なお、特別な単語として [CLS], [SEP], [MASK] を定義しておきます。
事前学習コーパスの作成
BERT [2] の事前学習ではコーパスに含まれる文に対してマスクをかけることで教師なし学習として言語モデルを訓練するので、そのための訓練データを作成しておきます。
Masked Language Model では、単語系列中の 15% を置き換えることにして、80% の確率で特別な単語 [MASK] に、10% の確率でランダムな単語に、残る 10% の確率でそのままにしておきます。
また、今回は Next Sentence Prediction ではなく Sentence-Order Prediction [3] を採用します。
実装
実行環境
ハードウェア
・CPU Intel(R) Core(TM) i7-7700 3.60GHz
ソフトウェア
・Windows 10 Pro 1909
・Python 3.6.6
・nltk 3.4.5
・numpy 1.17.3
・sentencepiece 0.1.91実行するプログラム
実装したプログラムは GitHub で公開しています。
bert_corpus.py結果
プログラムを実行すると、前処理を施した一文を各行に書き込み、各トピックを空白行で区切った日本語コーパスが作成されます。
次に Sentence Piece モデルを訓練して、jawiki.model と jawiki.vocab が作成されます。
最後に事前学習を行うための CTFDeserializer で読み込むテキストファイルが作成されます。
これで訓練を行う準備が整ったので、Part2 では CNTK を使って日本語の教師なし事前学習を行います。
参考
- Taku Kudo and John Richardson. "SentencePiece: A simple and language independent subword tokenizer and detokenizer for Neural Text Processing", arXiv preprint arXiv:1808.06226, (2018).
- Jacob Devlin, Ming-Wei Chang, Kenton Lee, Kristina Toutanova. "BERT: Pre-training of Deep Bidirectional Transformers for Language Understanding", arXiv preprint arXiv:1810.04805, (2018).
- Zhenzhong Lan, Mingda Chen, Sebastian Goodman, Kevin Gimpel, Piyush Sharma, and Radu Soricut. "ALBERT: A Lite BERT for self-supervised learning of language representations", arXiv preprint arXiv:1909.11942 (2019).
- 投稿日:2020-09-13T14:42:09+09:00
論文と公共データベースを使って無料で始めるAI創薬
1. はじめに
コンピュータ上で化合物の活性や物性を予測することは従来より行われてきたが、特に近年、蓄積データの増大や、Deep Learning技術の発展もあり、AI創薬というキーワードを耳にするようになってきた。
そこで今回、新型コロナウィルス(SARS-CoV-2)の治療薬の予測に関する "Computational Models Identify Several FDA Approved or Experimental Drugs as Putative Agents Against SARS-CoV-2" という論文の内容を自分で試してみることで、AI創薬を「無料で」疑似体験してみたので共有したい。
この論文を選んだのは、以下3つの理由による。
- 公共データを利用しており、解析手順、プログラムがgithubで公開されているため、再現しやすい。
- 従来型の解析手法(フィンガープリント、ランダムフォレスト)が用いられており、比較的再現しやすい。
- 新型コロナウィルスの治療薬は、世界中で開発が進められており、興味があった。
2. 今回の作業方針
まず初めに、論文の内容を試すための作業方針を検討した(というか試行錯誤してこうなったというのが正解)
実は本論文における収集データ、解析プログラムは全てgithub( https://github.com/alvesvm/sars-cov-mpro/tree/master/mol-inf-2020 )で公開されており、これを使えば何の苦労もなく論文の内容を再現することができる。
ただ、これをそのままダウンロードし、Jupyter上でリターンキーを連打したとしても、「なるほど」と理解した気になって終わるだけで、何も残らないだろう。
手を動かし、詰まり、調べ、考えることの繰り返しにより、自分の理解不足に気づき、今まで理解できなかったことが、少しずつ理解できるようになると思う。
このため以下の方針で進めることとした。
- 公共データベースからのデータの収集も 自分で調べながら やってみる。
- 収集したデータを用いて、自分で予測プログラムを書いてみる。
- 最終結果を論文と比較し、できれば考察もしてみる。
- 論文の内容の詳細の確認や、詰まった場合のヒントを得る場合、結果の比較のためにgithubを参照する。
次に論文の概要、今回利用する公共データベースについて記載する。
3.参考論文概要
論文では、新型コロナウィルスのウイルス複製に関与するメインプロテアーゼを薬物のターゲット分子としており、これに非常によく似た構造をもつ2003年に流行したSARSコロナウイルスのメインプロテアーゼに対する阻害活性を予測する予測モデルを構築している。
さらに、DrugBankより取得した市販/中止/実験、および治験薬データに対し、構築した予測モデルを適用することで、最終的に41の薬を新型コロナウィルスの候補治療薬として提示している。
なお、学習データとなるSARSコロナウイルスに対する阻害活性情報は、ChEMBLやPDB等の公共データベースより収集している。
ちなみにターゲット分子であるメインプロテアーゼについては、PDBj入門:PDBjの生体高分子学習ポータルサイト 242: コロナウイルスプロテアーゼ(Coronavirus Proteases)で詳しく解説されている。
4. 公共データベースの概要
次に本論文で使われている公共データベースについて簡単にまとめてみる。
ChEMBL (https://www.ebi.ac.uk/chembl/)
ChEMBLは、医薬品および医薬品候補となりうる低分子の生物活性情報について、手動でキュレーションされたものを収載したデータベースである。
欧州分子生物学研究所(EMBL)の欧州バイオインフォマティクス研究所(EBI)によって管理されている。収載データ件数は次の通りである(ダウンロードしたV27のテーブルのレコード件数より算出)。
項目 収載件数 化合物数 2,444,828 生物活性値数 16,066,124 アッセイ数 1,221,361 PDB (https://www.rcsb.org/)
PDB(Protein Data Bank)はタンパク質と核酸の3次元構造データを収載したデータベースである。Worldwide Protein Data Bankにより管理されている。
収載データ件数次のとおりである(2020/9/12現在)。
項目 収載件数 Structures 49012 Structures of Human Sequences 12216 Nucleic Acid Containing Structures 12216 DrugBank (https://www.drugbank.ca/)
DrugBankは、薬物と薬物標的に関する情報を収載したデータベースである。Metabolomics Innovation Center (TMIC)にて管理されている。
最新リリース(バージョン5.1.7、2020-07-02にリリース)の収載件数は以下の通りである。
項目 収載件数 薬物 13,715 承認済み小分子薬 2,649 承認済み生物製剤(タンパク質、ペプチド、ワクチン、およびアレルゲン) 1,405 栄養補助食品 131 実験 (発見段階)薬物 6,417 リンクされたタンパク質シーケンス 5,234 さて、次からいよいよ、論文を内容を試す作業を進める。
5. データ準備
5.1 学習データの準備
ここでは論文に記載されているSARSウィルスのメインプロテアーゼに対する阻害分子の実験データ(ChEMBLE91件、PDB22件)の収集を試みる。
5.1.1 ChEMBLデータの取得
ChEMBLはWEBブラウザでも利用できるが、データをダウンロードし、ローカルのデータベースに登録してSQLから利用することもできる。今回はPostgreSQLをインストールし、PostgreSQL用のダンプをロードして利用することとする。
① ChEMBLデータのダウンロード
ftp://ftp.ebi.ac.uk/pub/databases/chembl/ChEMBLdb/latest/をクリックするとダウンロード用のファイルの一覧が表示されるので、chembl_27_postgresql.tar.gzをダウンロードし展開する。② PostgreSQLのインストール
続いてUbuntu18.04にPostgreSQLをインストール 等を参考に、PostgreSQLをインストールする。
③ ChEMBLのロード
PostgreSQLにログインしてデータベースを作成する。
pgdb=# create database chembl_27;続いてデータを解凍したディレクトリに移動し以下を実行する。
しばらく時間はかかるが、PostgreSQLにデータが取り込まれる。pg_restore --no-owner -U postgres -d chembl_27 chembl_27_postgresql.dmp④ ChEMBLのテーブルの理解
次にpgAdmin等からSQLを発行してデータを取得するわけだが、まずはどのテーブルにどんなデータが格納されているのかを
ftp://ftp.ebi.ac.uk/pub/databases/chembl/ChEMBLdb/latest/schema_documentation.txtをざっと見てみる。またSchema Questions and SQL ExamplesにSQLの例があるのでこれを見て重要そうなテーブルに当たりをつける。あたりをつけたテーブルについて、先のschema_documentation.txtを調べた結果を整理したのが以下の表になる。
テーブル名 説明 compound_structures 化合物のさまざまな構造表現(Molfile、InChIなど)を格納するテーブル molecule_dictionary 関連する識別子を持つ化合物/生物学的治療薬の非冗長リスト compound_records 科学的ドキュメントより抽出された各化合物を表す。 docs アッセイが抽出されたすべての科学的ドキュメント(論文または特許)を保持する activities 科学的ドキュメントに記録されたアッセイの結果である活性値またはエンドポイントを格納するテーブル assay 各ドキュメントで報告されているアッセイのリストを保存する。異なるドキュメントからの同じアッセイは、別のアッセイとして表示される target_dictionary ChEMBLに含まれる全てのキュレーション済のターゲットの辞書。タンパク質ターゲットと非タンパク質ターゲット(例:生物、組織、細胞株)の両方が含まれる。 ⑤ データベースにSQLを発行しSARSの阻害データをゲット
ChEMBLでは、全てのデータにCHEMBL IDが割り当てられるため、まずはSARSコロナウィルスのCHEMBL ID検索してみる。このため前項で調べたtarget_dictionaryテーブルに対し以下のSQLを投げてみる。
select * from target_dictionary where upper(pref_name) like '%SARS%'以下の通り4つが得られる。
今回はメインプロテアーゼ(3C-like proteaseとしても知られる)の阻害データを収集したいので、pref_nameより一番上のCHEMBL3927であることが分かる。
ターゲット分子のCHEMBL IDが分かれば、Schema Questions and SQL Examplesの下部の "Retrieve compound activity details for all targets containing a protein of interest" というクエリにより、ターゲット分子に対する活性を持つ分子の一覧が得られるので、このSQLのCHEMBL ID部分を変更すればよい。
SELECT m.chembl_id AS compound_chembl_id, s.canonical_smiles, r.compound_key, d.pubmed_id, d.doi, a.description, act.standard_type, act.standard_relation, act.standard_value, act.standard_units, act.activity_comment FROM compound_structures s, molecule_dictionary m, compound_records r, docs d, activities act, assays a, target_dictionary t WHERE s.molregno = m.molregno AND m.molregno = r.molregno AND r.record_id = act.record_id AND r.doc_id = d.doc_id AND act.assay_id = a.assay_id AND a.tid = t.tid AND t.chembl_id = 'CHEMBL3927' AND (act.standard_type ='IC50' or act.standard_type ='Ki') order by compound_chembl_id;注意点としては今回、阻害データだけほしいので、standard_typeとしてIC50または Kiを指定している。IC50とKiの定義は厳密には異なると思われるが、論文ではともに学習データとして利用しているっぽかったので、ここでは準ずることとする。
結果をpgAdminからcsvにエクスポートしておく。
⑥ データの整理
さて、ここはデータサイエンティストっぽい泥臭い作業となる。
csvには以下のようなデータが得られている。
canonical_smilesは化合物の構造を示すデータであるため、説明変数を生成するために必要となる。
standard_valueはstandard_typeの値(ここでは以降、阻害値と呼ぶことにする)であり、standard_unitsは単位である。
standard_unitsは今回は全部同じnMなので、単位を変換して揃える必要はない。論文の予測モデル用のデータ作成のため、以下の処理をした。
- csvのstandard_relationに不等号が含まれている場合の対応として、このようなケースでは安全をみた数値として判断することが多いが(例えば10より大きいというデータであれば、倍の20ぐらいと想定する等)、今回は不等号は無視し、standard_valueをそのまま阻害値として採用した。
- 複数の実験等によりCHEMBL IDが重複して含まれる場合、阻害値はこれらの平均をとった。
- 最終的な目的変数として、論文では阻害値のしきい値を10uMとしているため、10uMより小さい値は1 (Active)、大きい値は0 (Inactive)として出力した。
以下にこれらの処理をしたスクリプト断片を示す。
prev_chembl.pyrows_by_id = defaultdict(list) # csvを読み込み、CHEMBLID毎にrowをまとめる with open(args.input, "r") as f: reader = csv.DictReader(f) for row in reader: rows_by_id[row["compound_chembl_id"]].append(row) # activeとみなす値 threshold = 10000 # CHEMBLID毎に値を処理しながら出力する with open(args.output, "w") as f: writer = csv.writer(f, lineterminator="\n") writer.writerow(["chembl_id", "canonical_smiles", "value", "outcome"]) # CHEMBLID毎に値を処理 for id in rows_by_id: # 合計を求める total = 0.0 for row in rows_by_id[id]: value = row["standard_value"] total += float(value) # 平均を求める mean = total/len(rows_by_id[id]) print(f'{id},{mean}') outcome = 0 if mean < threshold: outcome = 1 writer.writerow([id, rows_by_id[id][0]["canonical_smiles"], mean, outcome])このプログラムを実行した結果、Active/Inactiveの件数に1件の差はあったものの、論文と同じ91件の学習データが得られた。論文では取得手順は書かれていなかったので、このデータの収集がほぼ再現できたことが今回一番嬉しかった。
5.1.2 PDBからのデータ取得
次にPDBからの取得を行う。実は取得手順は以前に書いた記事 バイオ系公共データベースからスクレイピングにより機械学習データを収集するとほぼ同じである。唯一の違いは、メインプロテアーゼかどうかを判断するために、macromoleculeという項目も収集している点である。scrapyを使わない版の収集プログラムを以下に再掲する。
get_pdb.pyimport requests import json import time import lxml.html import argparse import csv def get_ligand(ligand_id): tmp_url = "https://www.rcsb.org" + ligand_id response = requests.get(tmp_url) if response.status_code != 200: return response.status_code, [] html = response.text root = lxml.html.fromstring(html) #print(html) print(tmp_url) smiles = root.xpath("//tr[@id='chemicalIsomeric']/td[1]/text()")[0] inchi = root.xpath("//tr[@id='chemicalInChI']/td[1]/text()")[0] inchi_key = root.xpath("//tr[@id='chemicalInChIKey']/td[1]/text()")[0] return response.status_code, [smiles, inchi, inchi_key] def get_structure(structure_id): structure_url = "https://www.rcsb.org/structure/" tmp_url = structure_url + structure_id print(tmp_url) html = requests.get(tmp_url).text root = lxml.html.fromstring(html) macromolecule_trs = root.xpath("//tr[contains(@id,'macromolecule-entityId-')]") macromolecule = "" for tr in macromolecule_trs: print(tr.xpath("@id")) macromolecule += tr.xpath("td[position()=1]/text()")[0] + "," print(f"macro={macromolecule}") binding_trs = root.xpath("//tr[contains(@id,'binding_row')]") datas = [] ids = [] for tr in binding_trs: print(tr.xpath("@id")) d1 = tr.xpath("td[position()=1]/a/@href") if d1[0] in ids: continue ids.append(d1[0]) status_code, values = get_ligand(d1[0]) ligand_id = d1[0][(d1[0].rfind("/") + 1):] print(ligand_id) if status_code == 200: smiles, inchi, inchi_key = values #item = tr.xpath("a/td[position()=2]/text()")[0] item = tr.xpath("td[position()=2]/a/text()")[0] item = item.strip() value = tr.xpath("td[position()=2]/text()")[0] value = value.replace(":", "") value = value.replace(";", "") value = value.replace(" ", "") value = value.replace("\n", "") print(value) values = value.split(" ", 1) print(values) value = values[0].strip() unit = values[1].strip() datas.append([ligand_id, smiles, inchi, inchi_key, item, value, unit, macromolecule]) time.sleep(1) return datas def main(): parser = argparse.ArgumentParser() parser.add_argument("-output", type=str, required=True) args = parser.parse_args() base_url = "https://www.rcsb.org/search/data" payloads = {"query":{"type":"group","logical_operator":"and","nodes":[{"type":"group","logical_operator":"and","nodes":[{"type":"group","logical_operator":"or","nodes":[{"type":"group","logical_operator":"and","nodes":[{"type":"terminal","service":"text","parameters":{"attribute":"rcsb_binding_affinity.value","negation":False,"operator":"exists"},"node_id":0},{"type":"terminal","service":"text","parameters":{"attribute":"rcsb_binding_affinity.type","operator":"exact_match","value":"IC50"},"node_id":1}],"label":"nested-attribute"},{"type":"group","logical_operator":"and","nodes":[{"type":"terminal","service":"text","parameters":{"attribute":"rcsb_binding_affinity.value","negation":False,"operator":"exists"},"node_id":2},{"type":"terminal","service":"text","parameters":{"attribute":"rcsb_binding_affinity.type","operator":"exact_match","value":"Ki"},"node_id":3}],"label":"nested-attribute"}]},{"type":"group","logical_operator":"and","nodes":[{"type":"terminal","service":"text","parameters":{"operator":"exact_match","negation":False,"value":"Severe acute respiratory syndrome-related coronavirus","attribute":"rcsb_entity_source_organism.taxonomy_lineage.name"},"node_id":4}]}],"label":"text"}],"label":"query-builder"},"return_type":"entry","request_options":{"pager":{"start":0,"rows":100},"scoring_strategy":"combined","sort":[{"sort_by":"score","direction":"desc"}]},"request_info":{"src":"ui","query_id":"e757fdfd5f9fb0efa272769c5966e3f4"}} print(json.dumps(payloads)) response = requests.post( base_url, json.dumps(payloads), headers={'Content-Type': 'application/json'}) datas = [] for a in response.json()["result_set"]: structure_id = a["identifier"] datas.extend(get_structure(structure_id)) time.sleep(1) with open(args.output, "w") as f: writer = csv.writer(f, lineterminator="\n") writer.writerow(["ligand_id", "canonical_smiles", "inchi", "inchi_key", "item", "value", "unit"]) for data in datas: writer.writerow(data) if __name__ == "__main__": main()これを実行すると以下のような形式のデータが21件得られる。一番右の項目にタンパク質の名前があるので、"3C like proteinase"や、"main proteinase"といったフレーズが含まれていないものは除外し、最終的に10件を学習データとした。
ligand_id,canonical_smiles,inchi,inchi_key,item,value,unit PMA,OC(=O)c1cc(C(O)=O)c(cc1C(O)=O)C(O)=O,"InChI=1S/C10H6O8/c11-7(12)3-1-4(8(13)14)6(10(17)18)2-5(3)9(15)16/h1-2H,(H,11,12)(H,13,14)(H,15,16)(H,17,18)",CYIDZMCFTVVTJO-UHFFFAOYSA-N,Ki,700,nM,"3C-like proteinase," TLD,Cc1ccc(S)c(S)c1,"InChI=1S/C7H8S2/c1-5-2-3-6(8)7(9)4-5/h2-4,8-9H,1H3",NIAAGQAEVGMHPM-UHFFFAOYSA-N,Ki,1400,nM,"Replicase polyprotein 1ab," ZU5,CC(C)C[C@H](NC(=O)[C@@H](NC(=O)OCc1ccccc1)[C@@H](C)OC(C)(C)C)C(=O)N[C@H](CCC(=O)C2CC2)C[C@@H]3CCNC3=O,"InChI=1S/C34H52N4O7/c1-21(2)18-27(31(41)36-26(14-15-28(39)24-12-13-24)19-25-16-17-35-30(25)40)37-32(42)29(22(3)45-34(4,5)6)38-33(43)44-20-23-10-8-7-9-11-23/h7-11,21-22,24-27,29H,12-20H2,1-6H3,(H,35,40)(H,36,41)(H,37,42)(H,38,43)/t22-,25+,26-,27+,29+/m1/s1",QIMPWBPEAHOISN-XSLDCGIXSA-N,Ki,99,nM,"3C-like proteinase," ZU3,CC(C)C[C@H](NC(=O)[C@H](CNC(=O)C(C)(C)C)NC(=O)OCc1ccccc1)C(=O)N[C@H](CCC(C)=O)C[C@@H]2CCNC2=O,"InChI=1S/C32H49N5O7/c1-20(2)16-25(28(40)35-24(13-12-21(3)38)17-23-14-15-33-27(23)39)36-29(41)26(18-34-30(42)32(4,5)6)37-31(43)44-19-22-10-8-7-9-11-22/h7-11,20,23-26H,12-19H2,1-6H3,(H,33,39)(H,34,42)(H,35,40)(H,36,41)(H,37,43)/t23-,24+,25-,26-/m0/s1",IEQRDAZPCPYZAJ-QYOOZWMWSA-N,Ki,38,nM,"3C-like proteinase," S89,OC[C@H](Cc1ccccc1)NC(=O)[C@H](Cc2ccccc2)NC(=O)/C=C/c3ccccc3,"InChI=1S/C27H28N2O3/c30-20-24(18-22-12-6-2-7-13-22)28-27(32)25(19-23-14-8-3-9-15-23)29-26(31)17-16-21-10-4-1-5-11-21/h1-17,24-25,30H,18-20H2,(H,28,32)(H,29,31)/b17-16+/t24-,25-/m0/s1",GEVQDXBVGFGWFA-KQRRRSJSSA-N,Ki,2240,nM,"3C-like proteinase,"残念ながらChEMBLのようにはうまくいかず論文の22件は得られなかった。また上の10件の中には、論文の22件に含まれていないものも何件かあった。
これについては、次のいずれかまたは、組み合わせが原因と考えている。
- PDBに対する検索の指定の仕方に誤り、漏れがあった。
- タンパク質の構造に紐づけられているLingadデータのみを収集対象したが、実は紐づけられていないものについても、リンク先の論文の中にデータが存在していた可能性がある。
原因は不明だが、今回の指定手順で得られたデータ自体が間違っているとも思えなかったので、PDBについてはこれら10件を採用することとした。
5.1.3 ChEMBLとPDBのデータのマージ
5.1.1, 5.1.2で得られたデータをマージし、最終的に101件を学習データとしてcsvに出力した。スクリプトは省略する。
5.2 バーチャルスクリーニング用データの準備
本論文では、市販/中止/実験、および治験薬に対し、構築した予測モデルを適用している。このために必要となる、市販/中止/実験、および治験薬の構造データをDrugBankより取得しておく。論文のgithubには整形されたデータがそのままあるが、しつこいくらいに自分でやってみる。
5.2.1 DrugBankからのデータ取得
まずXMLデータとして、
https://www.drugbank.ca/releases/latestの"DOWNLOAD (XML)"と記載のあるリンクから、xmlをダウンロードする。続いて
https://www.drugbank.ca/releases/latest#structuresからsdfをダウンロードする。アカウントは事前に作成しておく必要があったかもしれない。
これらを"full database.xml", "open structures.sdf"として保存する。
5.2.2 DrugBankデータの整形
sdfに構造データが格納されておりこれだけでも予測モデルを適用することはできるが、xmlには市販/中止/実験等の種別があるため、これらを関連づけてcsvに保存する。
以下にスクリプト断片を示す。make_drugbank_data.pydef get_group(groups_node, ns): ret = "" if groups_node: for i, child in enumerate(groups_node.iter(f"{ns}group")): if i > 0 : ret += "," ret += child.text return ret def get_id(drug_node, ns): for i, child in enumerate(drug_node.iter(f"{ns}drugbank-id")): for attr in child.attrib: if attr == "primary": return child.text return None def main(): parser = argparse.ArgumentParser() parser.add_argument("-input_xml", type=str, required=True) parser.add_argument("-input_sdf", type=str, required=True) parser.add_argument("-output", type=str, required=True) args = parser.parse_args() name_dict = {} smiles_dict = {} sdf_sup = Chem.SDMolSupplier(args.input_sdf) datas = [] for i, mol in enumerate(sdf_sup): if not mol: continue if mol.HasProp("DRUGBANK_ID"): id = mol.GetProp("DRUGBANK_ID") if mol.HasProp("COMMON_NAME"): name = mol.GetProp("COMMON_NAME") smiles = Chem.MolToSmiles(mol) name_dict[id] = name smiles_dict[id] = smiles print(f"{i} {id} {name} {smiles}") tree = ET.parse(args.input_xml) root = tree.getroot() ns = "{http://www.drugbank.ca}" ids = [] datas = [] for i, drug in enumerate(root.iter(f"{ns}drug")): id = get_id(drug, ns) category = get_group(drug.find(f"{ns}groups"), ns) if id and id in smiles_dict: print(f"{i}, {id}, {category}") ids.append(id) datas.append([name_dict[id], category, smiles_dict[id]]) df = pd.DataFrame(datas, index=ids, columns=[["name", "status", "smiles"]]) df.to_csv(args.output) if __name__ == "__main__": main()このスクリプトを実行して得られるデータはこんな感じになる。構造データはSMILESとして保存している。
id,name,status,smiles DB00006,Bivalirudin,"approved,investigational",CC[C@H](C)[C@H](NC(=O)[C@H](CCC(=O)O)NC(=O)[C@H](CCC(=O)O)NC(=O)[C@H](Cc1ccccc1)NC(=O)[C@H](CC(=O)O)NC(=O)CNC(=O)[C@H](CC(N)=O)NC(=O)CNC(=O)CNC(=O)CNC(=O)CNC(=O)[C@@H]1CCCN1C(=O)[C@H](CCCNC(=N)N)NC(=O)[C@@H]1CCCN1C(=O)[C@H](N)Cc1ccccc1)C(=O)N1CCC[C@H]1C(=O)N[C@@H](CCC(=O)O)C(=O)N[C@@H](CCC(=O)O)C(=O)N[C@@H](Cc1ccc(O)cc1)C(=O)N[C@@H](CC(C)C)C(=O)O DB00007,Leuprolide,"approved,investigational",CCNC(=O)[C@@H]1CCCN1C(=O)[C@H](CCCNC(=N)N)NC(=O)[C@H](CC(C)C)NC(=O)[C@@H](CC(C)C)NC(=O)[C@H](Cc1ccc(O)cc1)NC(=O)[C@H](CO)NC(=O)[C@H](Cc1c[nH]c2ccccc12)NC(=O)[C@H](Cc1c[nH]cn1)NC(=O)[C@@H]1CCC(=O)N1 DB00014,Goserelin,approved,CC(C)C[C@H](NC(=O)[C@@H](COC(C)(C)C)NC(=O)[C@H](Cc1ccc(O)cc1)NC(=O)[C@H](CO)NC(=O)[C@H](Cc1c[nH]c2ccccc12)NC(=O)[C@H](Cc1cnc[nH]1)NC(=O)[C@@H]1CCC(=O)N1)C(=O)N[C@@H](CCCN=C(N)N)C(=O)N1CCC[C@H]1C(=O)NNC(N)=O DB00027,Gramicidin D,approved,CC(C)C[C@@H](NC(=O)CNC(=O)[C@@H](NC=O)C(C)C)C(=O)N[C@@H](C)C(=O)N[C@@H](C(=O)N[C@H](C(=O)N[C@@H](C(=O)N[C@@H](Cc1c[nH]c2ccccc12)C(=O)N[C@H](CC(C)C)C(=O)N[C@@H](Cc1c[nH]c2ccccc12)C(=O)N[C@H](CC(C)C)C(=O)N[C@@H](Cc1c[nH]c2ccccc12)C(=O)N[C@H](CC(C)C)C(=O)N[C@@H](Cc1c[nH]c2ccccc12)C(=O)NCCO)C(C)C)C(C)C)C(C)C DB00035,Desmopressin,approved,N=C(N)NCCC[C@@H](NC(=O)[C@@H]1CCCN1C(=O)[C@@H]1CSSCCC(=O)N[C@@H](Cc2ccc(O)cc2)C(=O)N[C@@H](Cc2ccccc2)C(=O)N[C@@H](CCC(N)=O)C(=O)N[C@@H](CC(N)=O)C(=O)N1)C(=O)NCC(N)=O5.3 予測モデルの構築
次に学習データを使って予測モデルを構築する。
5.3.1 構造データからのフィンガープリント生成
ここでは、5.1.3 で生成されたcsvファイルから機械学習用の説明変数を生成する。
具体的には、csvファイルの"canonical_smiles"列にあるSMILESを読み取り、RDKitを使ってmolオブジェクトに変換する。その後、RDKitのAllChem.GetMorganFingerprintAsBitVectを使って2048ビットのフィンガープリントを生成する。フィンガープリントとはざっくり説明すると、化合物に特定の構造が含まれていれば、それに対応したビットが1、そうでない場合は0となるものがいくつかか集まったものである。今回は2048個の0または1で構成されるビット列が生成される。calc_descriptor.pyfrom rdkit import Chem from rdkit.Chem import AllChem from molvs.normalize import Normalizer, Normalization from molvs.tautomer import TAUTOMER_TRANSFORMS, TAUTOMER_SCORES, MAX_TAUTOMERS, TautomerCanonicalizer, TautomerEnumerator, TautomerTransform from molvs.fragment import LargestFragmentChooser from molvs.charge import Reionizer, Uncharger import argparse import csv import pandas as pd import numpy as np def normalize(smiles): # Generate Mol mol = Chem.MolFromSmiles(smiles) # Uncharge uncharger = Uncharger() mol = uncharger.uncharge(mol) # LargestFragmentChooser flagmentChooser = LargestFragmentChooser() mol = flagmentChooser(mol) # Sanitaize Chem.SanitizeMol(mol) # Normalize normalizer = Normalizer() mol = normalizer.normalize(mol) tautomerCanonicalizer = TautomerCanonicalizer() mol = tautomerCanonicalizer.canonicalize(mol) return Chem.MolToSmiles(mol) def main(): parser = argparse.ArgumentParser() parser.add_argument("-input", type=str, required=True) parser.add_argument("-output", type=str, required=True) args = parser.parse_args() #学習データの読み込み smiles_list = list() datas = [] with open(args.input, "r") as f: reader = csv.DictReader(f) for row in reader: org_smiles = row["canonical_smiles"] new_smiles = normalize(org_smiles) existFlag = False for tmp_smiles in smiles_list: if new_smiles == tmp_smiles: print("exist!") existFlag = True break if not existFlag: smiles_list.append(new_smiles) mol = Chem.MolFromSmiles(new_smiles) fp = AllChem.GetMorganFingerprintAsBitVect(mol, radius=3, nBits=2048, useFeatures=False, useChirality=False) fp = pd.Series(np.asarray(fp)).values data = [] data.append(row["id"]) data.append(int(row["outcome"])) data.extend(fp) datas.append(data) columns = list() columns.append("id") columns.append("outcome") columns.extend(["Bit_" + str(i + 1) for i in range(2048)]) df = pd.DataFrame(data=datas, columns=columns) df.set_index("id", inplace=True, drop=True) #保存 df.to_csv(args.output) if __name__ == "__main__": main()化合物の前処理、標準化を行い、念のため同じ化合物が存在した場合に除去するという処理も入れた。化合物から3次元座標を生成するという処理も行われことがあるが、今回利用したフィンガープリントの場合3次元座標は不要のため、処理はしていない。
実行後すると以下のようなデータが得られる。1列目がID、2列目が目的変数、3列目以降がフィンガープリントによる説明変数である。id,outcome,Bit_1,Bit_2,Bit_3,Bit_4,Bit_5,Bit_6,Bit_7,Bit_8,Bit_9,Bit_10,Bit_11,Bit_12,Bit_13,Bit_14,Bit_15,Bit_16,Bit_17,Bit_18,Bit_19,Bit_20,Bit_21,Bit_22,Bit_23,Bit_24,Bit_25,Bit_26,Bit_27,Bit_28,Bit_29,Bit_30,Bit_31,Bit_32,Bit_33,Bit_34,Bit_35,Bit_36,Bit_37,Bit_38,Bit_39,Bit_40,Bit_41,Bit_42,Bit_43,Bit_44,Bit_45,Bit_46,Bit_47,Bit_48,Bit_49,Bit_50,Bit_51,Bit_52,Bit_53,Bit_54,Bit_55,Bit_56,Bit_57,Bit_58,Bit_59,Bit_60,Bit_61,Bit_62,Bit_63,Bit_64,Bit_65,Bit_66,Bit_67,Bit_68,Bit_69,Bit_70,Bit_71,Bit_72,Bit_73,Bit_74,Bit_75,Bit_76,Bit_77,Bit_78,Bit_79,Bit_80,Bit_81,Bit_82,Bit_83,Bit_84,Bit_85,Bit_86,Bit_87,Bit_88,Bit_89,Bit_90,Bit_91,Bit_92,Bit_93,Bit_94,Bit_95,Bit_96,Bit_97,Bit_98,Bit_99,Bit_100,Bit_101,Bit_102,Bit_103,Bit_104,Bit_105,Bit_106,Bit_107,Bit_108,Bit_109,Bit_110,Bit_111,Bit_112,Bit_113,Bit_114,Bit_115,Bit_116,Bit_117,Bit_118,Bit_119,Bit_120,Bit_121,Bit_122,Bit_123,Bit_124,Bit_125,Bit_126,Bit_127,Bit_128,Bit_129,Bit_130,Bit_131,Bit_132,Bit_133,Bit_134,Bit_135,Bit_136,Bit_137,Bit_138,Bit_139,Bit_140,Bit_141,Bit_142,Bit_143,Bit_144,Bit_145,Bit_146,Bit_147,Bit_148,Bit_149,Bit_150,Bit_151,Bit_152,Bit_153,Bit_154,Bit_155,Bit_156,Bit_157,Bit_158,Bit_159,Bit_160,Bit_161,Bit_162,Bit_163,Bit_164,Bit_165,Bit_166,Bit_167,Bit_168,Bit_169,Bit_170,Bit_171,Bit_172,Bit_173,Bit_174,Bit_175,Bit_176,Bit_177,Bit_178,Bit_179,Bit_180,Bit_181,Bit_182,Bit_183,Bit_184,Bit_185,Bit_186,Bit_187,Bit_188,Bit_189,Bit_190,Bit_191,Bit_192,Bit_193,Bit_194,Bit_195,Bit_196,Bit_197,Bit_198,Bit_199,Bit_200,Bit_201,Bit_202,Bit_203,Bit_204,Bit_205,Bit_206,Bit_207,Bit_208,Bit_209,Bit_210,Bit_211,Bit_212,Bit_213,Bit_214,Bit_215,Bit_216,Bit_217,Bit_218,Bit_219,Bit_220,Bit_221,Bit_222,Bit_223,Bit_224,Bit_225,Bit_226,Bit_227,Bit_228,Bit_229,Bit_230,Bit_231,Bit_232,Bit_233,Bit_234,Bit_235,Bit_236,Bit_237,Bit_238,Bit_239,Bit_240,Bit_241,Bit_242,Bit_243,Bit_244,Bit_245,Bit_246,Bit_247,Bit_248,Bit_249,Bit_250,Bit_251,Bit_252,Bit_253,Bit_254,Bit_255,Bit_256,Bit_257,Bit_258,Bit_259,Bit_260,Bit_261,Bit_262,Bit_263,Bit_264,Bit_265,Bit_266,Bit_267,Bit_268,Bit_269,Bit_270,Bit_271,Bit_272,Bit_273,Bit_274,Bit_275,Bit_276,Bit_277,Bit_278,Bit_279,Bit_280,Bit_281,Bit_282,Bit_283,Bit_284,Bit_285,Bit_286,Bit_287,Bit_288,Bit_289,Bit_290,Bit_291,Bit_292,Bit_293,Bit_294,Bit_295,Bit_296,Bit_297,Bit_298,Bit_299,Bit_300,Bit_301,Bit_302,Bit_303,Bit_304,Bit_305,Bit_306,Bit_307,Bit_308,Bit_309,Bit_310,Bit_311,Bit_312,Bit_313,Bit_314,Bit_315,Bit_316,Bit_317,Bit_318,Bit_319,Bit_320,Bit_321,Bit_322,Bit_323,Bit_324,Bit_325,Bit_326,Bit_327,Bit_328,Bit_329,Bit_330,Bit_331,Bit_332,Bit_333,Bit_334,Bit_335,Bit_336,Bit_337,Bit_338,Bit_339,Bit_340,Bit_341,Bit_342,Bit_343,Bit_344,Bit_345,Bit_346,Bit_347,Bit_348,Bit_349,Bit_350,Bit_351,Bit_352,Bit_353,Bit_354,Bit_355,Bit_356,Bit_357,Bit_358,Bit_359,Bit_360,Bit_361,Bit_362,Bit_363,Bit_364,Bit_365,Bit_366,Bit_367,Bit_368,Bit_369,Bit_370,Bit_371,Bit_372,Bit_373,Bit_374,Bit_375,Bit_376,Bit_377,Bit_378,Bit_379,Bit_380,Bit_381,Bit_382,Bit_383,Bit_384,Bit_385,Bit_386,Bit_387,Bit_388,Bit_389,Bit_390,Bit_391,Bit_392,Bit_393,Bit_394,Bit_395,Bit_396,Bit_397,Bit_398,Bit_399,Bit_400,Bit_401,Bit_402,Bit_403,Bit_404,Bit_405,Bit_406,Bit_407,Bit_408,Bit_409,Bit_410,Bit_411,Bit_412,Bit_413,Bit_414,Bit_415,Bit_416,Bit_417,Bit_418,Bit_419,Bit_420,Bit_421,Bit_422,Bit_423,Bit_424,Bit_425,Bit_426,Bit_427,Bit_428,Bit_429,Bit_430,Bit_431,Bit_432,Bit_433,Bit_434,Bit_435,Bit_436,Bit_437,Bit_438,Bit_439,Bit_440,Bit_441,Bit_442,Bit_443,Bit_444,Bit_445,Bit_446,Bit_447,Bit_448,Bit_449,Bit_450,Bit_451,Bit_452,Bit_453,Bit_454,Bit_455,Bit_456,Bit_457,Bit_458,Bit_459,Bit_460,Bit_461,Bit_462,Bit_463,Bit_464,Bit_465,Bit_466,Bit_467,Bit_468,Bit_469,Bit_470,Bit_471,Bit_472,Bit_473,Bit_474,Bit_475,Bit_476,Bit_477,Bit_478,Bit_479,Bit_480,Bit_481,Bit_482,Bit_483,Bit_484,Bit_485,Bit_486,Bit_487,Bit_488,Bit_489,Bit_490,Bit_491,Bit_492,Bit_493,Bit_494,Bit_495,Bit_496,Bit_497,Bit_498,Bit_499,Bit_500,Bit_501,Bit_502,Bit_503,Bit_504,Bit_505,Bit_506,Bit_507,Bit_508,Bit_509,Bit_510,Bit_511,Bit_512,Bit_513,Bit_514,Bit_515,Bit_516,Bit_517,Bit_518,Bit_519,Bit_520,Bit_521,Bit_522,Bit_523,Bit_524,Bit_525,Bit_526,Bit_527,Bit_528,Bit_529,Bit_530,Bit_531,Bit_532,Bit_533,Bit_534,Bit_535,Bit_536,Bit_537,Bit_538,Bit_539,Bit_540,Bit_541,Bit_542,Bit_543,Bit_544,Bit_545,Bit_546,Bit_547,Bit_548,Bit_549,Bit_550,Bit_551,Bit_552,Bit_553,Bit_554,Bit_555,Bit_556,Bit_557,Bit_558,Bit_559,Bit_560,Bit_561,Bit_562,Bit_563,Bit_564,Bit_565,Bit_566,Bit_567,Bit_568,Bit_569,Bit_570,Bit_571,Bit_572,Bit_573,Bit_574,Bit_575,Bit_576,Bit_577,Bit_578,Bit_579,Bit_580,Bit_581,Bit_582,Bit_583,Bit_584,Bit_585,Bit_586,Bit_587,Bit_588,Bit_589,Bit_590,Bit_591,Bit_592,Bit_593,Bit_594,Bit_595,Bit_596,Bit_597,Bit_598,Bit_599,Bit_600,Bit_601,Bit_602,Bit_603,Bit_604,Bit_605,Bit_606,Bit_607,Bit_608,Bit_609,Bit_610,Bit_611,Bit_612,Bit_613,Bit_614,Bit_615,Bit_616,Bit_617,Bit_618,Bit_619,Bit_620,Bit_621,Bit_622,Bit_623,Bit_624,Bit_625,Bit_626,Bit_627,Bit_628,Bit_629,Bit_630,Bit_631,Bit_632,Bit_633,Bit_634,Bit_635,Bit_636,Bit_637,Bit_638,Bit_639,Bit_640,Bit_641,Bit_642,Bit_643,Bit_644,Bit_645,Bit_646,Bit_647,Bit_648,Bit_649,Bit_650,Bit_651,Bit_652,Bit_653,Bit_654,Bit_655,Bit_656,Bit_657,Bit_658,Bit_659,Bit_660,Bit_661,Bit_662,Bit_663,Bit_664,Bit_665,Bit_666,Bit_667,Bit_668,Bit_669,Bit_670,Bit_671,Bit_672,Bit_673,Bit_674,Bit_675,Bit_676,Bit_677,Bit_678,Bit_679,Bit_680,Bit_681,Bit_682,Bit_683,Bit_684,Bit_685,Bit_686,Bit_687,Bit_688,Bit_689,Bit_690,Bit_691,Bit_692,Bit_693,Bit_694,Bit_695,Bit_696,Bit_697,Bit_698,Bit_699,Bit_700,Bit_701,Bit_702,Bit_703,Bit_704,Bit_705,Bit_706,Bit_707,Bit_708,Bit_709,Bit_710,Bit_711,Bit_712,Bit_713,Bit_714,Bit_715,Bit_716,Bit_717,Bit_718,Bit_719,Bit_720,Bit_721,Bit_722,Bit_723,Bit_724,Bit_725,Bit_726,Bit_727,Bit_728,Bit_729,Bit_730,Bit_731,Bit_732,Bit_733,Bit_734,Bit_735,Bit_736,Bit_737,Bit_738,Bit_739,Bit_740,Bit_741,Bit_742,Bit_743,Bit_744,Bit_745,Bit_746,Bit_747,Bit_748,Bit_749,Bit_750,Bit_751,Bit_752,Bit_753,Bit_754,Bit_755,Bit_756,Bit_757,Bit_758,Bit_759,Bit_760,Bit_761,Bit_762,Bit_763,Bit_764,Bit_765,Bit_766,Bit_767,Bit_768,Bit_769,Bit_770,Bit_771,Bit_772,Bit_773,Bit_774,Bit_775,Bit_776,Bit_777,Bit_778,Bit_779,Bit_780,Bit_781,Bit_782,Bit_783,Bit_784,Bit_785,Bit_786,Bit_787,Bit_788,Bit_789,Bit_790,Bit_791,Bit_792,Bit_793,Bit_794,Bit_795,Bit_796,Bit_797,Bit_798,Bit_799,Bit_800,Bit_801,Bit_802,Bit_803,Bit_804,Bit_805,Bit_806,Bit_807,Bit_808,Bit_809,Bit_810,Bit_811,Bit_812,Bit_813,Bit_814,Bit_815,Bit_816,Bit_817,Bit_818,Bit_819,Bit_820,Bit_821,Bit_822,Bit_823,Bit_824,Bit_825,Bit_826,Bit_827,Bit_828,Bit_829,Bit_830,Bit_831,Bit_832,Bit_833,Bit_834,Bit_835,Bit_836,Bit_837,Bit_838,Bit_839,Bit_840,Bit_841,Bit_842,Bit_843,Bit_844,Bit_845,Bit_846,Bit_847,Bit_848,Bit_849,Bit_850,Bit_851,Bit_852,Bit_853,Bit_854,Bit_855,Bit_856,Bit_857,Bit_858,Bit_859,Bit_860,Bit_861,Bit_862,Bit_863,Bit_864,Bit_865,Bit_866,Bit_867,Bit_868,Bit_869,Bit_870,Bit_871,Bit_872,Bit_873,Bit_874,Bit_875,Bit_876,Bit_877,Bit_878,Bit_879,Bit_880,Bit_881,Bit_882,Bit_883,Bit_884,Bit_885,Bit_886,Bit_887,Bit_888,Bit_889,Bit_890,Bit_891,Bit_892,Bit_893,Bit_894,Bit_895,Bit_896,Bit_897,Bit_898,Bit_899,Bit_900,Bit_901,Bit_902,Bit_903,Bit_904,Bit_905,Bit_906,Bit_907,Bit_908,Bit_909,Bit_910,Bit_911,Bit_912,Bit_913,Bit_914,Bit_915,Bit_916,Bit_917,Bit_918,Bit_919,Bit_920,Bit_921,Bit_922,Bit_923,Bit_924,Bit_925,Bit_926,Bit_927,Bit_928,Bit_929,Bit_930,Bit_931,Bit_932,Bit_933,Bit_934,Bit_935,Bit_936,Bit_937,Bit_938,Bit_939,Bit_940,Bit_941,Bit_942,Bit_943,Bit_944,Bit_945,Bit_946,Bit_947,Bit_948,Bit_949,Bit_950,Bit_951,Bit_952,Bit_953,Bit_954,Bit_955,Bit_956,Bit_957,Bit_958,Bit_959,Bit_960,Bit_961,Bit_962,Bit_963,Bit_964,Bit_965,Bit_966,Bit_967,Bit_968,Bit_969,Bit_970,Bit_971,Bit_972,Bit_973,Bit_974,Bit_975,Bit_976,Bit_977,Bit_978,Bit_979,Bit_980,Bit_981,Bit_982,Bit_983,Bit_984,Bit_985,Bit_986,Bit_987,Bit_988,Bit_989,Bit_990,Bit_991,Bit_992,Bit_993,Bit_994,Bit_995,Bit_996,Bit_997,Bit_998,Bit_999,Bit_1000,Bit_1001,Bit_1002,Bit_1003,Bit_1004,Bit_1005,Bit_1006,Bit_1007,Bit_1008,Bit_1009,Bit_1010,Bit_1011,Bit_1012,Bit_1013,Bit_1014,Bit_1015,Bit_1016,Bit_1017,Bit_1018,Bit_1019,Bit_1020,Bit_1021,Bit_1022,Bit_1023,Bit_1024,Bit_1025,Bit_1026,Bit_1027,Bit_1028,Bit_1029,Bit_1030,Bit_1031,Bit_1032,Bit_1033,Bit_1034,Bit_1035,Bit_1036,Bit_1037,Bit_1038,Bit_1039,Bit_1040,Bit_1041,Bit_1042,Bit_1043,Bit_1044,Bit_1045,Bit_1046,Bit_1047,Bit_1048,Bit_1049,Bit_1050,Bit_1051,Bit_1052,Bit_1053,Bit_1054,Bit_1055,Bit_1056,Bit_1057,Bit_1058,Bit_1059,Bit_1060,Bit_1061,Bit_1062,Bit_1063,Bit_1064,Bit_1065,Bit_1066,Bit_1067,Bit_1068,Bit_1069,Bit_1070,Bit_1071,Bit_1072,Bit_1073,Bit_1074,Bit_1075,Bit_1076,Bit_1077,Bit_1078,Bit_1079,Bit_1080,Bit_1081,Bit_1082,Bit_1083,Bit_1084,Bit_1085,Bit_1086,Bit_1087,Bit_1088,Bit_1089,Bit_1090,Bit_1091,Bit_1092,Bit_1093,Bit_1094,Bit_1095,Bit_1096,Bit_1097,Bit_1098,Bit_1099,Bit_1100,Bit_1101,Bit_1102,Bit_1103,Bit_1104,Bit_1105,Bit_1106,Bit_1107,Bit_1108,Bit_1109,Bit_1110,Bit_1111,Bit_1112,Bit_1113,Bit_1114,Bit_1115,Bit_1116,Bit_1117,Bit_1118,Bit_1119,Bit_1120,Bit_1121,Bit_1122,Bit_1123,Bit_1124,Bit_1125,Bit_1126,Bit_1127,Bit_1128,Bit_1129,Bit_1130,Bit_1131,Bit_1132,Bit_1133,Bit_1134,Bit_1135,Bit_1136,Bit_1137,Bit_1138,Bit_1139,Bit_1140,Bit_1141,Bit_1142,Bit_1143,Bit_1144,Bit_1145,Bit_1146,Bit_1147,Bit_1148,Bit_1149,Bit_1150,Bit_1151,Bit_1152,Bit_1153,Bit_1154,Bit_1155,Bit_1156,Bit_1157,Bit_1158,Bit_1159,Bit_1160,Bit_1161,Bit_1162,Bit_1163,Bit_1164,Bit_1165,Bit_1166,Bit_1167,Bit_1168,Bit_1169,Bit_1170,Bit_1171,Bit_1172,Bit_1173,Bit_1174,Bit_1175,Bit_1176,Bit_1177,Bit_1178,Bit_1179,Bit_1180,Bit_1181,Bit_1182,Bit_1183,Bit_1184,Bit_1185,Bit_1186,Bit_1187,Bit_1188,Bit_1189,Bit_1190,Bit_1191,Bit_1192,Bit_1193,Bit_1194,Bit_1195,Bit_1196,Bit_1197,Bit_1198,Bit_1199,Bit_1200,Bit_1201,Bit_1202,Bit_1203,Bit_1204,Bit_1205,Bit_1206,Bit_1207,Bit_1208,Bit_1209,Bit_1210,Bit_1211,Bit_1212,Bit_1213,Bit_1214,Bit_1215,Bit_1216,Bit_1217,Bit_1218,Bit_1219,Bit_1220,Bit_1221,Bit_1222,Bit_1223,Bit_1224,Bit_1225,Bit_1226,Bit_1227,Bit_1228,Bit_1229,Bit_1230,Bit_1231,Bit_1232,Bit_1233,Bit_1234,Bit_1235,Bit_1236,Bit_1237,Bit_1238,Bit_1239,Bit_1240,Bit_1241,Bit_1242,Bit_1243,Bit_1244,Bit_1245,Bit_1246,Bit_1247,Bit_1248,Bit_1249,Bit_1250,Bit_1251,Bit_1252,Bit_1253,Bit_1254,Bit_1255,Bit_1256,Bit_1257,Bit_1258,Bit_1259,Bit_1260,Bit_1261,Bit_1262,Bit_1263,Bit_1264,Bit_1265,Bit_1266,Bit_1267,Bit_1268,Bit_1269,Bit_1270,Bit_1271,Bit_1272,Bit_1273,Bit_1274,Bit_1275,Bit_1276,Bit_1277,Bit_1278,Bit_1279,Bit_1280,Bit_1281,Bit_1282,Bit_1283,Bit_1284,Bit_1285,Bit_1286,Bit_1287,Bit_1288,Bit_1289,Bit_1290,Bit_1291,Bit_1292,Bit_1293,Bit_1294,Bit_1295,Bit_1296,Bit_1297,Bit_1298,Bit_1299,Bit_1300,Bit_1301,Bit_1302,Bit_1303,Bit_1304,Bit_1305,Bit_1306,Bit_1307,Bit_1308,Bit_1309,Bit_1310,Bit_1311,Bit_1312,Bit_1313,Bit_1314,Bit_1315,Bit_1316,Bit_1317,Bit_1318,Bit_1319,Bit_1320,Bit_1321,Bit_1322,Bit_1323,Bit_1324,Bit_1325,Bit_1326,Bit_1327,Bit_1328,Bit_1329,Bit_1330,Bit_1331,Bit_1332,Bit_1333,Bit_1334,Bit_1335,Bit_1336,Bit_1337,Bit_1338,Bit_1339,Bit_1340,Bit_1341,Bit_1342,Bit_1343,Bit_1344,Bit_1345,Bit_1346,Bit_1347,Bit_1348,Bit_1349,Bit_1350,Bit_1351,Bit_1352,Bit_1353,Bit_1354,Bit_1355,Bit_1356,Bit_1357,Bit_1358,Bit_1359,Bit_1360,Bit_1361,Bit_1362,Bit_1363,Bit_1364,Bit_1365,Bit_1366,Bit_1367,Bit_1368,Bit_1369,Bit_1370,Bit_1371,Bit_1372,Bit_1373,Bit_1374,Bit_1375,Bit_1376,Bit_1377,Bit_1378,Bit_1379,Bit_1380,Bit_1381,Bit_1382,Bit_1383,Bit_1384,Bit_1385,Bit_1386,Bit_1387,Bit_1388,Bit_1389,Bit_1390,Bit_1391,Bit_1392,Bit_1393,Bit_1394,Bit_1395,Bit_1396,Bit_1397,Bit_1398,Bit_1399,Bit_1400,Bit_1401,Bit_1402,Bit_1403,Bit_1404,Bit_1405,Bit_1406,Bit_1407,Bit_1408,Bit_1409,Bit_1410,Bit_1411,Bit_1412,Bit_1413,Bit_1414,Bit_1415,Bit_1416,Bit_1417,Bit_1418,Bit_1419,Bit_1420,Bit_1421,Bit_1422,Bit_1423,Bit_1424,Bit_1425,Bit_1426,Bit_1427,Bit_1428,Bit_1429,Bit_1430,Bit_1431,Bit_1432,Bit_1433,Bit_1434,Bit_1435,Bit_1436,Bit_1437,Bit_1438,Bit_1439,Bit_1440,Bit_1441,Bit_1442,Bit_1443,Bit_1444,Bit_1445,Bit_1446,Bit_1447,Bit_1448,Bit_1449,Bit_1450,Bit_1451,Bit_1452,Bit_1453,Bit_1454,Bit_1455,Bit_1456,Bit_1457,Bit_1458,Bit_1459,Bit_1460,Bit_1461,Bit_1462,Bit_1463,Bit_1464,Bit_1465,Bit_1466,Bit_1467,Bit_1468,Bit_1469,Bit_1470,Bit_1471,Bit_1472,Bit_1473,Bit_1474,Bit_1475,Bit_1476,Bit_1477,Bit_1478,Bit_1479,Bit_1480,Bit_1481,Bit_1482,Bit_1483,Bit_1484,Bit_1485,Bit_1486,Bit_1487,Bit_1488,Bit_1489,Bit_1490,Bit_1491,Bit_1492,Bit_1493,Bit_1494,Bit_1495,Bit_1496,Bit_1497,Bit_1498,Bit_1499,Bit_1500,Bit_1501,Bit_1502,Bit_1503,Bit_1504,Bit_1505,Bit_1506,Bit_1507,Bit_1508,Bit_1509,Bit_1510,Bit_1511,Bit_1512,Bit_1513,Bit_1514,Bit_1515,Bit_1516,Bit_1517,Bit_1518,Bit_1519,Bit_1520,Bit_1521,Bit_1522,Bit_1523,Bit_1524,Bit_1525,Bit_1526,Bit_1527,Bit_1528,Bit_1529,Bit_1530,Bit_1531,Bit_1532,Bit_1533,Bit_1534,Bit_1535,Bit_1536,Bit_1537,Bit_1538,Bit_1539,Bit_1540,Bit_1541,Bit_1542,Bit_1543,Bit_1544,Bit_1545,Bit_1546,Bit_1547,Bit_1548,Bit_1549,Bit_1550,Bit_1551,Bit_1552,Bit_1553,Bit_1554,Bit_1555,Bit_1556,Bit_1557,Bit_1558,Bit_1559,Bit_1560,Bit_1561,Bit_1562,Bit_1563,Bit_1564,Bit_1565,Bit_1566,Bit_1567,Bit_1568,Bit_1569,Bit_1570,Bit_1571,Bit_1572,Bit_1573,Bit_1574,Bit_1575,Bit_1576,Bit_1577,Bit_1578,Bit_1579,Bit_1580,Bit_1581,Bit_1582,Bit_1583,Bit_1584,Bit_1585,Bit_1586,Bit_1587,Bit_1588,Bit_1589,Bit_1590,Bit_1591,Bit_1592,Bit_1593,Bit_1594,Bit_1595,Bit_1596,Bit_1597,Bit_1598,Bit_1599,Bit_1600,Bit_1601,Bit_1602,Bit_1603,Bit_1604,Bit_1605,Bit_1606,Bit_1607,Bit_1608,Bit_1609,Bit_1610,Bit_1611,Bit_1612,Bit_1613,Bit_1614,Bit_1615,Bit_1616,Bit_1617,Bit_1618,Bit_1619,Bit_1620,Bit_1621,Bit_1622,Bit_1623,Bit_1624,Bit_1625,Bit_1626,Bit_1627,Bit_1628,Bit_1629,Bit_1630,Bit_1631,Bit_1632,Bit_1633,Bit_1634,Bit_1635,Bit_1636,Bit_1637,Bit_1638,Bit_1639,Bit_1640,Bit_1641,Bit_1642,Bit_1643,Bit_1644,Bit_1645,Bit_1646,Bit_1647,Bit_1648,Bit_1649,Bit_1650,Bit_1651,Bit_1652,Bit_1653,Bit_1654,Bit_1655,Bit_1656,Bit_1657,Bit_1658,Bit_1659,Bit_1660,Bit_1661,Bit_1662,Bit_1663,Bit_1664,Bit_1665,Bit_1666,Bit_1667,Bit_1668,Bit_1669,Bit_1670,Bit_1671,Bit_1672,Bit_1673,Bit_1674,Bit_1675,Bit_1676,Bit_1677,Bit_1678,Bit_1679,Bit_1680,Bit_1681,Bit_1682,Bit_1683,Bit_1684,Bit_1685,Bit_1686,Bit_1687,Bit_1688,Bit_1689,Bit_1690,Bit_1691,Bit_1692,Bit_1693,Bit_1694,Bit_1695,Bit_1696,Bit_1697,Bit_1698,Bit_1699,Bit_1700,Bit_1701,Bit_1702,Bit_1703,Bit_1704,Bit_1705,Bit_1706,Bit_1707,Bit_1708,Bit_1709,Bit_1710,Bit_1711,Bit_1712,Bit_1713,Bit_1714,Bit_1715,Bit_1716,Bit_1717,Bit_1718,Bit_1719,Bit_1720,Bit_1721,Bit_1722,Bit_1723,Bit_1724,Bit_1725,Bit_1726,Bit_1727,Bit_1728,Bit_1729,Bit_1730,Bit_1731,Bit_1732,Bit_1733,Bit_1734,Bit_1735,Bit_1736,Bit_1737,Bit_1738,Bit_1739,Bit_1740,Bit_1741,Bit_1742,Bit_1743,Bit_1744,Bit_1745,Bit_1746,Bit_1747,Bit_1748,Bit_1749,Bit_1750,Bit_1751,Bit_1752,Bit_1753,Bit_1754,Bit_1755,Bit_1756,Bit_1757,Bit_1758,Bit_1759,Bit_1760,Bit_1761,Bit_1762,Bit_1763,Bit_1764,Bit_1765,Bit_1766,Bit_1767,Bit_1768,Bit_1769,Bit_1770,Bit_1771,Bit_1772,Bit_1773,Bit_1774,Bit_1775,Bit_1776,Bit_1777,Bit_1778,Bit_1779,Bit_1780,Bit_1781,Bit_1782,Bit_1783,Bit_1784,Bit_1785,Bit_1786,Bit_1787,Bit_1788,Bit_1789,Bit_1790,Bit_1791,Bit_1792,Bit_1793,Bit_1794,Bit_1795,Bit_1796,Bit_1797,Bit_1798,Bit_1799,Bit_1800,Bit_1801,Bit_1802,Bit_1803,Bit_1804,Bit_1805,Bit_1806,Bit_1807,Bit_1808,Bit_1809,Bit_1810,Bit_1811,Bit_1812,Bit_1813,Bit_1814,Bit_1815,Bit_1816,Bit_1817,Bit_1818,Bit_1819,Bit_1820,Bit_1821,Bit_1822,Bit_1823,Bit_1824,Bit_1825,Bit_1826,Bit_1827,Bit_1828,Bit_1829,Bit_1830,Bit_1831,Bit_1832,Bit_1833,Bit_1834,Bit_1835,Bit_1836,Bit_1837,Bit_1838,Bit_1839,Bit_1840,Bit_1841,Bit_1842,Bit_1843,Bit_1844,Bit_1845,Bit_1846,Bit_1847,Bit_1848,Bit_1849,Bit_1850,Bit_1851,Bit_1852,Bit_1853,Bit_1854,Bit_1855,Bit_1856,Bit_1857,Bit_1858,Bit_1859,Bit_1860,Bit_1861,Bit_1862,Bit_1863,Bit_1864,Bit_1865,Bit_1866,Bit_1867,Bit_1868,Bit_1869,Bit_1870,Bit_1871,Bit_1872,Bit_1873,Bit_1874,Bit_1875,Bit_1876,Bit_1877,Bit_1878,Bit_1879,Bit_1880,Bit_1881,Bit_1882,Bit_1883,Bit_1884,Bit_1885,Bit_1886,Bit_1887,Bit_1888,Bit_1889,Bit_1890,Bit_1891,Bit_1892,Bit_1893,Bit_1894,Bit_1895,Bit_1896,Bit_1897,Bit_1898,Bit_1899,Bit_1900,Bit_1901,Bit_1902,Bit_1903,Bit_1904,Bit_1905,Bit_1906,Bit_1907,Bit_1908,Bit_1909,Bit_1910,Bit_1911,Bit_1912,Bit_1913,Bit_1914,Bit_1915,Bit_1916,Bit_1917,Bit_1918,Bit_1919,Bit_1920,Bit_1921,Bit_1922,Bit_1923,Bit_1924,Bit_1925,Bit_1926,Bit_1927,Bit_1928,Bit_1929,Bit_1930,Bit_1931,Bit_1932,Bit_1933,Bit_1934,Bit_1935,Bit_1936,Bit_1937,Bit_1938,Bit_1939,Bit_1940,Bit_1941,Bit_1942,Bit_1943,Bit_1944,Bit_1945,Bit_1946,Bit_1947,Bit_1948,Bit_1949,Bit_1950,Bit_1951,Bit_1952,Bit_1953,Bit_1954,Bit_1955,Bit_1956,Bit_1957,Bit_1958,Bit_1959,Bit_1960,Bit_1961,Bit_1962,Bit_1963,Bit_1964,Bit_1965,Bit_1966,Bit_1967,Bit_1968,Bit_1969,Bit_1970,Bit_1971,Bit_1972,Bit_1973,Bit_1974,Bit_1975,Bit_1976,Bit_1977,Bit_1978,Bit_1979,Bit_1980,Bit_1981,Bit_1982,Bit_1983,Bit_1984,Bit_1985,Bit_1986,Bit_1987,Bit_1988,Bit_1989,Bit_1990,Bit_1991,Bit_1992,Bit_1993,Bit_1994,Bit_1995,Bit_1996,Bit_1997,Bit_1998,Bit_1999,Bit_2000,Bit_2001,Bit_2002,Bit_2003,Bit_2004,Bit_2005,Bit_2006,Bit_2007,Bit_2008,Bit_2009,Bit_2010,Bit_2011,Bit_2012,Bit_2013,Bit_2014,Bit_2015,Bit_2016,Bit_2017,Bit_2018,Bit_2019,Bit_2020,Bit_2021,Bit_2022,Bit_2023,Bit_2024,Bit_2025,Bit_2026,Bit_2027,Bit_2028,Bit_2029,Bit_2030,Bit_2031,Bit_2032,Bit_2033,Bit_2034,Bit_2035,Bit_2036,Bit_2037,Bit_2038,Bit_2039,Bit_2040,Bit_2041,Bit_2042,Bit_2043,Bit_2044,Bit_2045,Bit_2046,Bit_2047,Bit_2048 CHEMBL365134,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,1,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,1,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0 CHEMBL187579,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1,0,0,0,0,0,0,0,0,0,0,0,0,05.3.2 予測モデルの構築
いよいよ、生成した説明変数(および目的変数)のデータを読み込み、予測モデルを作成する。論文に従いランダムフォレストを用いた。GridSearchによるハイパーパラメータ探索や、5フォールドクロスバリデーション、評価指標もできるかぎり論文を再現したかったので、githubのソースを一部流用している。また、githubのコードでは、scikit-learnのpermutation_test_scoreメソッドでY-randamiztionを行っていたため、これも流用した。
create_model.pyimport argparse import csv import pandas as pd import numpy as np import gzip import _pickle as cPickle from sklearn import metrics from sklearn.ensemble import RandomForestClassifier from sklearn.feature_selection import VarianceThreshold from sklearn.model_selection import train_test_split, cross_validate, GridSearchCV from sklearn.model_selection import permutation_test_score, StratifiedKFold def calc_metrics_derived_from_confusion_matrix(metrics_name, y_true, y_predict): tn, fp, fn, tp = metrics.confusion_matrix(y_true, y_predict).ravel() # PPV, precision # TP / TP + FP if metrics_name in ["PPV", "precision"]: return tp / (tp + fp) # NPV # TN / TN + FN if metrics_name in ["NPV"]: return tn / (tn + fn) # sensitivity, recall, TPR # TP / TP + FN if metrics_name in ["sensitivity", "recall", "TPR"]: return tp / (tp + fn) # specificity # TN / TN + FP if metrics_name in ["specificity"]: return tn / (tn + fp) def calc_metrics(metrics_name, y_true, y_predict): if metrics_name == "accuracy": return metrics.accuracy_score(y_true, y_predict) if metrics_name == "ba": return metrics.balanced_accuracy_score(y_true, y_predict) if metrics_name == "roc_auc": return metrics.roc_auc_score(y_true, y_predict) if metrics_name == "kappa": return metrics.cohen_kappa_score(y_true, y_predict) if metrics_name == "mcc": return metrics.matthews_corrcoef(y_true, y_predict) if metrics_name == "precision": return metrics.precision_score(y_true, y_predict) if metrics_name == "recall": return metrics.recall_score(y_true, y_predict) def main(): parser = argparse.ArgumentParser() parser.add_argument("-input", type=str, required=True) parser.add_argument("-output_model", type=str, required=True) parser.add_argument("-output_report", type=str, required=True) args = parser.parse_args() df = pd.read_csv(args.input, index_col=0) print(df.shape) y_train = df['outcome'].to_numpy() print(y_train) X_train = df.iloc[:, 1:] print(y_train.shape) print(X_train.shape) # Number of trees in random forest n_estimators = [100, 250, 500, 750, 1000] max_features = ['auto', 'sqrt'] criterion = ['gini', 'entropy'] class_weight = [None,'balanced', {0:.9, 1:.1}, {0:.8, 1:.2}, {0:.7, 1:.3}, {0:.6, 1:.4}, {0:.4, 1:.6}, {0:.3, 1:.7}, {0:.2, 1:.8}, {0:.1, 1:.9}] random_state = [24] # Create the random grid param_grid = {'n_estimators': n_estimators, 'max_features': max_features, 'criterion': criterion, 'random_state': random_state, 'class_weight': class_weight} # setup model building rf = GridSearchCV(RandomForestClassifier(), param_grid, n_jobs=-1, cv=5, verbose=1) rf.fit(X_train, y_train) print() print('Best params: %s' % rf.best_params_) print('Score: %.2f' % rf.best_score_) rf_best = RandomForestClassifier(**rf.best_params_, n_jobs=-1) rf_best.fit(X_train, y_train) #一旦できたモデルを保存 with gzip.GzipFile(args.output_model, 'w') as f: cPickle.dump(rf_best, f) with gzip.GzipFile(args.output_model, 'r') as f: rf_best = cPickle.load(f) # Params pred = [] ad = [] pred_proba = [] index = [] cross_val = StratifiedKFold(n_splits=5) # Do 5-fold loop for train_index, test_index in cross_val.split(X_train, y_train): fold_model = rf_best.fit(X_train.iloc[train_index], y_train[train_index]) fold_pred = rf_best.predict(X_train.iloc[test_index]) fold_ad = rf_best.predict_proba(X_train.iloc[test_index]) pred.append(fold_pred) ad.append(fold_ad) pred_proba.append(fold_ad[:, 1]) index.append(test_index) threshold_ad = 0.70 # Prepare results to export fold_index = np.concatenate(index) fold_pred = np.concatenate(pred) fold_ad = np.concatenate(ad) fold_pred_proba = np.concatenate(pred_proba) fold_ad = (np.amax(fold_ad, axis=1) >= threshold_ad).astype(str) five_fold_morgan = pd.DataFrame({'Prediction': fold_pred, 'AD': fold_ad, 'Proba': fold_pred_proba}, index=list(fold_index)) five_fold_morgan.AD[five_fold_morgan.AD == 'False'] = np.nan five_fold_morgan.AD[five_fold_morgan.AD == 'True'] = five_fold_morgan.Prediction five_fold_morgan.sort_index(inplace=True) five_fold_morgan['y_train'] = pd.DataFrame(y_train) # morgan stats all_datas = [] datas = [] datas.append(calc_metrics("accuracy", five_fold_morgan['y_train'], five_fold_morgan['Prediction'])) datas.append(calc_metrics("ba", five_fold_morgan['y_train'], five_fold_morgan['Prediction'])) datas.append(calc_metrics("roc_auc", five_fold_morgan['y_train'], five_fold_morgan['Proba'])) datas.append(calc_metrics("kappa", five_fold_morgan['y_train'], five_fold_morgan['Prediction'])) datas.append(calc_metrics("mcc", five_fold_morgan['y_train'], five_fold_morgan['Prediction'])) datas.append(calc_metrics("precision", five_fold_morgan['y_train'], five_fold_morgan['Prediction'])) datas.append(calc_metrics("recall", five_fold_morgan['y_train'], five_fold_morgan['Prediction'])) datas.append(calc_metrics_derived_from_confusion_matrix("sensitivity", five_fold_morgan['y_train'], five_fold_morgan['Prediction'])) datas.append(calc_metrics_derived_from_confusion_matrix("PPV", five_fold_morgan['y_train'], five_fold_morgan['Prediction'])) datas.append(calc_metrics_derived_from_confusion_matrix("specificity", five_fold_morgan['y_train'], five_fold_morgan['Prediction'])) datas.append(calc_metrics_derived_from_confusion_matrix("NPV", five_fold_morgan['y_train'], five_fold_morgan['Prediction'])) datas.append(1) all_datas.append(datas) # morgan AD stats morgan_ad = five_fold_morgan.dropna(subset=['AD']) morgan_ad['AD'] = morgan_ad['AD'].astype(int) coverage_morgan_ad = len(morgan_ad['AD']) / len(five_fold_morgan['y_train']) datas = [] datas.append(calc_metrics("accuracy", morgan_ad['y_train'], morgan_ad['AD'])) datas.append(calc_metrics("ba", morgan_ad['y_train'], morgan_ad['AD'])) datas.append(calc_metrics("roc_auc", morgan_ad['y_train'], morgan_ad['Proba'])) datas.append(calc_metrics("kappa", morgan_ad['y_train'], morgan_ad['AD'])) datas.append(calc_metrics("mcc", morgan_ad['y_train'], morgan_ad['AD'])) datas.append(calc_metrics("precision", morgan_ad['y_train'], morgan_ad['AD'])) datas.append(calc_metrics("recall", morgan_ad['y_train'], morgan_ad['AD'])) datas.append(calc_metrics_derived_from_confusion_matrix("sensitivity", morgan_ad['y_train'], morgan_ad['AD'])) datas.append(calc_metrics_derived_from_confusion_matrix("PPV", morgan_ad['y_train'], morgan_ad['AD'])) datas.append(calc_metrics_derived_from_confusion_matrix("specificity", morgan_ad['y_train'], morgan_ad['AD'])) datas.append(calc_metrics_derived_from_confusion_matrix("NPV", morgan_ad['y_train'], morgan_ad['AD'])) datas.append(coverage_morgan_ad) all_datas.append(datas) # print stats print('\033[1m' + '5-fold External Cross Validation Statistical Characteristcs of QSAR models developed morgan' + '\n' + '\033[0m') morgan_5f_stats = pd.DataFrame(all_datas, columns=["accuracy", "ba", "roc_auc", "kappa", "mcc", "precision", "recall", "sensitivity", "PPV", "specificity", "NPV", "Coverage"]) morgan_5f_stats.to_csv(args.output_report) print(morgan_5f_stats) # Y ramdomaization permutations = 20 score, permutation_scores, pvalue = permutation_test_score(rf_best, X_train, y_train, cv=5, scoring='balanced_accuracy', n_permutations=permutations, n_jobs=-1, verbose=1, random_state=24) print('True score = ', score.round(2), '\nY-randomization = ', np.mean(permutation_scores).round(2), '\np-value = ', pvalue.round(4)) if __name__ == "__main__": main()これを実行すると
output_modelで指定したパスに予測モデル本体が保存され、output_reportで指定したパスに構築された予測モデルの評価指標が出力される。その結果は次の通りとなった。accuracy balanced_accuracy roc_auc kappa mcc precision recall sensitivity PPV specificity NPV Coverage 0.801980 0.713126 0.807507 0.490156 0.551662 0.9375 0.441176 0.441176 0.9375 0.985075 0.776471 1.000000 0.852941 0.736842 0.812030 0.564661 0.627215 1.0000 0.473684 0.473684 1.0000 1.000000 0.830508 0.673267 True score = 0.71 Y-randomization = 0.48 p-value = 0.0476前半の1行目のスコアは、全学習データにおけるクロスバリデーションのスコアである。recallとsensitivityは同じことだが、scikit-learn版と自前で組み込んだ関数が一致しているかの比較のため、両方表示している。
前半の2行目は、論文の中で考慮されているものであり、予測信頼度(予測したクラスに対するscikit-learnのpredict_probaで得られる値といえばわかるだろうか)がしきい値(0.7)以上のデータに限定したクロスバリデーションのスコアであり、より信頼度の高いデータだけによる指標と考えられ、多少指標がよくなっている。
後半は、Y-randamizationの結果であり、scikit-learnのドキュメントによると、True scoreはターゲットを入れ替えない真のスコア、Y-randamizationはターゲットをランダマイズしたスコア、 p-valueはスコアが偶然に得られる確率であり、最良は
1 /(n_permutations + 1)で最悪は1.0らしい。今回、1 /(n_permutations + 1)= 1 / (20 + 1) = 0.0467であり、最良の値になっているため、0.71 というbalanced_accuracyは偶然に得られたものではなさそうだ。5.4 予測モデルを利用した候補薬物のスクリーニング
5.4.1 DrugBankデータに対する予測の実行
5.3で作成した予測モデル本体と、ChEMBL、PDBから抽出した学習データ(101件)、DrugBankから抽出したバーチャルスクリーニング用データ(10750件)をそれぞれ以下のスクリプト(断片のみ記載)に指定して、予測を行う。
predict_drugbank.pyparser = argparse.ArgumentParser() parser.add_argument("-input_train", type=str, required=True) parser.add_argument("-input_predict", type=str, required=True) parser.add_argument("-input_model", type=str, required=True) parser.add_argument("-output_csv", type=str, required=True) parser.add_argument("-output_report", type=str, required=True) args = parser.parse_args() #学習データ読み込み trains = list() with open(args.input_train, "r") as f: reader = csv.DictReader(f) for i, row in enumerate(reader): smiles = row["canonical_smiles"] new_smiles = normalize(smiles) trains.append((row["id"], new_smiles)) #予測データの読み込み smiles_list = list() datas = [] datas_other = [] with open(args.input_predict, "r") as f: reader = csv.DictReader(f) for row in reader: print(row["id"]) smiles = row["smiles"] new_smiles = normalize(smiles) dup_pred = None dup_train = None if new_smiles is None or Chem.MolFromSmiles(new_smiles) is None: print(f"error! {id}") new_smiles = smiles existFlag = False for db_id, tmp_smiles in smiles_list: if new_smiles == tmp_smiles: dup_pred = db_id print(f"{id}: same structure exist in predict data! - {db_id}, {tmp_smiles}") break for db_id, tmp_smiles in trains: if new_smiles == tmp_smiles: dup_train = db_id print(f"{id}: same structure exist in train data! - {db_id}, {tmp_smiles}") break smiles_list.append((row["id"], new_smiles)) mol = Chem.MolFromSmiles(new_smiles) fp = AllChem.GetMorganFingerprintAsBitVect(mol, radius=3, nBits=2048, useFeatures=False, useChirality=False) fp = pd.Series(np.asarray(fp)).values data = [] data.append(row["id"]) data.extend(fp) datas.append(data) datas_other.append((row["id"], row["name"], row["status"], dup_pred, dup_train)) columns = list() columns.append("id") columns.extend(["Bit_" + str(i+1) for i in range(2048)]) df = pd.DataFrame(data=datas, columns=columns) df.set_index("id", inplace=True, drop=True) df_other = pd.DataFrame(data=datas_other, columns=["id", "name", "status", "dup_predict", "dup_train"]) df_other.set_index("id", inplace=True, drop=True) #一旦保存 df.to_csv(args.output_csv) #df = pd.read_csv(args.input, index_col=0) X_vs = df.iloc[:, 0:] with gzip.GzipFile(args.input_model, 'r') as f: model = cPickle.load(f) ad_threshold = 0.70 y_pred = model.predict(X_vs) confidence = model.predict_proba(X_vs) confidence = np.amax(confidence, axis=1).round(2) ad = confidence >= ad_threshold pred = pd.DataFrame({'Prediction': y_pred, 'AD': ad, 'Confidence': confidence}, index=None) pred.AD[pred.AD == False] = np.nan pred.AD[pred.AD == True] = pred.Prediction.astype(int) pred_ad = pred.dropna().astype(int) coverage_ad = len(pred_ad) * 100 / len(pred) print('VS pred: %s' % pred.Prediction) print('VS pred AD: %s' % pred_ad.Prediction) print('Coverage of AD: %.2f%%' % coverage_ad) pred.index = X_vs.index predictions = pd.concat([df_other, pred], axis=1) for col in ['Prediction', 'AD']: predictions[col].replace(0, 'Inactive', inplace=True) predictions[col].replace(1, 'Active', inplace=True) print(predictions.head()) predictions.to_csv(args.output_report)
output_csvには予測データの説明変数の計算結果が出力される。また、output_reportにはDrugBankの各薬剤のDrugBank ID, Common Name, 市販/中止/実験等の種別、予測結果、しきい値を超える予測信頼度に対する予測結果、予測信頼度等が出力される。また、outptu_reportには、各予測データにおいて、同じ予測データの中で重複する構造があった場合および学習データに対して重複する構造があった場合、それぞれ重複したIDを出力するようにした。予測データの中で321件、学習データに対して6件、重複するデータ存在した。5.4.2 論文との比較
論文では、DrugBankに対するバーチャルスクリーニング結果は以下に記載されている。
https://github.com/alvesvm/sars-cov-mpro/tree/master/mol-inf-2020/datasets/screened_compounds
drugbank_hits_qsar_consensus.xlsx
ここに出力されているDrugBankの薬剤およびその結果と、前項で出力した予測結果を比較した。論文では、SiRIMS、Dragon, そして今回作成したRDKitのMorganの3パターンの記述子計算による予測モデルを作成し、そのうち複数のモデルがActiveとしたものを最終的なActiveとしている。
全体的な比較と、SiRIMS、Dragonそれぞれに対する比較をしてみた。
なお、github上のMorganの結果は気になる点を見つけたため、これとの比較は見送っている。全体的な結果との比較
論文では41件となっていたが、github上のエクセルでは最終的なActive件数は51件となっていた。
その51件に対し、今回作成したモデルがActiveとしたものは、19件であった。SiRIMSモデル、Dragonモデルとの比較
SiRIMSモデル、Dragonモデルと、Active/Inactive件数の比較は以下の通りである。
MODEL ACTIVE INACITVE TOTAL SiRMS 309 9305 9614 DRAGON 864 8750 9614 作成モデル 314 9300 9614 つづいてSiRMISモデル、DRragonモデルそれぞれと共通のActive数を比較してみた。
MODEL SiRMS DRAGON 作成モデル SiRMS - 39 79 DRAGON 39 - 35 作成モデル 79 35 - Active数はDRAGONが最も多いにもかかわらず、今回の作成したモデルと一致したActive件数は、SiRMSが79件でありDragonの倍になっている。このことから今回の作成モデルはDragonよりはSiRMSに類似したモデルと考えられる。
5.4.3 おまけ: 予測モデルの適用範囲の可視化
論文には記載がなかったものの、学習データ上に予測対象のデータがどのように分布するかを可視化してみた。機械学習では学習データと似ていないデータの場合、予測の信頼性が問題となるからである。方法としてはまず、学習データで次元削減モデルを作成し、そのモデルを用いて予測対象データの2次元座標を生成しプロットしてみた。
次元削減手法はPCAとUMAPを試した。データは作成モデルと同様、Morganフィンガープリントを利用した。
青が学習データ、赤が予測データである。〇がActive、XがInactiveになる。学習データで〇とXが重なっているところは、UMAPの方がうまく分離できているように見える。可視化したスクリプトのソースは以下の通りとなる。学習データでPCAまたはUMAPのモデルをfitで作成し、それに対し各予測データをtransformした結果を表示している。図をみると予測対象データは、学習データの範囲から大きく逸脱していないように見えるが、学習データから構成されるデータのみで次元削減していることも影響しているかもしれない。
view_ad.pyparser = argparse.ArgumentParser() parser.add_argument("-train", type=str, required=True) parser.add_argument("-predict", type=str) parser.add_argument("-result", type=str) parser.add_argument("-method", type=str, default="PCA", choices=["PCA", "UMAP"]) args = parser.parse_args() # all_train.csvの読み込み, fpの計算 train_datas = [] train_datas_active = [] train_datas_inactive = [] with open(args.train, "r") as f: reader = csv.DictReader(f) for row in reader: smiles = row["canonical_smiles"] mol = Chem.MolFromSmiles(smiles) fp = AllChem.GetMorganFingerprintAsBitVect(mol, radius=3, nBits=2048, useFeatures=False, useChirality=False) train_datas.append(fp) if int(row["outcome"]) == 1: train_datas_active.append(fp) else: train_datas_inactive.append(fp) if args.predict and args.result: result_outcomes = [] result_ads = [] # 予測結果読み込み with open(args.result, "r",encoding="utf-8_sig") as f: reader = csv.DictReader(f) for i, row in enumerate(reader): #print(row) if row["Prediction"] == "Active": result_outcomes.append(1) else: result_outcomes.append(0) result_ads.append(row["Confidence"]) # drugbank.csvの読み込み, fpの計算 predict_datas = [] predict_datas_active = [] predict_datas_inactive = [] predict_ads = [] with open(args.predict, "r") as f: reader = csv.DictReader(f) for i, row in enumerate(reader): print(i) smiles = row["smiles"] mol = Chem.MolFromSmiles(smiles) fp = AllChem.GetMorganFingerprintAsBitVect(mol, radius=3, nBits=2048, useFeatures=False, useChirality=False) predict_datas.append(fp) if result_outcomes[i] == 1: predict_datas_active.append(fp) else: predict_datas_inactive.append(fp) # 分析 model = None if args.method == "PCA": model = PCA(n_components=2) model.fit(train_datas) if args.method == "UMAP": model = umap.UMAP() model.fit(train_datas) result_train = model.transform(train_datas) result_train_active = model.transform(train_datas_active) result_train_inactive = model.transform(train_datas_inactive) plt.title(args.method) #plt.scatter(result_train[:, 0], result_train[:, 1], c="blue", alpha=0.1, marker="o") plt.scatter(result_train_active[:, 0], result_train_active[:, 1], c="blue", alpha=0.5, marker="o") plt.scatter(result_train_inactive[:, 0], result_train_inactive[:, 1], c="blue", alpha=0.5, marker="x") # 予測(predict) if args.predict and args.result: result_predict = model.transform(predict_datas) result_predict_active = model.transform(predict_datas_active) result_predict_inactive = model.transform(predict_datas_inactive) #plt.scatter(result_predict[:, 0], result_predict[:, 1], c=result_ads, alpha=0.1, cmap='viridis_r') plt.scatter(result_predict_active[:, 0], result_predict_active[:, 1], c="red", alpha=0.1, marker="o") plt.scatter(result_predict_inactive[:, 0], result_predict_inactive[:, 1], c="red", alpha=0.1, marker="x") plt.show()6. おわりに
6.1 感想
今回、論文の内容を試した過程を通じて、以下のことを得ることができた。今後自ら解析を行ったり、別の論文を読んだりする場合に多いに役立つと思う。
- 創薬分野におけるデータ収集から前処理、予測モデル作成、予測モデルの評価までを一通り体験することができた。特に予測モデルを作成する前のデータの収集や前処理、予測モデルを作成した後の適用・評価がいかに手間がかかるのかか身をもって体験することができた。
- 公共データべースについて、概要やデータ収集方法などの理解を深めることができた。
今回は従来手法による論文を試したが、Graph Convolution Networks(GCN) などDeep Learning を活用した論文も続々と出てきており、近いうちにそれらも試してみたい。
6.2 今回の論文に関連したその他のお勧めの論文
論文を探す過程で、他に見つかったコロナウィルス関連の予測モデルに関する論文を以下に2点あげる。いずれも、データセットが公開されているため試してみるにはよいと思う。
この論文では、本記事の論文とは異なるpapain-like protease (PLpro)いうターゲットに対する予測モデルを作成している。
これは、本記事の論文と同じメインプロテアーゼに対する予測モデルを作成している。OECD ガイドライン(https://www.oecd.org/chemicalsafety/risk-assessment/37849783.pdf )に沿ってモデルが評価されており、化合物を対象とした予測モデルの評価に関する知見を得ることができそうである。
6.3 本記事で利用したライブラリ
本記事で利用した主なライブラリは以下のとおりである。
- Python 3.7
- RDkit 2020.03.5
- scikit-learn 0.23.2
- MolVS 0.1.1
6.5 本記事のプログラムについて
本記事に掲載したプログラムは https://github.com/kimisyo/sars_trace_1 にて公開している。