- 投稿日:2020-02-28T23:58:28+09:00
AWS EC2でMemoryErrorとNo space left on deviceで詰まった話
対処
MemoryError →
--no-cache-dirをつければ通る。
No space left on device → ファイル容量を減らせ!以下余談
MemoryErrorの対処
AWSのEC2でpipからtensorflowを入れようとしたところ。。。
pip install tensorflow ~ 真っ赤なエラー... ~ MemoryErrorMemoryErrorで入れることができない。。。
そんな時は--no-cache-dirを付けてキャッシュを無効化すればいい!ということで再度実行。pip install tensorflow --no-cache-dir ~ やっぱりエラー... ~ ERROR: Could not install packages due to an EnvironmentError: [Errno 28] No space left on deviceエラーが
[Errno 28] No space left on deviceに変わった。こいつの原因はなんだ?No space left on deviceの対処
容量を確認してみる
どうやら容量が足りないらしい。そこで
df -hで確認してみる。ファイルシス サイズ 使用 残り 使用% マウント位置 devtmpfs 475M 0 475M 0% /dev tmpfs 492M 0 492M 0% /dev/shm tmpfs 492M 412K 492M 1% /run tmpfs 492M 0 492M 0% /sys/fs/cgroup /dev/xvda1 8.0G 5.0G 3.1G 63% / tmpfs 99M 0 99M 0% /run/user/1000そんなに圧迫してるんか...?他に原因がないらしいのでとりあえず使用率30%くらいまで減らしてみる。
find . -xdev -type f | cut -d "/" -f 2 | sort | uniq -c | sort -nr
余談:このコマンド叩けば各ファイルの容量がわかる。
とりあえずcloneしたレポジトリと、pipの中身を消して容量を無理やり減らして再実行!したら通りました。めでたしめでたし
- 投稿日:2020-02-28T22:33:06+09:00
1日10時間株価予測AIを作り続ける 4か月目
4記事目なので過去の記事も参照ください
3か月目当初の目的を考える
このプロジェクトの元々の目的は、深層学習を使って株式市場またはもっと広義に金融市場で収益を上げることでした。その目的を達成するためには以下に示すように関係するいくつかの要素があり、収益可能性はそれらの要素を少しずつまた一つずつ向上させることによって上昇していくと考えました。今回は最初にそれらの要素をここに記し、どのような方法を取ってきたか、またそれらを改良するためにどのような計画が立てられるかについて記したいと思います。
1. 学習データの量と質
2. 学習データに対する深層学習の”層“の適応度
3. 完成したAIの運用方法1. 学習データの量と質
より良い結果を出すには、過学習を防ぐ必要がある。過学習を防ぐには主に2つの方法があり、一つにはデータの量を増やすこと、もう一つは正則化を導入することであるが後者は2.学習データに対する深層学習の”層”の適応度に関するものである。
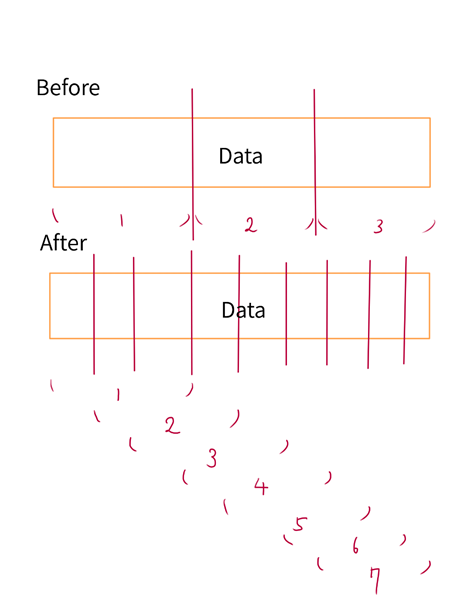
データの量を増やすことに関して難しい面もある。単純に色々な株価データを学習データに追加することでその量を増やすことはできるが、米国株、日本株その他のデータをごちゃまぜにしたデータを学ばせるのは賛否両論がある上に経験上あまり結果が良くない。一方で、データの種類の範囲を一部の種類(例えば日本株)のみに絞るまたはETFのみに絞ると、今度はデータの量が足りずに困る。学習データの多様化を防ぎながらデータ量を増やす方法はあるだろうか。この図を見るとわかるように今までは元のデータを単に分割して学習データにしていたため、学習データの量に限界があった。一方で新しい方法を使えば、一つの元データからより多くの学習データを取り出し、適宜学習データ量を増やすことができる。尚、画像で抽象的に示した”Data”とは、具体的には例えば次のような画像データである。
この方法にはもう一ついい点がある。学習データの質を向上させるために価格変動が少なかったデータを除外することができるが、それによってデータ量が減るデメリットを緩和することができる。
今後は、データの種類を絞り、かつ学習に不要と思われるデータを適宜排除しつつ、データ量は上記の方法で増やしデータの可能性を最大限に引き出すことを目標にします。2. 学習データに対する深層学習の”層“の適応度
この部分が一番難しいかつ勉強する価値があると思います。
最初はどこかから拾ってきたSequential modelを訳もわからず使って学習させていましたが、それでは限界があったので背景にある理論について少し学びました。
おそらく一番重要なのはレイヤーの基本的な順番でそれらは以下のようになっています。
- Convolution
- Relu
- Pooling
- Flatten
- Fully Connected(Dense)
- Softmaxまた、Convolution, ReluそしてPoolingはひとまとまりにして繰り返すことが可能です。理由がわかりませんが、以下のようにこれらを繰り返すと結果がよくなる傾向にあります。
未だわからないことが多くめちゃくちゃなSequential modelを使っている可能性がありますのでご注意ください。
今後、レイヤーに係る理論をなるべく深く理解し、より目的にあった効率的なSequential modelを自分で構築できるように頑張ります。3. 完成したAIの運用方法
今まで実用的な運用、バックテストをサボっていたのでここがあまり進んでいません。
AIによる民主主義的意思決定を導入し、AIの判断の信憑性と精確性をある程度確保する方法を考案しましたがどのタイミングでAIを起動するか(起動したタイミングで株価チャートを読み込み判断を下す)、またその結果どのような売買行動を起こさせるかということが収益に大きく影響すると思われます。今回、コロナウィルスの影響で株価が暴落していたので直近で以下の範囲の価格チャートを3人のAIに見せ、多数決によってどのような結果が出るのか見てみました。
3分の2のAIが上がると予測し、結果は上がるでした。来週、株式市場はどうなってしまうのでしょうか。
- 投稿日:2020-02-28T22:28:48+09:00
【PyTorchチュートリアル②】Autograd: 自動微分
はじめに
前回に引き続き、PyTorch 公式チュートリアル の第2弾です。
今回は Autograd: Automatic Differentiation を進めてみたいと思います。目次
1.Autograd(自動微分)
2.Tensor
3.勾配
4.You can do many crazy things with autograd!
5.最後に
履歴1.Autograd(自動微分)
PyTorch には Autograd(自動微分)の機能が実装されています。
Tensor に勾配情報を保持しておき、定義した計算グラフ(式)に対して backward() メソッドで勾配が計算されます。
以下で具体例をあげながら Autograd について見ていきましょう。2.Tensor
PyTorch の Tensor は、requires_grad 属性を True にすることで勾配が記録されるようになります。
backward() で勾配を計算すると、Tensor の grad 属性に勾配が保持されます。以下の記述で Tensor を定義します。
requires_grad=True を指定し、勾配が記録されるようにします。import torch x = torch.ones(2, 2, requires_grad=True) print(x)tensor([[1., 1.], [1., 1.]], requires_grad=True)計算グラフ(式) y を作成します。
y = x + 2 print(y)tensor([[3., 3.], [3., 3.]], grad_fn=<AddBackward0>)print すると、出力に grad_fn があります。
これは、勾配を計算するための計算グラフが構築されていることを示しています。print(y.grad_fn)<AddBackward0 object at 0x7f8cc977e5c0>y を利用してさらに計算グラフ(式) z、 out を作成します。
z = y * y * 3 out = z.mean() print(z, out)tensor([[27., 27.], [27., 27.]], grad_fn=<MulBackward0>) tensor(27., grad_fn=<MeanBackward0>)[参考情報]
tensor.requires_grad_() で requires_grad 属性を変更することができます。a = torch.randn(2, 2) a = ((a * 3) / (a - 1)) print(a.requires_grad) a.requires_grad_(True) print(a.requires_grad) b = (a * a).sum() print(b.grad_fn)False True <SumBackward0 object at 0x7fcb2ba0a3c8>3.勾配
out.backward() で勾配を計算します。
out.backward()out の x による偏微分、d(out)/dx を出力します。
print(x.grad)tensor([[4.5000, 4.5000], [4.5000, 4.5000]])out は out = z.mean() 、 z は z = y * y * 3 ですので、以下の式になります。
out = \frac{1}{4}\sum_{i=1}^{4}z_i 、 z_i = 3(x_i + 2)^2 、 z_i\bigr\rvert_{x_i=1} = 27従って out を x で偏微分すると
\begin{align} \frac{\partial out}{\partial x_i} &= \frac{3}{2}(x_i+2)\\ &= \frac{9}{2} = 4.5 \end{align}になり、自動微分されていることが分かります。
4.You can do many crazy things with autograd!
以下のコードの意味するところがよく分からないのですが、見ていきます。
x = torch.randn(3, requires_grad=True) y = x * 2 while y.data.norm() < 1000: y = y * 2 print(y)tensor([ -492.4446, -1700.8485, -339.7951], grad_fn=<MulBackward0>)x は標準化(平均0、標準偏差1)されたランダム値です。
y.data.norm() はウィキペディアにも説明がありますが、ベクトル空間における距離のことで、以下のユークリッドノルムのことです。ユークリッドノルム = \sqrt{|x_1|^2+\cdots+|x_n|^2} \\2次元の場合、2点間の距離と同じ式になりますので、まさに距離のことです。
実際、x の値と norm() を出力してみると、上記の計算式の結果がノルムとして返却されます。\begin{eqnarray} ユークリッドノルム &=& \sqrt{|-0.9618|^2+|-3.3220|^2+|-0.6637|^2}\\ &=& 3.5215 \end{eqnarray}print(x) print(x.data.norm())tensor([-0.9618, -3.3220, -0.6637], requires_grad=True) tensor(3.5215)ですので、上記のコードは、x のノルムが 1,000 になるまで x を2倍し続けるという式を表します。
y の勾配を y.backward() で計算したいのですが、y はスカラーではないため、そのまま計算することができません。
実際、y.backward() を実行するとエラーが発生します。y.backward()RuntimeError: grad can be implicitly created only for scalar outputs適当なベクトルを設定することで、勾配が計算されます。
gradients = torch.tensor([0.1, 1.0, 0.0001], dtype=torch.float) y.backward(gradients) print(x.grad)tensor([5.1200e+01, 5.1200e+02, 5.1200e-02])このチュートリアルの意味するところを考えてみます。
このチュートリアルにも記載されていますが、勾配はヤコビ行列で表すことができます。\begin{split}J=\left(\begin{array}{ccc} \frac{\partial y_{1}}{\partial x_{1}} & \cdots & \frac{\partial y_{1}}{\partial x_{n}}\\ \vdots & \ddots & \vdots\\ \frac{\partial y_{m}}{\partial x_{1}} & \cdots & \frac{\partial y_{m}}{\partial x_{n}} \end{array}\right)\end{split}また、autograd はヤコビ行列と与えられたベクトルの積を計算するためのエンジンであるとの記載もあります。
\begin{split}J^{T}\cdot v=\left(\begin{array}{ccc} \frac{\partial y_{1}}{\partial x_{1}} & \cdots & \frac{\partial y_{m}}{\partial x_{1}}\\ \vdots & \ddots & \vdots\\ \frac{\partial y_{1}}{\partial x_{n}} & \cdots & \frac{\partial y_{m}}{\partial x_{n}} \end{array}\right)\left(\begin{array}{c} \frac{\partial l}{\partial y_{1}}\\ \vdots\\ \frac{\partial l}{\partial y_{m}} \end{array}\right)=\left(\begin{array}{c} \frac{\partial l}{\partial x_{1}}\\ \vdots\\ \frac{\partial l}{\partial x_{n}} \end{array}\right)\end{split}この情報をもとに、今回のケースに当てはめてみます。
ここからは、自分の想像も入っているので正しくないかもしれません。x = torch.randn(3, requires_grad=True)x はランダムの3変数ですので x の添え字 n は 3 になります。
x_1 , x_2 , x_3y を考えます。
y の定義は以下です。y = x * 2 while y.data.norm() < 1000: y = y * 2まず、最初の y = x * 2 を見てみます。
変数の数に注目すると、y は x * 2 で、単純に2倍するだけですので、変数の数は変わりません。
ですので、y で変換後も 値は 3つのままです。
従って m も 3 になり、ヤコビ行列のサイズは 3 × 3 です。{\begin{split}J=\left(\begin{array}{ccc} \frac{\partial y_{1}}{\partial x_{1}} & \frac{\partial y_{1}}{\partial x_{2}} & \frac{\partial y_{1}}{\partial x_{3}}\\ \frac{\partial y_{2}}{\partial x_{1}} & \frac{\partial y_{2}}{\partial x_{2}} & \frac{\partial y_{2}}{\partial x_{3}}\\ \frac{\partial y_{3}}{\partial x_{1}} & \frac{\partial y_{3}}{\partial x_{2}} & \frac{\partial y_{3}}{\partial x_{3}}\\ \end{array}\right)\end{split} }上の print(x)の値 [-0.9618, -3.3220, -0.6637] が [x1, x2, x3 ] に当てはまります。
また、y = x * 2 にこの数値を当てはめると [-1.9236, -6.644, -1.3274] になり、これが [y1, y2, y3] になります。
数値を当てはめるまでもありませんでしたが、x と y の変換式を書くと以下になります。y_1 = 2x_1\\ y_2 = 2x_2\\ y_3 = 2x_3\\この式を、それぞれ x1, x2, x3 で偏微分すると以下のようになります。
\frac{\partial y_{1}}{\partial x_{1}} = 2 , \frac{\partial y_{1}}{\partial x_{2}} = 0 , \frac{\partial y_{1}}{\partial x_{3}} = 0\\ \frac{\partial y_{2}}{\partial x_{1}} = 0 , \frac{\partial y_{2}}{\partial x_{2}} = 2 , \frac{\partial y_{2}}{\partial x_{3}} = 0\\ \frac{\partial y_{3}}{\partial x_{1}} = 0 , \frac{\partial y_{3}}{\partial x_{2}} = 0 , \frac{\partial y_{3}}{\partial x_{3}} = 2\\従ってヤコビ行列は以下になります。
while 前の1回目の変換(y = x * 2)を表すため、J1 としました。{\begin{split}J_1=\left(\begin{array}{ccc} 2 & 0 & 0\\ 0 & 2 & 0\\ 0 & 0 & 2\\ \end{array}\right)\end{split} }2回目の y を考えます。
while の後の y = y * 2 が2回目の y です。
式が1回目と同じですので、2回目の y のヤコビ行列も先ほどと同じで以下になります。
2回目の y のヤコビ行列を表すため、J2 とします。{\begin{split}J_2=\left(\begin{array}{ccc} 2 & 0 & 0\\ 0 & 2 & 0\\ 0 & 0 & 2\\ \end{array}\right)\end{split} }これを繰り返します。
初期値である x.data.norm() が「3.5215」で、y.data.norm() < 1000 でループさせるため、ループは8回実行され、y は9回定義されます。
全体としては、以下のようになります。
式 x1の値 x2の値 x3の値 - x1 x2 x3 1回目のyで変換 2 * x1 2 * x2 2 * x3 2回目のyで変換 4 * x1 4 * x2 4 * x3 3回目のyで変換 8 * x1 8 * x2 8 * x3 4回目のyで変換 16 * x1 16 * x2 16 * x3 5回目のyで変換 32 * x1 32 * x2 32 * x3 6回目のyで変換 64 * x1 64 * x2 64 * x3 7回目のyで変換 128 * x1 128 * x2 128 * x3 8回目のyで変換 256 * x1 256 * x2 256 * x3 9回目のyで変換 512 * x1 512 * x2 512 * x3 最終的に y はこの9個の変換の合成関数になります。
こちらの数学サイト にも記載がありますが、合成関数のヤコビ行列は行列の式で表せるので
y 全体のヤコビ行列 J は\begin{eqnarray} J &=& J_9 \times J_8 \times J_7 \times J_6 \times J_5 \times J_4 \times J_3 \times J_2 \times J_1\\\\ &=& \left( \begin{array}{ccc} 2 & 0 & 0 \\ 0 & 2 & 0 \\ 0 & 0 & 2 \end{array} \right) \left( \begin{array}{ccc} 2 & 0 & 0 \\ 0 & 2 & 0 \\ 0 & 0 & 2 \end{array} \right) \cdots \left( \begin{array}{ccc} 2 & 0 & 0 \\ 0 & 2 & 0 \\ 0 & 0 & 2 \end{array} \right) \left( \begin{array}{ccc} 2 & 0 & 0 \\ 0 & 2 & 0 \\ 0 & 0 & 2 \end{array} \right) \\\\ &=& \left( \begin{array}{ccc} 512 & 0 & 0 \\ 0 & 512 & 0 \\ 0 & 0 & 512 \end{array} \right) \end{eqnarray}になります。
これに当てはめて以下の計算を考えてみましょう。
gradients = torch.tensor([0.1, 1.0, 0.0001], dtype=torch.float) y.backward(gradients) print(x.grad)gradients はヤコビ行列に掛けるベクトルです。
上記で計算したヤコビ行列に当てはめると以下のようになります。{\begin{split}x.grad=\left(\begin{array}{ccc} 512 & 0 & 0\\ 0 & 512 & 0\\ 0 & 0 & 512\\ \end{array}\right)\left(\begin{array}{c} 0.1\\ 1.0\\ 0.0001 \end{array}\right)=\left(\begin{array}{c} 51.2\\ 512\\ 0.0512 \end{array}\right)\end{split} }まとめると、Autograd(自動微分)は以下のようなイメージでしょうか。
- 関数(式)を定義する度にヤコビ行列を保持しておく。
- backward メソッドで保持したヤコビ行列から「微分」を計算する。
ここで話は変わって、以下のように、torch.no_grad() ブロックで記述することにより、関数の変化を追跡しなくなります。
(x ** 2)は追跡されません。print(x.requires_grad) print((x ** 2).requires_grad) with torch.no_grad(): print((x ** 2).requires_grad)True True Falseまた、detach() は、tensorの変数はコピーされますが、勾配は引き継がれません。
print(x.requires_grad) y = x.detach() print(y.requires_grad) print(x.eq(y).all())True False tensor(True)5.最後に
以上が、PyTorch の2つ目のチュートリアル「Autograd: Automatic Differentiation」の内容です。
1つ目のチュートリアルとは次元が違う内容でした。
後半は、想像の部分もありますので、間違っているかもしれません。
ご指摘いただければ幸いです。次回は3つ目のチュートリアル「NEURAL NETWORKS」を進めてみたいと思います。
履歴
2020/02/28 初版公開
- 投稿日:2020-02-28T22:05:51+09:00
機械学習のアルゴリズム(単純パーセプトロン)
はじめに
以前、「機械学習の分類」で取り上げたアルゴリズムについて、その理論とpythonでの実装、scikit-learnを使った分析についてステップバイステップで学習していく。個人の学習用として書いてるので間違いなんかは大目に見て欲しいと思います。
今回から分類問題(Classification)にとりかかるつもりです。まずは基本のパーセプトロンから。
今回参考にしたのは以下のサイト。ありがとうございます。
2クラス分類とは
2クラス分類は、ある入力に対し「1」か「0」か(または「1」か「-1」)を出力することを言います。「60%の確率で故障するかも」ではなく故障するかしないか白黒つけます。2クラス分類にもいろいろあり、パーセプトロンは最も基本の分類機になります。
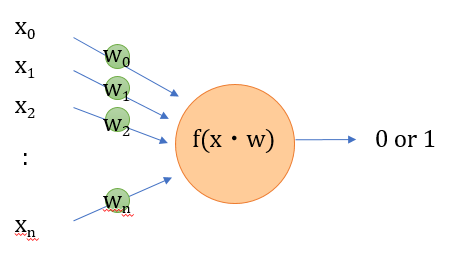
パーセプトロンの概要
パーセプトロンは、神経細胞に着想を得たモデルで、多数の入力に重み付けしたものを足し合わせて、ある閾値をこえた場合に1が出力されます。
図示するとよく見るあの絵です。数式表現
n個の入力を$\boldsymbol{x}=(x_0,x_1,\cdots, x_{n}) $、重みを$\boldsymbol{w}=(w_0,w_1,\cdots, w_{n}) $とし、すべてを足し合わせると、
w_0x_0+w_1x_1+\cdots+w_{n}x_{n} \\\ =\sum_{i=0}^{n}w_ix_i \\\ = \boldsymbol{w}^T\boldsymbol{x}と表される。Tは転置行列です。そして、この値が正なら1、負なら-1を出力します。こういう-1か1かの値を示す関数をステップ関数と呼びます。
入力に無関係な初期値をバイアス項と呼ぶのですが、バイアス項を$w_0$とすると$x_0=1$とすれば上の式がそのまま使えます。
ここまでをpythonで書いてみる
pythonは行列の積を「@」で計算できるので、パーセプトロンへ入力input、を出力をoutputとすると
import numpy as np w = np.array([1.,-2.,3.,-4.]) x = np.array([1.,2.,3.,4.]) input = w.T @ x output = 1 if input>=0 else -1簡単ですね。
パーセプトロンの学習
パーセプトロンはいわゆる「教師あり学習」です。与えられた$\boldsymbol{x}$に対し、正解ラベル$\boldsymbol{t}=(t_0,t_1,\cdots,t_n)$があったとして、$\boldsymbol{w}^T\boldsymbol{x}$が正しく正解ラベルを返すような$\boldsymbol{w}$を求める必要があります。
これは、回帰のときと同じように教師データを使って学習していく必要があります。パーセプトロンにおいても、同じように損失関数を決めて、パラメータ$\boldsymbol{w}$を更新しながら損失を最小化するアプローチが有効です。
パーセプトロンの損失関数
では、どのような損失関数を設定すればいいでしょうか。考え方としては、「2つのクラスを分類する境界を基準として正解であれば損失無し、不正解であれば境界からの距離に応じて損失を与える」という考え方です。
そういう要望に応えるにはヒンジ関数というのががよく使われます。scikit-learnのパーセプトロンもヒンジ関数が使われているみたいです。ヒンジ関数については、
こちらにもあるように、ある値から増加して行く関数で、$h(x)$とすると、$$ h(x) = \max(0,x-a)$$と書くことができます。ヒンジ関数はSVM(サポートベクターマシン)でも使われるそうです。SVMは大事なのでまたいずれ。
損失関数について、要素ごとの正解ラベル$t_n$と予測値$ step(w_nx_n)$が同じ場合、$t_nw_nx_n$は 正の値を示し、異なる場合は負の値となる。損失関数は小さいほどよいので、損失関数を$L$とすると、$$L=\sum_{i=0}^{n}\max(0,-t_nw_nx_n)$$である。損失関数を最小にする$w_n$を、勾配降下法を用いて求める。
$L$を$w_n$について偏微分すると、
\frac{\partial L}{\partial w_n}=-t_nx_nであるので、$w_n$を更新する漸化式は
w_{i+1}=w_{i}+\eta t_nx_nと書ける。なお、$\eta$は学習率です。
パーセプトロンのpython実装
実際にpythonで実装してみます。使用するデータはおなじみscikit-learnからアヤメ(iris)の分類。データセットの詳しい説明は以下を参照してください。
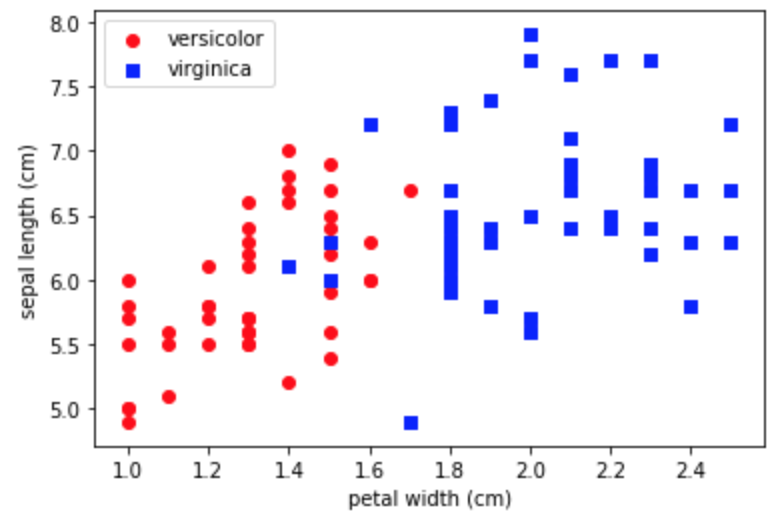
まずは2クラス分類なので、そこに特化します。使用するデータは別になんでもいいんですけど、独断と偏見で、ラベルには"versicolor"と "virginica"。特徴量には"sepal length (cm)"と"petal width (cm)"を選択しました。
まずはデータを可視化します。
mport numpy as np import pandas as pd import matplotlib.pyplot as plt %matplotlib inline from sklearn.datasets import load_iris iris = load_iris() df_iris = pd.DataFrame(iris.data, columns=iris.feature_names) df_iris['target'] = iris.target_names[iris.target] fig, ax = plt.subplots() x1 = df_iris[df_iris['target']=='versicolor'].iloc[:,3].values y1 = df_iris[df_iris['target']=='versicolor'].iloc[:,0].values x2 = df_iris[df_iris['target']=='virginica'].iloc[:,3].values y2 = df_iris[df_iris['target']=='virginica'].iloc[:,0].values ax.scatter(x1, y1, color='red', marker='o', label='versicolor') ax.scatter(x2, y2, color='blue', marker='s', label='virginica') ax.set_xlabel("petal width (cm)") ax.set_ylabel("sepal length (cm)") ax.legend() plt.plot()なんとなく分類できそうですね(そういうデータを選んだとも言う)。
パーセプトロンクラスの実装
Perceptronクラスを実装します。バイアス項を意図的に追加しています。
class Perceptron: def __init__(self, eta=0.1, n_iter=1000): self.eta=eta self.n_iter=n_iter self.w = np.array([]) def fit(self, x, y): self.w = np.ones(len(x[0])+1) x = np.hstack([np.ones((len(x),1)), x]) for _ in range(self.n_iter): for i in range(len(x)): loss = np.max([0, -y[i] * self.w.T @ x[i]]) if (loss!=0): self.w += self.eta * y[i] * x[i] def predict(self, x): x = np.hstack([1., x]) return 1 if self.w.T @ x>=0 else -1 @property def w_(self): return self.w各データごとにヒンジ損失関数を計算し、不正解だった場合に重みを最急勾配法で更新しています。決められた更新回数になったら計算を打ち切っていますが、これは誤差が一定値以下になった打ち切るとしてもいいかもしれません。
実際に分類してみる
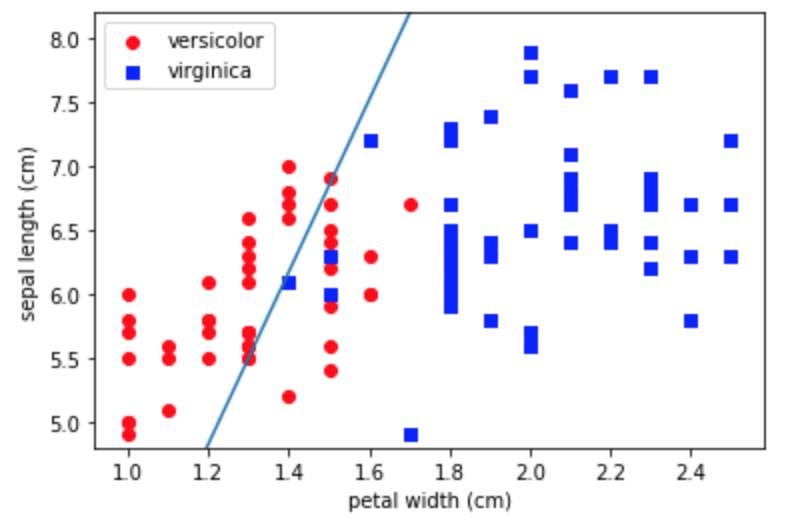
先ほどのクラスにデータを入れて学習させた後に、境界を引いてみます。
df = df_iris[df_iris['target']!='setosa'] df = df.drop(df.columns[[1,2]], axis=1) df['target'] = df['target'].map({'versicolor':1, 'virginica':-1}) x = df.iloc[:,0:2].values y = df['target'].values model = Perceptron() model.fit(x, y) # グラフの描画 fig, ax = plt.subplots() x1 = df_iris[df_iris['target']=='versicolor'].iloc[:,3].values y1 = df_iris[df_iris['target']=='versicolor'].iloc[:,0].values x2 = df_iris[df_iris['target']=='virginica'].iloc[:,3].values y2 = df_iris[df_iris['target']=='virginica'].iloc[:,0].values ax.scatter(x1, y1, color='red', marker='o', label='versicolor') ax.scatter(x2, y2, color='blue', marker='s', label='virginica') ax.set_xlabel("petal width (cm)") ax.set_ylabel("sepal length (cm)") # 分類境界を描画する w = model.w_ x_fig = np.linspace(1.,2.5,100) y_fig = [-w[2]/w[1]*xi-w[0]/w[1] for xi in x_fig] ax.plot(x_fig, y_fig) ax.set_ylim(4.8,8.2) ax.legend() plt.show()virginicaは正しく分類できているみたいですが、visicolorが分類できていないですね。こんなもんなんですかね。
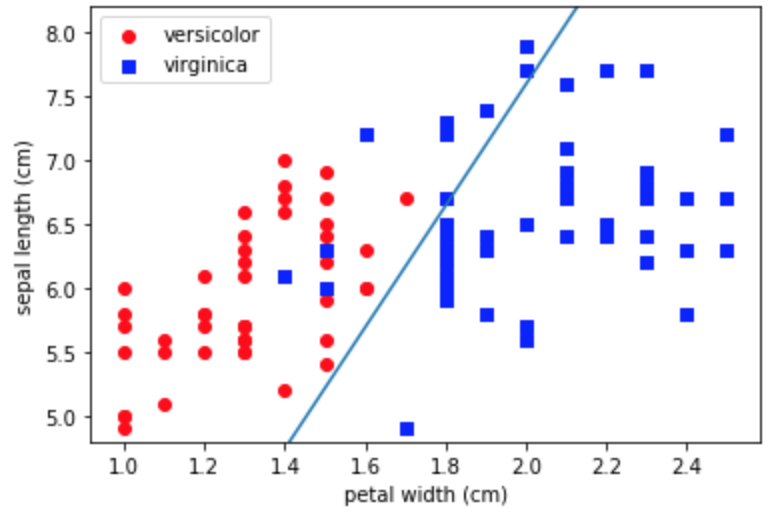
scikit-learnでやってみる
df = df_iris[df_iris['target']!='setosa'] df = df.drop(df.columns[[1,2]], axis=1) df['target'] = df['target'].map({'versicolor':1, 'virginica':-1}) x = df.iloc[:,0:2].values y = df['target'].values from sklearn.linear_model import Perceptron model = Perceptron(max_iter=40, eta0=0.1) model.fit(x,y) # グラフ部分は省略あれ、さっきと逆でversicolorが分類できています。損失関数のあたりが少し違うのかもしれませんが検証できていません。
まとめ
分類機の基本であるパーセプトロンについて考えてみました。ディープラーニングはパーセプトロンを大量に組み合わせたモデルになっているので、パーセプトロンの理解はあとあと大事になってきます。
- 投稿日:2020-02-28T21:44:25+09:00
画像をスクレイピングして学習させたい
特定分野の画像をアップサンプリングするのに使えるんじゃないかなーと思って集めました。
週末にやってみたい!これを試したい
コード必要なし!画像を高解像度化するPythonコード(RAISR)
https://blog.capilano-fw.com/?p=282画像を集めるならこれを参考にすればよさそう
PythonでGoogle画像検索をして画像をフォルダに保存する
https://qiita.com/Yuki-Takatsu/items/3f30727d5b21a83ea4edPythonでGoogleの画像を大量スクレープする
https://qiita.com/Jixjia/items/881c03c50c6f07b0b6abPythonでBing Image Search API v7を使って画像収集する
https://qiita.com/m-shimao/items/74ee036fff8fac01566eBingの画像検索APIを使って画像を大量に収集する
https://qiita.com/ysdyt/items/49e99416079546b65dfcPythonでGoogleとBingから画像を取得するWEBスクレイピングツールを作った
https://nekodeki.com/python%E3%81%A7%E7%94%BB%E5%83%8F%E3%82%92%E5%8F%96%E5%BE%97%E3%81%99%E3%82%8B%E3%82%B9%E3%82%AF%E3%83%AC%E3%82%A4%E3%83%94%E3%83%B3%E3%82%B0%E3%83%84%E3%83%BC%E3%83%AB/
- 投稿日:2020-02-28T21:38:31+09:00
PyOCRでエラーが出た話
tesseractって「テッサラクト」ですよね?ここ読んで理解してください。
Jupyterはジュピターだし、Tensorflowはテンサーフロウです。
元英語教師としてここは譲れない。エラーの内容
-psmっていう引数がダメってお叱りを受けました。
File "C:\Users\hogehoge\Anaconda3\envs\pyocr\lib\site-packages\pyocr\tesseract.py", line 362, in image_to_string
raise TesseractError(status, errors)
pyocr.error.TesseractError: (1, b"Error, unknown command line argument '-psm'\r\n")
直したこと
-psmっていう引数がダメだけど、--psmならOKらしい。ほえー。
C:\Users\hoge\Anaconda3\envs\pyocr\Lib\site-packages\pyocr\lib\site-packages\pyocr\
tesseract.pycommand = [TESSERACT_CMD, "input.bmp", 'stdout', "--psm", "0"]C:\Users\hoge\Anaconda3\envs\pyocr\Lib\site-packages\pyocr\
builders.pytess_flags = ["--psm", str(tesseract_layout)]ここのページを参考にしたのですが、Googleのキャッシュなんですよね。
ブログ全体を見てみるとハック系の記事ばかりなので、カテゴリを統一したいという意図でしょうかね?
(いやまぁこの問題は解決したっぽいんですけど、本来のOCRを用いて解決したい問題は解決していないのがなんとも。。。)
- 投稿日:2020-02-28T21:20:00+09:00
AWS CDK(Python)開発からAWSリソース構築までの手順※開発初心者向け
自分がほぼインフラ一本でやってきて、開発経験が乏しかったことから
開発初心者がCDKを使ってAWSリソースを構築するために行ったことや手順を纏めてみました。
※バージョンによってはこの通り動かないこともあると思いますのでご了承ください
私は2019年12月時点の最新版のバージョンを利用していますコーディング環境
環境用意は下記の記事を参照
WindowsにCDK環境構築する手順(Python)
https://qiita.com/toma_shohei/items/985916e1a95ec4c38121CDK(Python)コーディング
◾️まず概要をつかむ
https://pages.awscloud.com/rs/112-TZM-766/images/B-3.pdf◾️環境用意したらWorkShopやってみる
・Workshop(英語)
https://cdkworkshop.com/30-python.html・Workshop(日本語:ダウンロード版)
http://bit.ly/cdkworkshopjprequirements.txt編集(扱うリソースをinstall)
=======
aws-cdk.coreaws-cdk.aws-autoscaling
aws-cdk.aws-ec2
aws-cdk.aws-elasticloadbalancingv2
aws-cdk.aws-rds
aws-cdk.aws-ssm
aws-cdk.aws-route53aws-cdk.aws-autoscaling-common
====================
pip install -r requirements.txtフォルダ構成
cdk init した段階で以下のようなフォルダ構成が勝手に出来上がります。
https://cdkworkshop.com/30-python/20-create-project/300-structure.html自分はpyyaml導入してパラメータをコードから分離しています。
フォルダ構成は下記の様なイメージになります。・フォルダ構成イメージ
CDK-WORKSHOP
□┣config
□┃┣prd
□┃┃┣ec2.yaml
□┃┃┣vpc.yaml
□┃┃┣・・・
□┃┗stg
□┃□┣ec2.yaml
□┃□┣vpc.yaml
□┃□┣・・・
□┣cdk-workshop_stack.py
□┣・・・※CDKのTOP直下にconfigというフォルダを切っている。
その配下に環境別(prd、stgなど)のフォルダを切り、
その配下にリソース別(ec2やvpcなど)にパラメータを記述している。(cdk.jsonで読み込むディレクトリを切り替える)
cdk-workshop_stack.pyに全リソース構築するコードを記載(config配下のパラメータを読み取る)。
複数リージョンへの構築は同一フォルダではできなかったのでフォルダを分けること。下記のようなやり方はpyyamlを利用しているとできない(バグと思われるが必ず出力がJSONになってしまう)
https://dev.classmethod.jp/cloud/aws-cdk-multi-account-region/暫定対応(今後修正予定、現状では解決手段不明)
!Refをパラメータに書くときは「\!Ref」とし、
出力されたyamlを一括変換「\!Ref」⇒「!Ref」する。
※!GetAttも同様
※pyyamlでyaml.safe_loadを利用しているが「!」をエスケープする方法がわからない
yamlに書かずに.py上に"!Ref " + yamlから読み込んだ文字 とすると 文字列全体に" "がかかり、!Refまで文字列扱いされてしまうCloudFormation実行
・CloudFormationテンプレート出力
cdk synth --version-reporting false --path-metadata false > C:\Users\test\Desktop\test.txt・上記暫定対応実施
・CloudFormationデプロイコマンド(チェンジセット作成まで)
aws cloudformation deploy --stack-name testStack --template-file C:\Users\test\Desktop\test.txt --no-fail-on-empty-changeset --no-execute-changeset・変更セット確認⇒実行
マネジメントコンソールからCloudFormationを開くと
作成したスタックの変更セットを確認し、問題なければ実行を押すよく見るドキュメント
・AWS CDK Python Reference
https://docs.aws.amazon.com/cdk/api/latest/python/index.html・Python examples
https://github.com/aws-samples/aws-cdk-examples課題
・High-level constructsの利用
手動で構築済の既存のリソース上に新たに構築するリソースをコードで構築していく運用(?)のため
Low-level constructs(CfnXXXという名前のライブラリ)しか利用できていない
※本来ならコード量を少なくするため、High-level constructsを使いたいが、
High-level constructsで作ったリソース上にしか作れないようになっている?(⇒これは調査中)・テストコードがかけていない
そもそも何を確認すれば良いのかが未検討所感
・IaCでの構築について
品質は間違いなく上がります。(ある程度の規模の構築案件なら必ずミスする自分がミスしなかった)
検証環境作ったら本番環境はほぼコピペで作成できるので再現性はかなり高いです。・CDKの特徴
TerraformでもAWS構築経験はありますが、CDKの方がはまりポイントが少ない印象です。
後発のツールであるため、バージョンアップの間隔が短く(毎日のようにコミットされている)、
最新版が後方互換しない場合があるので、リスクはありますが現状はそれで悩まされたことは少ししかないです。
- 投稿日:2020-02-28T21:00:29+09:00
ESPCNを使ってみた
はじめに
今回の記事はこちらの記事をqiita用に書き直したものです。
超解像度化
超解像度化とは一言で言うと、「画像の解像度を上げる」ことです。そう聞くとただ画像を引き延ばすだけと思う人もいるかもしれませんが、画像を縮小する際には元の画像の情報がどうしても抜け落ちてしまいます。簡単な検証をしてみます。まずは適当な画像を二分の一のサイズに縮小します。その後bilinear法を使って、画像のサイズがもとの画像と同じになるように拡大します。その結果が下の画像です。
左が下の画像、右が一度縮小した後拡大した画像一目では分かりづらいですが解像度は同じでも、ぼやけてしまい細かい特徴が消えてしまっています。解像度をただ上げるだけではなく細かい特徴を補いながら解像度の高い画像へと変換するのが、超解像度化のタスクです。
ESPCNについて
ESPCN(efficient sub-pixel convolutional neural network)は2016年に発表されたディープニューラルネットワークベースの超解像度化モデルです。
これに類似のモデルとして、2015年に発表されたSRCNN(Super-Resolution Convolutional Neural Network)があります。これは、超解像度のタスクにニューラルネットワークを用いたもので、三つの畳み込み層によって高解像度の画像へと変換するモデルです。
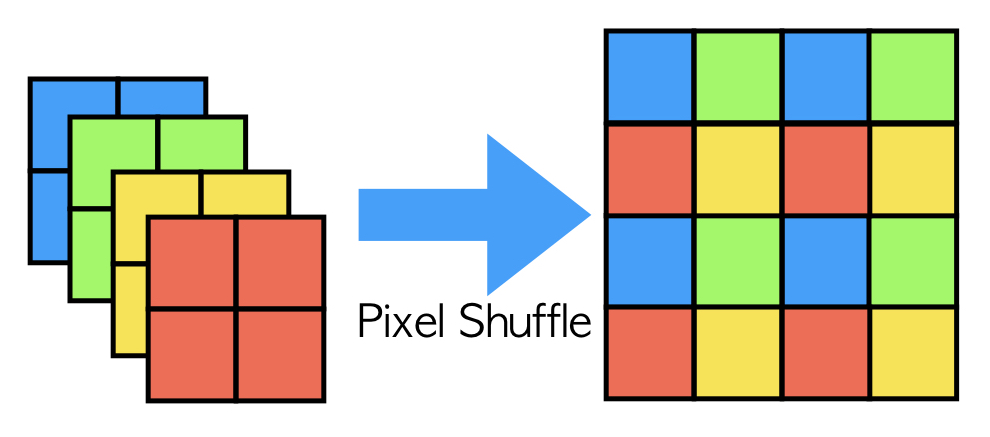
ESPCNの特殊な点として、通常は解像度を上げる際には逆畳み込みという操作を加えるのですがこの操作によって、格子状のノイズが発生することがあります。これを克服するためにESPCNではPixel Shuffleという操作によって解像度を上げています。例えば画像を二倍のサイズにしたい時には、出力の直前で四枚の画像を作りその四枚を決まった位置関係で組み合わせて出力にします。
実装と結果
こちらのコードとスクレイピングして得た画像を用いて学習を行いました。ちなみに学習に用いた画像は10000枚ほどです。
左が下画像、中央が一度縮小したのち拡大した画像、右がESPCNを用いた画像。若干元画像と比べると細かい部分で違和感がありますが、ほとんど復元できていますね。これを使えば動画でも解像度を高くすることもできそうです。
終わりに
今回はESPCNを用いて超解像度化を試してみました。こういうモデルを使って、APIなどを作ってみるのも面白そうです。他にもGANなどを用いて生成した画像をこのモデルを使うことで解像度を上げるなどといった使い方もできそうです。
以下が今回の記事の作成の際に参考にしたサイトと論文です。
画像の超解像度化: ESPCN の pytorch 実装 / 学習
Image Super-Resolution Using Deep Convolutional Networks(2015)
Real-Time Single Image and Video Super-Resolution Using an Efficient Sub-Pixel Convolutional Neural Network(2016)
- 投稿日:2020-02-28T20:03:12+09:00
cGANをACGANに進化させたかった
はじめに
この記事は「cGAN(conditional GAN)でくずし字MNIST(KMNIST)の生成」の続きです。
cGANを元にACGANをやろうとしてあれこれやってみた際の記録になります。cGANからの進化という意味では自然な発想かなと思いますが、実装してみるとなかなか...
ACGANとは
cGANについては前回の記事に簡単に紹介したので、ACGANについて簡単に説明します。
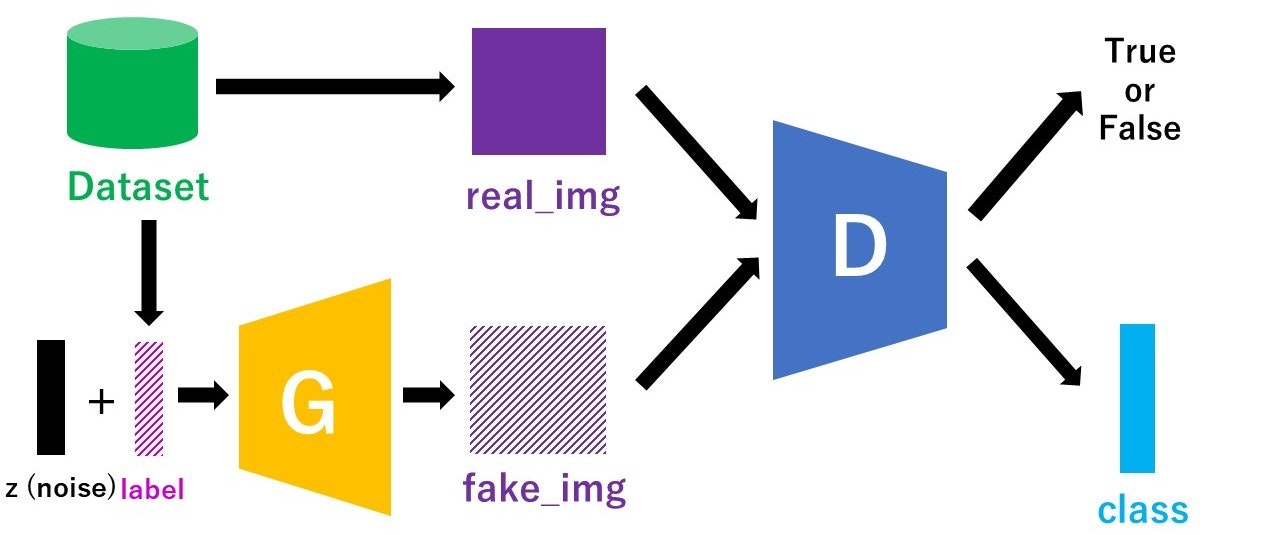
ACGANとは、一言だと「Discriminatorがクラス分類タスクも行うcGAN」です。
よりバリエーションの多い画像の出力を可能とする手法になっています。元論文はこちら
A. Odena, C. Olah, J. Shlens. Conditional Image Synthesis With Auxiliary Classifier GANs. CVPR, 2016
ACGANの論文については、元論文を記事化されている方がいらっしゃいますので、そちらが参考になります。
<参考記事>
AC-GAN(Conditional Image Synthesis with Auxiliary Classifier GANs)の論文解説ACGANのモデル構造
cGANでは、Discriminatorに本物/偽物画像とラベル情報を入力して、本物or偽物の識別を出力としていました。一方ACGANでは、Discriminatorの入力が画像のみで、本物or偽物の識別だけでなくそれがどのクラスかを当てるクラス判定も出力に加わります。図で書くと以下のような感じです。
図中のclassの部分が、Discriminatorが予測するクラス分類の出力です。labelと同じく、クラス数次元のベクトルの形をしています。とりあえず実装
ACGANはGitHubにPyTorch実装があります。
これを参考にしながら、前回の記事で書いたcGANの実装に手を加えてみます。やることは
- Discriminatorの入力からラベル情報をなくす
- Discriminatorの出力にクラス分類を足す
- 2種類のlossを計算し、伝播させる
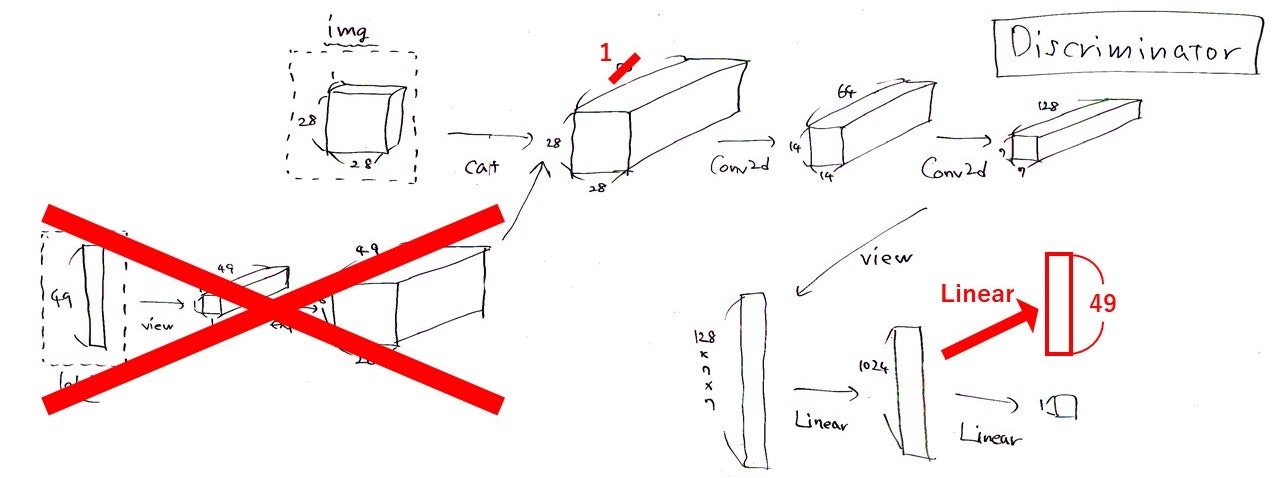
がほぼすべてです。そうすると、Discriminatorの構造はこんな感じです。
これは前回の記事に載せたcGANのDiscriminatorの構造の図にお絵描きしたものですが、赤で示された部分がACGANでの変更点になります。Discriminatorの実装です。
pythonclass Discriminator(nn.Module): def __init__(self, num_class): super(Discriminator, self).__init__() self.num_class = num_class self.conv = nn.Sequential( nn.Conv2d(1, 64, kernel_size=4, stride=2, padding=1), #入力は1チャネル(白黒だから), フィルターの数64, フィルターのサイズ4*4 nn.LeakyReLU(0.2), nn.Conv2d(64, 128, kernel_size=4, stride=2, padding=1), nn.LeakyReLU(0.2), nn.BatchNorm2d(128), ) self.fc = nn.Sequential( nn.Linear(128 * 7 * 7, 1024), nn.BatchNorm1d(1024), nn.LeakyReLU(0.2), ) self.fc_TF = nn.Sequential( nn.Linear(1024, 1), nn.Sigmoid(), ) self.fc_class = nn.Sequential( nn.Linear(1024, num_class), nn.LogSoftmax(dim=1), ) self.init_weights() def init_weights(self): for module in self.modules(): if isinstance(module, nn.Conv2d): module.weight.data.normal_(0, 0.02) module.bias.data.zero_() elif isinstance(module, nn.Linear): module.weight.data.normal_(0, 0.02) module.bias.data.zero_() elif isinstance(module, nn.BatchNorm1d): module.weight.data.normal_(1.0, 0.02) module.bias.data.zero_() elif isinstance(module, nn.BatchNorm2d): module.weight.data.normal_(1.0, 0.02) module.bias.data.zero_() def forward(self, img): x = self.conv(img) x = x.view(-1, 128 * 7 * 7) x = self.fc(x) x_TF = self.fc_TF(x) x_class = self.fc_class(x) return x_TF, x_classクラス分類の出力をどう加えるかは色々やり方がありそうです。先ほど貼ったリンクのPyTorch実装で最後に
Linear層を二股に分岐させていたので、ここでは同じように実装しています。この変更に合わせると、1エポック当たりの関数はこんな感じ。
pythondef train_func(D_model, G_model, batch_size, z_dim, num_class, TF_criterion, class_criterion, D_optimizer, G_optimizer, data_loader, device): #訓練モード D_model.train() G_model.train() #本物のラベルは1 y_real = torch.ones((batch_size, 1)).to(device) D_y_real = (torch.rand((batch_size, 1))/2 + 0.7).to(device) #Dに入れるノイズラベル #偽物のラベルは0 y_fake = torch.zeros((batch_size, 1)).to(device) D_y_fake = (torch.rand((batch_size, 1)) * 0.3).to(device) #Dに入れるノイズラベル #lossの初期化 D_running_TF_loss = 0 G_running_TF_loss = 0 D_running_class_loss = 0 D_running_real_class_loss = 0 D_running_fake_class_loss = 0 G_running_class_loss = 0 #バッチごとの計算 for batch_idx, (data, labels) in enumerate(data_loader): #バッチサイズに満たない場合は無視 if data.size()[0] != batch_size: break #ノイズ作成 z = torch.normal(mean = 0.5, std = 1, size = (batch_size, z_dim)) #平均0.5の正規分布に従った乱数を生成 real_img, label, z = data.to(device), labels.to(device), z.to(device) #Discriminatorの更新 D_optimizer.zero_grad() #Discriminatorに本物画像を入れて順伝播⇒Loss計算 D_real_TF, D_real_class = D_model(real_img) D_real_TF_loss = TF_criterion(D_real_TF, D_y_real) CEE_label = torch.max(label, 1)[1].to(device) D_real_class_loss = class_criterion(D_real_class, CEE_label) #DiscriminatorにGeneratorにノイズを入れて作った画像を入れて順伝播⇒Loss計算 fake_img = G_model(z, label) D_fake_TF, D_fake_class = D_model(fake_img.detach()) #fake_imagesで計算したLossをGeneratorに逆伝播させないように止める D_fake_TF_loss = TF_criterion(D_fake_TF, D_y_fake) D_fake_class_loss = class_criterion(D_fake_class, CEE_label) #2つのLossの和を最小化 D_TF_loss = D_real_TF_loss + D_fake_TF_loss D_class_loss = D_real_class_loss + D_fake_class_loss D_TF_loss.backward(retain_graph=True) D_class_loss.backward() D_optimizer.step() D_running_TF_loss += D_TF_loss.item() D_running_class_loss += D_class_loss.item() D_running_real_class_loss += D_real_class_loss.item() D_running_fake_class_loss += D_fake_class_loss.item() #Generatorの更新 G_optimizer.zero_grad() #Generatorにノイズを入れて作った画像をDiscriminatorに入れて順伝播⇒見破られた分がLossになる fake_img_2 = G_model(z, label) D_fake_TF_2, D_fake_class_2 = D_model(fake_img_2) #Gのloss(max(log D)で最適化) G_TF_loss = -TF_criterion(D_fake_TF_2, y_fake) G_class_loss = class_criterion(D_fake_class_2, CEE_label) #Gからすると、Dが本物だと思い込んでかつクラスもあててくれた方がうれしい G_TF_loss.backward(retain_graph=True) G_class_loss.backward() G_optimizer.step() G_running_TF_loss += G_TF_loss.item() G_running_class_loss -= G_class_loss.item() D_running_TF_loss /= len(data_loader) D_running_class_loss /= len(data_loader) D_running_real_class_loss /= len(data_loader) D_running_fake_class_loss /= len(data_loader) G_running_TF_loss /= len(data_loader) G_running_class_loss /= len(data_loader) return D_running_TF_loss, G_running_TF_loss, D_running_class_loss, G_running_class_loss, D_running_real_class_loss, D_running_fake_class_loss先ほど述べた変更点に加えて、入れるノイズも少し変えました。
前回は30次元・平均0.5・標準偏差0.2の正規分布でしたが、今回は100次元・平均0.5・標準偏差1の正規分布にしています。クラス分類のlossは
torch.nn.NLLLoss()です。これも先ほどのリンクの実装に合わせました。結果
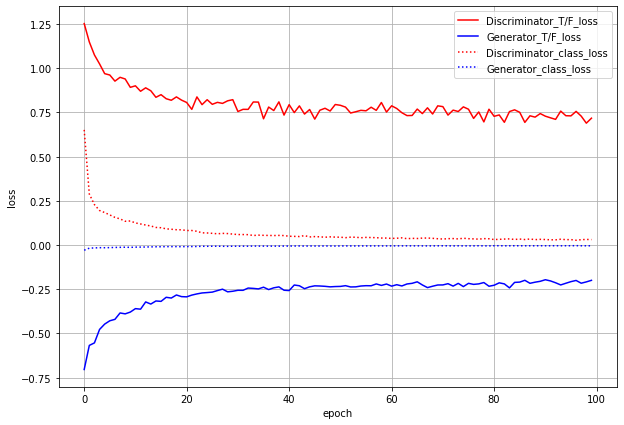
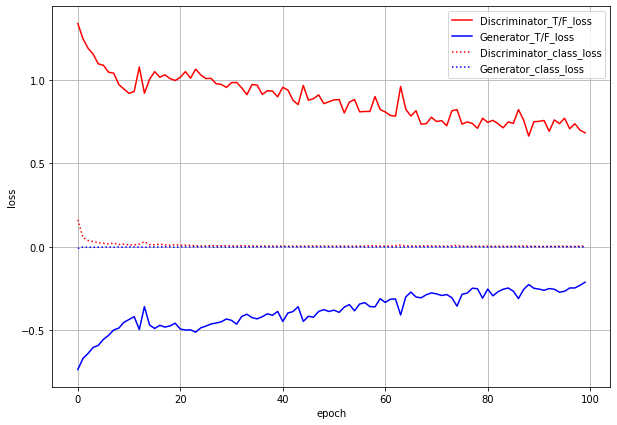
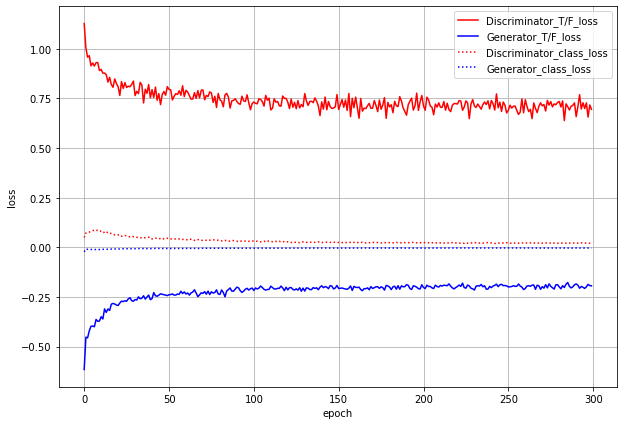
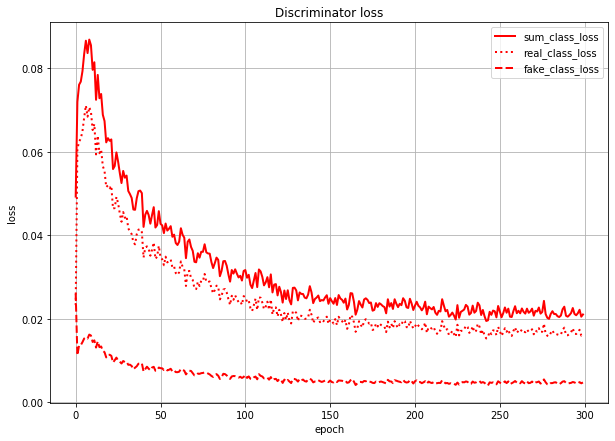
まずはlossのグラフです。
ACGANでは、本物or偽物識別のlossとクラス分類のlossの2つがあり、GeneratorにもDiscriminatorにも両方のlossを伝播させます。グラフでもわけてプロットしています。
T/F_lossが本物/偽物識別のloss(実線)、class_lossがクラス分類のloss(点線)です。これだけ見るとうまくいっているように見えます。しかし...
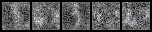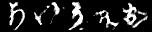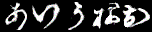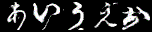
これは1epochごとに各ラベルの画像を1枚ずつ生成したときのgifです。
一番上の行が左から「あ、い、う・・・」と続いて、右下が「・・・を、ん、ゝ」となるようにラベル情報を入力していますが、与えたラベルと生成する画像がほとんど対応していません。でも、全く意味のない画像というよりは「別のラベルの文字」を生成しているように見えます。cGANと同様に、100epoch訓練後のGeneratorで「あ」~「ゝ」を5枚ずつ生成してみました。
ラベルちゃんと対応してそうなのは「け」くらいじゃないでしょうか。
(というか完全にモード崩壊・・・)ちなみに、同じ条件で100epoch訓練後のcGANの生成結果がこれです。
明らかにcGANの方がラベルに近い文字が出力されています。うまくいかない原因・・・?
パッと出力を見た感想ですが、ACGANではGeneratorとDiscriminator双方が、本来とは異なる形の文字をそのラベルの文字だと思い込む(Ex: DiscriminatorとGeneratorが両方とも「い」の形に似ているものを「あ」のラベル扱いしている)のではないか?と思いました。
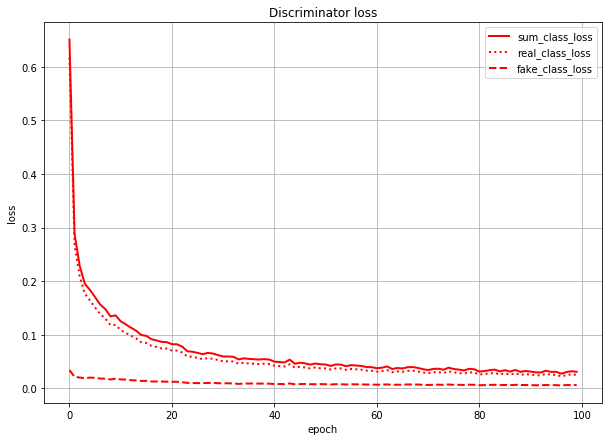
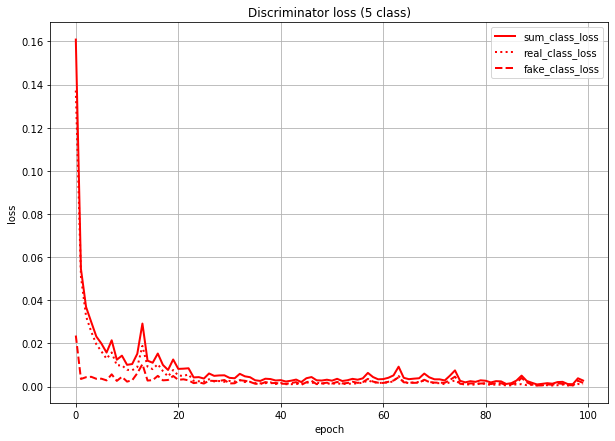
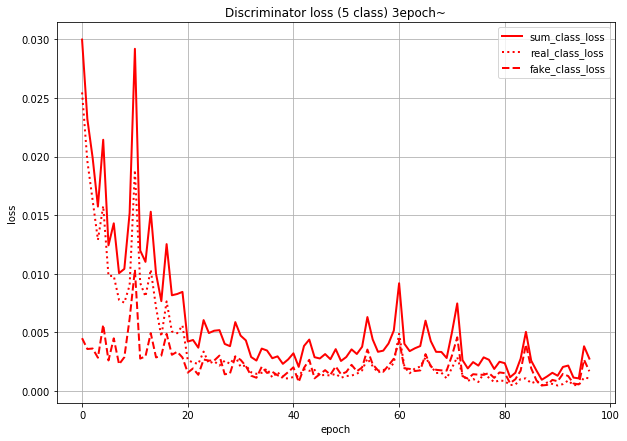
これは、Discriminatorのクラス分類のlossを本物画像由来のlossと偽物画像(=Generatorが作った画像)由来のlossに分けて描いたグラフです。sum_class_lossは合計値(=さっきのグラフの赤点線と同じ)になってます。
このグラフを見ると、Discriminatorは(特に学習序盤で)本物画像の判定を間違え、偽物画像の判定を当てまくっていることになります。
(数値で言うと、real_class_lossはfake_class_lossの序盤で20倍、終盤で5倍くらいの値になっている)つまり、Generatorが「あ」のラベルで作った画像は、実際の形は「あ」とはだいぶ異なる形になっていても、Discriminatorでも「あ」扱いされている、というようなことが想像できます。
おそらく理想としては、クラス分類のlossは本物画像由来も偽物画像由来も同じくらいの値になってほしいのではと思います。
うまくいってそうなものと比較してみる
ACGANの元論文でも言及されていることですが、クラス数が多すぎると同一ネットワークでは出力画像の質が落ちるらしいです。元論文でもImageNet(1000クラス)を10クラス×100ケースに分けて実験しています。
そこで、こちらも一度5クラスでやってみることにしました。
ネットワーク構造は同じにして、「あ」~「お」の5文字生成をやってみます。
lossのグラフは同じような感じです。T/F_lossの方はまだ下がる余地がありそうではあります。
こちらも若干のムラはありますが、後半はかなり綺麗に出来ています。
続いて、100epoch訓練後のもので5枚ずつ画像を生成してみます。
なんとモード崩壊もしてなさそうです。では、Discriminatorのクラス分類のlossです。
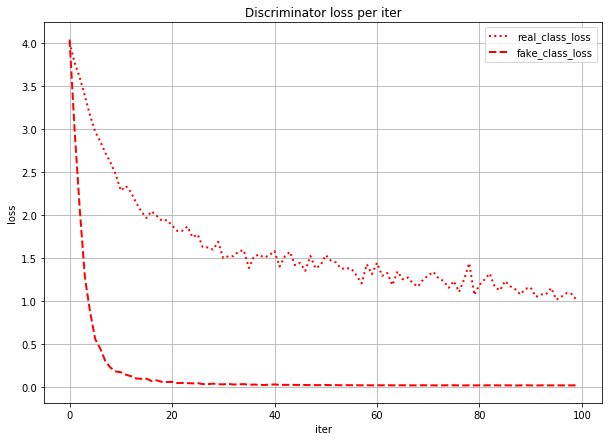
数値ベースで言うと、序盤は10倍くらい差があったのが、終盤ではほぼ同じ値になっているのですが、このグラフだと見づらいので3epoch以降だけ表示してみます。
こう見るとreal_class_lossとfake_class_lossがかなり近い値になっていくのがよくわかります。iterごとのloss
そもそも学習の序盤で1epochめから本物分類と偽物分類で10倍~20倍の差がでるの??と思ったので、iterごと(ミニバッチごと)のlossを表示してみました。
確かに最初はreal_class_lossとfake_class_lossでlossの値は変わりませんが、fake_class_lossの方が急激に下がっているのがわかります。事前学習を試す
最初の数エポックで本物画像だけを学習させるなどしてみましたが、それでもほぼ意味がなかったので、分類タスクだけを事前学習させてみることにしました。
Discriminatorだけを取ってきて分類タスクだけ解かせます。
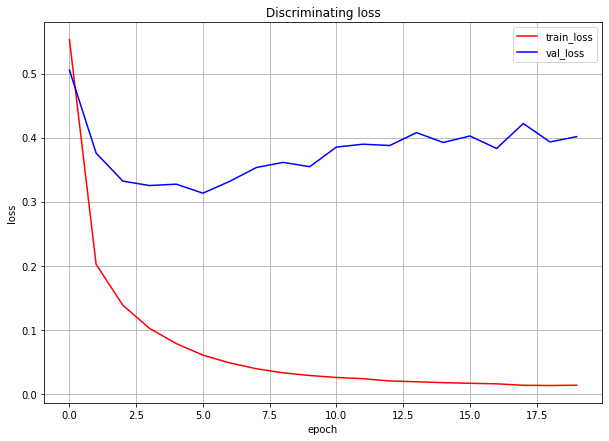
分類タスクの結果
収束がかなり速いので、20epochしかやっていません。
なんか結果的には微妙ですが、とりあえずこの20epoch訓練後のDiscriminatorを使うことにします。事前学習適用時の結果
True/Falseのlossは事前学習しないときとほとんど変化がありません。分類のloss
については、序盤からかなり小さくなっています。では、本物画像由来と偽物画像由来の分類lossを見てみます。
300epochまで学習させてみました。事前学習しないときに比べると、本物画像由来のlossの値もかなり下がってはいます。偽物画像由来のlossと比べても4倍程度に収まっていますが、それでも同じくらいの値にはなっていません。この300epoch訓練後のACGANによる生成画像を見てみます。
うーん。。
効果が見られません。うまく行った文字が増えたということもなく、モード崩壊も起きています。感想
くずし字データセットは、1文字当たりのデータ数が多くは6000で300~400程度しかないものも数個あります。1クラス当たりのデータ数が多い方がうまくいくと思うので、CIFAR-10よりもデータ数が多いくずし字ならうまくいく可能性はあると思いましたが、ダメでした。
個人的には、潜在空間での各ラベルの文字どうしの距離が近い(=異なるラベルの文字でも潜在空間内ではかなり近いところにいる)のでは?と思います。
元論文の実験ではCIFAR-10やImageNetを10クラスごとで実験していましたが、くずし字だと10クラスでは半分強しかうまくいく文字がなく、5クラスにして初めてうまくいきました。いずれにしても、49クラスをACGANで狙って出力させるのはかなり難しそうなのであきらめることにします・・・
- 投稿日:2020-02-28T19:34:54+09:00
AnsibleのVmware系モジュールを使ったのでまとめ
はじめに
タイトルのようにAnsibleでVmwareのGuestVMを作ったり色々便利に使いたい、というケースがあったのでこれを機に触ってみました。
ついでにDynamic Inventoryも使えると便利かなと思い、こちらも触ってみました。動作検証環境
- Vmware側
- VMware vCenter Server Appliance, 6.7.0, 42000
- VMware ESXi, 6.7.0, 13006603
- クライアント(Ansible実行環境)側
- OS: CentOS 7
- 一応Dockerのpython:latestでも試しました
- Python 3.8.1
- python2系でもvmware系モジュールは動きますが、Dynamic Inventoryを使おうとするとsetuptoolsという依存パッケージがPython 3.5以上でないと対応してなかったため3.5以上のほうが幸せになれそうです
- ansible==2.9.5
- pyvmomi==6.7.3
使用したモジュール
Vmware系のモジュールは歴史的には大きく分けて2種類あったようで、例えばGuestVMの作成・削除をするモジュールとしては
- vmware_guestモジュール
- pythonのPyVmomiパッケージを利用
- 推奨
- vsphere_guestモジュール
- pythonのPysphereパッケージを利用
- 長らく
DEPRECATEDだったが、ansible 2.9でついに削除されたの二通りの書き方があったようですが、上記のようにvmware_guestに統合されたようなので、こちらを使っていきます。
(細かい歴史的経緯はよく知りません。。)各種準備
Vmware側
vmware系モジュールの多くはvCenterサーバ経由でのAPIを利用しているようで、vCenterサーバがほぼ必須となります。
(vsphere系モジュールにはESXiに対してアクションできるものもあったとか何とか)
私が検証した環境にはvCenterサーバがなかったので仮想アプライアンスをデプロイするところから始めましたが、今回はこちらの内容は割愛します。vCenterの各種情報をパラメータとして指定する必要があるのであらかじめ整理・取得しておきましょう。
今回は例として以下とします。
vCenter情報 例 ホスト名 192.168.0.1 (IPアドレスで可) ユーザ名 administrator@vsphere.localパスワード passwordデータセンター名 Datcenterクライアント(Ansible実行環境)側
今回は簡単に検証したかったのでansibleも入っていない状況からバージョン指定せずにインストールしていきます。
$ pip install ansible pyvmomi実際にPlaybook等を書いていく
ここからPlaybookなどを書いていきますが、その前に以降のPlaybookで使い回しするであろうvCenterの情報をあらかじめ変数としてファイルに書いておきましょう。
vars_vmware.ymlvcenter_hostname: 192.168.0.1 vcenter_username: administrator@vsphere.local vcenter_password: password vcenter_datacenter: DatacenterVmwareへの接続確認
細かい操作・変更を始める前にちゃんとVmwareに接続できているか確認してみましょう。
とりあえずシンプルそうなvmware_about_infoモジュールを使ってました。
tasksの中身はリンクにあるサンプルほぼそのままです。
一般的なモジュールはssh接続したリモートサーバで実行するものが多いですが、
vmware系のモジュールは大抵delegate_to: localhostと書かれているようにpyvmomiパッケージを持つansible実行ホストで実行します。test.yml- hosts: local gather_facts: false vars_files: - vars_vmware.yml tasks: - name: Provide information about vCenter vmware_about_info: hostname: "{{ vcenter_hostname }}" username: "{{ vcenter_username }}" password: "{{ vcenter_password }}" validate_certs: false delegate_to: localhost register: cluster_info - debug: var: cluster_infoインベントリも今回はlocalhostを設定しました。
local.ini[local] localhostPlaybookを実行してみます。
$ ansible-playbook -i local.ini test.yml PLAY [local] *********************************************************************************************************************************************************************************************** TASK [Provide information about vCenter] ******************************************************************************************************************************************************************* ok: [localhost -> localhost] TASK [debug] *********************************************************************************************************************************************************************************************** ok: [localhost] => { "cluster_info": { "about_info": { "api_type": "VirtualCenter", "api_version": "6.7.3", "build": "15129973", "instance_uuid": "*****-******", "license_product_name": "VMware VirtualCenter Server", "license_product_version": "6.0", "locale_build": "000", "locale_version": "INTL", "os_type": "linux-x64", "product_full_name": "VMware vCenter Server 6.7.0 build-15129973", "product_line_id": "vpx", "product_name": "VMware vCenter Server", "vendor": "VMware, Inc.", "version": "6.7.0" }, "ansible_facts": { "discovered_interpreter_python": "/usr/bin/python" }, "changed": false, "failed": false } } PLAY RECAP ************************************************************************************************************************************************************************************************* localhost : ok=2 changed=0 unreachable=0 failed=0 skipped=0 rescued=0 ignored=0エラーなく接続して値が取得できてますね!
GuestVMの作成(From Template)
今度はvmware_guestモジュールを使ってGuestVMを作成します。
おそらく一番使われていそうなモジュールだと思いますが、実際に動かす前に諸々準備をする必要があります。VMテンプレートの作成
vmware_guestモジュールで最小限のパラメータでゲストVMを作成した場合はOSがインストールされる前の空っぽの状態になります。
せっかくAnsibleでゲストVMを作成したのにそのままパッケージインストールやアプリのデプロイができないのは片手落ち感がありました。
KickStart用のISOイメージを作成・マウントして...みたいな方法もできそうですが、
ここはスマートに(?)VMテンプレートの作成してクローンとしてGuestVMを作成しようと思います。VMテンプレートとしてはお好きなものを作ってもらえればいいので詳細は省きますが、
私の例ではCentOS7を最小構成インストールしたものを使用しました。
また、その状態のテンプレートを使用した場合、VMNICが有効にならないなどの事象があったため、
追加でopen-vm-toolsパッケージをインストールする必要があるようなので、これとopen-vm-toolsを動かすperlだけははインストールしておきましょう。$ sudo yum install -y open-vm-tools perl※ Debian系の場合でも同名のパッケージがあるようなのでapt等でインストールしましょう。
今回は
Template_CentOSという名前の仮想マシンを作成し、テンプレートに変換しました。
※ ここではテンプレートに変換していますが、起動中のVMを指定しても特に問題なくクローン作成されるようです。folderの確認
また
vmware_guestモジュールでGuestVMの作成をする場合にはfolderというパラメータを入れる必要があるので、このfolderの値を確認します。
Vmware側のドキュメントやvSphereWebClient上から確認する方法が分かりませんでしたが(ご指摘いただけると幸いです)、
vmware_folder_infoモジュールで取得するのが手っ取り早かったです。get_folders.yml- hosts: local gather_facts: false vars_files: - vars_vmware.yml tasks: - name: Provide information about vCenter folders vmware_folder_info: hostname: "{{ vcenter_hostname }}" username: "{{ vcenter_username }}" password: "{{ vcenter_password }}" datacenter: "{{ vcenter_datacenter }}" validate_certs: false delegate_to: localhost register: folder_info - debug: var: folder_info$ ansible-playbook -i local.ini get_folders.yml PLAY [local] *********************************************************************************************************************************************************************************************** TASK [Provide information about vCenter folders] *********************************************************************************************************************************************************** ok: [localhost -> localhost] TASK [debug] *********************************************************************************************************************************************************************************************** ok: [localhost] => { "folder_info": { "ansible_facts": { "discovered_interpreter_python": "/usr/bin/python" }, "changed": false, "failed": false, "folder_info": { "datastoreFolders": { "path": "/Datacenter/datastore", "subfolders": {} }, "hostFolders": { "path": "/Datacenter/host", "subfolders": {} }, "networkFolders": { "path": "/Datacenter/network", "subfolders": {} }, "vmFolders": { "path": "/Datacenter/vm", "subfolders": {} } } } } PLAY RECAP ************************************************************************************************************************************************************************************************* localhost : ok=2 changed=0 unreachable=0 failed=0 skipped=0 rescued=0 ignored=0この中の
vmFoldersに記載された/Datacenter/vmが仮想マシン用のfolderのようです。やっとGuestVMの作成
これらのパラメータを使って、ようやくvmware_guestモジュールを使用できます。
仮想マシン名は
test1としています。
仮想マシン名は(厳密には一意ではないようですが)一意の名前を指定しましょう。
既存の仮想マシン名を指定した場合はGuestVMの作成は起こらず、既存の仮想マシンのstateで指定した電源状態に変化するだけのようです。(いわゆる冪等性ってやつだと思います)なお今回は
Clusterという名前のvSphereクラスタを作成し、その配下にGuestVMを作成しましたが、
クラスタがない場合や作成するESXiホストを指定したい場合はclusterの代わりにesxi_hostnameを指定してください。その他パラメータはドキュメントを確認してください。
create_guestvm.yml- hosts: local gather_facts: false vars_files: - vars_vmware.yml tasks: - name: Create a virtual machine on given ESXi hostname vmware_guest: name: test1 # VM名 hostname: "{{ vcenter_hostname }}" username: "{{ vcenter_username }}" password: "{{ vcenter_password }}" datacenter: "{{ vcenter_datacenter }}" folder: /Datacenter/vm # vmware_folder_infoで取得したfolder名 validate_certs: false template: Template_CentOS # 準備で作成したテンプレート名 state: poweredon # 起動状態にする # guest_id: centos7_64Guest # 仮想マシンの作成時には必須であり、テンプレートからの作成時には不要 cluster: Cluster # esxi_hostname: 192.168.*.* # clusterないしesxi_hostnameのいずれかを指定する disk: - size_gb: 16 type: thin datastore: datastore hardware: memory_mb: 1024 num_cpus: 1 scsi: paravirtual networks: - name: VM Network start_connected: true wait_for_ip_address: yes customization: hostname: test1 delegate_to: localhost register: vm_info - debug: var: vm_info$ ansible-playbook -i local.ini create_guestvm.yml PLAY [local] *********************************************************************************************************************************************************************************************** TASK [Create a virtual machine on given ESXi hostname] ***************************************************************************************************************************************************** changed: [localhost -> localhost] TASK [debug] *********************************************************************************************************************************************************************************************** ok: [localhost] => { "vm_info": { "ansible_facts": { "discovered_interpreter_python": "/usr/bin/python" }, "changed": true, "failed": false, "instance": { "annotation": "", "current_snapshot": null, "customvalues": {}, "guest_consolidation_needed": false, "guest_question": null, "guest_tools_status": "guestToolsRunning", "guest_tools_version": "10336", "hw_cluster": "Cluster", "hw_cores_per_socket": 1, "hw_datastores": [ "datastore" ], "hw_esxi_host": "192.168.*.*", "hw_eth0": { "addresstype": "assigned", "ipaddresses": [ "192.168.*.*", ~~~~ 中略 ~~~~ "instance_uuid": "****^****", "ipv4": "192.168.*.*", "ipv6": null, "module_hw": true, "moid": "vm-***", "snapshots": [], "vimref": "vim.VirtualMachine:vm-***", "vnc": {} } } } PLAY RECAP ************************************************************************************************************************************************************************************************* localhost : ok=2 changed=1 unreachable=0 failed=0 skipped=0 rescued=0 ignored=0スペックによってはそれなりの時間がかかると思いますが、これでGuestVMが作成されましたね!
.instance.ipv4にIPアドレスの情報もあるので、add_hostモジュールで追加してその後のタスクを実行!なんてことも可能です。オマケ:VmwareのDynamic Inventoryを使ってみる
AnsibleはVmwareについてDynamic Inventoryを使用することができます。
Dynamic Inventory自体については以下を参考してもらえるのがよいですが、簡単にいうと
タスクを実行したい対象が数台ぐらいであればVMのホスト名(IP)をインベントリに書けばいいのですが、対象が3桁・4桁と増えていくとそれも大変になります。
そこでホスト情報を直接記載するのではなく、API等からホスト情報を取得することで動的にインベントリに必要な情報を取ってこよう、というものです。
https://docs.ansible.com/ansible/latest/user_guide/intro_dynamic_inventory.htmlこちらのページを参考に、ついでにDynamic Inventoryを使ってみました。
https://docs.ansible.com/ansible/latest/scenario_guides/vmware_scenarios/vmware_inventory.html準備として、先にインストールした
pyvmomiモジュールに加えて、vSphere Automation SDKが必要なようです。
こちらの手順にしたがってインストールします。
なお、動作検証環境で記載したようにsetuptoolsパッケージがpython3.5未満ではインストールできなかったため、python3系で実行しています。
(setuptoolsはバージョン指定をすればインストールできたのですが、その後のsdkのインストールには失敗したのでそれ以降は追いかけてません。回避方法等があればご指摘ください)$ pip install --upgrade pip setuptools $ pip install --upgrade git+https://github.com/vmware/vsphere-automation-sdk-python.gitまず
ansible.cfgでVmwareのDynamic Inventoryに必要なプラグインを有効化します。ansible.cfg[inventory] enable_plugins = vmware_vm_inventory続いて、インベントリですが、vCenterの情報を記載します。またファイル名の末尾が
.vmware.ymlor.vmware.yamlである必要があります。inventory.vmware.ymlplugin: vmware_vm_inventory strict: False hostname: 192.168.0.1 username: administrator@vsphere.local password: password validate_certs: False with_tags: Trueここで確認で
ansible-inventoryコマンドを実行するとJSON形式のインベントリ情報が出力されます。$ ansible-inventory -i inventory.vmware.yml --list { "_meta": { "hostvars": { "test1_****-****": { "ansible_host": "192.168.*.*", "config.cpuHotAddEnabled": false, "config.cpuHotRemoveEnabled": false, "config.hardware.numCPU": 1, "config.instanceUuid": "****-****", "config.name": "test1", "config.template": false, "guest.guestId": "centos7_64Guest", "guest.guestState": "running", "guest.hostName": "localhost.localdomain", "guest.ipAddress": "192.168.*.*", "name": "test1", "runtime.maxMemoryUsage": 1024 }, ~~~ 中略 ~~~ } }, "all": { "children": [ "centos7_64Guest", "other3xLinux64Guest", "poweredOff", "poweredOn", "ungrouped" ] }, "centos7_64Guest": { "hosts": [ "test1_****-****", "", ~~~ 中略 ~~~ ] }, ~~~ 中略 ~~~ }
allグループのchildrenとしてguest_idや電源状態のグループがあり、hostsの中に配列でホスト名が記載されており、ホスト毎の詳細な情報はhost_varsとして記載されているようです。
なお、ホスト名はVM名にhw_product_uuidというUUIDをつけた形式のようです。最後に例としてvmware_guest_tools_upgradeモジュールを使ってVmwareToolのアップグレードをやってみたいと思います。
今回は他のVMに影響を与えたくなかったのでtest1のみに対してアップグレードを実行しますが、
hostsをallやグループ名にすれば複数台に対して実行されるはずです。vmtool_upgrade.yml- hosts: test1_***-**** gather_facts: false vars_files: - vars_vmware.yml tasks: - name: Upgrade VMware Tools using VM name vmware_guest_tools_upgrade: hostname: "{{ vcenter_hostname }}" username: "{{ vcenter_username }}" password: "{{ vcenter_password }}" datacenter: "{{ vcenter_datacenter }}" name: "{{ name }}" validate_certs: false delegate_to: localhost実行した結果、作成したばかりのVMだったためVmwareToolも最新版だったようで変化はありませんでしたが、
ある程度の期間と台数を運用している環境で実行すれば中々強力な作業自動化に繋がると思います。$ ansible-playbook -i inventory.vmware.yml play.yml -v Using /work/ansible.cfg as config file PLAY [test1_***-***] ********************************************************************************************************************************************************** TASK [Upgrade VMware Tools using VM name] ****************************************************************************************************************************************************************** ok: [test1_***-*** -> localhost] => {"changed": false, "msg": "VMware tools is already up to date"} PLAY RECAP ************************************************************************************************************************************************************************************************* test1_***-*** : ok=1 changed=0 unreachable=0 failed=0 skipped=0 rescued=0 ignored=0
- 投稿日:2020-02-28T19:26:11+09:00
Pythonによる累乗近似のお話
EXCELで累乗近似曲線を引いたことで話が始まる
ある減衰していく時系列のデータがあって、普通にEXCELで近似曲線を引いてみたら累乗近似がいい感じだったので、業務の都合上EXCELではなくPythonで再現しようと思った。
やってみた結果、なぜか微妙に合わない…。なぜだろうと不思議に思ったところ以下の情報にたどり着いた。
Pythonによる累乗近似
ほとんどこのサイトを見れば解決するけども、自分の備忘録のために記事にする。scipyのcurve_fitによる累乗近似とEXCELの累乗近似の違い
累乗の式を以下のように定義。
$y = bx^{a}$
scipyのcurve_fitは上記の式でデータと最も近似する$a$と$b$を返す非線形回帰を行っている。ではEXCELではどうなのか。
上記の式は以下のような変換ができる。
$y = bx^{a}$
$\Rightarrow \ln y = \ln (bx^{a}) ・・・(対数をとる)$
$\Rightarrow \ln y = a\ln x + \ln b ・・・(右辺を分解)$
$\Rightarrow Y = aX + B ・・・(対数部分を新たな変数としてまとめる)$
このように累乗式の対数をとると$Y=aX+B$の線形回帰を行うことできる。
つまりEXCELの累乗近似は対数変換して線形回帰した結果を出力しているということらしい。Pythonで実践してみる
ではPythonで実際どうなるのかやってみる。
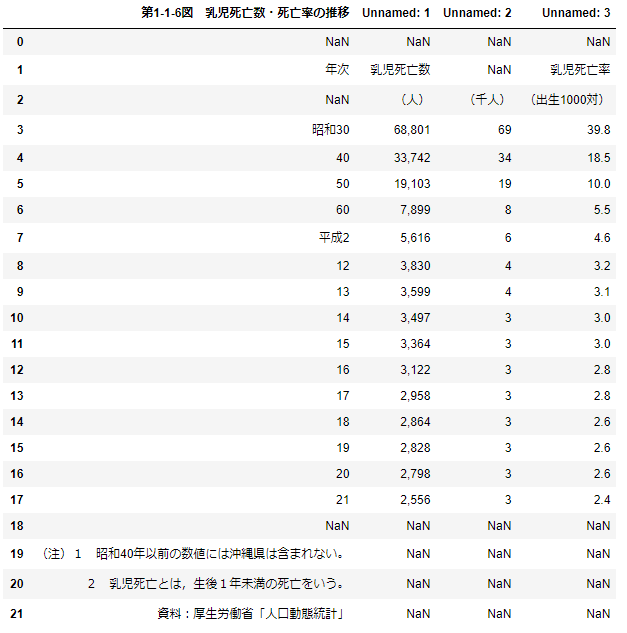
データは内閣府のページにある「平成23年版 子ども・若者白書」の乳児死亡数・死亡率の推移のデータを使用する。
「平成23年版 子ども・若者白書」乳児死亡数・死亡率の推移まずデータの読み込み
read_data.pyimport numpy as np from scipy.optimize import curve_fit import matplotlib.pyplot as plt import pandas as pd df=pd.read_csv('乳児死亡率.csv',encoding='cp932') # 文字化け防止でencoding='cp932' display(df)変な構成になっているので成形
adjust_data.pydf=df.iloc[3:18,:].rename(columns={'第1-1-6図 乳児死亡数・死亡率の推移':'年次'\ ,'Unnamed: 1':'乳児死亡数(人)'\ ,'Unnamed: 2':'乳児死亡数(千人)'\ ,'Unnamed: 3':'乳児死亡率'}) # 後々の処理のために連番カラム作成 rank=range(1,len(df)+1) df['rank']=rank # すべてのカラムがobject型なので乳児死亡率をfloat型に df['乳児死亡率']=df['乳児死亡率'].astype(float) df['乳児死亡数(人)']=df['乳児死亡数(人)'].str.replace(',','').astype(np.int) display(df)乳児死亡率をplot
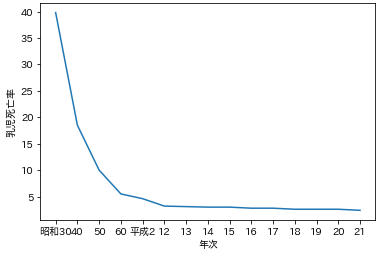
plot.pyx=df['年次'] y=df['乳児死亡率'] ax=plt.subplot(1,1,1) ax.plot(x,y) ax.set_xlabel('年次') ax.set_ylabel('乳児死亡率') plt.show()
赤ん坊の死亡率は60年前から比べるとかなり下がってるんだなぁ。医療の進歩すごい。
ではいよいよ近似のパラメーターを求める段階へ。scipyのcurve_fitで非線形回帰
func.pydef exp_func(x, a, b): return b*(x**a) def exp_fit(val1_quan, val2_quan): # maxfev:関数の呼び出しの最大数, check_finite:Trueの場合NaNが含まれている場合はValueError発生 l_popt, l_pcov = curve_fit(exp_func, val1_quan, val2_quan, maxfev=10000, check_finite=False) return exp_func(val1_quan, *l_popt),l_poptexp_funcのパラメータ$a$と$b$をexp_fitを使って求める。
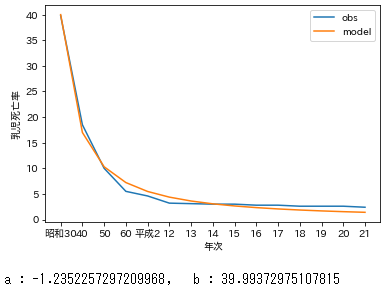
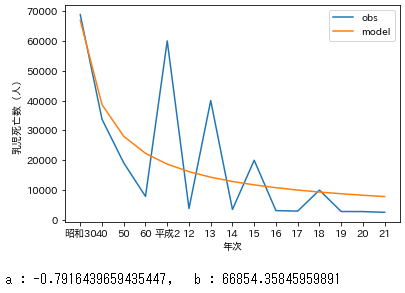
culc_params.pyx=df['年次'] x2=df['rank'] y=df['乳児死亡率'] y_fit,l_popt=exp_fit(x2,y) ax=plt.subplot(1,1,1) ax.plot(x,y,label='obs') ax.plot(x,y_fit,label='model') ax.set_xlabel('年次') ax.set_ylabel('乳児死亡率') plt.legend() plt.show() print('a : {}, b : {}'.format(l_popt[0],l_popt[1]))#求めたパラメータa,bを確認良い感じ。
EXCELの累乗近似(対数変換して線形回帰)の再現
func2.pydef exp_func_log(x, a, b): return a*np.log(x) + np.log(b) def exp_func_log_fit(val1_quan, val2_quan): l_popt, l_pcov = curve_fit(exp_func_log, val1_quan, np.log(val2_quan), maxfev=10000, check_finite=False) return exp_func_log(val1_quan, *l_popt),l_popt def log_to_exp(x,a,b): return np.exp(a*np.log(x) + np.log(b))exp_func_logのパラメータ$a$と$b$をexp_func_log_fitを使って求める。
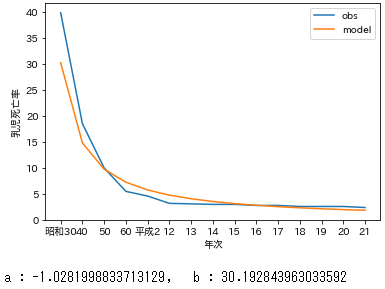
求めたパラメータ$a$と$b$を使って近似した$Y$は$\ln y$なのでlog_to_expで対数から変換して戻す。culc_params2.pyx=df['年次'] x2=df['rank'] y=df['乳児死亡率'] y_fit,l_popt=exp_func_log_fit(x2,y) y_fit=log_to_exp(x2,l_popt[0],l_popt[1]) ax=plt.subplot(1,1,1) ax.plot(x,y,label='obs') ax.plot(x,y_fit,label='model') ax.set_xlabel('年次') ax.set_ylabel('乳児死亡率') plt.legend() plt.show() print('a : {}, b : {}'.format(l_popt[0],l_popt[1])) #求めたパラメータa,bを確認
良い感じだけど、直接非線形回帰した方が当てはまりがよかった気がする。まとめ
どっちが適切とかはよくわからない。
けど「EXCELではできたのにPythonで同じにならない!」っていう状況に陥ったらこれを思い出してみてもいいかもしれない。追記(2020/02/28執筆直後)
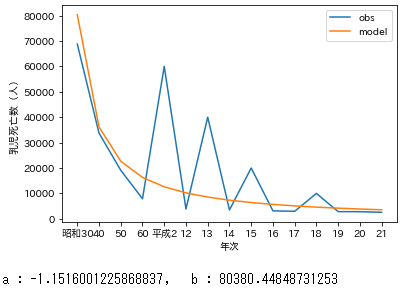
データの数値が大きい かつ ぶれている時、非線形回帰での近似は数値の大きいぶれに引っ張られやすい。
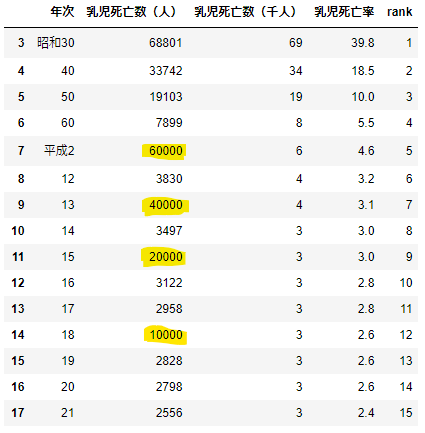
汎化能力的な観点で言うと、対数変換して線形回帰の方が良いのかもしれない。乳児死亡数(人)にダミーの数値を挿入する
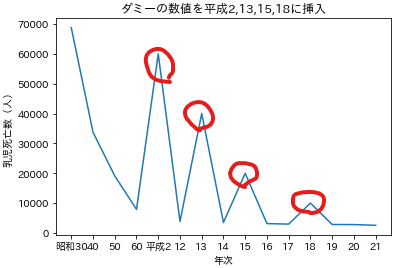
dummydata.pydf=pd.read_csv('乳児死亡率.csv',encoding='cp932') df=df.iloc[3:18,:].rename(columns={'第1-1-6図 乳児死亡数・死亡率の推移':'年次'\ ,'Unnamed: 1':'乳児死亡数(人)'\ ,'Unnamed: 2':'乳児死亡数(千人)'\ ,'Unnamed: 3':'乳児死亡率'}) # 後々の処理のために連番カラム作成 rank=range(1,len(df)+1) df['rank']=rank # すべてのカラムがobject型なので乳児死亡率をfloat型に df['乳児死亡率']=df['乳児死亡率'].astype(float) df['乳児死亡数(人)']=df['乳児死亡数(人)'].str.replace(',','').astype(np.int) # ダミーデータを挿入する df2=df.copy() df2.loc[df2['年次']=='平成2', '乳児死亡数(人)']=60000 df2.loc[df2['年次']=='13', '乳児死亡数(人)']=40000 df2.loc[df2['年次']=='15', '乳児死亡数(人)']=20000 df2.loc[df2['年次']=='18', '乳児死亡数(人)']=10000 display(df2) x=df2['年次'] y=df2['乳児死亡数(人)'] ax=plt.subplot(1,1,1) ax.plot(x,y) ax.set_xlabel('年次') ax.set_ylabel('乳児死亡数(人)') ax.set_title('ダミーの数値を平成2,13,15,18に挿入') plt.show()本編で使った関数を再度使用して近似曲線を引く
dummydata.py# 非線形回帰 x=df2['年次'] x2=df2['rank'] y=df2['乳児死亡数(人)'] y_fit,l_popt=exp_fit(x2,y) ax=plt.subplot(1,1,1) ax.plot(x,y,label='obs') ax.plot(x,y_fit,label='model') ax.set_xlabel('年次') ax.set_ylabel('乳児死亡数(人)') plt.legend() plt.show() print('a : {}, b : {}'.format(l_popt[0],l_popt[1])) # 対数変換線形回帰 x=df2['年次'] x2=df2['rank'] y=df2['乳児死亡数(人)'] y_fit,l_popt=exp_func_log_fit(x2,y) y_fit=log_to_exp(x2,l_popt[0],l_popt[1]) ax=plt.subplot(1,1,1) ax.plot(x,y,label='obs') ax.plot(x,y_fit,label='model') ax.set_xlabel('年次') ax.set_ylabel('乳児死亡数(人)') plt.legend() plt.show() print('a : {}, b : {}'.format(l_popt[0],l_popt[1]))明らかに非線形回帰で近似した方がダミーで入れた数値のぶれに引っ張られている。
区別して状況によって使い分けることが重要だと思われる。
- 投稿日:2020-02-28T18:29:59+09:00
datetime と ファイル書き込み と バックアップ
import os import shutil import datetime now = datetime.datetime.now() file_name = 'test.txt' #test.txtというファイルがあれば、 #日時をファイル名に含めたバックアップファイルをつくる if os.path.exists(file_name): shutil.copy(file_name, '{}.{}'.format( file_name, now.strftime('%Y_%m_%d_%H_%M_%S'))) #test.txtという名前で中身がtestであるファイルをつくる with open(file_name, 'w') as f: f.write('test')
- 投稿日:2020-02-28T17:55:25+09:00
高校数学でわかる単回帰分析〜ムーアの法則の検証
はじめに
単回帰分析についてはscikit-learnのlinear_model.LinearRegression()モデルを用いれば簡単に行うことができます。ここではは単回帰分析の原理をおさらいしたうえでLinearRegression()を使わずに地道に単回帰分析を行います。具体的にはCPUの一覧データを用いて「半導体回路の集積密度は1年半~2年で2倍となる」というムーアの法則が経験的に成り立つことを示して行きたいと思います。
単回帰分析の原理
単回帰分析は目的変数(y)を1つの説明変数(x)で予測するもので、それらの間の関係性をy=ax+bという一次式の形で表します。例えば、生徒たちの学習時間(x)と成績(y)のように、サンプルデータとしてxとyの組が複数得られている場合、これらのデータに基づき、例えば、新たな学習時間(x)が分かったとき、対応する成績(y)予測を可能にするのが単回帰分析です。
単回帰分析を行うことは、直線y=ax+bを求めることに相当します。では、傾きa及びy切片bはどのように求めるのでしょうか。
ここで最小二乗法の概念が登場してきます。最小二乗法という手法を用いるとa,bを求めることができるのです。求める回帰直線を以下のように表現すると、
\hat{y}= ax+b最小二乗法は以下のEを最小にするa, bを求めることに相当します。
E = \sum_{i=1}^{N} (y_i - \hat{y_i})^2 = \sum_{i=1}^{N} (y_i - (ax_i + b))^2 ・・・ (1)(y_iはサンプルの各データポイントのy座標を、\hat{y_i}は直線\hat{y}= ax+b上の予測値のy座標を意味します。)
(余談ですが、以下のように単にデータポイントと予測値の差の和をとり、それを最小にする手法ではだめなのでしょうか。\sum_{i=1}^{N} (y_i - \hat{y_i})少し考えればわかるかもしれませんが、データポイントが予測値の上下に存在する場合、お互いキャンセルアウトしてしまい、見かけより全体の差分が少なく算出されてしまうため、二乗する必要があるのですね。)
さて、本題に戻りましょう。では上記(1)のEを最小にするa,bのペアを求めるためにはどうしたらいいでしょうか。(1)式はa,bを2変数とした2次式とみなすことができます(xi,yiは既知のため定数とみなすことができます)。
すなわち、a,bについて(1)式をそれぞれ偏微分し、それらが=0になるa,bのペアを求めてやればEを最小にするa,bを求めることができるのです。
では早速計算していきましょう。
\frac{\partial E}{\partial a} = \sum_{i=1}^{N} 2(y_i - (ax_i+b))(-x_i) =2(-\sum_{i=1}^{N} x_iy_i + a\sum_{i=1}^{N}x_i^2+b\sum_{i=1}^{N}x_i)=0\frac{\partial E}{\partial b} = \sum_{i=1}^{N} 2(y_i - (ax_i+b))(-1) =2(-\sum_{i=1}^{N} y_i + a\sum_{i=1}^{N}x_i+b\sum_{i=1}^{N}1)=0すなわち、
a\sum_{i=1}^{N}x_i^2+b\sum_{i=1}^{N}x_i=\sum_{i=1}^{N} x_iy_i ・・・ (2)a\sum_{i=1}^{N}x_i+bN=\sum_{i=1}^{N} y_i ・・・ (3)ここで
A =\sum_{i=1}^{N}x_i^2,\quad B =\sum_{i=1}^{N}x_i,\quad C =\sum_{i=1}^{N}x_iy_i,\quad D =\sum_{i=1}^{N}y_iとおくと(2), (3)は
aA+bB=C ・・・ (2)'aB+bN=D ・・・ (3)'とできます。この連立方程式を地道に解いてみましょう(見づらさ解消のため以下ではΣの添字を省略します)。
(2)'\times N - (3)'\times B より\\ a(AN-B^2)=CN-BD\\ \therefore a = \frac{CN-BD}{AN-B^2}=\frac{N\sum x_iy_i-\sum x_i\sum y_i}{N\sum x_i^2-(\sum x_i)^2}\\=\frac{\bar{xy}-\bar{x}\bar{y}}{\bar{x^2}-\bar{x}^2} \quad(分母分子をN^2で割った) ・・・ (4)同様にして、
(2)'\times B - (3)'\times A より\\ b(B^2-AN)=BC-AD\\ \therefore b = \frac{BC-AD}{B^2-AN}=\frac{\sum x_i\sum x_iy_i-\sum x_i^2\sum y_i}{(\sum x_i)^2-N\sum x_i^2}=\frac{\sum x_i^2\sum y_i-\sum x_i\sum x_iy_i}{N\sum x_i^2-(\sum x_i)^2}\\=\frac{\bar{x^2}\bar{y}-\bar{x}\bar{xy}}{\bar{x^2}-\bar{x}^2} \quad(分母分子をN^2で割った) ・・・ (5)これで、Eを最小にするa,bのペアが求まったことになります。サンプルデータのデータポイントのx座標やy座標を(4),(5)の式に代入してあげれば簡単にa,bを求めることができるのですね。
求める回帰直線は最終的に(4), (5)から\hat{y}=ax+b=\frac{\bar{xy}-\bar{x}\bar{y}}{\bar{x^2}-\bar{x}^2}x+\frac{\bar{x^2}\bar{y}-\bar{x}\bar{xy}}{\bar{x^2}-\bar{x}^2} ・・・ (6)と表すことができます。
ムーアの法則
ここまで長々と単回帰分析の原理を見てきました。
さてここで単回帰分析を用いて「半導体回路の集積密度は1年半~2年で2倍となる」というムーアの法則が経験的に成り立っているのかを見ていきたいと思います。まずはこちらを参考にCPUのプロセッサー数の歴史的変遷を示した一覧表のCSVをスクレイピングにより取得してみましょう。下準備
データが取得できたら、早速コーディングしていきましょう。まずは取得したデータを見てみましょう。
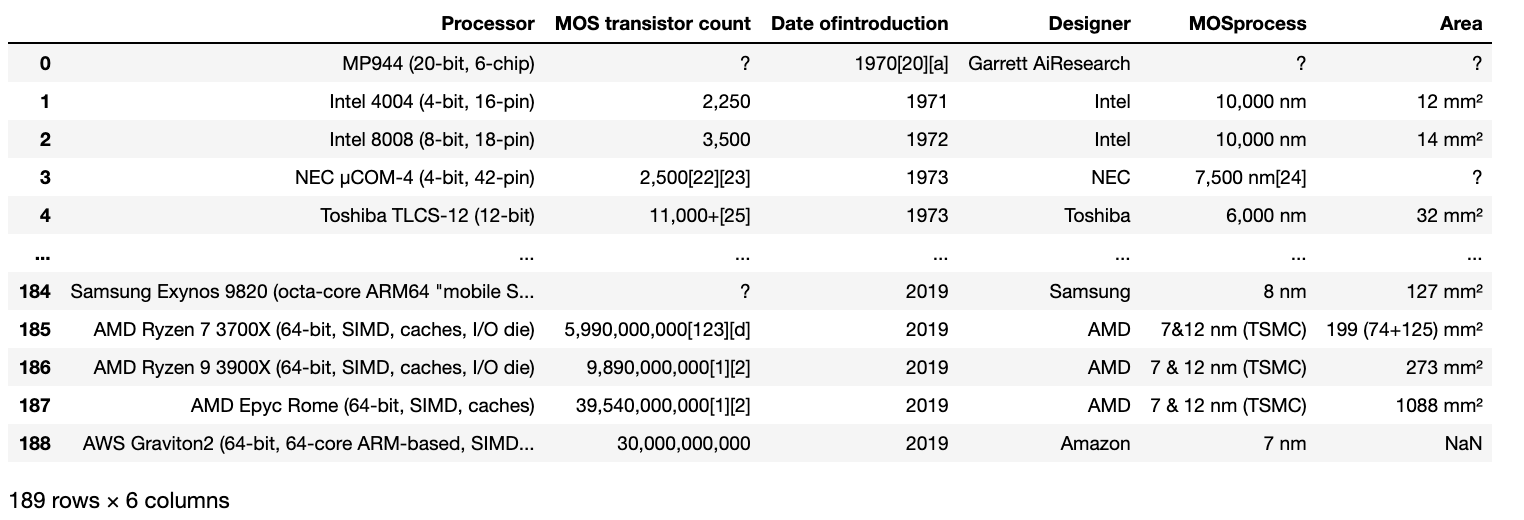
import re import pandas as pd import numpy as np import matplotlib.pyplot as plt df = pd.read_csv('processor.csv', sep='\t') df続いて、CPUのリリース年とトランジスタ数のみ抽出し新たにdf1として出力してみましょう。
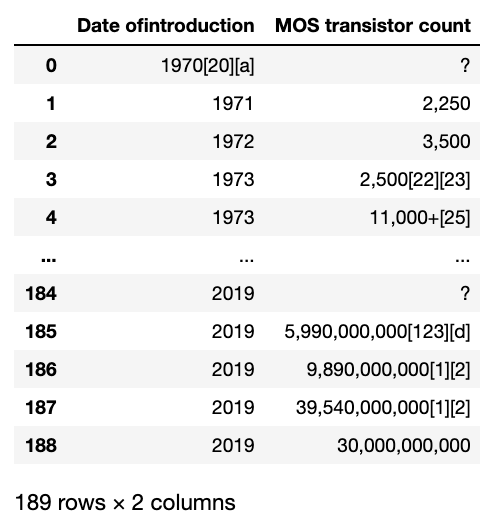
df1 = pd.concat([df['Date ofintroduction'], df['MOS transistor count'] ], axis=1) df1以下のようになります。
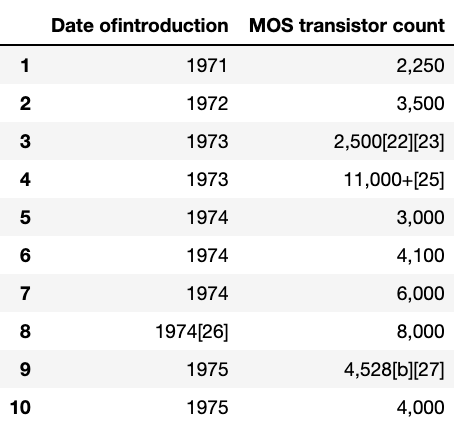
transistor countのデータのうち、「?」となっているものは利用できませんので排除してしまいましょう。df2 = df1[df1['MOS transistor count'] != '?'] df2.head(10)データをみると、リリース年やトランジスタ数のあとに注釈の[]が残っていてしまっていますね。また、トランジスタ数の3桁区切りのカンマが邪魔なのでこれらを以下の操作により除去していきましょう。
# 数字以外のもの(3桁区切りカンマ)除去のために指定 decimal = re.compile(r'[^\d]') # リリース年について注釈の「[」の左側部分のみ抽出し、「year」カラムとしてあらたにdf2に追加 df2['year'] = df2['Date ofintroduction'].apply(lambda x: int(x.split('[')[0])) # トランジスタ数について最初にマッチする数字部分(カンマ含む)を抽出しその後カンマ除去、「transistor count」カラムとしてあらたにdf2に追加 df2['transistor count'] = df2['MOS transistor count'].apply(lambda x: int(decimal.sub('', re.match(r'[\d, \,]+', x).group() ))) # 出力 df2結果は以下のようになり、あらたに「year」、「transistor count」カラムが追加されきれいなデータが入っているのがわかると思います。
プロット
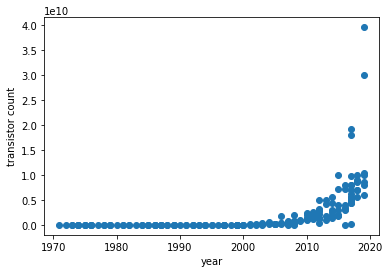
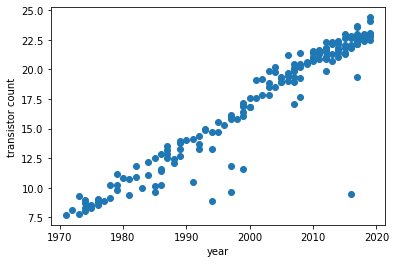
きれいにしたデータをもとに横軸「year」、縦軸「トランジスタ数」の散布図を描いてみましょう。
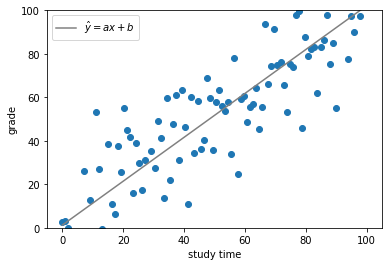
# サンプルデータの散布図 X = df2['year'] Y = df2['transistor count'] plt.scatter(X, Y) plt.xlabel('year') plt.ylabel('transistor count') plt.show()トランジスタ数が指数関数的に増加しているのが見て取れますね。
さて、ここでトランジスタ数の対数をとりプロットし直してみましょう。
# サンプルデータの対数散布図 Y = np.log(Y) plt.scatter(X, Y) plt.xlabel('year') plt.ylabel('transistor count') plt.show()ここでこのプロットに単回帰分析を適用してみましょう。「単回帰分析の原理」で導出した(6)式をコードに落とし込んでみましょう。
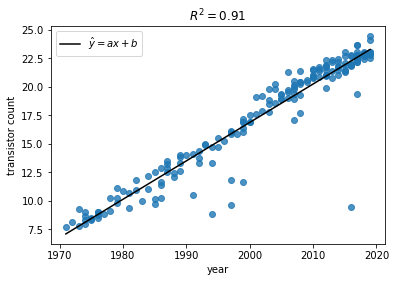
# サンプルデータの対数散布図 Y = np.log(Y) plt.scatter(X, Y ,alpha=.8) # 回帰直線の導出 denom = (X**2).mean() - (X.mean())**2 # a, bの分母 a = ( (X*Y).mean() - X.mean()*Y.mean() ) / denom b = ( Y.mean()*(X**2).mean() - X.mean()*(X*Y).mean() ) / denom # 回帰直線の式 pred_Y = a*X + b # R-squaredの計算 SSE_l = (Y - pred_Y).dot( (Y - pred_Y) ) SSE_m = (Y - Y.mean()).dot( (Y - pred_Y.mean()) ) r2 = 1 - SSE_l / SSE_m plt.plot(X, pred_Y, 'k', label='$\hat{y}=ax+b$') plt.title('$R^2 = %s$'%round(r2,2)) plt.ylabel('transistor count') plt.xlabel('year') plt.legend() plt.show()結果は以下のようになりました。(R-squared=0.91)
(ここでR-squaredについての補足説明をしますと、R-squaredは決定係数とも呼ばれ、回帰直線の予測モデルとしての妥当性を示す指標です。数式で表すと以下のように表すことができます(SqEはsquared errorの略です)。
R^2 = 1 - \frac{\sum SqE_{line}}{\sum SqE_{mean}}= 1 - \frac{\sum (y_i - \hat{y_i})^2}{\sum (y_i - \bar{y})^2}この式ではmean、つまりサンプルデータの平均ラインを基準としたときのデータポイントのばらつきと、回帰直線を基準としたときのデータポイントのばらつきを比較がなされます。例えば、ΣSqElineの値がΣSqEmeanの値と等しくなる時、R2は0となり、回帰直線はデータポイントの平均値直線に一致し、予測モデルとして全くといっていいほど機能していないことを表します(回帰直線にはデータの代表値である平均直線以上としての機能が求められるべきです)。一方、ΣSqElineが0のとき、回帰直線上には全てのデータポイントが乗っており、予測モデルはデータポイントを完全に予測できていることになります。この時のR2は1となります。)
ムーアの法則の検証
さて、得られた回帰直線をもとにムーアの法則をざっくりと検証してみましょう。
今まで見てきたように、トランジスタ数(tc)と回帰直線の関係は以下のように表すことができます。log(tc) = ax + b \\ \therefore tc = \exp(ax + b)よってある年(x2)にある年(x1)のトランジスタ数の2倍のトランジスタ数に到達するとすると、
2 = \frac{\exp(ax_2 + b)}{\exp(ax_1 + b)} = exp(a(x_2-x_1))\\ \therefore log2 = a(x_2-x_1)\\ \therefore x_2 - x_1 = \frac{log2}{a}つまり回帰直線の傾きaから計算したlog2/aはトランジスタ数が2倍にになるまでにかかる年数を意味することになります。早速これをもとに年数を計算してみましょう。
print("time to double:", round(np.log(2)/a,1), "years")outputtime to double: 2.1 yearsトランジスタ数が2倍になるまでは約2年という結果になりました。
ムーアさん、すごいですね。
- 投稿日:2020-02-28T17:51:31+09:00
将来の売上予測への挑戦:③PyFluxのパラメーターチューニング
はじめに
前回、「将来の売上予測への挑戦:②PyFluxを用いた時系列分析」で、PyFluxを用いたARIMA, ARIMAXのモデル構築を行いました。
ただ、あまり良い精度が出ませんでした。
パラメーターである、ARやMAの次元数などを手探りで行っていったのですが、そこがイマイチだったのでしょうか。
とはいえ、「統計的に見てこれがいい!」というのは、私には中々ハードルが高いもの(汗そこで、scikit-learnのGridSearchみたいなものがないかと調べてみました。
すると、「TV朝日の視聴率推移をSARIMAモデルで予測してみる」というページで、StatsModelsを用いた時系列分析のパラメーターチューニングを実装されていたので、そちらを参考に作ってみました。分析環境
Google Colaboratory
対象とするデータ
前回同様に、データは日別の売上と説明変数として気温(平均・最高・最低)を用います。
日付 売上金額 平均気温 最高気温 最低気温 2018-01-01 7,400,000 4.9 7.3 2.2 2018-01-02 6,800,000 4.0 8.0 0.0 2018-01-03 5,000,000 3.6 4.5 2.7 2018-01-04 7,800,000 5.6 10.0 2.6 パラメーターチューニング
下記が、前回のプログラムです。
ar, ma, integがパラメーターです。import pyflux as pf model = pf.ARIMA(data=df, ar=5, ma=5, integ=1, target='売上金額', family=pf.Normal()) x = model.fit('MLE')と、ここまでパラメーターチューニングと偉そうに申してきましたが、基本的には各パラメーターともに整数を取るので、数値をループでグルグル回しています。
def optimisation_arima(df, target): import pyflux as pf df_optimisations = pd.DataFrame(columns=['p','d','q','aic']) max_p=4 max_d=4 max_q=4 for p in range(0, max_p): for d in range(0, max_d): for q in range(0, max_q): model = pf.ARIMA(data=df, ar=p, ma=q, integ=d, target=target, family=pf.Normal()) x = model.fit('MLE') print("AR:",p, " I:",d, " MA:",q, " AIC:", x.aic) tmp = pd.Series([p,d,q,x.aic],index=df_optimisations.columns) df_optimisations = df_optimisations.append( tmp, ignore_index=True ) return df_optimisationsこれで、次のように呼び出すと
df_output = optimisation_arima(df, "売上金額")結果が表示されます。PyFluxの評価基準はいくつかありますが、AIC(小さい方がいいモデル)を用いています。
AR: 0 I: 0 MA: 0 AIC: 11356.163772323638 AR: 0 I: 0 MA: 1 AIC: 11262.28357561013 AR: 0 I: 0 MA: 2 AIC: 11218.453940684196 AR: 0 I: 0 MA: 3 AIC: 11171.121950637687 AR: 0 I: 1 MA: 0 AIC: 11462.586538415879それで、最もAICが小さかったAR/I/MAの組み合わせを、最適なパラメーターとして選択することができます。
df_optimisations[df_optimisations.aic == min(df_optimisations.aic)]おわりに
前回の精度が惨敗だったため、総当りのパラメーターチューニングを、行いました。
次なる改善策を考えないといけないですね。
- 投稿日:2020-02-28T17:02:56+09:00
AtCoder Beginner Contest 047 過去問復習
所要時間
感想
簡単すぎて違和感がすごいです。やはり、最近のABCはレベルが高すぎますね…。
A問題
小さいやつを足して大きいやつにできるかどうか
answerA.pyx=list(map(int,input().split())) x.sort() if x[0]+x[1]==x[2]: print("Yes") else: print("No")B問題
長方形の面積を求めるには左下のx,y座標と右上のx,y座標がわかれば良く、与えられる情報から長方形は狭まっていくのでaの値を元に場合分けをすれば良い。また、a2-a1とa4-a3はそれぞれ-になる可能性があるが、その場合は長方形ができないので0にする必要がある。
answerB.pyw,h,n=map(int,input().split()) a1,a2,a3,a4=0,w,0,h for i in range(n): x,y,a=map(int,input().split()) if a==1: a1=max(a1,x) elif a==2: a2=min(a2,x) elif a==3: a3=max(a3,y) else: a4=min(a4,y) print(max(a2-a1,0)*max(a4-a3,0))C問題
一回の操作により一つの連続する色の区間が反転することがわかるので、この操作により区間の数が一つ減ることがわかる。したがって、この操作を繰り返して全てを一つの区間にすれば良いので、区間の数-1が求める石の個数となる。また、区間の数は自作のgroupby関数を用いることで容易に求めることができる。
answerC.pydef groupby(a): a2=[[a[0],1]] for i in range(1,len(a)): if a2[-1][0]==a[i]: a2[-1][1]+=1 else: a2.append([a[i],1]) return a2 s=groupby(input()) print(len(s)-1)D問題
問題文が非常に長いので難問かと身構えたのですが、何も難しくはなく拍子抜けしてしまいました。(身構えすぎで時間がかかったので直していきたいです。長文に慣れる!)
まず、初めに持っているリンゴが0個であり、T個合計で売り買いすることから、金額の差が最大になる街でまとめてT//2個売り買いすれば利益が最大になることが分かります。
したがって、調べたいのは金額の差が最大になる街の組(i,j)になります。ここで街の組の両方とも動かすと計算量が多くなりそうなので、二つの街のうちリンゴを売る街jに注目しました。この時、リンゴを買うべき街iは一意に決まります。なぜなら街1~j-1のうちリンゴが一番安い街であるからです。また、この際、それぞれの街jについて街1~j-1のうちリンゴが一番安い街を求めていると計算量が0($n^2$)で間に合わないので、累積和のように隣り合うものの最小を求めるようにしました("街1~jでの最小"は"街1~j-1での最小と街jの間での最小"に等しい。)。
よって、金額の差が最大になる街の組について片一方のリンゴの値段を(一円だけ)下げるor上げることで利益を下げることができるので、このような街の組の個数を数え上げれば良いです(それぞれの街でリンゴの値段が異なるので単純に組の個数を数え上げれば良いです。同じ値段のリンゴがあるとより少ないコストで利益を下げることができますが、制約により異なる街で同じ値段のリンゴは存在しません。)。answerD.pyn,t=map(int,input().split()) a=list(map(int,input().split())) x=[0]*n x[0]=a[0] for i in range(1,n): x[i]=min(x[i-1],a[i]) d=dict() for i in range(1,n): y=a[i]-x[i-1] if y>0: if y in d: d[y]+=1 else: d[y]=1 d=list(d.items()) d.sort(reverse=True) print(d[0][1])
- 投稿日:2020-02-28T16:48:20+09:00
【Python初心者】結局どうやってPython開発して実行すればいいのさ?
本稿で伝えたいこと
これは備忘録ですが、私と重なる人がいれば参考になる!
かもしれません。私はだあれ?
- Pythonやりたい
- ネットからの情報収集ルーティーンがめんどい
- とりあえずPythonインストールしてみた
- とりあえずVisualStudioCodeをインストールしてみた
- とりあえずATOMインストールしてみた
- とりあえずDjangoインストールしてみた
- とりあえずAnacondaインストールしてみた
- とりあえずPyCharm Professionalインストールしてみたけど、あまり触ることなく30日間の無料トライアル期間が終わった
- で、結局どの環境でPythonやったらいいか分からない
3つくらい当てはまる人がいれば、とりあえず見てみても損ないかもしれません。
とりあえず全部一回消す
いろいろインストールしてしまったせいで、PC内はごちゃごちゃです。
なので、一旦Python絡みで入れたものはアンインストールします。はい、これで一旦スッキリしました。
とりあえずAnaconda入れとこ
適当にAnacondaに決めたわけではないです。
Anacondaさえ入れとけば、とりあえず漏れなく操作できそうだから。
パッケージ(要は定食みたいなもの)なので。「食うもん迷ったら、とりあえず定食頼め!」って、
おじいちゃんが言ってたので。ライブラリもpipに比べると少ないようですが、当面は困らないでしょうし、
コード記述&即実行で確認できて、かつ、勉強ノートとして保存できるJupyterNotebookもあるし、
知名度高い統合開発環境「Spyder」もありますしね。今の私がやってみたいことは、Anaconda入れとけば網羅してそうなんです。
一息ついたら触ってみよう
こんな感じで進めようと思っています。
- JupyterNotebookでお勉強(コード書いて実行して~の繰り返し)
- Spyderで情報収集するやつを作る
- 作ったWebアプリをタスク実行とかで馬車馬のように働いてもらう!まとめ
Pythonのにわか知識が渋滞したら、一旦リセットしてシンプルに行こう!ってことです。
最後に備忘録ぽいこと、「Pythonやるときは必ず仮想環境でね」。
でないと、ライブラリ同士が干渉してしまったり、余計なエラーで時間とられちゃうかもしれないですよ。ってどこかで見かけました。
- 投稿日:2020-02-28T15:48:37+09:00
NumPyのzerosは大きさ0でも定義できるよという話
概要
- NumPyでは
np.zeros((0, 4))みたいに大きさ0のzerosを定義できる- これを使うと空リストを定義してappend()で追加していくみたいなことがarrayでもできる
NumPyのarrayでもlist.append()みたいなことがやりたい
リストはappendが使える
Pythonでリストを作るときによくやるのが
a = [] for i in range(10): a.append(1)みたいなやつ
Arrayにはappendはない
NumPyのArrayで同じことをやろうとするとnumpy.vstack()やnumpy.hstack()を使うことになる
→ くっつける2つのArrayの行数(or 列数)が合わないといけないので、初めにshape=(1, 4)のArrayを定義してましたダメな方import numpy as np a = np.zeros((1, 4)) for i in range(10): ones = np.ones((1, 4)) a = a.vstack((a, ones)) a = a[1:, :] # 最初に定義した部分だけ除いてる大きさ0のArrayが定義できる
初めにshape=(1, 4)のArrayを定義してから除く操作が気持ち悪く感じていたので、試しに大きさ0のzerosを定義したらいけた
見やすい方import numpy as np a = np.zeros((0, 4)) # 大きさ0のzeros for i in range(10): ones = np.ones((1, 4)) a = a.vstack((a, ones))
- 投稿日:2020-02-28T15:43:08+09:00
コンテナ上のpython27/pyodbcでホストのSQL Serverにアクセスする
概要
先日の以下の記事でmysqlにアクセスする方法をご紹介しましたが、
コンテナ上のpython3/mysql-connector-pythonでホストのmysqlにアクセスする
SQL serverバージョンをご紹介します。前提
ホストSQL Server
ODBC Driver 17 for SQL Server
SQLServer認証でポート1443で動作している手順
ODBCが動くDockerコンテナの作り方の記事内にある以下のリポジトリをcloneします。
https://github.com/Microsoft/mssql-docker/tree/master/oss-drivers/pyodbc> git clone https://github.com/microsoft/mssql-docker.git > cd mssql-docker/oss-drivers/pyodbc以下のツリーになっていると思います。
pyodbc ├── Dockerfile ├── README.md ├── docker-compose.yml *docker-composeで立ち上げるために追加 ├── entrypoint.sh └── sample.py *編集します。もしくは別ファイルを作ってもよいです。touch入ってませんが...Dockerfile# mssql-python-pyodbc # Python runtime with pyodbc to connect to SQL Server FROM ubuntu:16.04 # apt-get and system utilities RUN apt-get update && apt-get install -y \ curl apt-utils apt-transport-https debconf-utils gcc build-essential g++-5\ && rm -rf /var/lib/apt/lists/* # adding custom MS repository RUN curl https://packages.microsoft.com/keys/microsoft.asc | apt-key add - RUN curl https://packages.microsoft.com/config/ubuntu/16.04/prod.list > /etc/apt/sources.list.d/mssql-release.list # install SQL Server drivers RUN apt-get update && ACCEPT_EULA=Y apt-get install -y msodbcsql unixodbc-dev # install SQL Server tools RUN apt-get update && ACCEPT_EULA=Y apt-get install -y mssql-tools RUN echo 'export PATH="$PATH:/opt/mssql-tools/bin"' >> ~/.bashrc RUN /bin/bash -c "source ~/.bashrc" # python libraries RUN apt-get update && apt-get install -y \ python-pip python-dev python-setuptools \ --no-install-recommends \ && rm -rf /var/lib/apt/lists/* # install necessary locales RUN apt-get update && apt-get install -y locales \ && echo "en_US.UTF-8 UTF-8" > /etc/locale.gen \ && locale-gen RUN pip install --upgrade pip # install SQL Server Python SQL Server connector module - pyodbc RUN pip install pyodbc # install additional utilities RUN apt-get update && apt-get install gettext nano vim -y # add sample code RUN mkdir /sample ADD . /sample WORKDIR /sample CMD /bin/bash ./entrypoint.shdocker-compose.yamlversion: '3' services: pyodbc: restart: always build: . container_name: 'pyodbc' working_dir: '/sample' tty: true volumes: - .:/sample extra_hosts: - "(ホストのCOMPUTERNAME):(ホストのIPアドレス)" ports: - 1443:1443sample.pyimport pyodbc server = '(ホストのIPアドレス),(ホストのSQLServer動作ポート)' username = 'sa' password = 'password' cnxn = pyodbc.connect('DRIVER={ODBC Driver 17 for SQL Server};SERVER='+server+';database=(データベース名);UID='+username+';PWD='+password) cursor = cnxn.cursor() print ('Using the following SQL Server version:') tsql = "SELECT @@version;" with cursor.execute(tsql): rows = cursor.fetchall() for row in rows: print(str(row))> docker-compose up -d --build ... > docker container ps CONTAINER ID IMAGE COMMAND CREATED STATUS PORTS NAMES 2c51574bfd36 pyodbc_pyodbc "/bin/sh -c '/bin/ba…" 51 minutes ago Up 51 minutes 0.0.0.0:1443->1443/tcp pyodbc > docker exec -it 2c51574bfd36 /bin/bash $ python --version Python 2.7.12 $ python sample.py Using the following SQL Server version: (u'Microsoft SQL Server 2017 (RTM) - 14.0.1000.169 (X64) \n\tAug 22 2017 17:04:49 \n\tCopyright (C) 2017 Microsoft Corporation\n\tExpress Edition (64-bit) on Windows 10 Pro 10.0 <X64> (Build 18362: ) (Hypervisor)\n', )結果
無事SQL ServerにアクセスしDB参照できました。
後日談(2/28追記内容)
ヒアドキュメントを文字列代入した時にエラー発生
変更内容
sample.pytsql = '''\ SELECT * FROM ~ INNER JOIN ~ ON '''エラー内容
$ python sample.py File "sample.py", line 17 SyntaxError: Non-ASCII character '\xe7' in file sample.py on line 18, but no encoding declared; see http://python.org/dev/peps/pep-0263/ for details File "sample.py", line 54, in <module> with cursor.execute(tsql): UnicodeDecodeError: 'ascii' codec can't decode byte 0xe7 in position 48: ordinal not in range(128)試行した手順
以下の記事で解決しましませんでした。
http://shu223.hatenablog.com/entry/20111201/1328334689以下の記事で解決しませんでした。
https://qiita.com/chatrate/items/eb4b05cd1a6652529fd9root@2c51574bfd36:/sample# python -c "import site; print (site.getsitepackages())"
['/usr/local/lib/python2.7/dist-packages', '/usr/lib/python2.7/dist-packages']解決した手順
以下を参考にしたら解決しました。
https://kaworu.jpn.org/python/Python%E3%81%AEUnicodeDecodeError%E3%81%AE%E5%AF%BE%E5%87%A6%E6%96%B9%E6%B3%95
with cursor.execute(tsql.decode('utf-8')):
- 投稿日:2020-02-28T15:10:49+09:00
UnionFind(相当)を10行で実装する
素集合データ構造(Union-Find)は、要素を互いに交わらないグループに分類するデータ構造です。標準的な実装方法はUnion-Find木です。・・・というのは皆さんすでにご存知と思います。
Union-Find木はQiitaにも実装例がたくさんあるのですが、私は以前から疑問に思っていました。
find()の再帰呼び出しがあって分かりにくい。もっと分かりやすく・短く書けないか?- 組み込みのコレクション型を利用した方が、実は高速なのではないか?
10行実装
そこで、Pythonでdictとfrozensetを使って短い実装を書いてみました。空白を含めても10行です。
class EasyUnionFind: def __init__(self, n): self._groups = {x: frozenset([x]) for x in range(n)} def union(self, x, y): group = self._groups[x] | self._groups[y] self._groups.update((c, group) for c in group) def groups(self): return frozenset(self._groups.values())比較してみた
Union-Find木による実装と比較してみます。
比較対象は、Qiitaで「Union Find Python」で検索して見つけた一番いいね数が多い記事で紹介されていた実装です。今回の10行実装の方が遅いという結果になりました。また、要素数が増えるに従って差が開いているようです。残念。
要素数 Union-Find木実装 今回の10行実装 所要時間の比 1000 0.72秒 1.17秒 1.63 2000 1.46秒 2.45秒 1.68 4000 2.93秒 5.14秒 1.75 8000 6.01秒 11.0秒 1.83 ただ、遅いといってもUnion-Find木の2倍弱程度なので、場合によっては利用価値がある・・・かもしれません。
比較用コードと実行結果
コード:
import random import timeit import sys import platform class EasyUnionFind: """ dict と frozenset を使った実装。 """ def __init__(self, n): self._groups = {x: frozenset([x]) for x in range(n)} def union(self, x, y): group = self._groups[x] | self._groups[y] self._groups.update((c, group) for c in group) def groups(self): return frozenset(self._groups.values()) class UnionFind(object): """ 典型的なUnion-Find木による実装。 https://www.kumilog.net/entry/union-find の実装例をコピーしたが、 今回不要なメンバ関数を削除し .groups() を追加した。 """ def __init__(self, n=1): self.par = [i for i in range(n)] self.rank = [0 for _ in range(n)] self.size = [1 for _ in range(n)] self.n = n def find(self, x): if self.par[x] == x: return x else: self.par[x] = self.find(self.par[x]) return self.par[x] def union(self, x, y): x = self.find(x) y = self.find(y) if x != y: if self.rank[x] < self.rank[y]: x, y = y, x if self.rank[x] == self.rank[y]: self.rank[x] += 1 self.par[y] = x self.size[x] += self.size[y] def groups(self): groups = {} for i in range(self.n): groups.setdefault(self.find(i), []).append(i) return frozenset(frozenset(group) for group in groups.values()) def test1(): """2つの実装の結果が同じかどうかテストする。差異があれば AssertionError を送出する。""" print("===== TEST1 =====") random.seed(20200228) n = 2000 for _ in range(1000): elements = range(n) pairs = [ (random.choice(elements), random.choice(elements)) for _ in range(n // 2) ] uf1 = UnionFind(n) uf2 = EasyUnionFind(n) for x, y in pairs: uf1.union(x, y) uf2.union(x, y) assert uf1.groups() == uf2.groups() print('ok') print() def test2(): """ 要素数を増やしながら、2つの実装の所要時間を出力する。 """ print("===== TEST2 =====") random.seed(20200228) def execute_union_find(klass, n, test_datum): for pairs in test_datum: uf = klass(n) for x, y in pairs: uf.union(x, y) timeit_number = 1 for n in [1000, 2000, 4000, 8000]: print(f"n={n}") test_datum = [] for _ in range(1000): elements = range(n) pairs = [ (random.choice(elements), random.choice(elements)) for _ in range(n // 2) ] test_datum.append(pairs) t = timeit.timeit(lambda: execute_union_find(UnionFind, n, test_datum), number=timeit_number) print("UnionFind", t) t = timeit.timeit(lambda: execute_union_find(EasyUnionFind, n, test_datum), number=timeit_number) print("EasyUnionFind", t) print() def main(): print(sys.version) print(platform.platform()) print() test1() test2() if __name__ == "__main__": main()実行結果:
3.7.6 (default, Dec 30 2019, 19:38:28) [Clang 11.0.0 (clang-1100.0.33.16)] Darwin-18.7.0-x86_64-i386-64bit ===== TEST1 ===== ok ===== TEST2 ===== n=1000 UnionFind 0.7220867589999997 EasyUnionFind 1.1789850389999987 n=2000 UnionFind 1.460918638999999 EasyUnionFind 2.4546459260000013 n=4000 UnionFind 2.925022847000001 EasyUnionFind 5.142797402000003 n=8000 UnionFind 6.01257184 EasyUnionFind 10.963117657000005
- 投稿日:2020-02-28T14:48:42+09:00
ISSが見える時間を予測する
はじめに
前回、PyEphemの機能を見てて、ISSを目視できる日時を予測できるんじゃないかなぁとなんとなく思ったんだけど、でもISSが見える条件ってなんだろう。
物理的な位置関係とかでごちゃごちゃ悩んで、こりゃ思ったより難しいていうか無理! って一瞬なったけど、よく考えるともしかして次の3つの条件を満たせばいいんじゃなかろうか。
- 観測者から見たISSの仰角が正
- 観測者から見た太陽の仰角が負
- ISSが食じゃない
試してみる。
import ephem import requests import datetime from math import degrees as deg tle_url = 'https://spaceflight.nasa.gov/realdata/sightings/SSapplications/Post/JavaSSOP/orbit/ISS/SVPOST.html' loc = ('35.689922','139.692122',41) # 都庁 home = ephem.Observer() home.lat = loc[0] home.lon = loc[1] home.elevation = loc[2] sun = ephem.Sun() doc = requests.get(tle_url).text lines = doc.split('\n') for idx in range(len(lines)): if lines[idx].strip() == 'TWO LINE MEAN ELEMENT SET': iss = ephem.readtle(lines[idx+2], lines[idx+3], lines[idx+4]) break utcnow = datetime.datetime.utcnow().replace(second=0,microsecond=0) utcnow += datetime.timedelta(days=0) # 確認用 for after in range(0, 60*24*10, 1): # 予測期間(m),予測間隔(m) when = utcnow + datetime.timedelta(minutes=after) home.date = when sun.compute(home) alt_sun = deg(sun.alt) iss.compute(home) alt_iss = deg(iss.alt) az_iss = deg(iss.az) iss.compute(when) ecl = iss.eclipsed if alt_sun <= -4 and alt_iss >= 10 and not(ecl): jst = when+datetime.timedelta(hours=9) print('%s - %2.2f %3.2f' % (jst, alt_iss, az_iss))直近10日間を予測してみると、
2020-03-07 05:38:00 - 11.98 9.07 2020-03-07 05:39:00 - 12.73 27.92 2020-03-07 05:40:00 - 11.22 46.14 2020-03-09 05:38:00 - 12.83 338.71 2020-03-09 05:39:00 - 19.49 353.95 2020-03-09 05:40:00 - 25.41 19.47 2020-03-09 05:41:00 - 24.90 51.33
- 3月7日5時38分から3分間程度 (最大12.73度)
- 3月9日5時38分から4分程度 (最大25.41度)
を予測した。
答え合わせ
以下の予報サイトで確認してみる。
https://spotthestation.nasa.gov/sightings/view.cfm?country=Japan®ion=None&city=Fuchu#.XlcICsvng2x
位置やTLE最新化サボりの誤差を考慮すると、ほぼほぼ正解と言って良さそう。(ISSの仰角を10、太陽の仰角を-4としたのはNASAの予測に合わせてチューニングした)
これでもうISSを見逃すことはない。
- 投稿日:2020-02-28T14:39:20+09:00
VSCodeでNumpyのインテリセンス(入力補完)が不完全で解決まで軽くハマった話
問題
MacでDockerコンテナを利用して深層学習のための環境を作ったが、
NumPyの入力補完(以下、IntelliSense)が不完全で、ちょっと不便。環境
- VSCode 1.42.1
- MacOS Catalina 10.15.3
- VSCodeの拡張機能に
ms-python.pythonは導入済み
(これさえ入れればpythonのIntelliSenseは問題なく動作する)- Python3.6.9 64bit
- Numpy 1.18.1
その他の環境は本記事のテーマにあまり関係ないので割愛。結論
ディレクトリ
.vscodeにあるsetting.jsonに設定を追加すれば良い。手順
NumPyなどのパッケージがどこの場所にあるかを確認する
import numpy as np print(np.__file__) # 出力結果 # /usr/local/lib/python3.6/dist-packages/numpy/__init__.py私の環境では以上のディレクトリだったので、このパスを確認して次の手順へ移ります。
setting.jsonに設定を追加する
setting.json{ "python.autoComplete.extraPaths": [ "/usr/local/lib/python3.6/dist-packages" ], }今回はNumPyの話に絞っているが、これ以外のライブラリも同様のことが言える。
よく見たらちゃんとVSCodeの公式でも説明されている。カスタムパッケージの場所でIntelliSenseを有効にする
おわりに
分かれば何てことはないが、拡張機能に頼ってあまり
setting.jsonを直接いじらないような方はこの機会に少し覚えておくと損はない。
- 投稿日:2020-02-28T14:11:23+09:00
Python学習1日の初学者が作る「Twitterでデータベースにまとめたアカウントを一括ブロックするやつ」
まえがき
前回の記事で特定のアカウントをRTした人をリスト化してまとめる方法を書きました:
Python学習1秒の初学者が作る「Twitterで特定のアカウントをRTした人を探すやつ」なんか2chに貼られてたみたいです:
【朗報】プログラミング初心者、DAPPIをリツイートした愛国戦士をデータベース化するプログラムを作ってしまう初学者という意味で「1秒」と誇張したタイトルにしたところ2chの民を怒らせてしまったようなので今回から訂正します。すみませんでした。
今回やること
スレッドを見たらこういう書き込みがありました。
ということで、前回記事で作成したDBを利用し、アカウントを一括ブロックするものを作ってみようというのが本記事です。コード
前回の記事よりもかなり簡単です。
ex2.py# -*- coding:utf-8 -*- import tweepy import pyodbc # APIの秘密鍵 CK = '****' # コンシューマーキー CKS = '****' # コンシューマーシークレット AT = '****' # アクセストークン ATS = '****' # アクセストークンシークレット #メインルーチン def main(total): conn_str = 'Driver={{Microsoft Access Driver (*.mdb, *.accdb)}};Dbq={0};'.format("****.accdb") #DB操作のための定型文的なやつ conn = pyodbc.connect(conn_str) cur = conn.cursor() #twitter APIに接続するための定型文的な奴 auth = tweepy.OAuthHandler(CK, CKS) auth.set_access_token(AT, ATS) api = tweepy.API(auth, wait_on_rate_limit=True) sql = "SELECT screen_name FROM RTers ;" cur.execute(sql) row=cur.fetchone() while row: print("ブロック:" +str(row[0][1:])) api.create_block(str(row[0][1:])) total=total+1 row=cur.fetchone() if total==10: break print(str(total)+"件をブロック") #DBをクローズ cur.close() conn.close() return(total) if __name__ == '__main__': total=0 total=main(total) #ここでmain関数をコールほとんど前回使用したソースコードから今回不要な処理を削っただけのものです。
データベースとTwitter APIに接続し、データベースから引っ張ってきた情報をもとに
Twitter APIで対象アカウントをブロックしています。今回新たに使用したものはこれだけです。
api.create_block(str(row[0][1:]))create_block()の使い方は簡単で、ブロック対象のアカウントのID(@の後ろの部分)を
引数として渡してあげるだけです。(例: api.create_block("chili_in") )実行するとどうなる
今回はテストということで10件処理したところでプログラムの実行を止めています。
コード内の以下の部分を削除するとDBに記録されているアカウントを全てブロックすることができます。if total==10: break
- 投稿日:2020-02-28T14:11:23+09:00
Python学習1日の初学者が作る「データベースにまとめたTwitterアカウントを一括ブロックするやつ」
まえがき
前回の記事で特定のアカウントをRTした人をデータベース化する方法を書きました:
Python学習1秒の初学者が作る「Twitterで特定のアカウントをRTした人をデータベースにまとめるやつ」なんか2chに貼られてたみたいです:
【朗報】プログラミング初心者、DAPPIをリツイートした愛国戦士をデータベース化するプログラムを作ってしまう初学者という意味で「1秒」と誇張したタイトルにしたところ2chの民を怒らせてしまったようなので今回から訂正します。すみませんでした。
今回やること
スレッドを見たらこういう書き込みがありました。
ということで、前回記事で作成したDBを利用し、アカウントを一括ブロックするものを作ってみようというのが本記事です。コード
前回の記事よりもかなり簡単です。
ex2.py# -*- coding:utf-8 -*- import tweepy import pyodbc # APIの秘密鍵 CK = '****' # コンシューマーキー CKS = '****' # コンシューマーシークレット AT = '****' # アクセストークン ATS = '****' # アクセストークンシークレット #メインルーチン def main(total): conn_str = 'Driver={{Microsoft Access Driver (*.mdb, *.accdb)}};Dbq={0};'.format("****.accdb") #DB操作のための定型文的なやつ conn = pyodbc.connect(conn_str) cur = conn.cursor() #twitter APIに接続するための定型文的な奴 auth = tweepy.OAuthHandler(CK, CKS) auth.set_access_token(AT, ATS) api = tweepy.API(auth, wait_on_rate_limit=True) sql = "SELECT screen_name FROM RTers ;" cur.execute(sql) row=cur.fetchone() while row: print("ブロック:" +str(row[0][1:])) api.create_block(str(row[0][1:])) total=total+1 row=cur.fetchone() if total==10: break print(str(total)+"件をブロック") #DBをクローズ cur.close() conn.close() return(total) if __name__ == '__main__': total=0 total=main(total) #ここでmain関数をコールほとんど前回使用したソースコードから今回不要な処理を削っただけのものです。
データベースとTwitter APIに接続し、データベースから引っ張ってきた情報をもとに
Twitter APIで対象アカウントをブロックしています。今回新たに使用したものはこれだけです。
api.create_block(str(row[0][1:]))create_block()の使い方は簡単で、ブロック対象のアカウントのID(@の後ろの部分)を
引数として渡してあげるだけです。(例: api.create_block("chili_in") )実行するとどうなる
今回はテストということで10件処理したところでプログラムの実行を止めています。
コード内の以下の部分を削除するとDBに記録されているアカウントを全てブロックすることができます。if total==10: break
- 投稿日:2020-02-28T13:33:12+09:00
言語処理100本ノック-37(pandas使用):頻度上位10語
言語処理100本ノック 2015「第4章: 形態素解析」の37本目「頻度上位10語」記録です。
今回はグラフ表示ということでmatplotlibを使います。matplotlibで誰もが陥ると思われる「豆腐問題」(日本語が表示される豆腐っぽい文字がグラフ上に表示される現象)に対応します。参考リンク
リンク 備考 037.頻度上位10語.ipynb 回答プログラムのGitHubリンク 素人の言語処理100本ノック:37 多くのソース部分のコピペ元 MeCab公式 最初に見ておくMeCabのページ 環境
種類 バージョン 内容 OS Ubuntu18.04.01 LTS 仮想で動かしています pyenv 1.2.16 複数Python環境を使うことがあるのでpyenv使っています Python 3.8.1 pyenv上でpython3.8.1を使っています
パッケージはvenvを使って管理していますMecab 0.996-5 apt-getでインストール 上記環境で、以下のPython追加パッケージを使っています。通常のpipでインストールするだけです。
種類 バージョン matplotlib 3.1.3 pandas 1.0.1 第4章: 形態素解析
学習内容
夏目漱石の小説『吾輩は猫である』に形態素解析器MeCabを適用し,小説中の単語の統計を求めます.
形態素解析, MeCab, 品詞, 出現頻度, Zipfの法則, matplotlib, Gnuplot
ノック内容
夏目漱石の小説『吾輩は猫である』の文章(neko.txt)をMeCabを使って形態素解析し,その結果をneko.txt.mecabというファイルに保存せよ.このファイルを用いて,以下の問に対応するプログラムを実装せよ.
なお,問題37, 38, 39はmatplotlibもしくはGnuplotを用いるとよい.
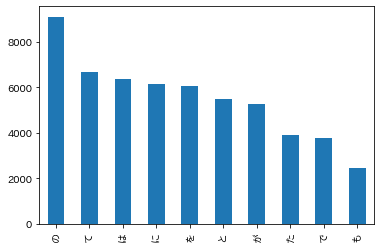
37. 頻度上位10語
出現頻度が高い10語とその出現頻度をグラフ(例えば棒グラフなど)で表示せよ.
回答
回答プログラム 037.頻度上位10語.ipynb
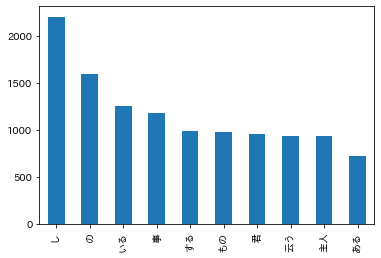
import pandas as pd import matplotlib.pyplot as plt plt.rcParams['font.family'] = 'IPAexGothic' def read_text(): # 0:表層形(surface) # 1:品詞(pos) # 2:品詞細分類1(pos1) # 7:基本形(base) df = pd.read_table('./neko.txt.mecab', sep='\t|,', header=None, usecols=[0, 1, 2, 7], names=['surface', 'pos', 'pos1', 'base'], skiprows=4, skipfooter=1 ,engine='python') return df[(df['pos'] != '空白') & (df['surface'] != 'EOS') & (df['pos'] != '記号')] df = read_text() df['surface'].value_counts()[:10].plot.bar() # 助詞と助動詞を除外 df[~df['pos'].str.startswith('助')]['surface'].value_counts()[:10].plot.bar()回答解説
豆腐(グラフ文字化け対応)対応
以下の記事を参考にして豆腐(グラフ文字化け対応)対応をしました。対応方法はOSおよびPython環境(pyenv使用など)に大きく依存するので注意ください。
1. フォントインストール
apt-getでフォントをインストールしますapt-get install fonts-ipaexfont2. キャッシュ削除
matplotlibのフォントキャッシュである以下のファイルを物理削除します。2つの違いがわかっていないのですが、「キャッシュは消してもいいや」の感覚で削除しました。
- /Users/ユーザー名/.cache/matplotlib/fontlist-v300.json
- /Users/ユーザー名/.cache/matplotlib/fontlist-v310.json
3. Pythonでフォント指定
Python上の以下の設定でグラフ出力するフォントを指定します。これで「豆腐」対応完了です。
plt.rcParams['font.family'] = 'IPAexGothic'グラフ出力
pandasだとplotを使ってそのまま出力できるので非常に便利です。df['surface'].value_counts()[:10].plot.bar() # 助詞と助動詞を除外 df[~df['pos'].str.startswith('助')]['surface'].value_counts()[:10].plot.bar()出力結果(実行結果)
プログラム実行すると以下の結果が出力されます。やはり数字だけで見るよりグラフ化した方がわかりやすいですね。
全単語
助詞と助動詞を除外した単語
- 投稿日:2020-02-28T13:16:03+09:00
【Python】if文を使ったファイル操作
問題
フォルダが100個あり、その中に2つの画像が入っているべきである。
しかし、フォルダによっては1つしかない場合がある。目的
1つしか画像が入っていないフォルダのみを”mada”フォルダに集める。
コード
import os, glob import shutil if not os.path.isdir('mada'): os.makedirs('mada') path = os.getcwd() # ファイルもフォルダも混ざったリスト files = os.listdir(path) # フォルダのみのリスト dirs = [f for f in files if os.path.isdir(os.path.join(path, f))] # フォルダごとにファイルの数を確認し、1であればmadaフォルダへコピー for f in dirs: files = os.listdir(os.path.join(path, f)) if len(files) == 1: shutil.copytree(f,("./mada/" + f))
- 投稿日:2020-02-28T12:51:05+09:00
responder 2.0.0 以降で jinja2 の filter を追加する
TL;DR
Responder が Ver 2.0.0 になって api から jinja_env が消えて custom filter が使えず困ったので、取り敢えず動作するように解消する話
cutom filter と html
custom_filters.pydef css_filter(path): return f"./static/css/{path}"_layout.html... <link rel="stylesheet" href="{{ 'style.css' | css }}"> ...1.3.2 まで
1.3.2までは以下の書き方でfilterの追加が行えていた
app.pyimport responder import custom_filters api = responder.API() api.jinja_env.filters.update( css=custom_filters.css_filter )2.0.0 以降
2.0.0 以降は
jinja_envがいないので、取り敢えず以下のように編集したapp.pyimport responder import custom_filters api = responder.API() api.templates._env.filters.update( css=custom_filters.css_filter )_ 付きなので responder 的には、あまり参照することを推奨していないかもしれない...
- 投稿日:2020-02-28T12:38:19+09:00
Python3.7系でvimやzshにエラーが出た
起きた問題
zshはPythonのトレースバックメッセージが表示され、vim(私の場合SpaceVim)がinsertしようとすると、大量にエラーを吐かれて使いものにならなかった。vimの方のエラー内容は忘れてしまいました...
解消方法
Pythonのバージョンを3.6.0に戻してプラグインをそれぞれ再インストールしました。
vim
私はSpaceVimを使っているので、vimを開いて、
:SPUpdateを行いました。zsh
zplugでプラグインを管理しているため、
zplug updateで再インストール上記で解決しました。
かなり内容が薄くなってしまいましたが、とりあえずバージョンを戻せば元のように動くようになるようです
- 投稿日:2020-02-28T11:52:26+09:00
Python FlowFishMaster
- 投稿日:2020-02-28T11:38:51+09:00
2015年、コロナウイルスの流行を予測した論文
はじめに
今日は、2015年にコロナウイルス(Covid-19)の流行を予測した論文を紹介します。中国とアメリカの研究者たちの共同研究の結果です。下記に論文のタイトルとリンクを記します。論文のPDFがダウンロードも可能です。
A SARS-like cluster of circulating bat coronaviruses shows potential for human emergence
https://www.nature.com/articles/nm.3985研究目的は、治療法とワクチンの開発目的として、ウイルスモデル(実際のコロナウイルス)を作成したものだそうです。面白い(ある意味怖いですが)ことは、論文の中に、現在のようなコロナウイルスの流行が正確に予測していることです。
論文の著者
2015年に中国の武漢ウイルス研究所(Wuhan Institute of Virology)と米国のノースカロライナ大学が共同研究した内容で、Nature誌に掲載されています。そうです。震源地の武漢海鮮市場の隣にあるあの武漢ウイルス研究所です。今回のコロナウイルス論文の責任著者(Corresponding Author)は、下記のお二方です。ちなみに、Shi Zengli博士は、この分野の研究では世界的に権威がある方だそうです。そして、ノースカロライナ大学も免疫学分野でアメリカトップクラスだそうです。
論文の内容
5年前、中国に生食するコウモリ(Horseshoe Bat)からSARSと類似するウイルスが発見され、遺伝子組み換えで、キメラウイルスが作られました。このウイルスを実験したところ、毒性が極めて強く、肺炎を起こす能力が高いことが発見されました。
しかし、どの治療法も予防効果が確認できませんでした。中国に生食するコウモリ(Horseshoe Bat)の中から、SARSと類似するウイルス(SHC014-CoV)のスパイクたんぱく質と野生ネズミのコロナウイルス(mouse-adapted SARS-Cov)を遺伝子組み換え(Reverse Genetics)を利用し、新型コロナウイルスと非常に類似するキメラウイルスを生産する。
予防のため、人の呼吸器の上皮細胞及び実験用ネズミに注入したところ、毒性が極めて強く、肺炎を起こす能力が高いことが確認されました。
予防のため、このキメラウイルスの治療法及びワクチンの開発(単核抗体でAce2をブロックする抗体を製造)を試みたんですが、どの方法も効果が確認されなかったことが結論です。
この論文の内容から見ると、今後のワクチンの開発が極めて難しいことが予測されます。そして、この論文の最後には、近年にコウモリから発生する新しいSARSウイルスの大流行を警告しています。
この記事は、新型コロナウイルスが武漢ウイルス研究所で作られた陰謀論を言うのではなく、5年前に同様なウイルスが研究され、治療とワクチン開発が極めて難しい結論になったことを紹介する目的に書きましたものです。
参考文献
- A SARS-like cluster of circulating bat coronaviruses shows potential for human emergence. https://www.nature.com/articles/nm.3985
- Dr. Shi Zhengli https://ja.wikipedia.org/wiki/%E7%9F%B3%E6%AD%A3%E9%BA%97
- Wuhan Institute of Virology () http://english.whiov.cas.cn/
- What is China not telling the world about the novel coronavirus? https://www.scmp.com/comment/letters/article/3049845/what-china-not-telling-world-about-novel-coronavirus
- 投稿日:2020-02-28T11:38:51+09:00
2015年にコロナウイルスの流行を予測した論文の紹介
はじめに
今日は、2015年にコロナウイルス(Covid-19)の流行を予測した論文を紹介します。中国とアメリカの研究者たちの共同研究の結果です。下記に論文のタイトルとリンクを記します。論文のPDFがダウンロードも可能です。
A SARS-like cluster of circulating bat coronaviruses shows potential for human emergence
https://www.nature.com/articles/nm.3985新しいウイルスが発生した時を想定し、治療法とワクチンの開発を目的として、新しいウイルスモデル(実際のコロナウイルス)を作成したそうです。面白いことは、論文の中に、現在のコロナウイルスの流行が正確に予測していることです。(ある意味怖いですが)
論文の著者
この論文は、2015年に中国の武漢ウイルス研究所(Wuhan Institute of Virology)と米国のノースカロライナ大学が共同研究したもので、Nature誌に掲載されています。そうです。震源地の武漢市の海鮮市場の隣にある「武漢ウイルス研究所」です。今回のコロナウイルス論文の責任著者(Corresponding Author)は、下記のお二方です。ちなみに、Shi Zengli博士は、この分野の研究では世界的に権威がある方だそうです。そして、ノースカロライナ大学も免疫学分野でアメリカトップクラスです。
論文の内容
5年前、中国のコウモリ(Horseshoe Bat)からSARSと類似するウイルスが発見され、遺伝子組み換えで、キメラウイルスが作られました。このウイルスを実験したところ、毒性が極めて強く、肺炎を起こす能力が高いことが発見されました。
しかし、どの治療法も予防効果が確認できませんでした。中国に生食するコウモリ(Horseshoe Bat)の中から、SARSと類似するウイルス(SHC014-CoV)が発見されました、このウイルスのスパイクたんぱく質と野生ネズミのコロナウイルス(mouse-adapted SARS-Cov)を遺伝子組み換え(Reverse Genetics)で、2020年の新型コロナウイルスと非常に類似するキメラウイルスを生産しました。
このキメラウイルスを、人の呼吸器の上皮細胞及び実験用ネズミに注入したところ、毒性が極めて強く、肺炎を起こす能力が高いことが確認されました。
予防のため、このキメラウイルスの治療法及びワクチンの開発(単核抗体でAce2をブロックする抗体を製造)を試みたんですが、どの方法も効果が確認されなかったことが結論です。(すべての試みが失敗に終わってしまった。)この論文の結果を見ると、今後のワクチンの開発が極めて難しいことが予測されます。
そして、この論文の最後には、近年にコウモリから発生する新しいSARSウイルスの大流行を警告しています。
Our work suggests a potential risk of SARS-CoV re-emergence from viruses currently circulating in bat populations.
この記事は、新型コロナウイルスが武漢ウイルス研究所で作られた陰謀論を言うのではなく、5年前に同様なウイルスが研究され、治療とワクチン開発が極めて難しい結論になったことを紹介する目的に書きましたものです。
参考文献
- A SARS-like cluster of circulating bat coronaviruses shows potential for human emergence. https://www.nature.com/articles/nm.3985
- Dr. Shi Zhengli https://ja.wikipedia.org/wiki/%E7%9F%B3%E6%AD%A3%E9%BA%97
- Wuhan Institute of Virology () http://english.whiov.cas.cn/
- What is China not telling the world about the novel coronavirus? https://www.scmp.com/comment/letters/article/3049845/what-china-not-telling-world-about-novel-coronavirus