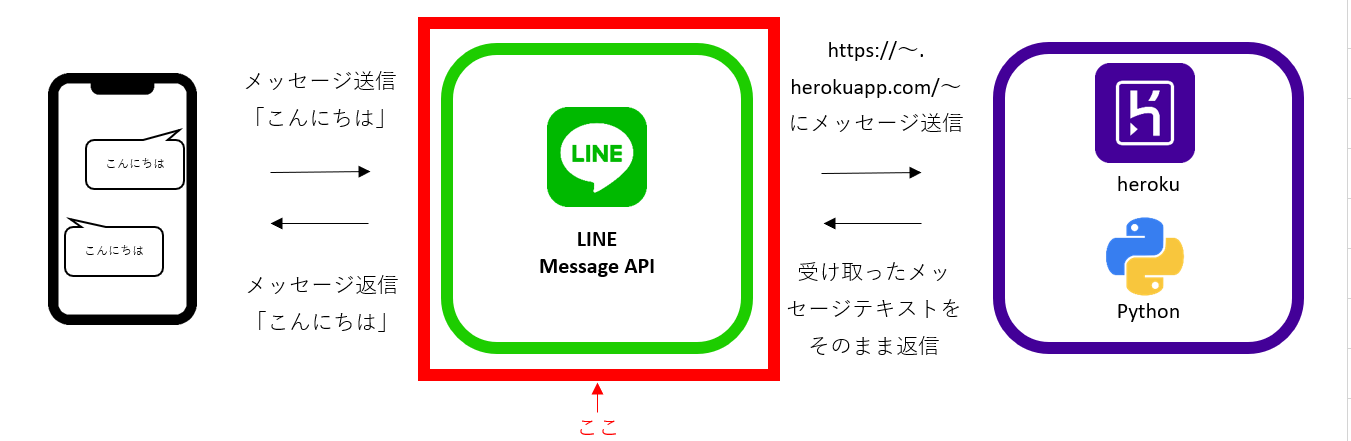
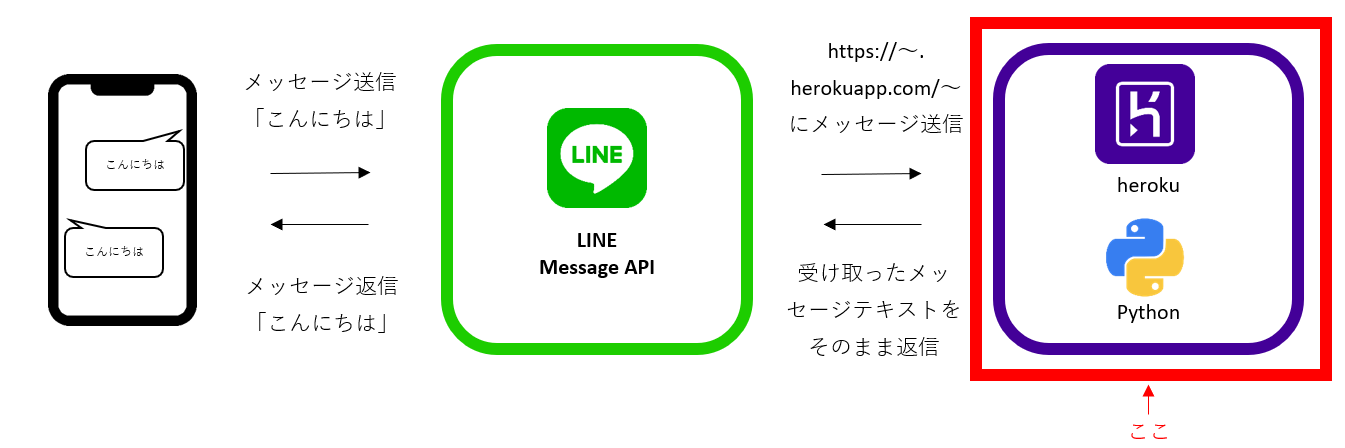
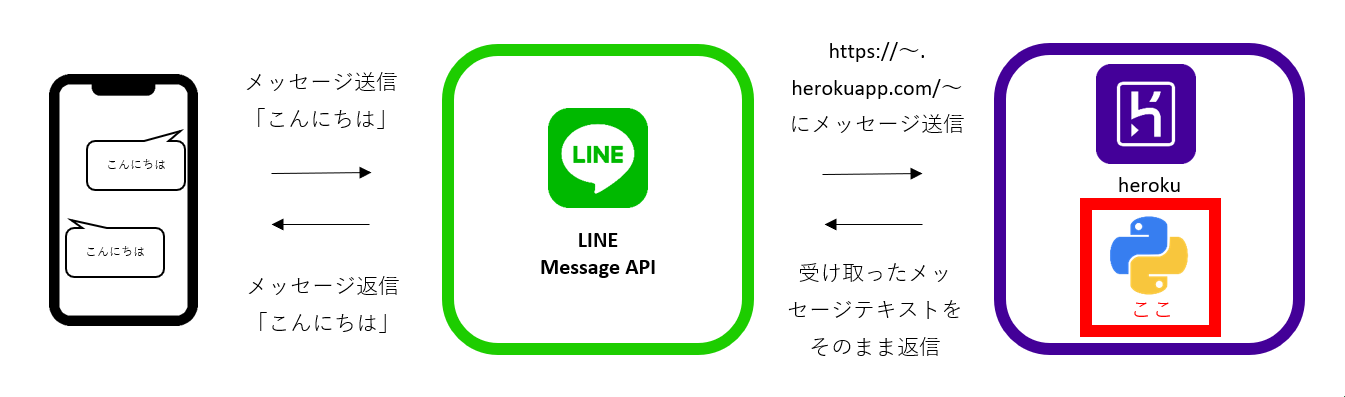
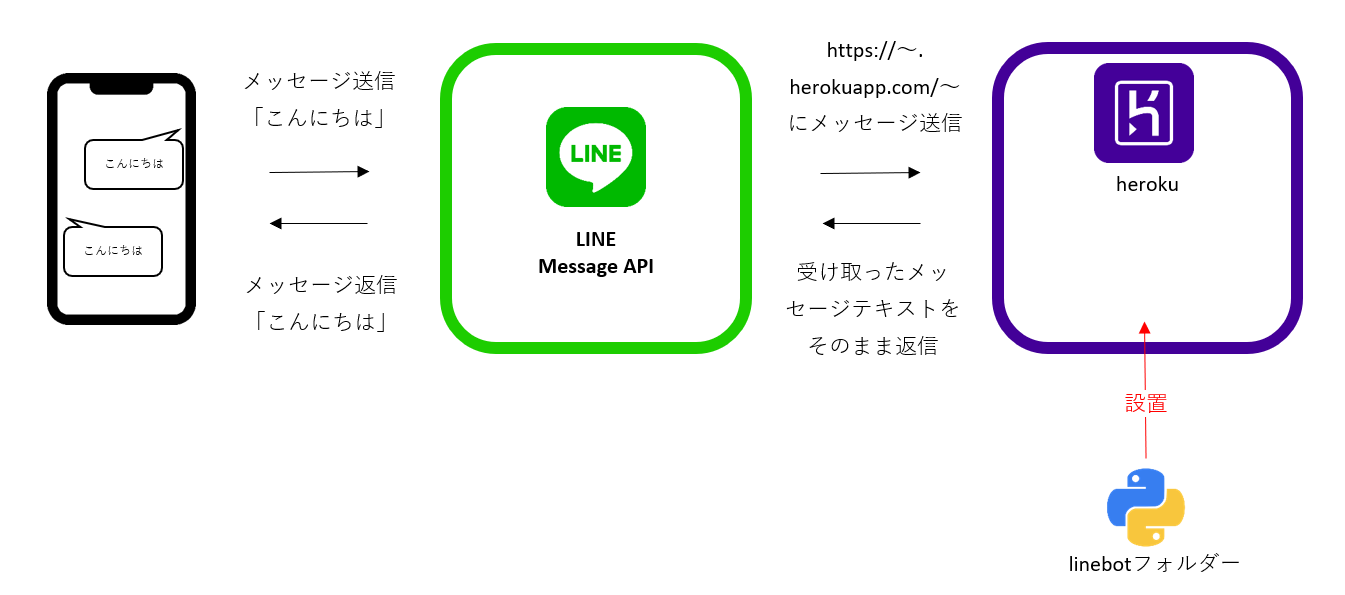
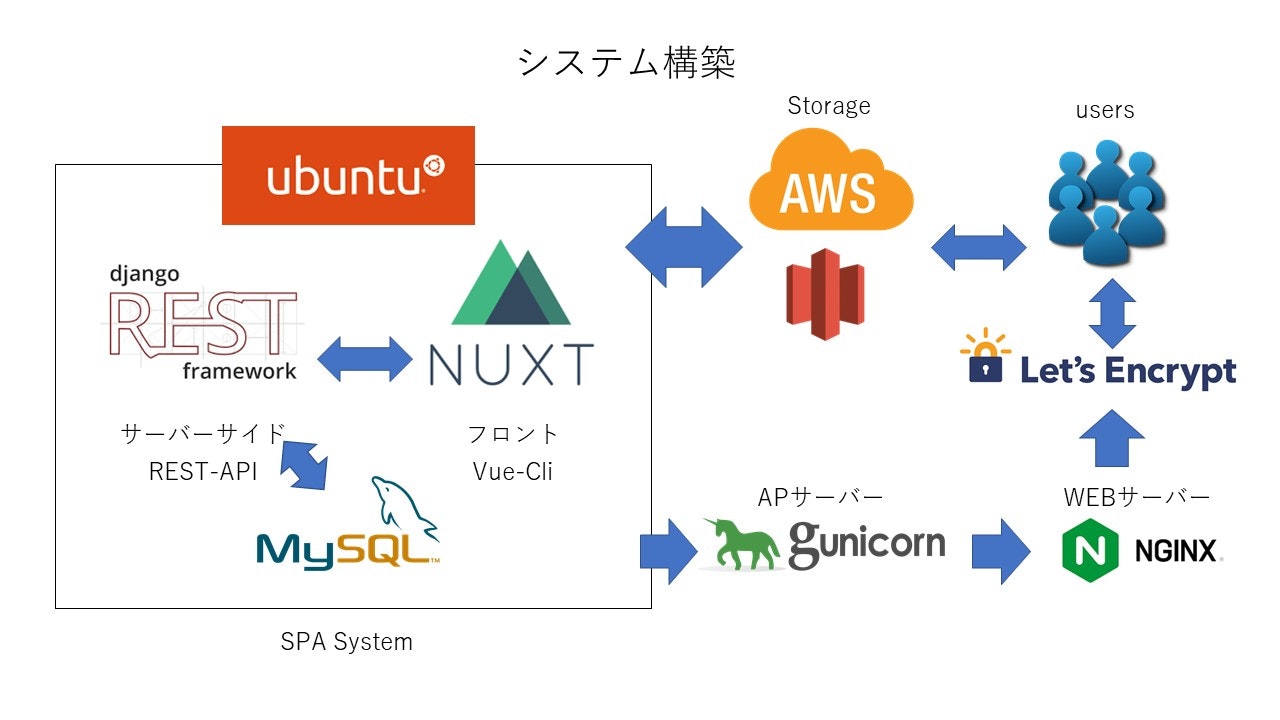
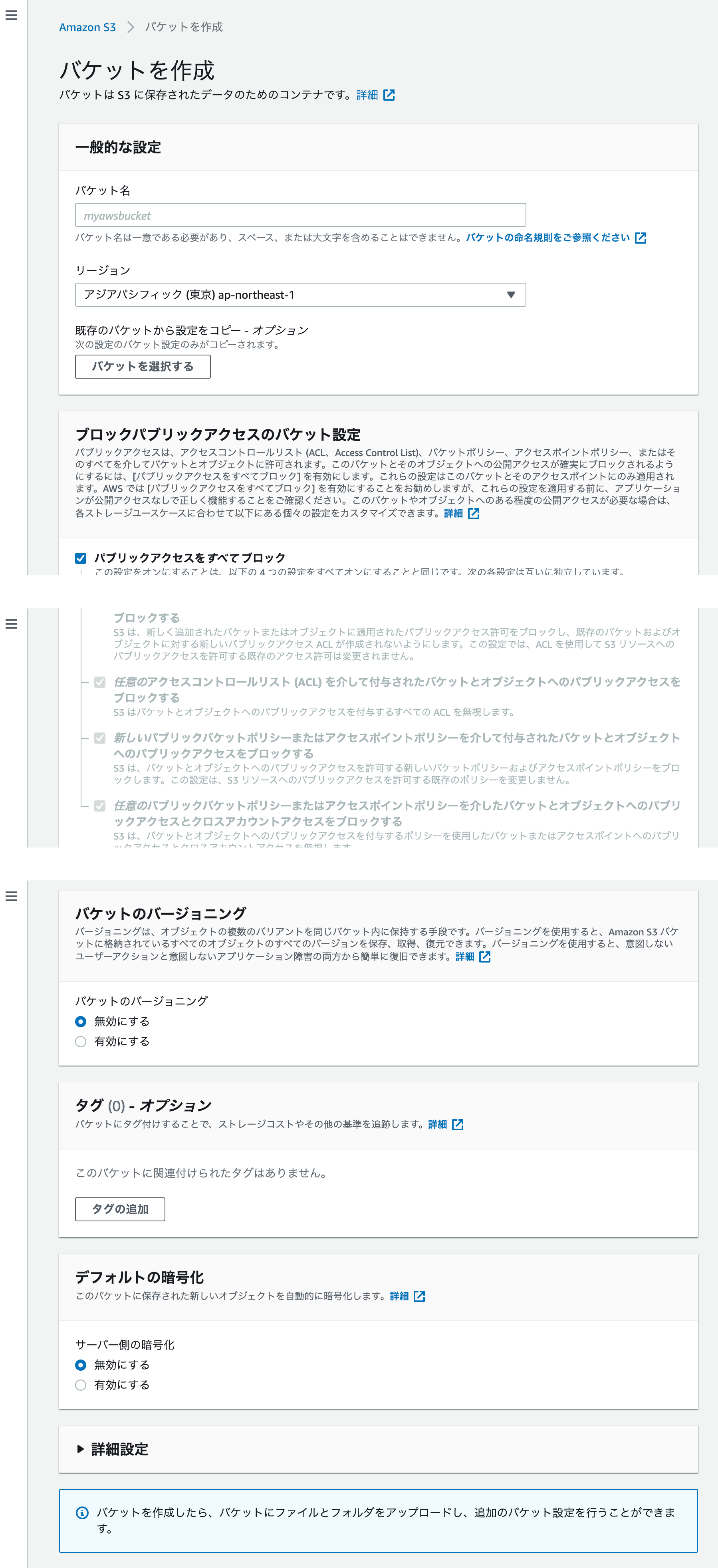
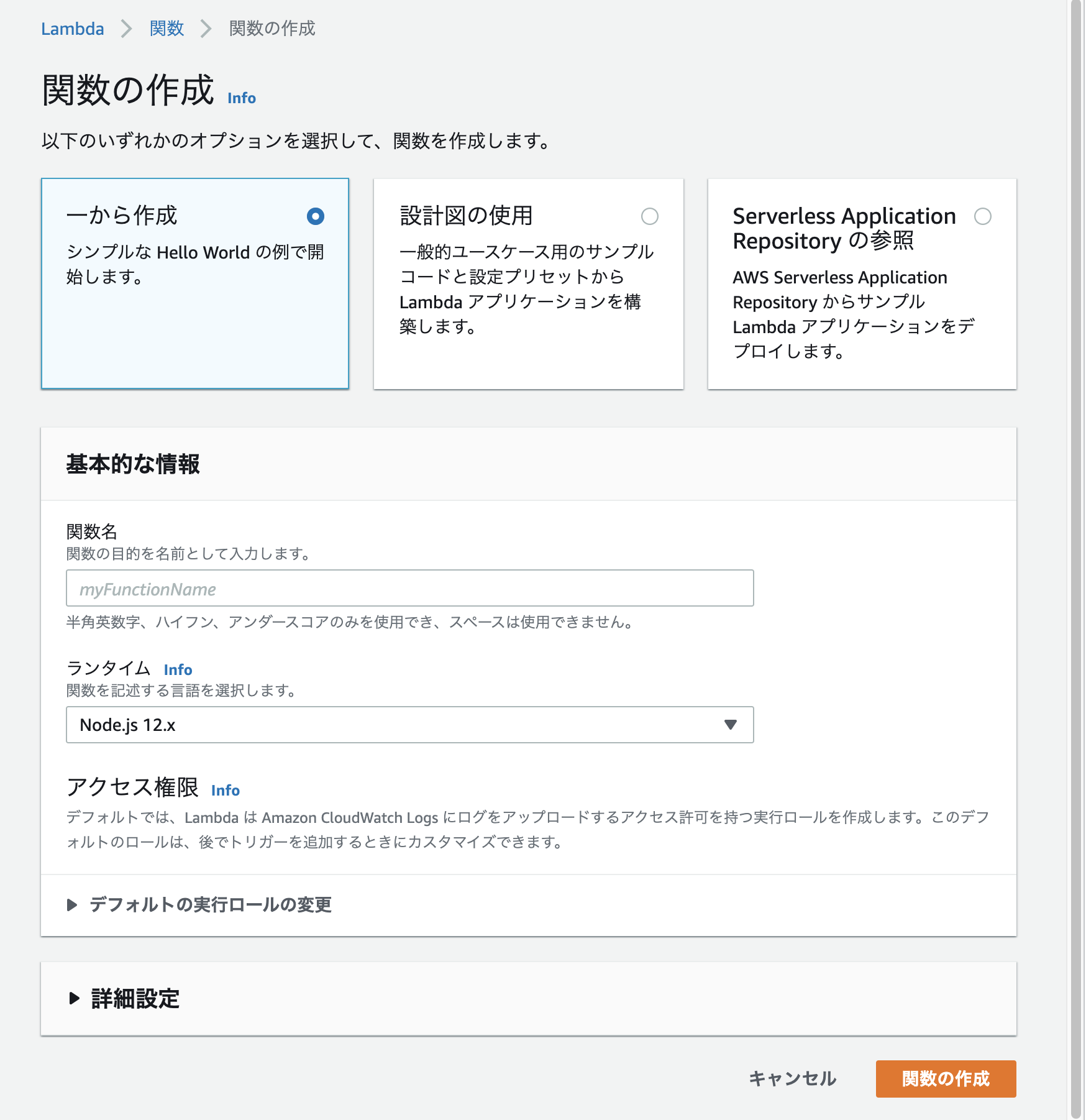
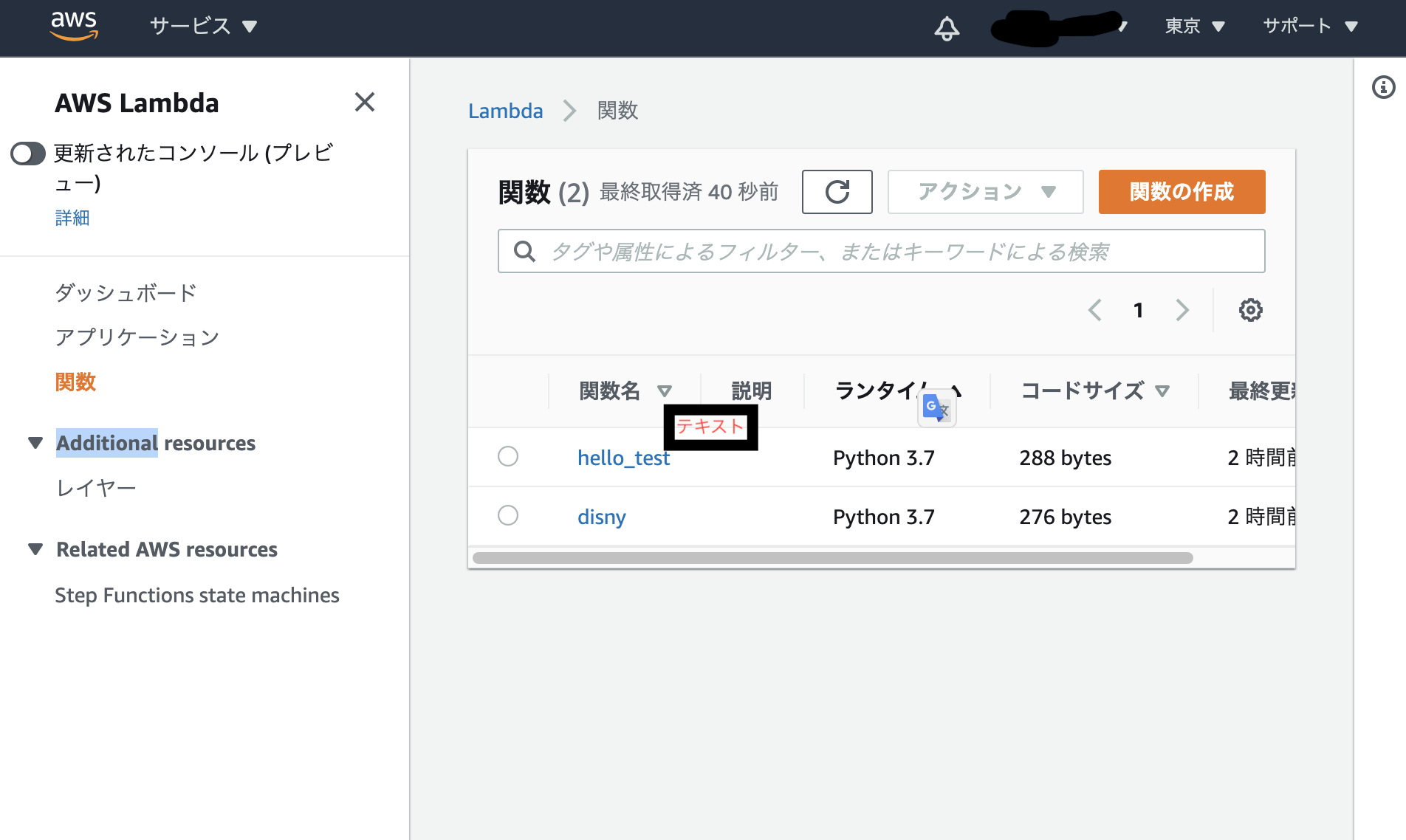
- 投稿日:2020-11-24T23:28:19+09:00
Python制御構文(備忘録)
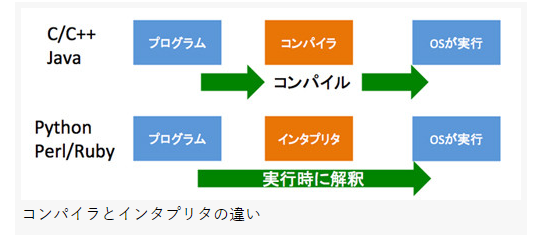
Pythonの制御構文周りの備忘録です。
随時追加・修正を行っていきます。比較演算子
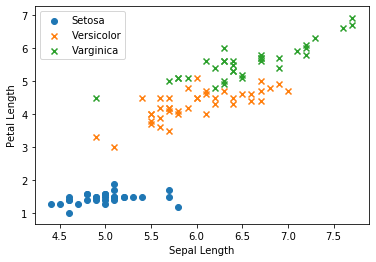
演算子 説明 A == B AとBが等しい A != B AとBが等しくない A < B AがBよりも小さい A > B AがBよりも大きい A <= B AがB以下 A >= B AがB以上 A in [LIST] [LIST]の中にAがある A not in [LIST] [LIST]の中にAがない A is None AがNoneである A is not None AがNoneでない 条件A and 条件B 条件Aと条件Bのどちらも満たす 条件A or 条件B 条件Aまたは条件Bのどちらかを満たす if
num = 5 if num == 0: print('数値は0') elif num < 0: print('数値は0より小さい') else: print('数値は0より大きい')while
limit = input('Enter:') # 入力を受け付ける count = 0 while True: if count >= limit: break # countが10以上ならループを抜ける if count == 5: count += 1 continue # countが5なら次のループへ print(count) count += 1 else: # breakされずにループが終了したら実行する print('Done')for
for i in [1, 2, 3, 4, 5, 6, 7, 8, 9, 10]: if i == 5: # iが5なら次のループへ continue: if i == 8: # iが8ならループを抜ける break: print(i) # breakされずにループが終了したら実行する else: print('Done') # 指定回数処理したいが値を取り出す必要が無い時に_を利用する for _ in range(10): print('hello') # 2から3つ飛ばしで10を超えるまで処理を行う for i in range(2, 10, 3): print('hello') # インデックスも取得したい場合 for i, animal in enumerate(['dog', 'cat', 'bird']): print(i, animal) # 複数のリストを同時に展開して取得したい場合 animals = ['dog', 'cat', 'bird'] foods = ['meat', 'fish', 'bean'] for animal, food in zip(animals, foods): print(animal, food) # 辞書のループ処理 data = {'x': 10, 'y': 20} for k, v in d.items(): print(k, ':', v)
- 投稿日:2020-11-24T23:28:19+09:00
Python備忘録(制御構文)
Pythonの制御構文周りの備忘録です。
随時追加・修正を行っていきます。演算子
演算子 説明 A == B AとBが等しい A != B AとBが等しくない A < B AがBよりも小さい A > B AがBよりも大きい A <= B AがB以下 A >= B AがB以上 A in [LIST] [LIST]の中にAがある A not in [LIST] [LIST]の中にAがない A is None AがNoneである A is not None AがNoneである 条件A and 条件B 条件Aと条件Bのどちらも満たす 条件A or 条件B 条件Aまたは条件Bのどちらかを満たす if
num = 5 if num == 0: print('数値は0') elif num < 0: print('数値は0より小さい') else: print('数値は0より大きい')while
limit = input('Enter:') # 入力を受け付ける count = 0 while True: if count >= limit: break # countが10以上ならループを抜ける if count == 5: count += 1 continue # countが5なら次のループへ print(count) count += 1 else: # breakされずにループが終了したら実行する print('Done')for
for i in [1, 2, 3, 4, 5, 6, 7, 8, 9, 10]: if i == 5: # iが5なら次のループへ continue: if i == 8: # iが8ならループを抜ける break: print(i) # breakされずにループが終了したら実行する else: print('Done') # 指定回数処理したいが値を取り出す必要が無い時に_を利用する for _ in range(10): print('hello') # 2から3つ飛ばしで10を超えるまで処理を行う for i in range(2, 10, 3): print('hello') # インデックスも取得したい場合 for i, animal in enumerate(['dog', 'cat', 'bird']): print(i, animal) # 複数のリストを同時に展開して取得したい場合 animals = ['dog', 'cat', 'bird'] foods = ['meat', 'fish', 'bean'] for animal, food in zip(animals, foods): print(animal, food) # 辞書のループ処理 data = {'x': 10, 'y': 20} for k, v in d.items(): print(k, ':', v)
- 投稿日:2020-11-24T22:50:01+09:00
Python2からPython3の切り替えたらdict valuesに気をつけよう!
概要
Python2のサポート終了からそろそろ1年経とうとしていますが、Python3に切り替えたことによって
dict.valuesの挙動が変わりましたので、改めて紹介したいと思います。
というのも、書き方によってはPython2時代と同じ挙動するので、そもそも以下で説明する問題に気付いていない可能性もあります。ざっくり公式ページの内容を要約しますと、
Python2で辞書型の変数をvalues()、items()、keys()メソッドで取得すると結果はリスト型で取得できていたが、Python3では、辞書ビューオブジェクトが返されるようになりました。
なので、listフィルタを追加することでPython2と同じ結果が得られるようになります。さっそくコード
Python3ではブロックスタイルと通常のスタイルで結果が異なるので、大丈夫だと思っていても、書き方(通常のスタイル)を変えただけで意図しない結果を招く可能性があります。
ちなみにansible_python_interpreterで挙動が変わるわけではなく、ansibleそのものがPython2.xかPython3.xによって挙動が変わります。Python3実行環境# 設定なし > ansible-config view ERROR! Invalid or no config file was supplied # Python3版 ansible > ansible --version ansible 2.9.14 config file = None configured module search path = ['/root/.ansible/plugins/modules', '/usr/share/ansible/plugins/modules'] ansible python module location = /usr/local/lib/python3.9/site-packages/ansible executable location = /usr/local/bin/ansible python version = 3.9.0 (default, Nov 18 2020, 13:28:38) [GCC 8.3.0]Python2実行環境# 設定なし > ansible-config view ERROR! Invalid or no config file was supplied # Python2版 ansible > ansible --version ansible 2.9.14 config file = None configured module search path = [u'/root/.ansible/plugins/modules', u'/usr/share/ansible/plugins/modules'] ansible python module location = /usr/local/lib/python2.7/site-packages/ansible executable location = /usr/local/bin/ansible python version = 2.7.18 (default, Apr 20 2020, 19:27:10) [GCC 8.3.0]dict_values.yml--- - name: Dict values hosts: localhost connection: local gather_facts: False vars: mydict: {"a": {"aa": 1 }, "b": {"bb": 2 }, "c": {"cc": 3 }} tasks: - name: block style debug debug: msg: >- {%- for d in mydict.values() -%} {{ d }} {%- endfor -%} - name: normal style debug debug: msg: "{{ item }}" with_items: "{{ mydict.values() }}"ブロックスタイルと通常の結果が全く同じ
実行結果(python2)TASK [debug] ************************************************************************* ok: [localhost] => { "msg": "{u'aa': 1}{u'cc': 3}{u'bb': 2}" } TASK [debug] ************************************************************************* ok: [localhost] => (item={u'aa': 1}) => { "msg": { "aa": 1 } } ok: [localhost] => (item={u'cc': 3}) => { "msg": { "cc": 3 } } ok: [localhost] => (item={u'bb': 2}) => { "msg": { "bb": 2 } }通常の結果が
dict_valuesで出力されています。
なので、この結果をループしたりすると動作が変わるので、要注意です。実行結果(python3)TASK [debug] ************************************************************************* ok: [localhost] => { "msg": "{'aa': 1}{'bb': 2}{'cc': 3}" } TASK [debug] ************************************************************************* ok: [localhost] => (item=dict_values([{'aa': 1}, {'bb': 2}, {'cc': 3}])) => { "msg": "dict_values([{'aa': 1}, {'bb': 2}, {'cc': 3}])" }対策
listフィルタを追加することで解決します。
全体のコードの整合性を保つためにブロックスタイルにもlistフィルタを追加するのも良いかと思います。(出力結果は変わりません)dict_values.yml--- - name: Dict values hosts: localhost connection: local gather_facts: False vars: mydict: {"a": {"aa": 1 }, "b": {"bb": 2 }, "c": {"cc": 3 }} tasks: - name: block style debug debug: msg: >- {%- for d in mydict.values() | list -%} {{ d }} {%- endfor -%} - name: normal style debug debug: msg: "{{ item }}" with_items: "{{ mydict.values() | list }}"
- 投稿日:2020-11-24T21:54:57+09:00
Pythonチュートリアルまとめ
print文
print文%s # 文字列 %r・・・ %d # 整数 %f # 固定小数点表記 %1.5f # 固定小数点表記(,5のところは小数以下桁) %e # 指数表記print("数値1=%f, 数値2=%.3f" % (1/3, 1/3)) 数値1=0.333333, 数値2=0.333引数"end"に値を入れることで、末尾に何をつけるかを設定
print("a", end=",") print("b", end=",") print("c")対話モード時に最後に表示した式を格納している変数
>>> tax = 12.5 / 100 >>> price= 100.5 >>> price * tax 12.5625 >>> price + _ 113.0625 >>> round(_,2) 113.06モジュールの検索パス
1.ビルトインモジュールの中
2.sys.path変数で得られるディレクトリのリスト
3.入力スクリプトのあるディレクトリ
4.PYTHONPATH
5.インストールごとのデフォルトコーディングスタイル
Pythonのコーディングスタイルを PEP8 という
コーディングスタイル・インデントはスペース4つ ・タブ禁止 ・4スペースは狭いインデントと広いインデントのちょうどよい妥協点。 ・コメント行の独立。 ・docstring ・エンコーディングは、UTF-8(default)・ASCII ・演算子の周囲やカンマの後ろにスペースを入れる ・カッコのすぐ内側にはスペースを入れないシーケンスオブジェクトの比較
・どちらかのシーケンスがなくなるまで比較 ・同じ要素は比較しない ・違う要素の比較結果クラス変数とインスタンス変数
・クラス変数とは・・・すべてのインスタンスが共有するメモリ
・インスタンス変数とは・・・各インスタンスに固有のメモリ以下は間違いの例・・・クラス変数にmutable(変更可)を使ってはいけない
クラス変数とインスタンス変数class Sample: c_list = []・・・クラス変数の使い方を間違えた例 def add_c_list(self,data): self.c_list.append(data) print("出力結果:", end=" ") sample1 = Sample() sample1.add_c_list("データ1") sample2 = Sample() sample2.add_c_list("データ2") for item_data in sample1.c_list: print(item_data, end=" ") ============================= 出力結果: データ1 データ2エスケープシーケンスの無効化
len関数
エスケープシーケンスは1文字でカウント 改行の\nは1文字としてカウントrange関数
range関数>>>print(range(5)) range(0,5) ・range関数は反復可能体(イテラブル) ・range関数はオブジェクトビルトイン関数
ビルトイン関数>>>dir(モジュール名) #dirがビルトイン関数。モジュールが定義している名前をすべて表示。1章 食欲をそそってみようか
・データ型の一般性が高く、問題領域は、Awk・Perlより広く、他言語と同等以上2章 インタープリタの使い方
インタープリタの起動>>> python -c コマンド >>> python -m モジュール名 >>>3章 気楽な入門編
演算子・べき乗演算子は、他の演算子より優先順位が高いため例外的に右から左へと評価される ・演算対象の型が混同していた場合(int,float)、整数は浮動小数点に変換される文字列の特徴>>> word[10000] # 大きすぎるインデックスを指定するとエラー IndexError Traceback (most recent call last) <ipython-input-4-47f442646512> in <module> ----> 1 Zen[50] IndexError: string index out of range >>> word[10000:20000] # 範囲外のスライシングはうまく処理 ''複数行にまたがる改行文字を取り消すprint("""\ Usage:thingy[options] -h Display this usage message -H hostname Hostname to connect to """)対話モードの電卓。アンダースコア最後に表示した式を変数「_」(アンダースコア)に代入してある。 >>> tax = 12.5/100 >>> price = 100.50 >>> price * tax 12.5625 >>> price + _ 113.0625 >>> round(_,2) 113.006対話モード列挙された文字リテラルは自動的に連結される >>> 'Py' 'thon' 'Python'4章 制御構造ツール
リストのアンパック>>> list(range(3,6)) # 一般的な個別の引数を使ったコール [3,4,5] >>> args = [3,6] # ここからは特殊な方法 >>> list(range(*arg)) # *arg を使うとアンパックされる。ただの 3,6 になる。 [3,4,5] # range(3,6) と同じ >>>ドキュメンテーション(docstring)・1行目:簡潔な要約。大文字始まる、ピリオド終わり。 ・2行目:空白 ・3行目:関数アノテーションdef 関数名(arg1: 'arg1の説明', arg2: 'arg2の説明', , ,)->'戻り値の説明': 処理docstringと関数アノテーションの例def my_func(n: 'この値から足し始める', m:'この値まで足す') -> 'nからmの合計値': """ nからmまでの合計を返す関数 """ ret = 0 for i in range(n, m+1): ret += i return ret5章 データ構造
リストをキューとして使う>>> from collections import deque >>> queue = deque(["A","B","C"]) # キューを作った >>> queue.append("D") # Dを追加 >>> queue.popleft() # 最初を取出 >>> queue.pop() # 最後を取出 >>> queue.pop(idx) # idx番目を取出ディクショナリ・キー:変更不能型 ・値:変更可能型 ・キーkeyの存在を確認、取得(検索): in演算子 ・値valueの存在を確認、取得(検索): in演算子, values() ・キーkeyと値valueの組み合わせの存在を確認: in演算子, items()多次元リストのソート演算子比較演算子 < <= == != is is not in not in条件についての補足・比較演算子 in および not in ・・・シーケンスの値の有無 ・演算子 is および is not ・・・オブジェクトの比較 ・ブール演算子 and および or・・・短絡演算子 ・比較の組み合わせ(複数条件) if x<y and x>z演算子の優先順位数値演算子 > 比較演算子シーケンスの比較、その他の型の比較・2つが基本的に同じシーケンスで、片方の長さが短い時は、この短い方が小となる。 ・文字列の辞書的順序には、個々の文字のUnicodeコードポイント番号で比較6章 モジュールとパッケージ
dir関数>>> import sys,fibo >>> dir(fibo) ・モジュールが定義している名前を確認するのに使う。モジュールの検索は、○○モジュールの場合、○○.pyを以下の順番で検索する
モジュールの検索パス1.ビルトインモジュール内で探す 2.sys.path変数で得られるディレクトリのリストを使ってspam.pyを検索 2-1.入力スクリプトのあるディレクトリ 2-2.PYTHONPATH 2-3.インストールごとのデフォルトmodule・モジュールは、ファイルである ・モジュールのファイルは、「.py」package・パッケージはフォルダーである >>> from パッケージ名 import モジュール名 #これを使うとモジュールを参照する際に短くてフルネームにする必要がない >>> import パッケージ名.モジュール名 #これを使うとモジュールを参照する際に長くてフルネームにする必要がある。[ドット区切りモジュール名]と呼ぶ。package実装例>>> import sound.effects.echo >>> sound.effects.echo.echofilter()#サブモジュールのロード。参照はフルネーム。長くてダメ!! >>> from sound.effects import echo >>> echo.echofilter()#参照が短くて良い!!コンパイル済みPythonファイル・Pythonは .py 以外に、Pythonコードをコンパイルした .pyc というファイルも実行できる ・インタプリタなので1行ごとにバイナリファイルに変換 ・コンパイラはまとめてバイナリファイルに変換8章 エラーと例外
概要・”エラー”は、「構文エラー」と「例外」に大別される ・"構文エラー"は「パース上のエラー」「構文解釈エラー」と呼ばれる ・”例外”は「文や式が正しくても、実行すると起こるエラー」である例外ZeroDivisionError NameError TypeError KeyboardInterrupt # キーボード割込例外 [Ctrl]+[c]9章 クラス
10章 標準ライブラリめぐり(モジュール)
モジュール名を参照短縮>>> from パッケージ名.サブモジュール名 import モジュール名 # これを使うとモジュールを参照する際に短くてフルネームにする必要がない >>> import パッケージ名.モジュール名 #これを使うとモジュールを参照する際に長くてフルネームにする必要がある。[ドット区切りモジュール名]と呼ぶ。moduleimport os # オペレーティングシステムとやり取りする関数 import glob # ファイルをワイルドカード検索 import sys # コマンドライン引数を処理する import re # 正規表現 import math # 浮動小数点数学 from struct import * # バイナリー import random # ランダム import collections # リスト import logging # ログ取りコマンドライン引数>>> import sys >>> print(sys.argv)randomモジュール・・・無作為抽出のツール>>> import random >>> random.choice(['apple','banana','lemon']) # choice はリスト内から選択 'apple' >>> random.sample(range(100),10) # sample は、第1引数から第2引数個を重複なく抽出 >>> random.random() # ランダムな浮動小数点 >>> radom.randrange(6) # range(6)からランダムに選んだ整数11章 標準ライブラリめぐり
ログ取り・ログ出力なのか、ユーザ(プログラム実行者)に伝えたい情報としての出力なのかを切り分けられる ・ログの種類をErrorやDebugのように、レベル分けが可能 ・フォーマットを指定すれば、簡単に統一化された出力が可能ログの優先順位(左から優先順位が低い)低い<- ->高い DEBUG、INFO、WARNING、ERROR、CRITICAL12章 仮想環境とパッケージ
pipによるパッケージ管理>>> pip install パッケージ名 # 最新バージョンのパッケージをインストール >>> pip install パッケージ名 ==2.6.0 # 特定バージョンのパッケージをインストール >>> pip install --upgrade パッケージ名 # 最新バージョンにアップグレード >>> pip uninstall パッケージ名 # パッケージをアンインストール >>> pip list # インストール済み確認 >>> pip freeze # インストール済み確認(出力形式がpip install) >>> pip show パッケージ名 # パッケージの詳細表示 version,author,summar,説明hp仮想環境>>> deactivate # 仮想環境終了14章 対話環境での入力行編集とヒストリ置換
ソースコード・エンコーディング・Pythonソースコードのエンコーディング:UTF-8 ・敢えてエンコードを変える時は以下になる #-*- coding:エンコーディング名 -*-インタープリタの終了ctrl+d >>>exit() >>>quit()タブ補完とヒストリ編集対話型インタープリタbpython IPythonアクティベート状態から抜ける対話モードのプライマリプロンプト/セカンダリプロンプト>>> ...
- 投稿日:2020-11-24T21:03:57+09:00
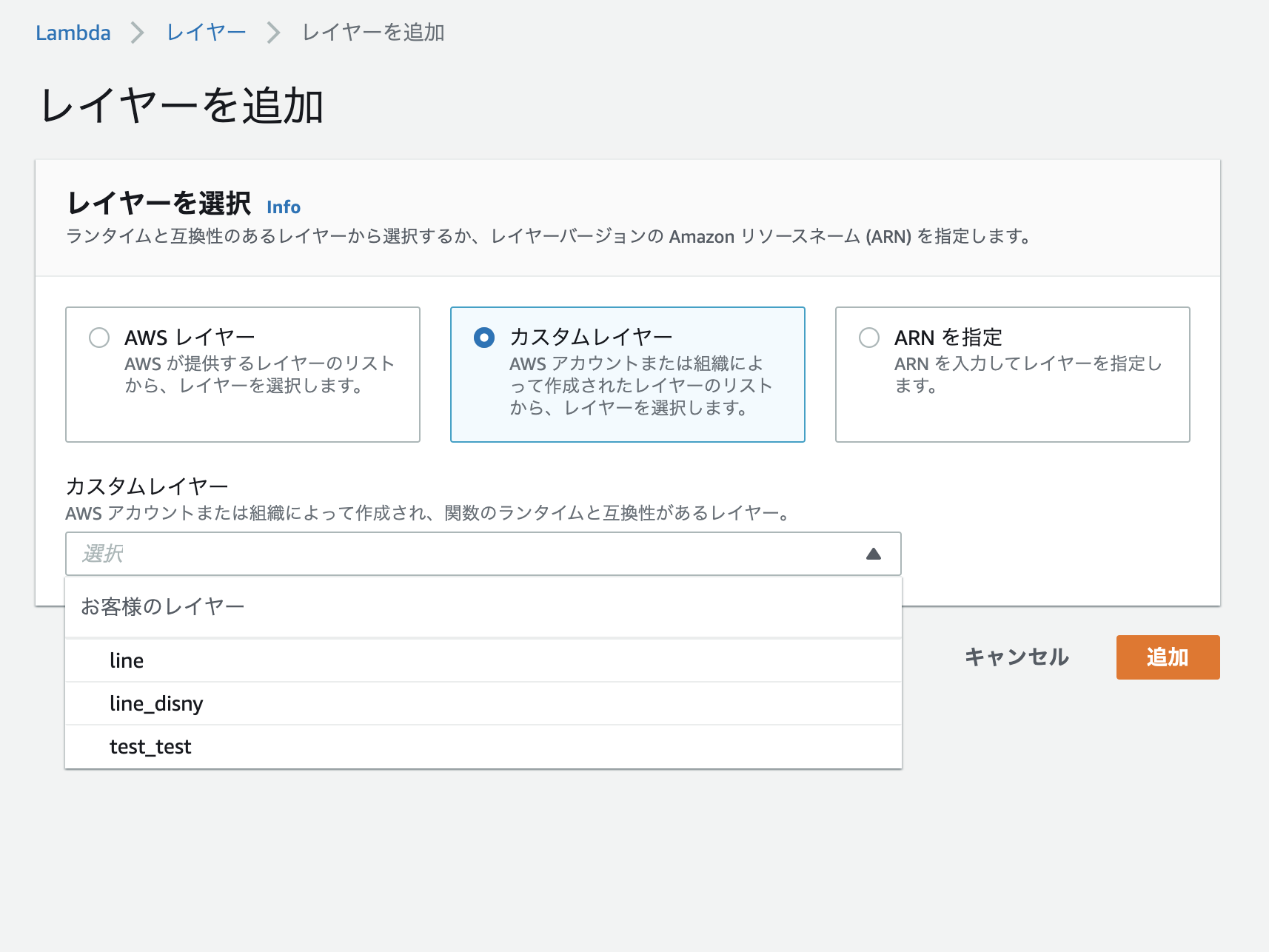
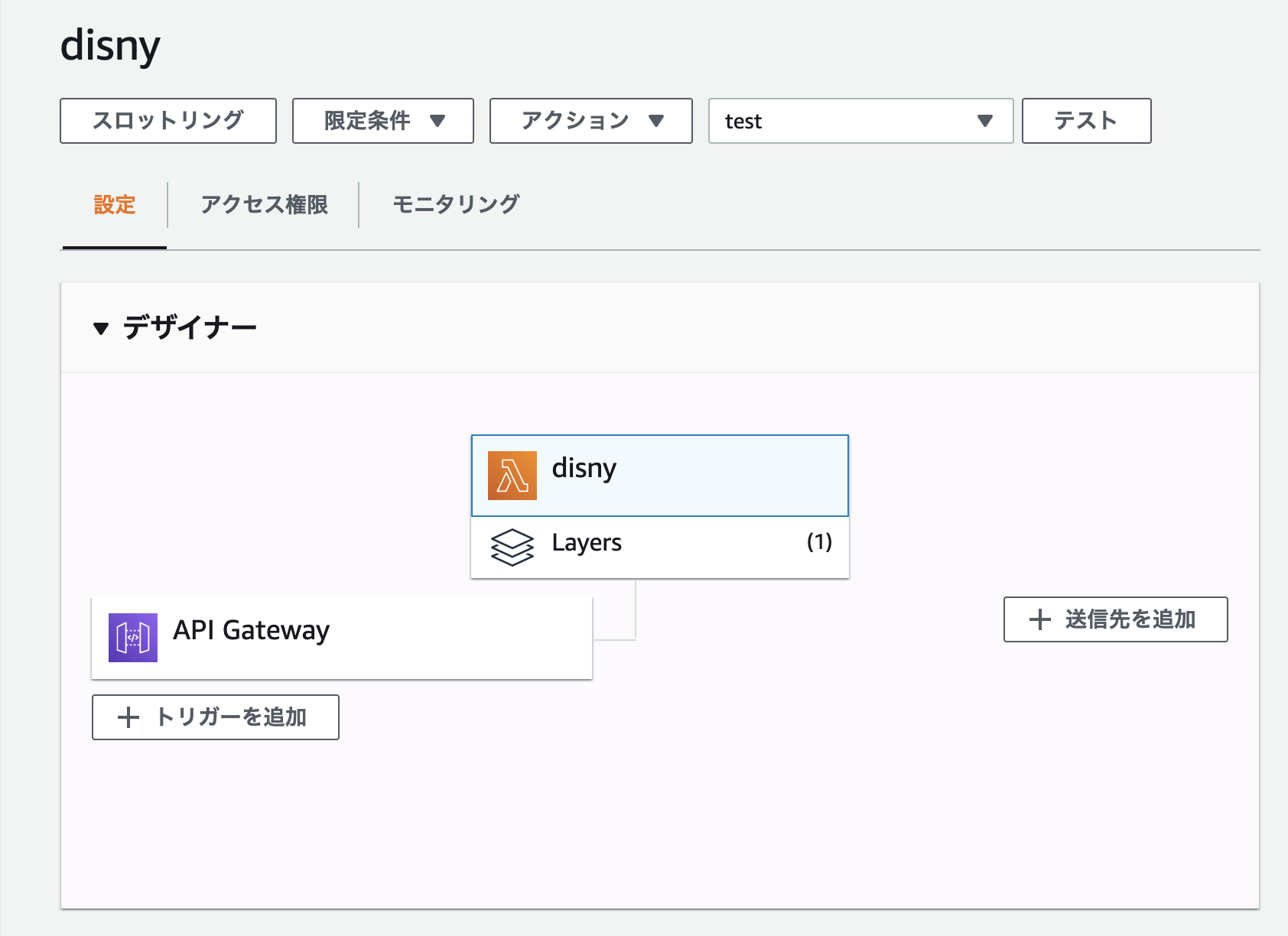
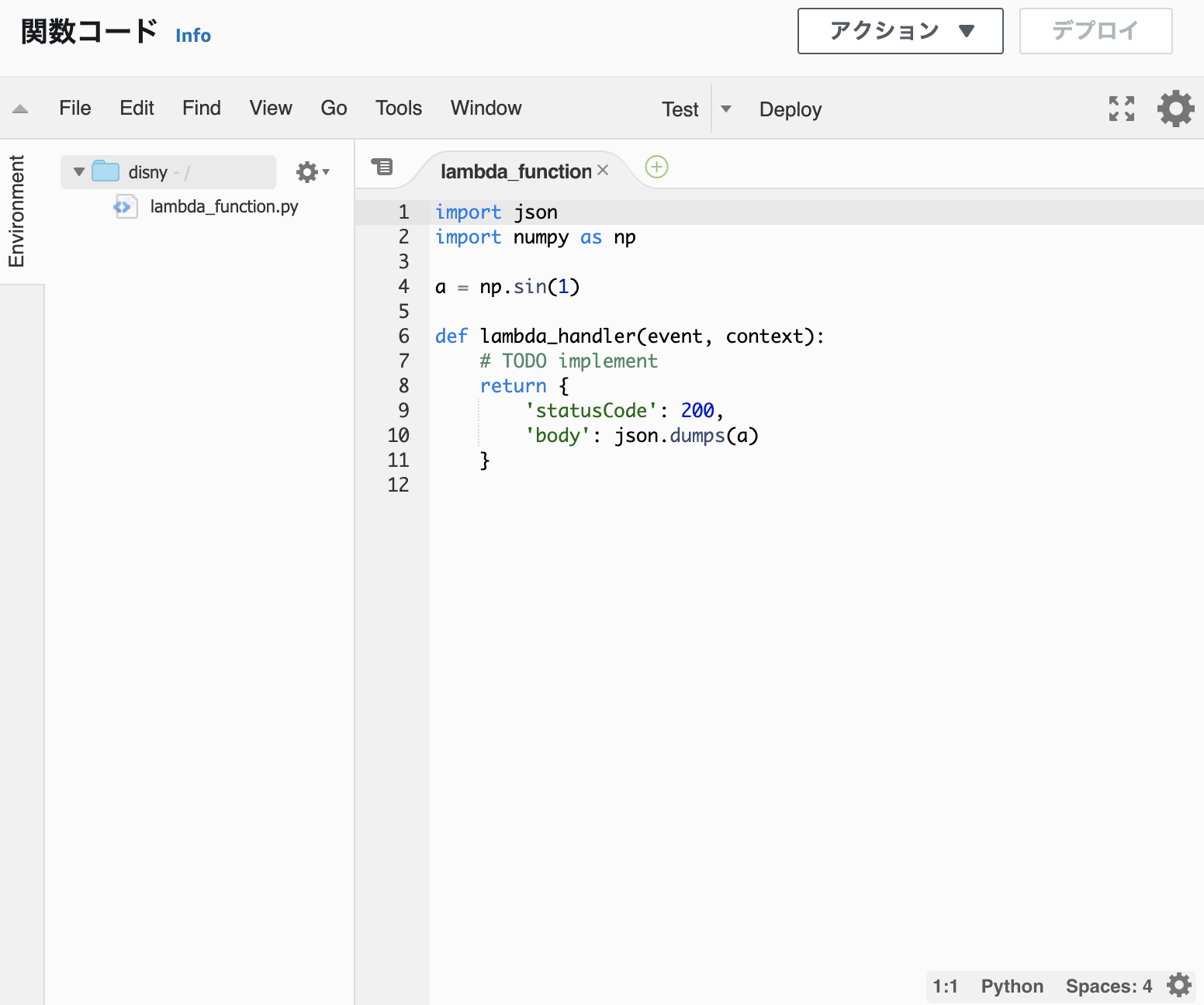
Pythonの関数を辞書(dict)のValueに格納して、Keyに応じた関数呼出しをする方法
1. メソッド(関数)を宣言する
Python3def sample_func_1(text : str) -> str: add_text = "追加した文字列" return (text + add_text) def sample_func_2(text : str) -> str: add_text2 = "追加した文字列" return (text + add_text2)2. 辞書型(dict)オブジェクトのValueに、関数を括弧を付けずに記述する
Python3func_dict = { 'label_1' : sample_func_1, 'label_2' : sample_func_2 }3. 辞書型オブジェクトKeyラベル名で、関数を呼び出して実行する
辞書(dict)のvalue値として、格納されているのは、関数(メソッド)オブジェクトそのものです。
そのため、関数(メソッド)を実行するには、辞書(func_dict)のvalue値に、引数を受け取る丸括弧("( )")を付ける必要があります。丸括弧("( )")を付けないで、辞書(dict)のvalue値として格納されている関数(メソッド)「それ自体」を呼び出した場合の結果は、以下になります。
Python3func_dict['label_1'] <function sample_func_1 at 0x100bafb80>上記のコードで辞書から呼出した関数(メソッド)を実行するには、引数を受け取る丸括弧("( )")をつける必要があります。
Python3func_dict['label_1']()なお、funct_dictが引数を1つ以上、受け取るメソッドである場合は、引数を与えないで関数呼出しをすると、エラーが出ます。
定義したメソッド(関数)は、str型のオブジェクトを引数を受け取る関数でした。
引数に適当な文字列を渡して、func_dict内に格納されているメソッド(関数)を実行してみます。Python3func_dict['label_1']("vxw") # 実行結果 'vxw追加した文字列'( 参考 )
Python3func_dict # 実行結果 {'label_1': <function sample_func_1 at 0x100bafaf0>, 'label_2': <function get_text_from_ppt at 0x100bafa60>} func_dict['label_1'] <function sample_func_1 at 0x100bafb80> type(func_dict['label_1']) # 実行結果 <class 'function'> type(func_dict['label_1']()) # 実行結果 Traceback (most recent call last): File "<stdin>", line 1, in <module> TypeError: sample_func_1() missing 1 required positional argument: 'text' type(func_dict['label_1']("giho")) # 実行結果 <class 'str'> func_dict['label_1']("giho") # 実行結果 'giho追加した文字列' returned_value = func_dict['label_1']("giho") returned_value # 実行結果 'giho追加した文字列'( 参考にしたWebページ )
( 使いみち )
今回、紹介した方法を用いることで、辞書型オブジェクトに定義されたKeyの値に応じて、そのKeyに対応する関数(メソッド)を呼び出すことができます。
データを処理する流れのなかで、条件に応じて、呼び出すべき適切な関数(メソッド)が切り替わるロジックが必要な場面で、役に立ちます。
基本的な例として、読み込んだファイルの拡張子の内容に応じて、そのファイルに所要の処理を行う関数(メソッド)を動的に選び出して呼出して実行する場面、などが考えられます。
PDFファイルやWordファイル、Excelファイルから、テキスト文字列のみを切り抜いてデータ取得するコードは、以下のWebページで紹介されています。
- deecode blog 「[Python] Word/Excel/PowerPoint/PDFからテキスト抽出するライブラリ・サンプルコード」
- @butadaさんのQiita記事 「pdf/docxファイルからのテキストマイニング」
Python3def get_text_from_txt(filename : str) -> str: filenameとして受け取ったパスに格納されているテキストファイルをreadして、テキストを*str*オブジェクトとして取得して、返り値として出力する処理 return text def get_text_from_pdf(filename : str) -> str: filenameとして受け取ったパスに格納されているPDFファイルをreadして、テキスト文字列を抜き出して、ひとつの*str*オブジェクトに連結して、返り値として出力する処理 return text def get_text_from_word(filename : str) -> str: filenameとして受け取ったパスに格納されているWordファイルをreadして、テキスト文字列を抜き出して、ひとつの*str*オブジェクトに連結して、返り値として出力する処理 return text def get_text_from_excel(filename : str) -> str: filenameとして受け取ったパスに格納されているExcelファイルをreadして、テキスト文字列を抜き出して、ひとつの*str*オブジェクトに連結して、返り値として出力する処理 return text func_dict = { 'txt' : get_text_from_txt, 'pdf' : get_text_from_pdf, 'word' : get_text_from_word, 'excel' : get_text_from_excel }( 備考 )
辞書のValueに関数(メソッド)を格納するときに、関数に括弧を付けると、以下のエラーが発生する。
関数の実行を行おうとしていると、解釈されてしまうため。PZython3func_dict = { 'label_1' : sample_func_1(), 'label_2' : sample_func_2() } # 実行結果 Traceback (most recent call last): File "<stdin>", line 2, in <module> TypeError: sample_func_1() missing 1 required positional argument: 'text'辞書を定義する上のコードのなかで、sample_func_1()の括弧のなかに特定の引数を与えると、辞書は定義できる(エラーは起きない)。
しかし、辞書のValueに格納された関数は、辞書を定義する際に受け取った引数しか、受け取ることのできない関数になってしまう。
( 辞書の定義 )
Python3func_dict = { 'label_1' : sample_func_1("abc"), 'label_2' : sample_func_2("def") }( 辞書内の関数の呼出し実行 )
func_dict辞書内で、Key"label_1"に対応するValueとして定義されている、関数sample_func_1("abc")が、引数"abc"を与えられた状態で、実行された。
Python3func_dict['label_1'] # 実行結果 'abc追加した文字列'func_dict['label_1'](を評価すると)は、すでに返り値として、str型のインスタンス'abc追加した文字列'を返している。そのため、func_dict['label_1']にさらに括弧を付けると、TypeError: 'str' object is not callableエラーが起きる。
Python3func_dict['label_1']() # 実行結果 Traceback (most recent call last): File "<stdin>", line 1, in <module> TypeError: 'str' object is not callablePython3func_dict['label_1']("abc") Traceback (most recent call last): File "<stdin>", line 1, in <module> TypeError: 'str' object is not callablePython3unc_dict['label_1']('abc') Traceback (most recent call last): File "<stdin>", line 1, in <module> TypeError: 'str' object is not callable
( 備考2 )
辞書のValにテキスト文字列を格納して、evalとexecを用いて、関数をよびだせる。
しかし、キーボードのストローク数が増えてしまい、エレガントではない。
Python3func_dict = { 'label_1' : "sample_func_1({arg})", 'label_2' : "sample_func_2({arg})" }Python3func_dict # 実行結果 {'label_1': 'sample_func_1({arg})', 'label_2': 'sample_func_2({arg})'} func_dict['label_1'] # 実行結果 'sample_func_1({arg})' expression = func_dict['label_1'].format(arg="'123'") expression # 実行結果 "sample_func_1('123')" eval(expression) # 実行結果 '123追加した文字列' result = eval(expression) result # 実行結果 '123追加した文字列' add_text = "result2=" text = add_text + expression text # 実行結果 "result2=sample_func_1('123')" exec(text) result2 # 実行結果 '123追加した文字列'
- 投稿日:2020-11-24T20:57:52+09:00
〔備忘録〕四則演算に関する演算子
Pythonで利用可能な四則演算一覧
数値型で使用可能な四則演算の一覧まとめです。数値型と言って急に出てきました。もう急に難しい感じです。要は「各データは型に分類されるよ、データ取り扱いの際は注意してね」ってことで納得しました。詳しくは下記を参照してください。最初の数行で挫折しそうですが目を通しました。脳みそをすべっていく感じがします。
データ型とはためしに
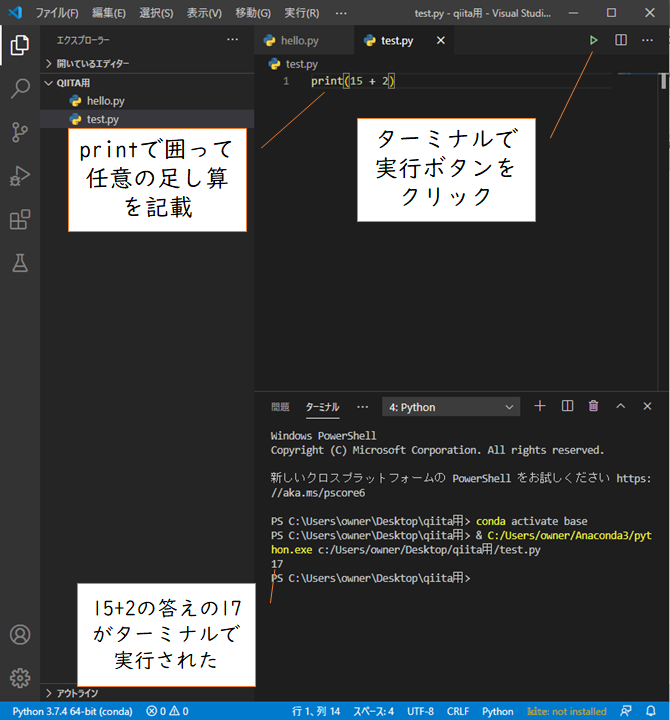
足し算のことです。これを記載する前にtest.pyにひたすら足し算の式を記入しターミナルで実行してもなにも表示されなくて途方にくれましたが、きちんとprintでくくらないとターミナルに表示されないんですね。もしくはターミナルに「python」て入力してインタラクティブモードで行う必要があります。
一覧
出力しないのでprintは記載しません。ターミナルに出力した場合はprintで囲ってください。普通の四則演算はできます。
test.pyx + y #xとyの和 x - y #xとyの差 x * y #xとyの積 x / y #xとyの商 x % y #xをyで割った余り x ** y #xのy乗型の異なる演算
これらは何も数値型だけのものではなく、文字列型でも適用可能です。ためしに"apple"と"banana"を足してみるとターミナルにはapplebananaと結果が表示されます。
しかし数値型と文字列型はそのままでは演算できません。
試しに文字列型appleと数値型2をそのまま足し算するとエラーがでました。
数値型の2を文字列型の"2"とするとapple2とエラーなく計算されました。pythonのような動的型付け言語の場合はそこまで意識することはありませんが、意識して考えていくとその他の言語を勉強する際にも役に立つかもしれません。
Javaの本とかを読んでいると「そんなのみればわかるだろうよ!!」という部分にも型を指定しなければならないので大変そうでした。
- 投稿日:2020-11-24T19:47:14+09:00
[備忘録]anaconda&VScodeでpythonのimportエラーに手こずった話
はじめに
ターミナルでpythonを実行するとモジュールのインポートでエラーが出てしまいました。今回はその備忘録です。
環境
mac:Big Sur
エディタ:VScode
Python:3.8
トラブル内容
VScodeで書いたpythonファイルをターミナルで実行するとそんなモジュールねぇよって怒られる。
でもインポートしてるのnumpyだし、ないわけないじゃん!!てことでひたすらググったら
どうやらターミナルから使用するpythonとVScodeで設定したpythonが異なるとインポートエラーが起こる模様解決方法
ターミナルで
pip show numpyと入力してLocationのところを見る。自分の場合は
anaconda3/lib/python3.8/site-packagesとなっていてanaconda3の中のpythonを使用していた。なのにVScodeで/usr/bin/python3を指定
していたのでエラーが出てしまった。わかれば簡単なことだったなぁ。後日談
このトラブルを解決した数日後にまたしても同じエラーが起きた。今回は使用するpythonも一致しているのにどしてと思ってまたひたすらググる。結果としては、モジュールのバージョンが古かったらしい。
なのでpip install -U numpyとすることで解決。numpyのところをエラーの出ているモジュール名に変えれば汎用できます。
ちなみにpip list -oとするとバージョンの古いやつが一覧で表示されます。
- 投稿日:2020-11-24T19:32:52+09:00
ポケモンホームAPIからランクバトルの環境の中心を可視化する
ポケモンの環境とは
ポケモン対戦で使われる環境という言葉は,ポケットモンスター ソード・シールドのランクバトルにおいてどんなポケモン・戦略・構築が使われているかを指す言葉です.環境を知ることで,それに対するメタを考察することで現環境で勝ちやすい構築を作っていくのがポケモン対戦の醍醐味と言えます.
環境の中心
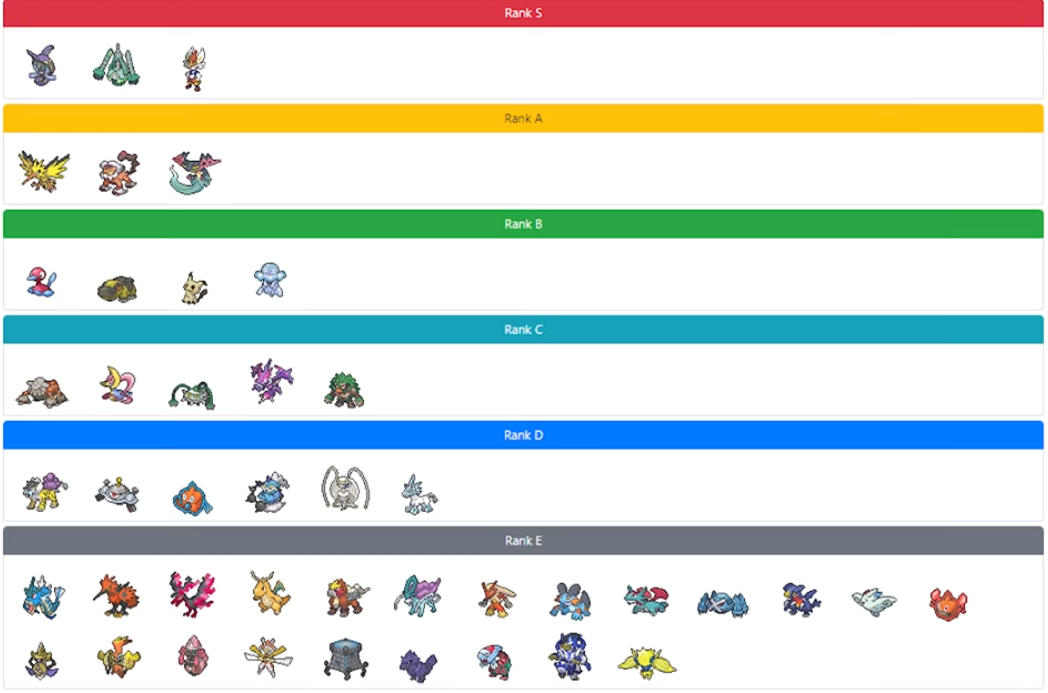
ガチ勢向けポケモン対戦情報共有サイトのポケモンソルジャーさんが運営するキャラランクメーカーではS ~ Eランクまでの評価を個人的に設定することができ,有名な実況者や解説動画投稿者が主観的に評価を下したキャラランクが参考にされています.
ポケモンソルジャーさん自身も定期的にキャラランクを公開しています.
https://youtu.be/TMXQ497foRgこのキャラランクのSランクについて,ポケモンソルジャーさんでは「環境の中心」と動画内で定義しています.
一方でその環境の中心の評価については主観的であることが問題であると考え,定量的な評価手法が必要であると考えました.目標
目標はずばり,「環境の中心を定量的に評価する」ことです.この記事では,可能な限り環境の中心をデータに基づいて考察したいと思います.
ポケモンホーム
ポケモンホームはスマホとSwitchで利用可能なポケモンの管理アプリです.ソフト内に保管しておけるポケモンの数に限りがあるため,こちらに不要なポケモンを一時的に保管するなどして使うことができます.
このポケモンホームにはそれ以外には交換機能やランクバトルでのポケモンの環境情報・プレイヤーのランキング機能なども備わっていて,ガチ勢の間ではこまめに環境調査に利用されています.
API
コチラの記事で紹介されている通り,ポケモンホームはUser-Agentを偽装することでアプリ内で利用されているAPIを直接叩いてjsonデータを取得することができます.今回使用するのは以下のAPIです.詳細についてはリンク先からご確認ください.
# シーズン・タームの情報の取得 https://api.battle.pokemon-home.com/cbd/competition/rankmatch/list # 該当シーズン・タームの対戦データの取得 https://resource.pokemon-home.com/battledata/ranking/$id/$rst/$ts2/pokemon # 該当シーズン・タームにおけるポケモンのデータ https://resource.pokemon-home.com/battledata/ranking/$id/$rst/$ts2/pdetail-$j評価手法
環境を視覚的にもわかりやすく,かつ分析がしやすい形に加工するために,今回はグラフを用いた評価を検討します.
グラフについては過去に書いた記事があるので,コチラを参照してください.この記事では,使用率ランキング上位のポケモンとの同時採用率の高さを評価することにしました.
環境の中心は,単体の性能ではなく他のポケモンに与える影響やパーティへの組み込みやすさを重視すべきだと考えたためです.
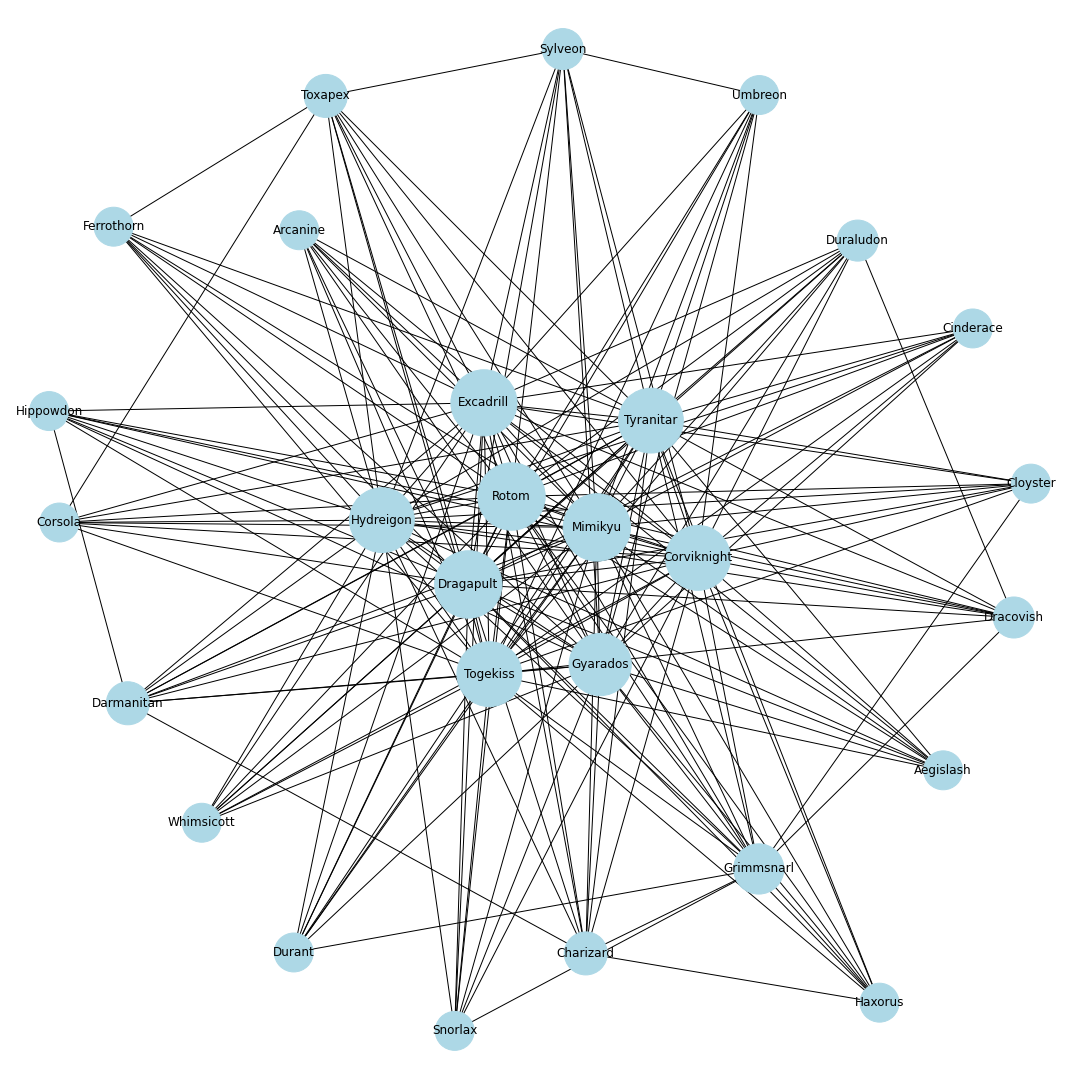
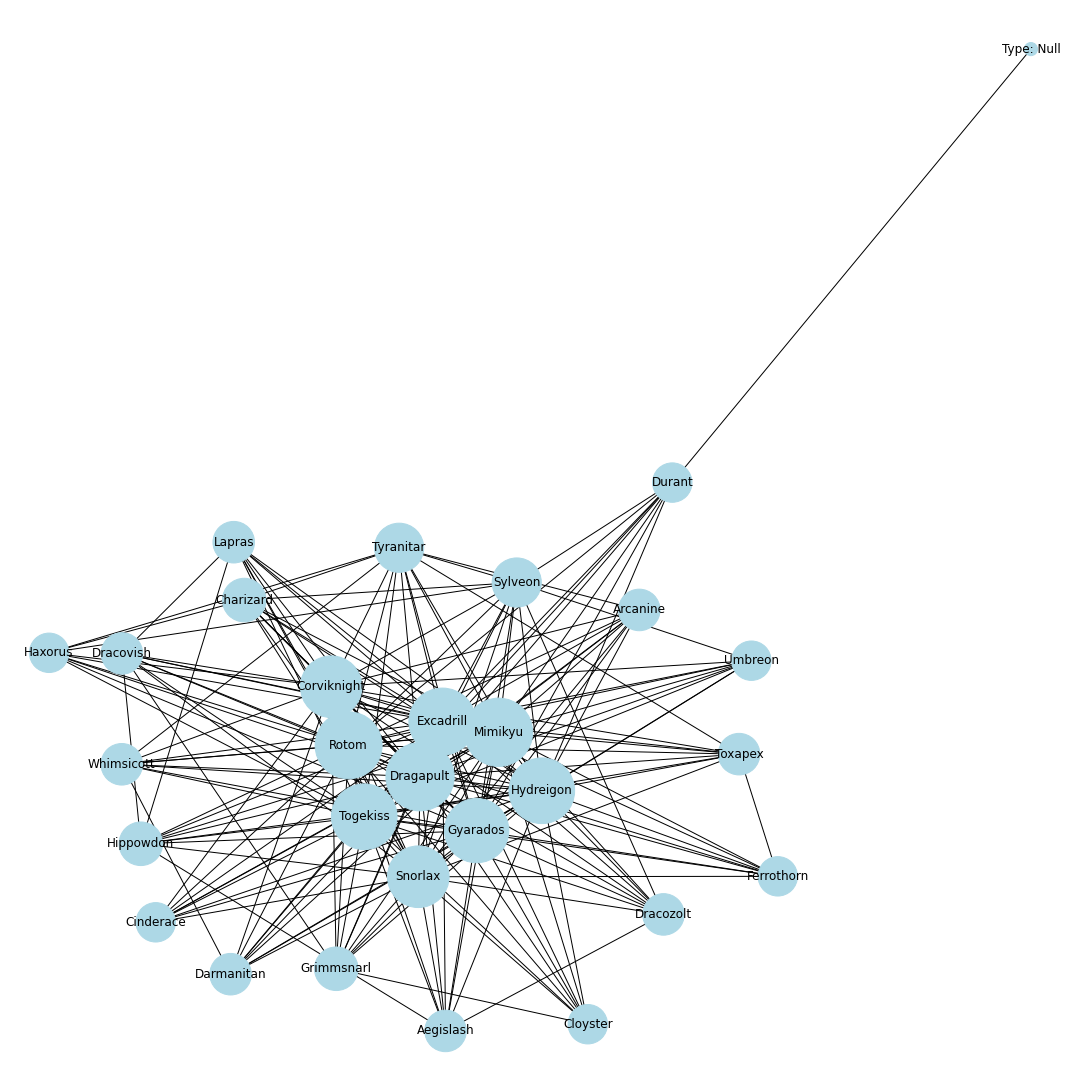
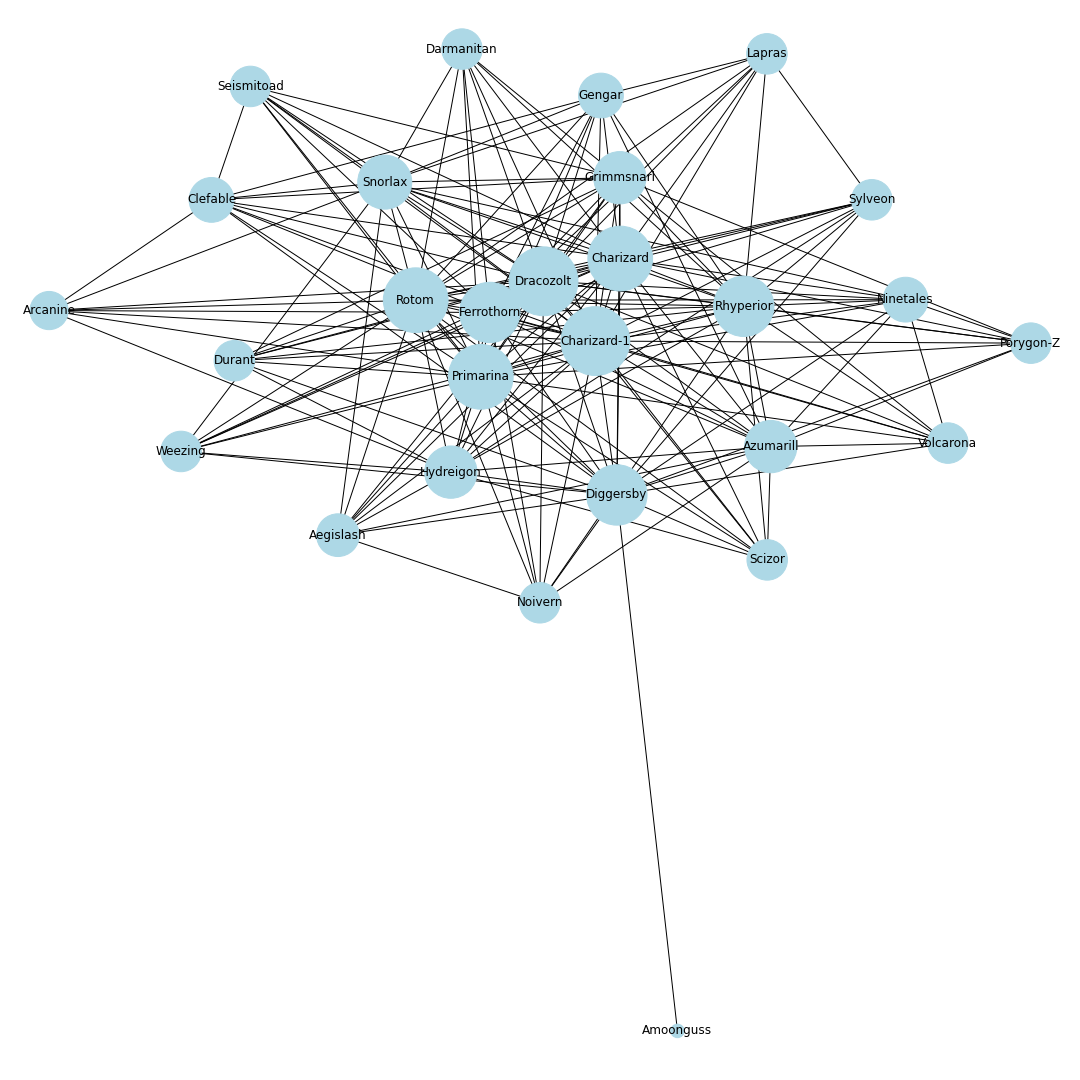
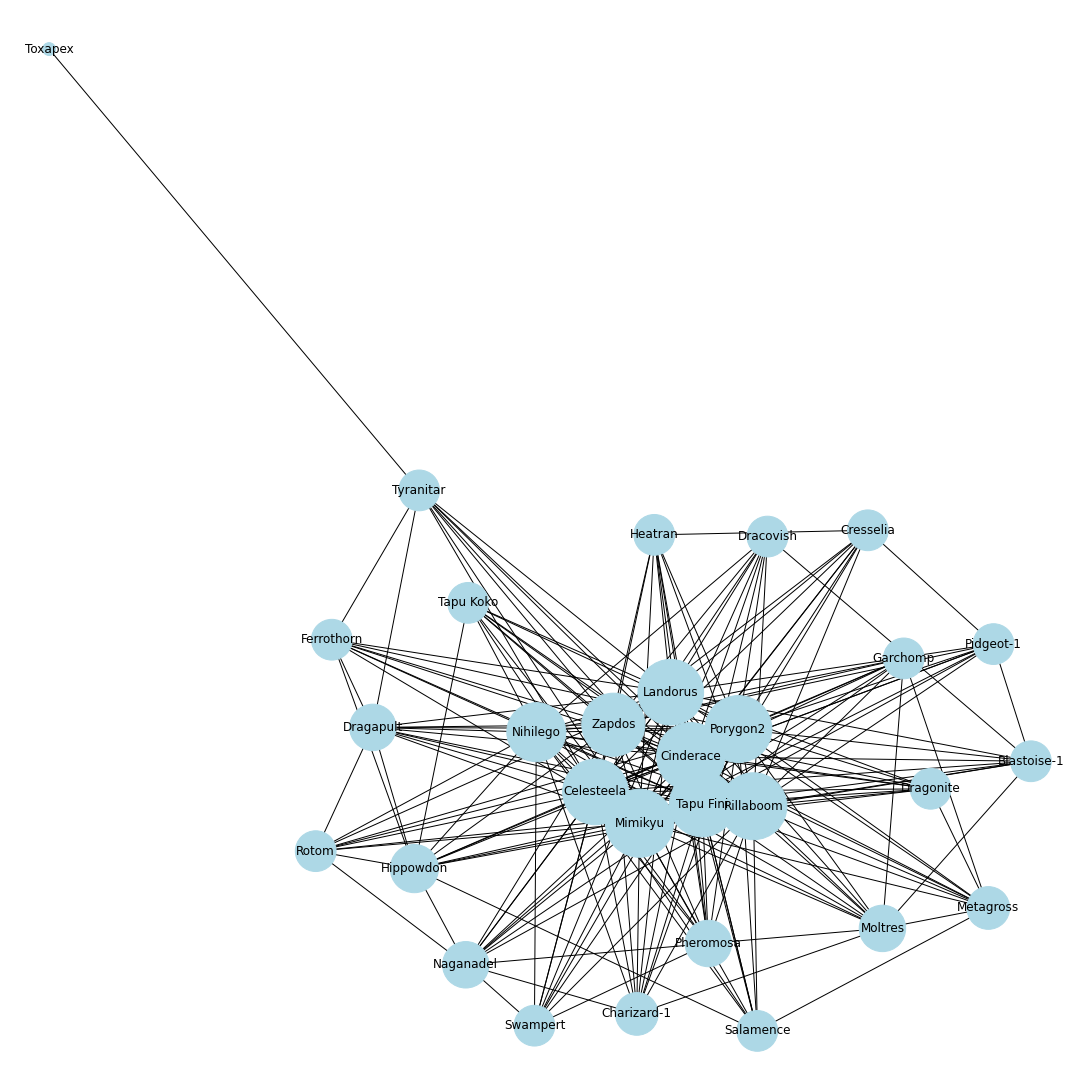
ポケモンホームの使用率ランキング上位30匹までの同時採用率が高いポケモンTOP10までを考慮し,その関係性を無向グラフで可視化することにします.ノードとエッジ
無向グラフではノードとエッジを定義する必要があります.
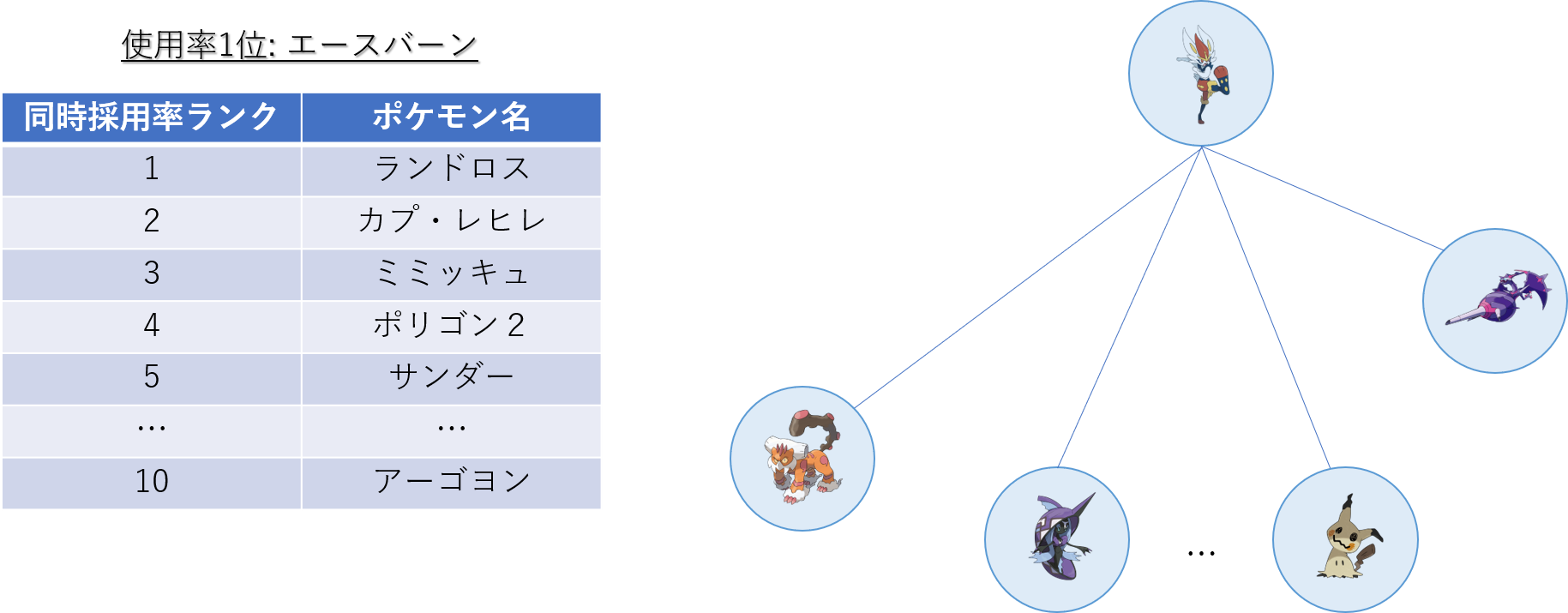
今回はノードをポケモン,エッジを同時採用率上位の有無としてグラフを作成します.
例えば,エースバーンと同時採用率が高い10匹を考えるとすると,エースバーンのノードから10本のエッジをそれぞれのポケモンのノードに伸ばすことになります.
これを上位30匹のポケモンに対して繰り返し,次数が多いノードに君臨するポケモンが環境上位のポケモンと相互に関係が強く影響を与えるポケモンであると仮定しました.
データ収集
必要なデータは以下の通りです.
- ポケモン図鑑(図鑑番号と名前の対応づけ)
- シーズン・タームのID情報(rst・ts1・ts2)
- 当該シーズン・タームの対戦環境情報(使用率上位のポケモンリスト)
- 当該シーズン・タームのポケモンに関する情報(同時採用率上位のポケモンリスト)
ポケモン図鑑
8世代に対応したポケモン図鑑を探したところ,jsonファイルで配布してくださっている方がいたのでコチラをお借りしました.まだ鎧の孤島以降に追加されたウーラオス・ブリザポス等のポケモンの情報が更新されていないため,最新の環境で試すと図鑑にないポケモンのせいで少し結果にノイズが入ります.
> git clone https://github.com/dayu282/pokemon-data.jsonimport json pokedex = {} for i in range(8): # すべての世代のポケモンを追加 j = i + 1 with open("pokemon-data.json/en/gen" + str(j) + "-en.json", "r", encoding = "utf-8") as f: pokemons = json.loads(f.read()) for pokemon in pokemons: pokedex[pokemon["no"]] = pokemon["name"]シーズン・タームのID情報
# シーズンの情報を丸ごと全取得してjson形式で保存 > curl https://api.battle.pokemon-home.com/cbd/competition/rankmatch/list \ -H 'accept: application/json, text/javascript, */*; q=0.01' \ -H 'countrycode: 304' \ -H 'authorization: Bearer' \ -H 'langcode: 1' \ -H 'user-agent: Mozilla/5.0 (Linux; Android 8.0; Pixel 2 Build/OPD3.170816.012) AppleWebKit/537.36 (KHTML, like Gecko) Chrome/83.0.4103.61 Mobile Safari/537.36' \ -H 'content-type: application/json' \ -d '{"soft":"Sw"}' \ -o season.jsonwith open("season.json", "r", encoding = "utf-8") as f: data = json.load(f)["list"] # タームごとに必要な情報だけを配列にまとめる terms = [] for season in data: for id in data[season]: terms.append({"id" : id, "season" : data[season][id]["season"], "rule" : data[season][id]["rule"], "rst" : data[season][id]["rst"], "ts1" : data[season][id]["ts1"], "ts2" : data[season][id]["ts2"]})当該シーズン・タームの対戦環境情報・ポケモンに関する情報
Jupyterを使ってコマンドに変数を埋め込むことでループを使って取得していく.ポケモン情報については5分割されているので,pdetail-{1から5}すべてを取得して連結する
for term in terms: if term["rule"] == 0: # 対戦環境の取得 id = term["id"] rst = term["rst"] ts1 = term["ts1"] ts2 = term["ts2"] pokemons_file = str(id) + "-pokemons.json" !curl -XGET https://resource.pokemon-home.com/battledata/ranking/$id/$rst/$ts2/pokemon -H 'user-agent: Mozilla/5.0 (Linux; Android 8.0; Pixel 2 Build/OPD3.170816.012) AppleWebKit/537.36 (KHTML, like Gecko) Chrome/83.0.4103.61 Mobile Safari/537.36' -H 'accept: application/json' -o $pokemons_file for i in range(5): # 当該タームのポケモン情報を取得 j = i + 1 pokeinfo_file = str(id) + "-pokeinfo-" + str(j) + ".json" !curl -XGET https://resource.pokemon-home.com/battledata/ranking/$id/$rst/$ts2/pdetail-$j -H 'user-agent: Mozilla/5.0 (Linux; Android 8.0; Pixel 2 Build/OPD3.170816.012) AppleWebKit/537.36 (KHTML, like Gecko) Chrome/83.0.4103.61 Mobile Safari/537.36' -H 'accept: application/json' -o $pokeinfo_fileid = 10011 # シーズン1のシングルバトル # 上位のポケモンリスト with open(str(id) + "-pokemons.json", "r", encoding = "utf-8") as f: top_pokemons = json.load(f)[:30] # ポケモンの情報 pokemons_info = {} for i in range(5): j = i + 1 with open(str(id) + "-pokeinfo-" + str(j) + ".json") as f: data = json.load(f) for index in data.keys(): pokemons_info[index] = data[index]グラフの描画と次数ランキングの作成
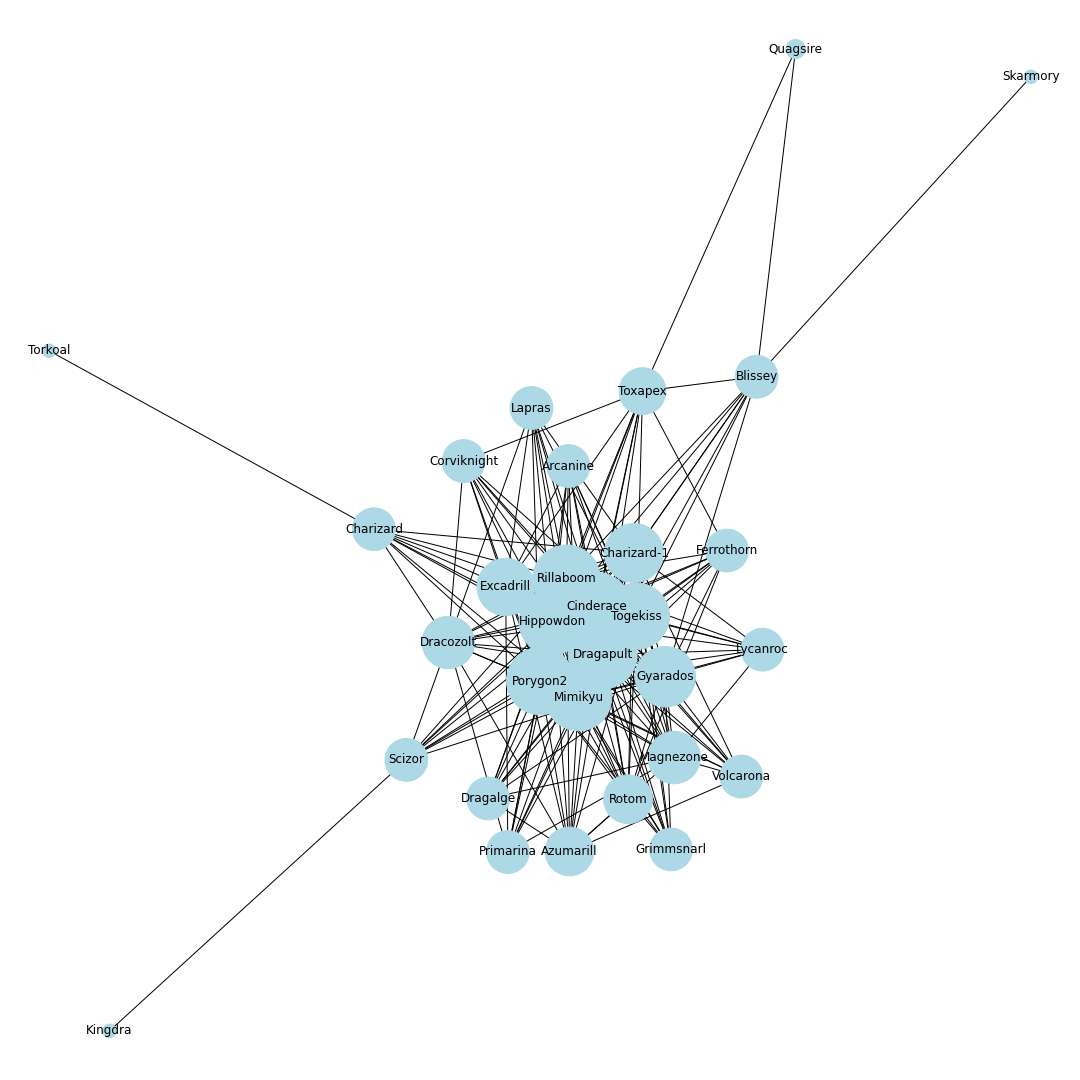
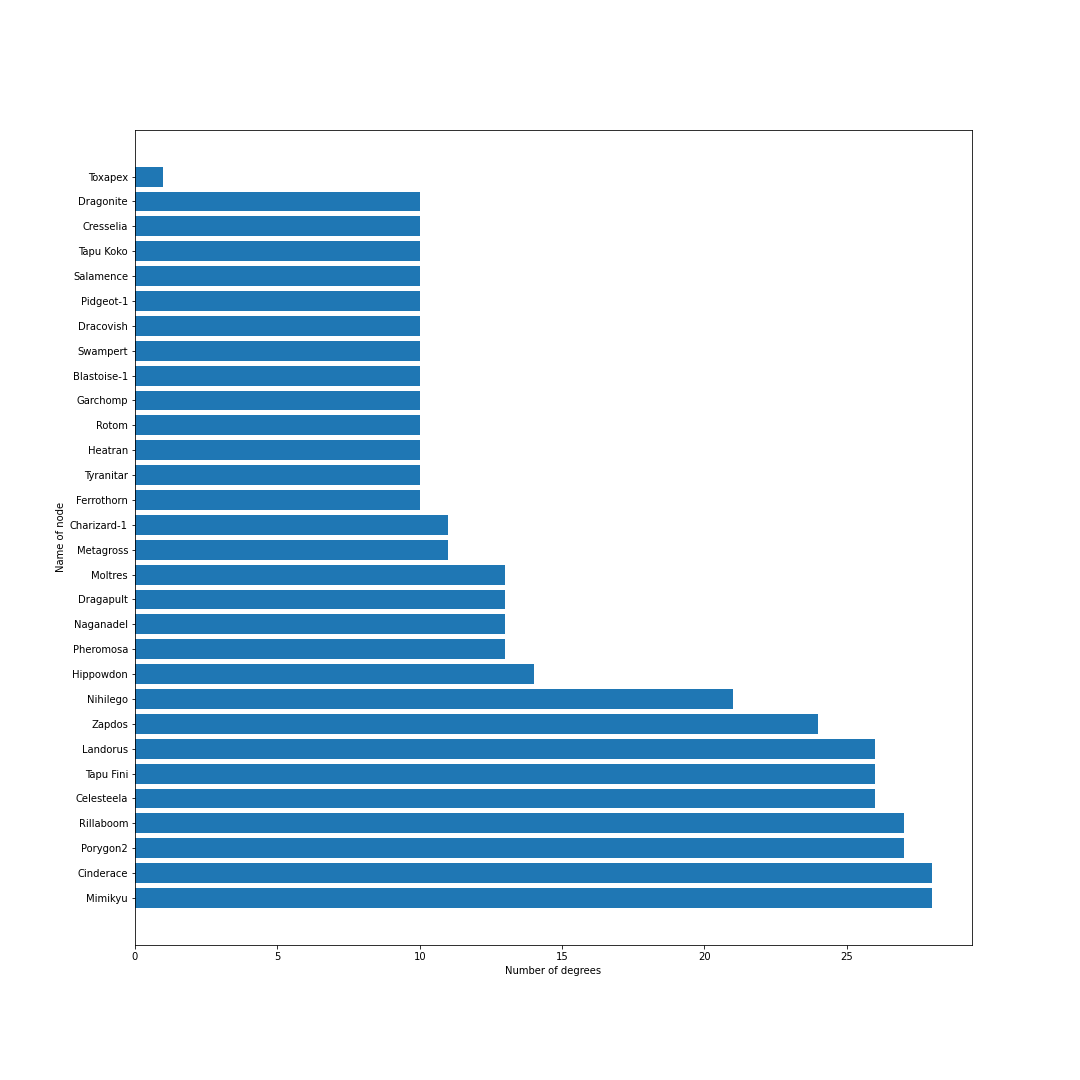
必要なデータが集まったので,グラフを描画していきます.
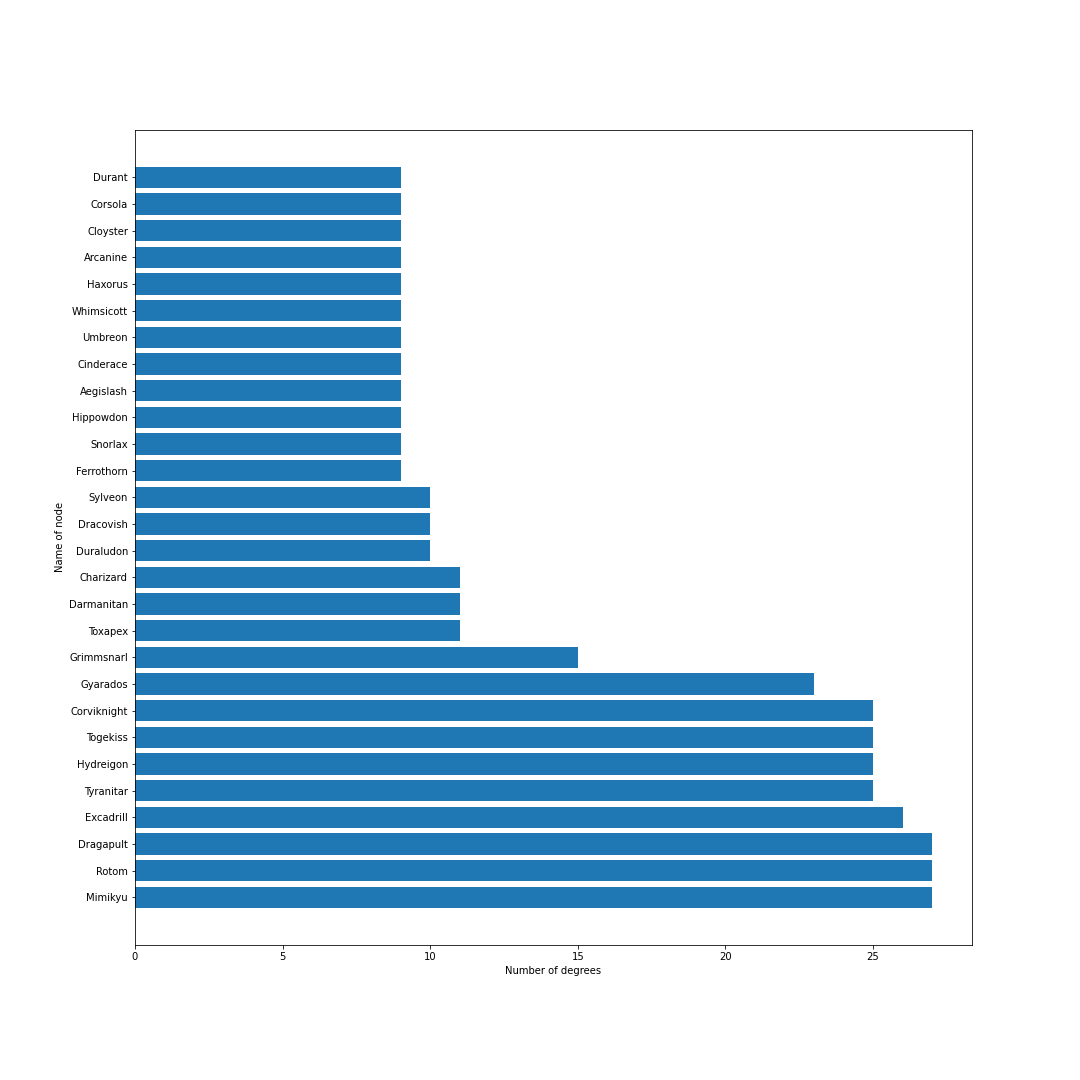
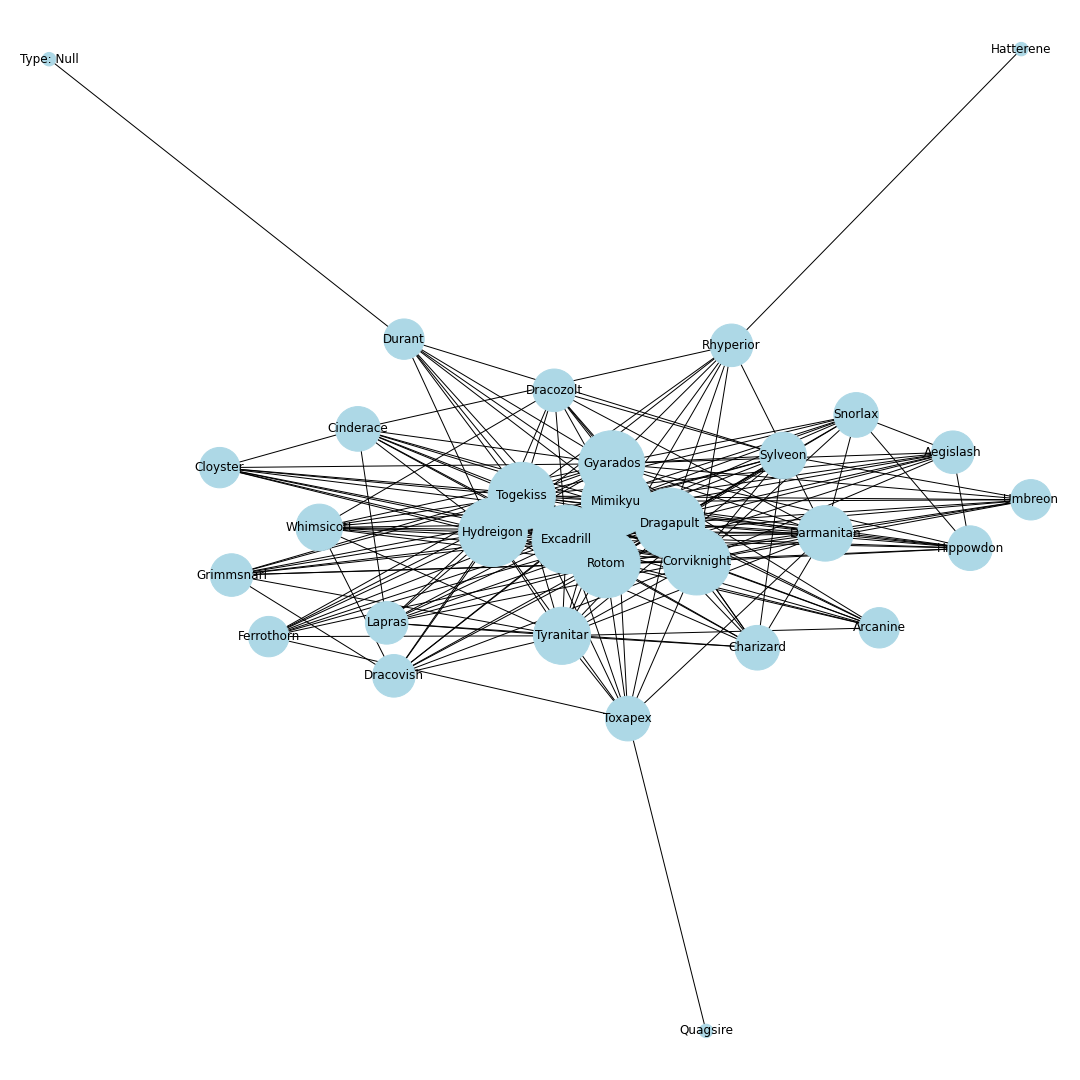
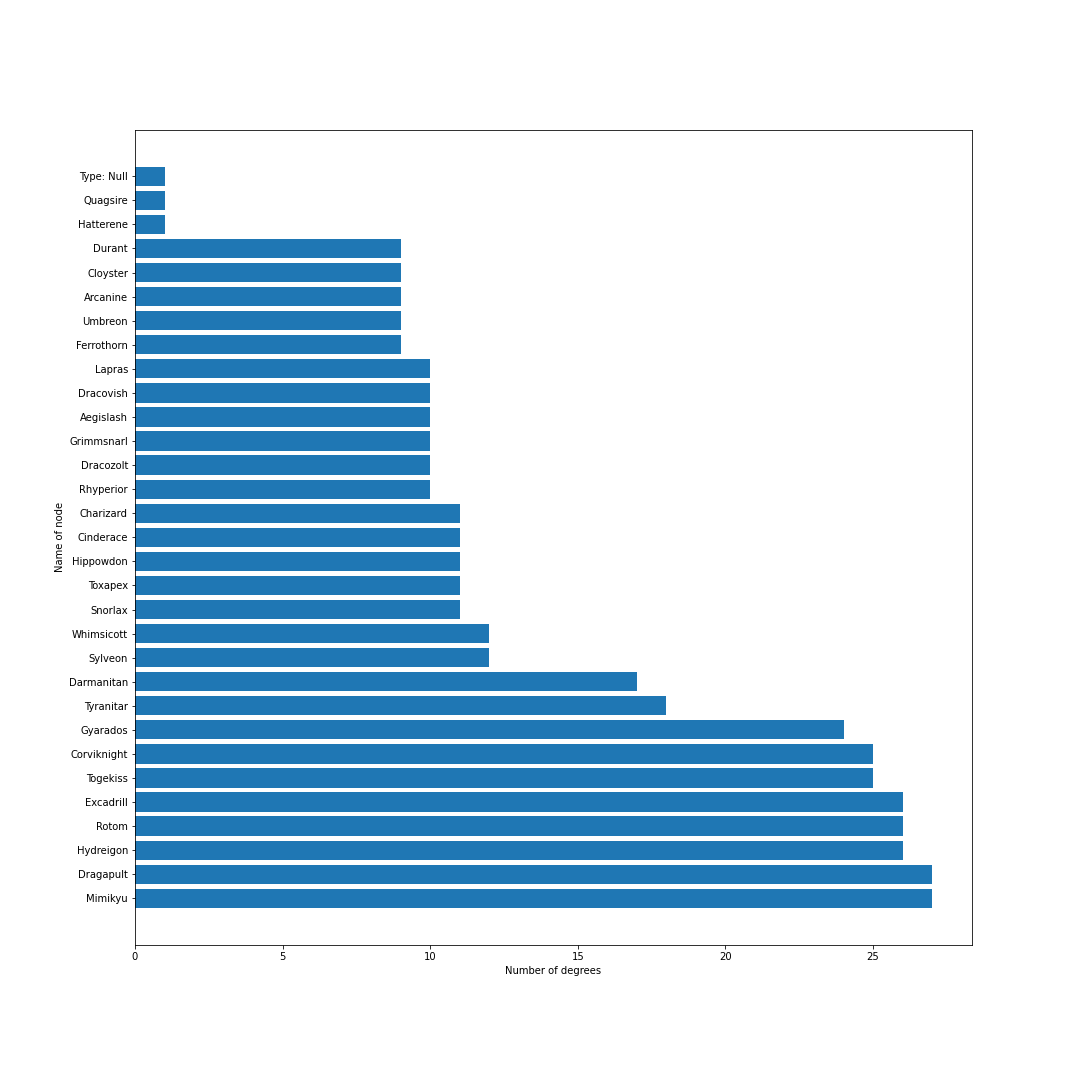
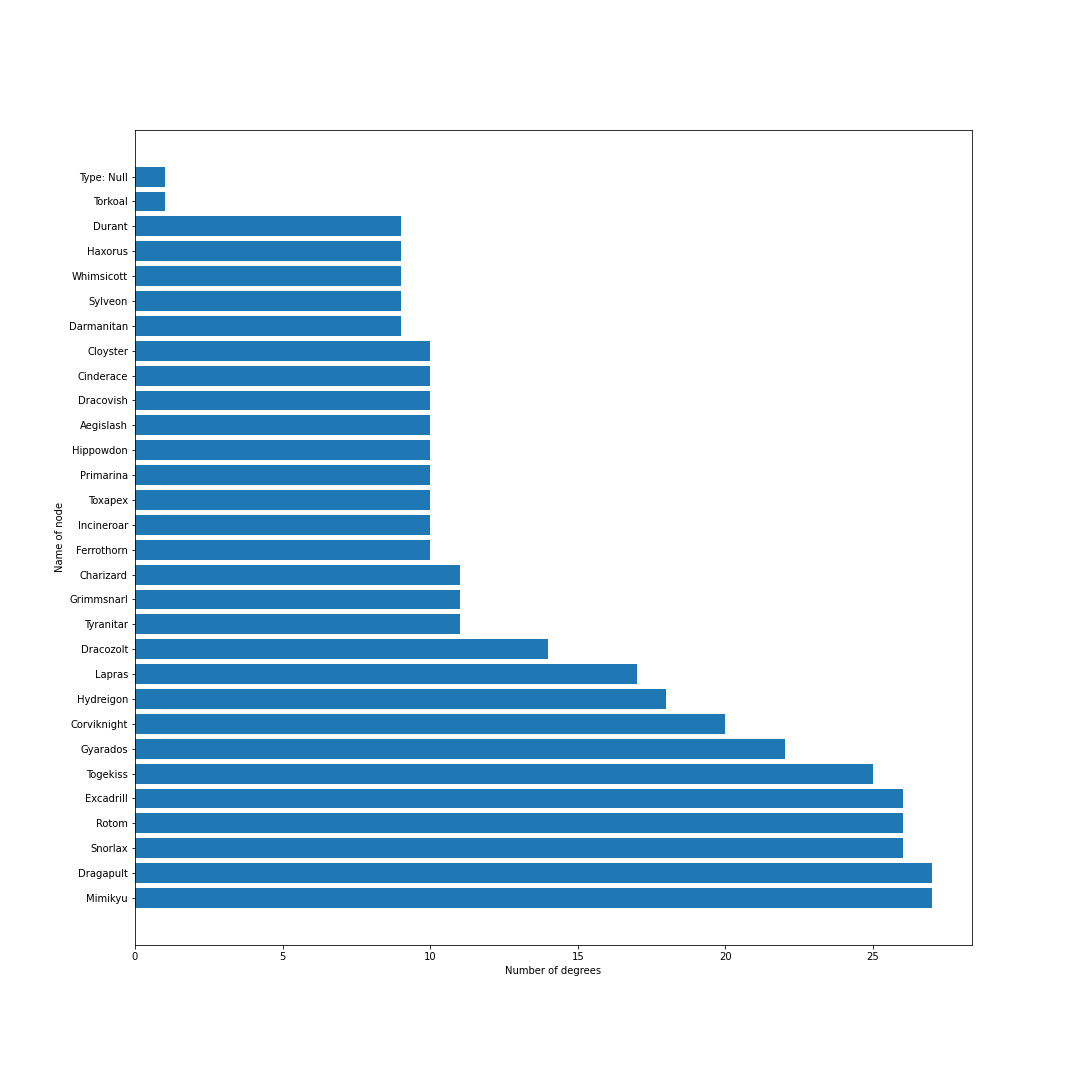
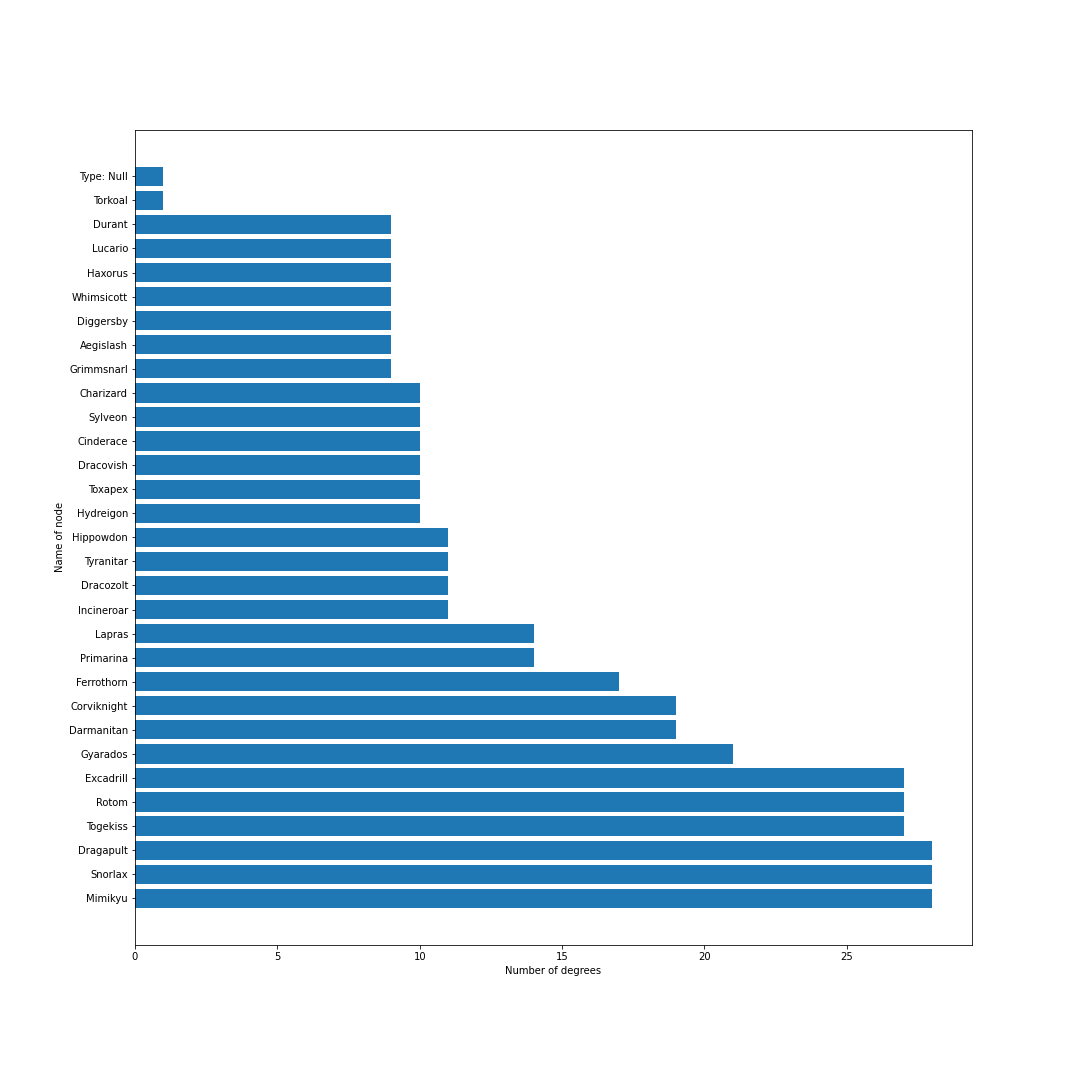
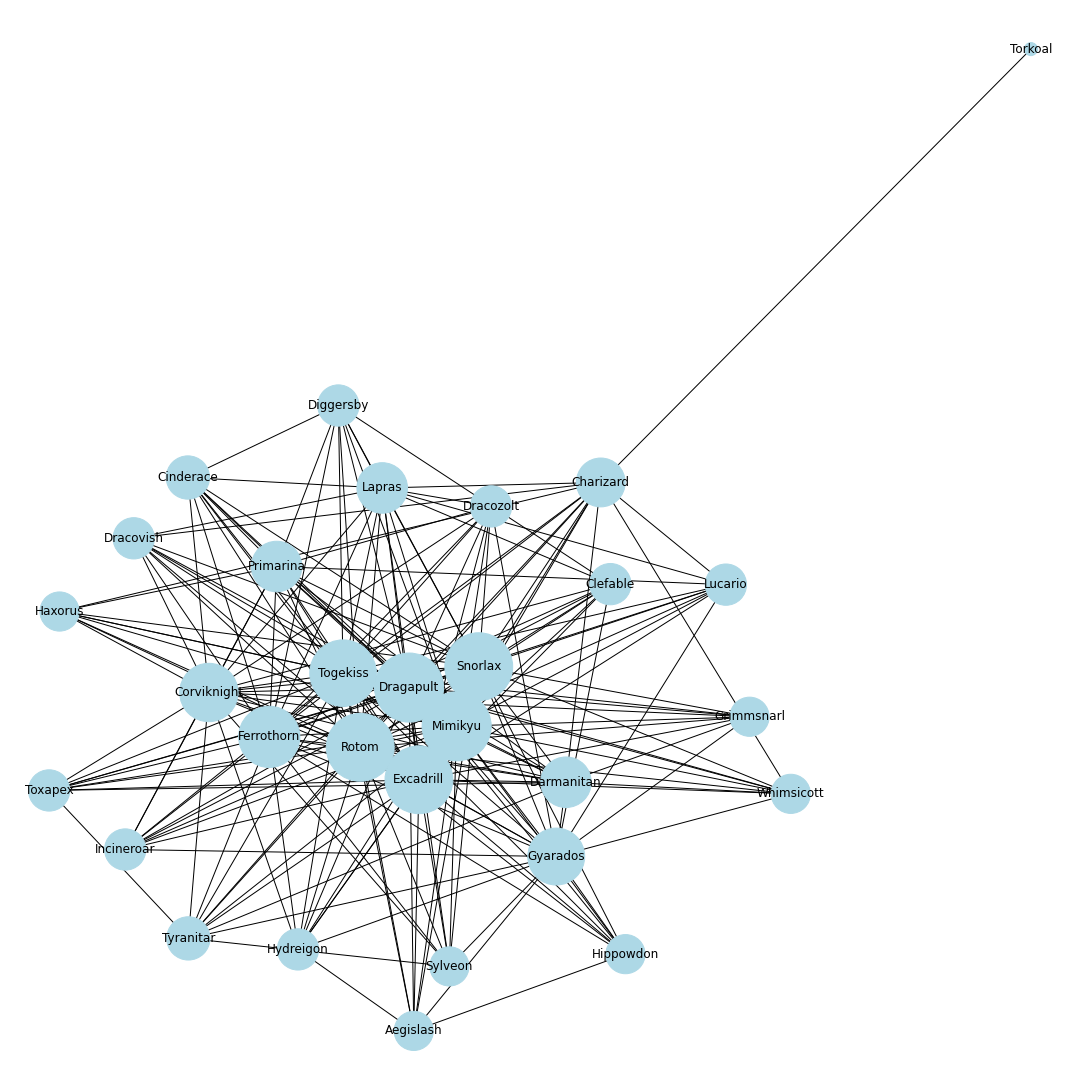
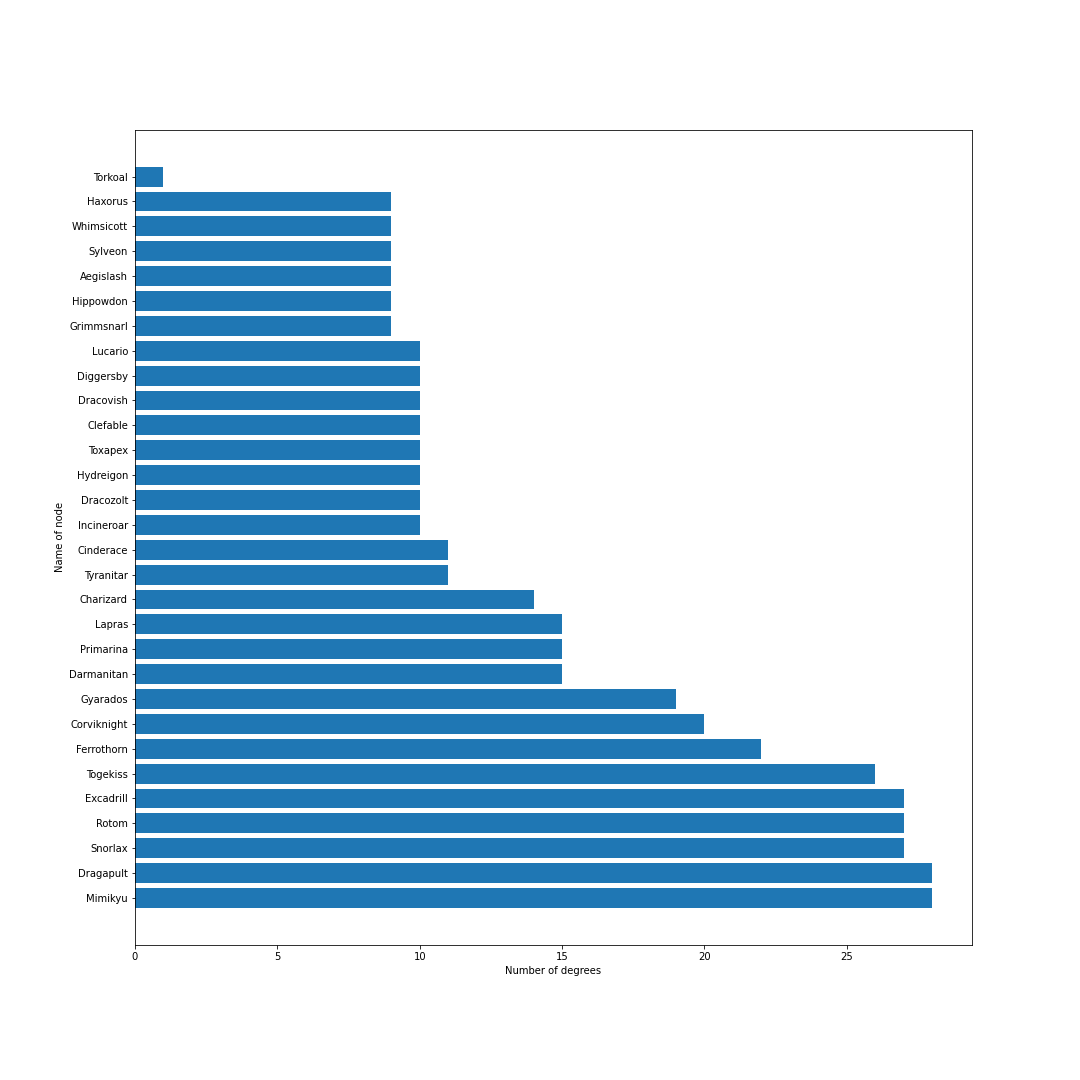
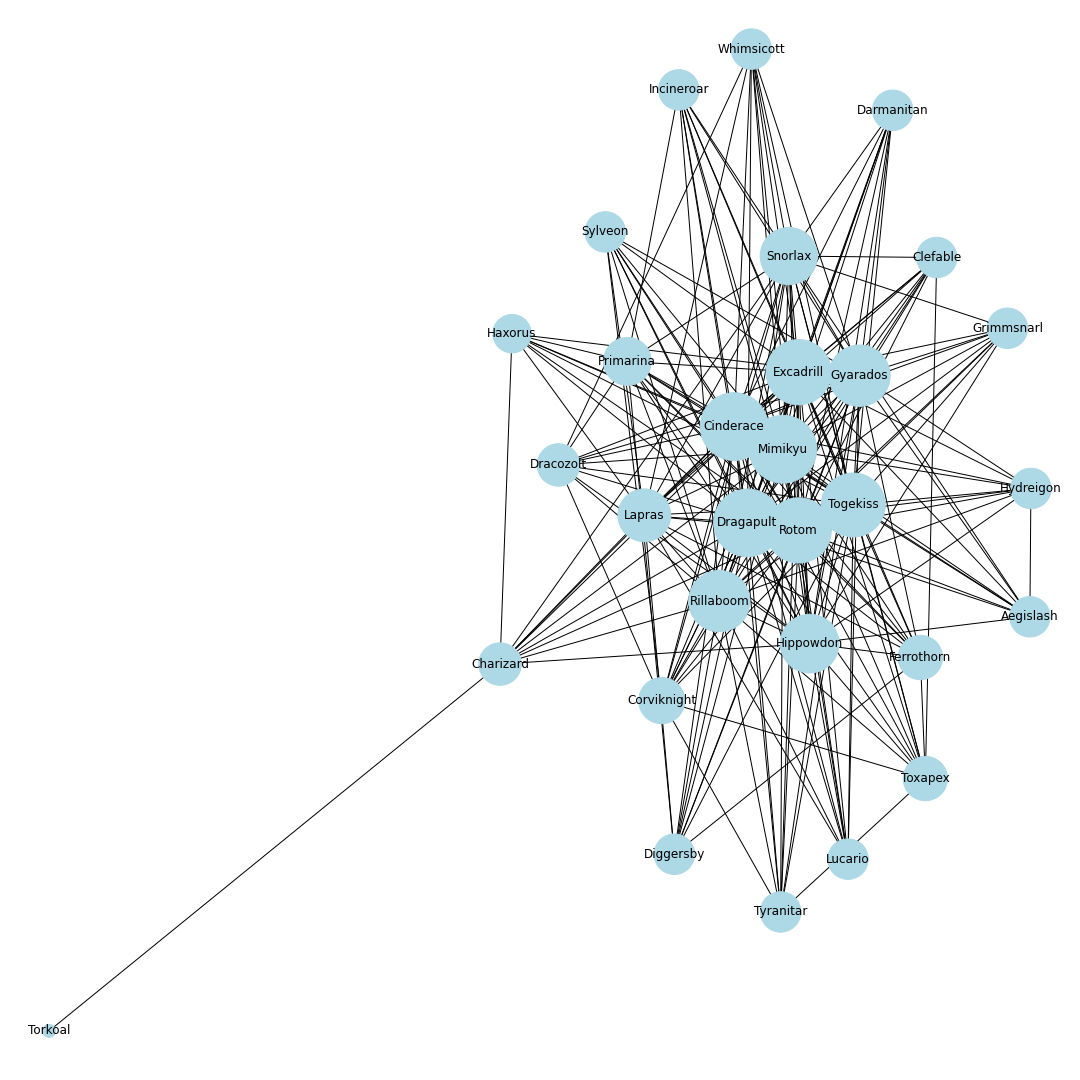
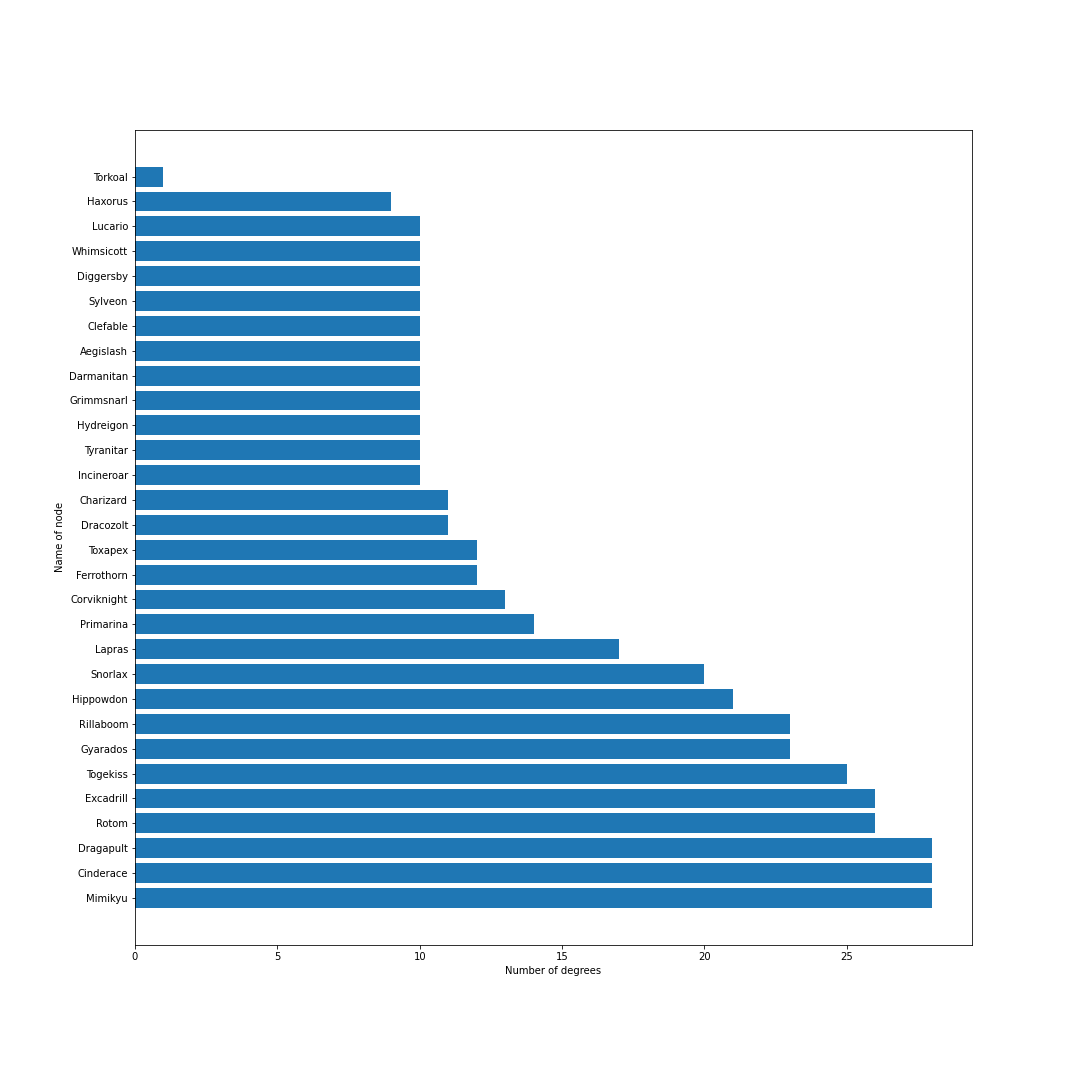
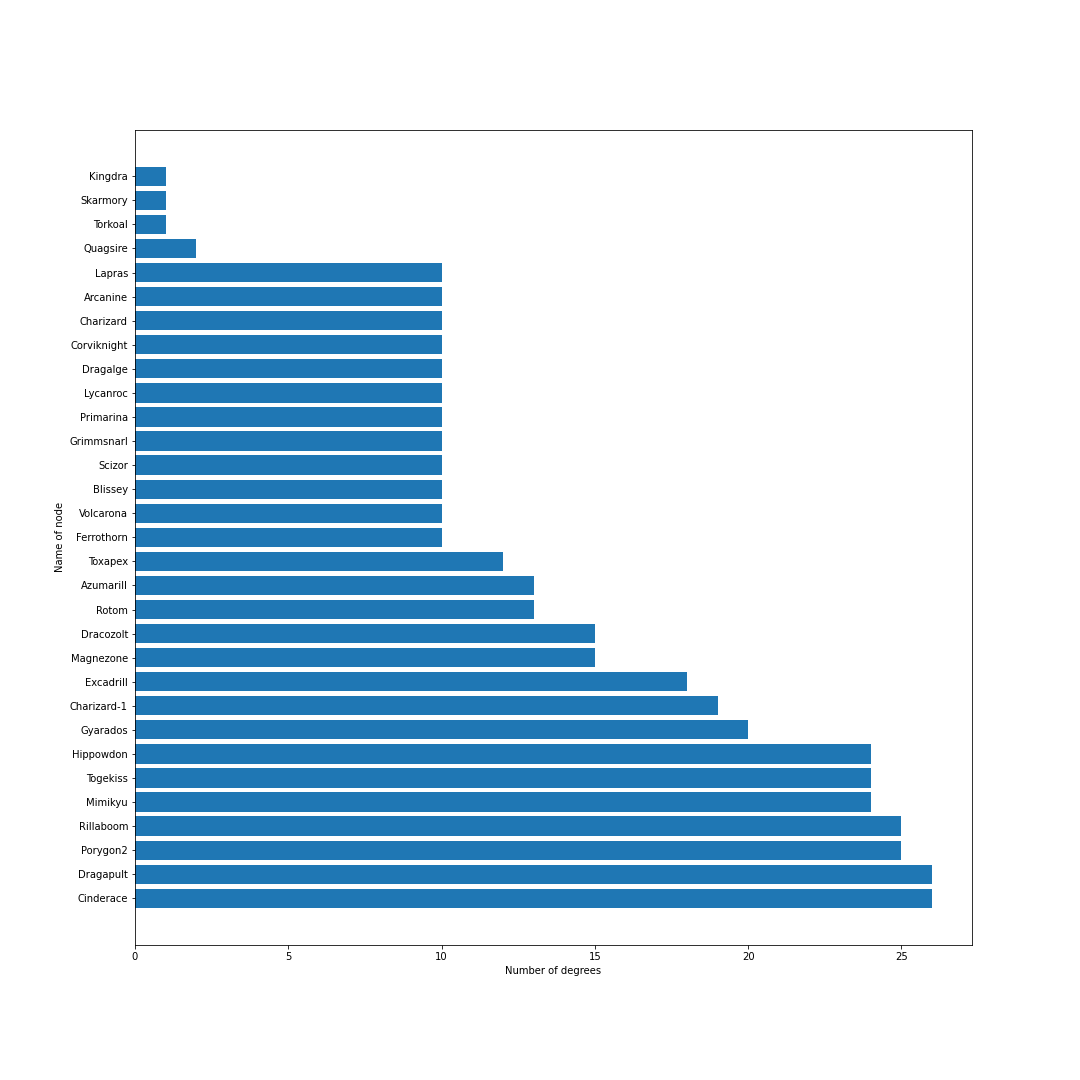
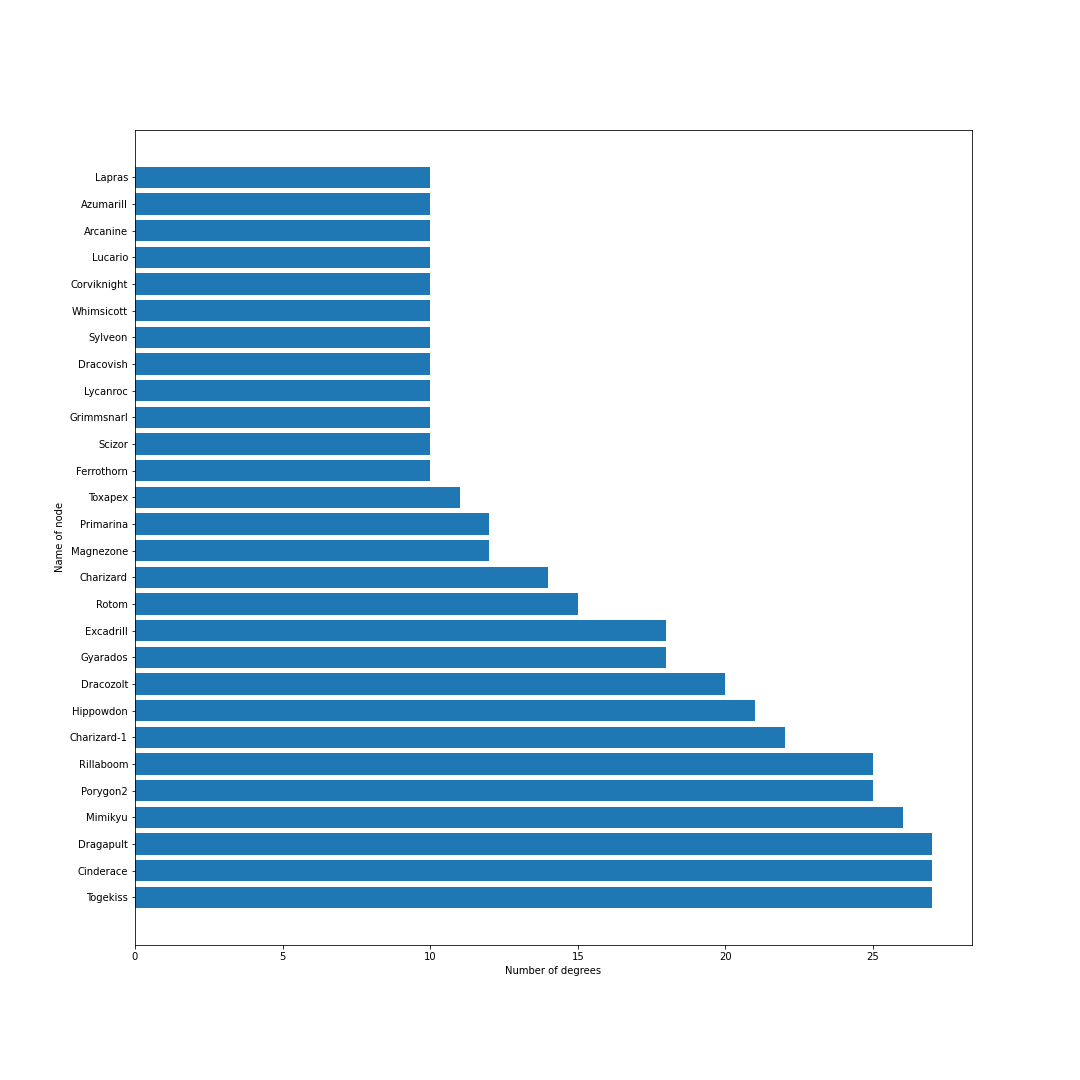
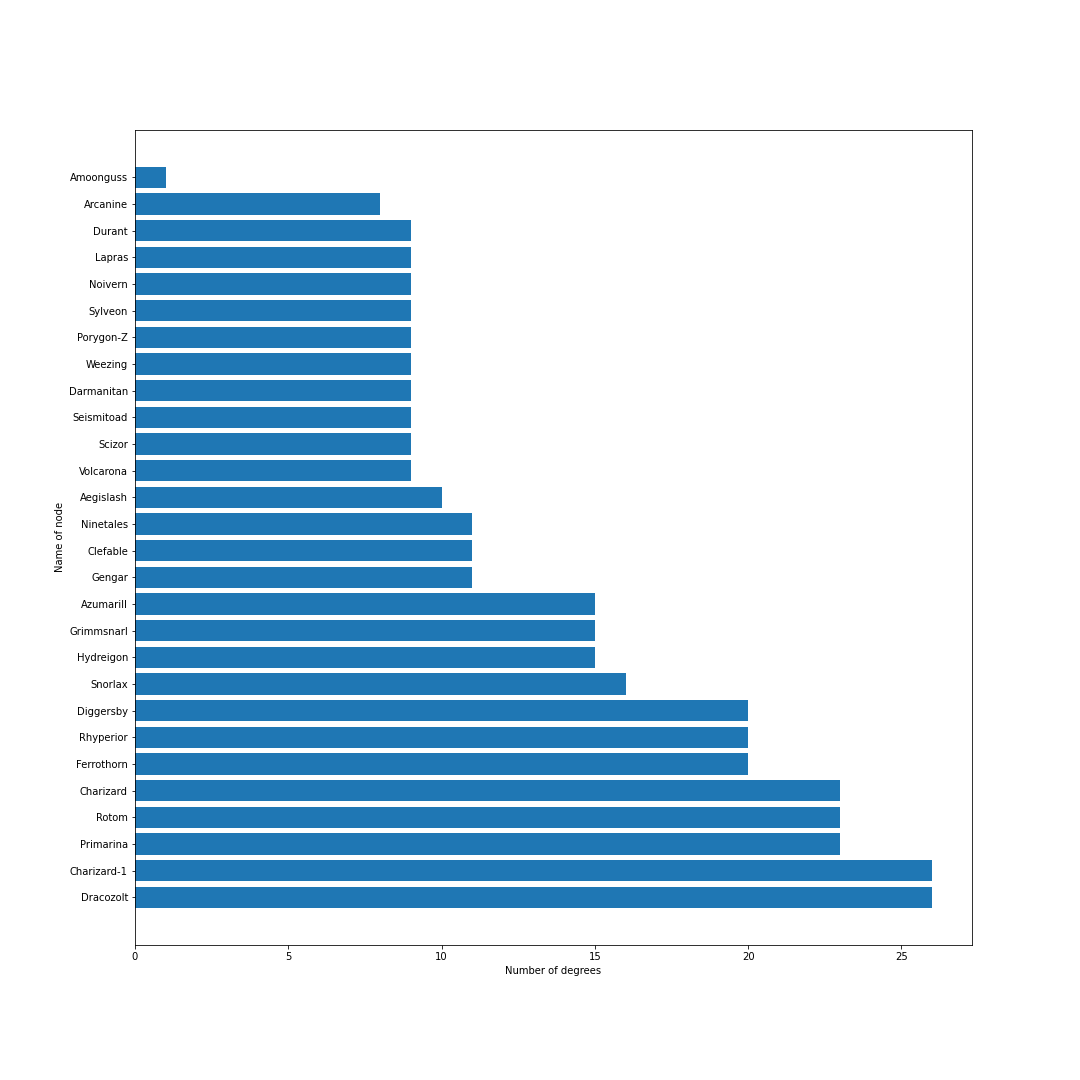
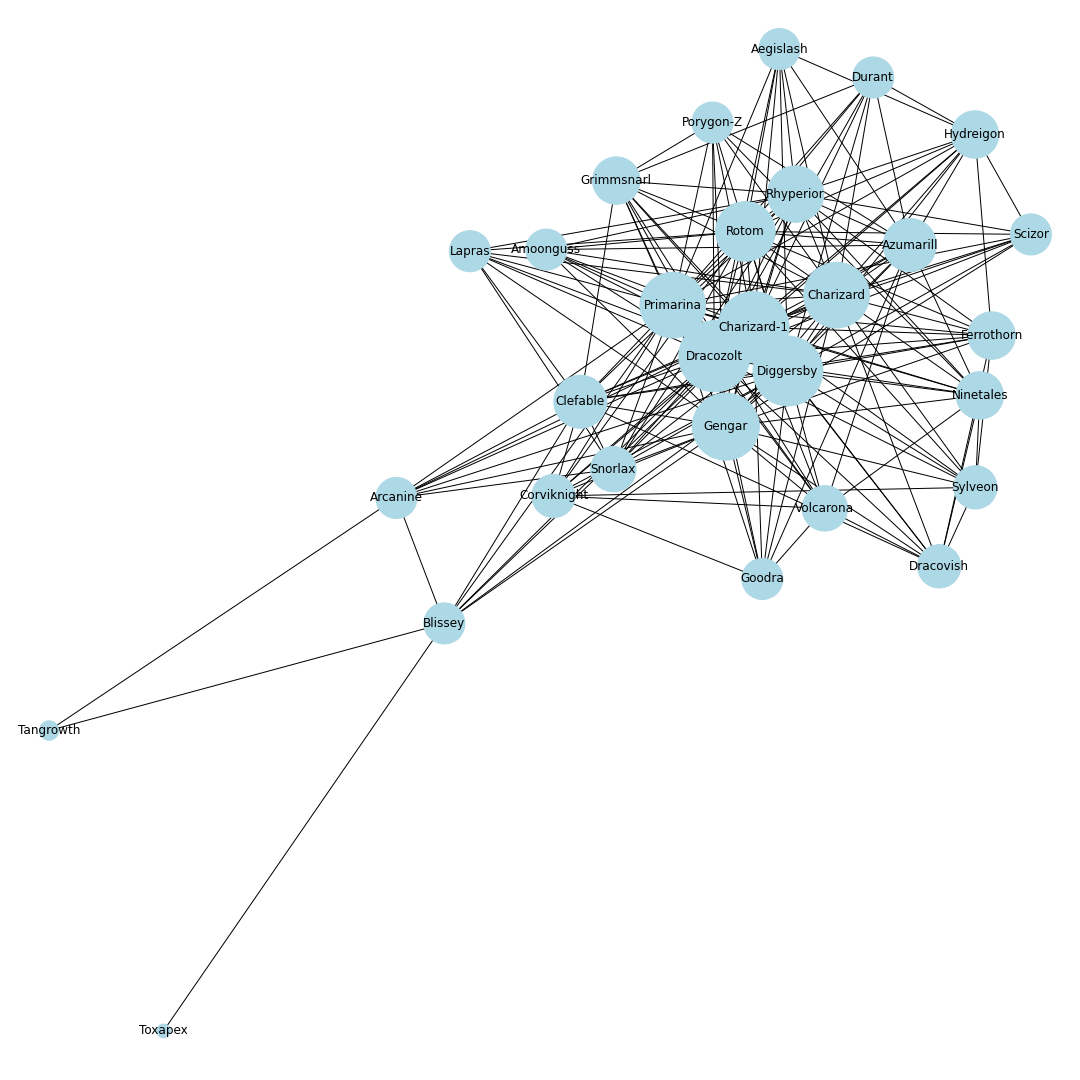
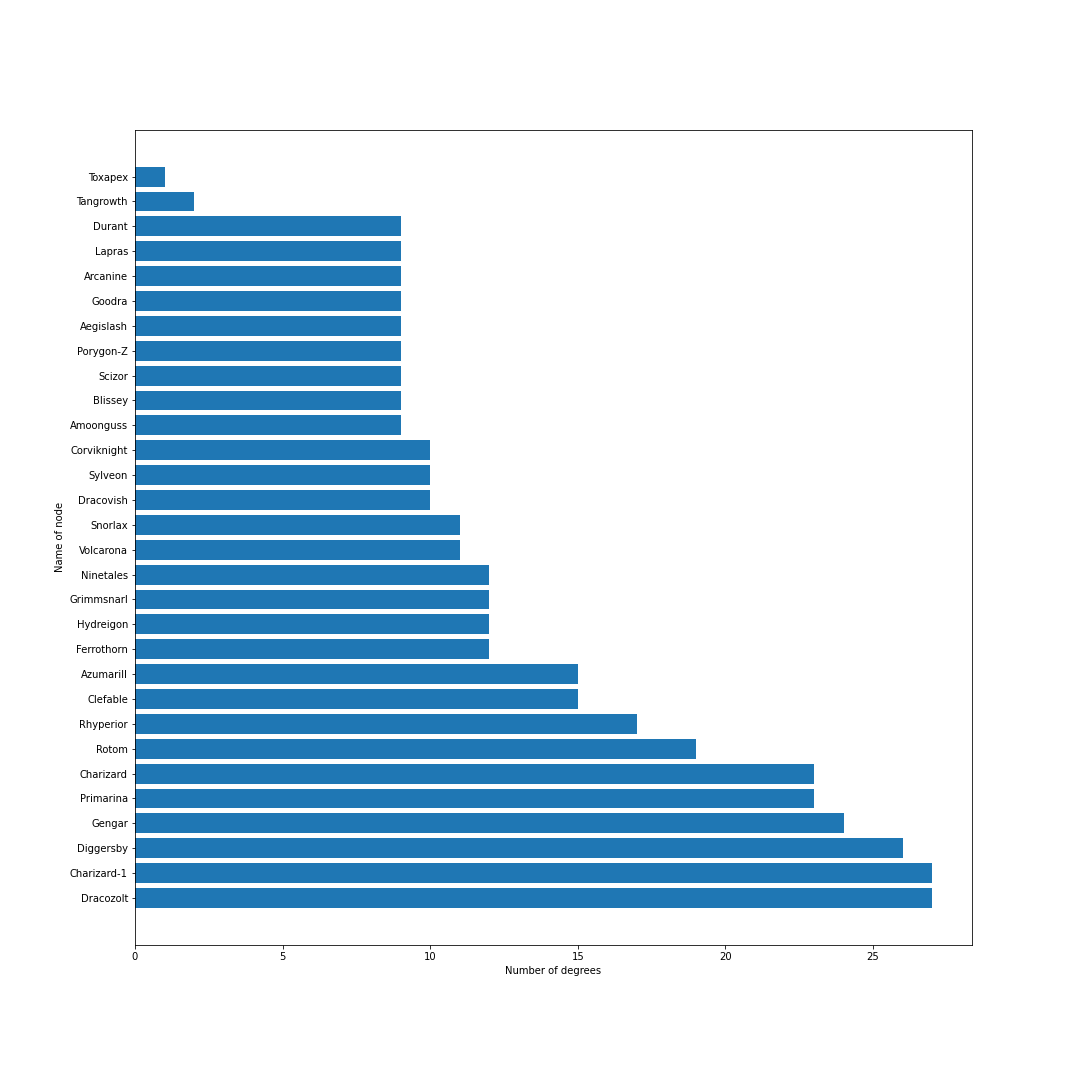
nodes = [] # ノードリスト edges = [] # エッジリスト # トップ30匹を考慮 for pokemon in top_pokemons: # 当該ポケモンをノードに追加 nodes.append(pokedex[pokemon["id"]]) # 一緒に採用されるポケモンを追加 with_poke_list = pokemons_info[str(pokemon["id"])][str(pokemon["form"])]["temoti"]["pokemon"] for with_poke in with_poke_list: nodes.append(pokedex[with_poke["id"]]) if (pokedex[with_poke["id"]], pokedex[pokemon["id"]]) not in edges: # 順番による重複をなくす edges.append((pokedex[pokemon["id"]], pokedex[with_poke["id"]])) nodes = list(set(nodes)) # 一度集合に変換して重複なしにするimport networkx as nx import matplotlib.pyplot as plt from matplotlib import font_manager # 無効グラフ作成 G = nx.Graph() G.add_nodes_from(nodes) G.add_edges_from(edges) # ネットワーク全体の次数の平均値を計算 average_deg = sum(d for n, d in G.degree()) / G.number_of_nodes() # ノードの次数に比例するようにサイズを設定 sizes = [2500*deg/average_deg for node, deg in G.degree()] plt.figure(figsize=(15, 15)) nx.draw(G, with_labels=True, node_color = "lightblue", node_size = sizes, edge_color = "black") plt.axis('off') plt.savefig(str(id) + '-graph.png') plt.show()# 次数の多い順番でnodeを並び替える nodes_sorted_by_degree = sorted(G.degree(), key=lambda x: x[1], reverse=True) print(nodes_sorted_by_degree) # 次数ランキングの出力 x, height = list(zip(*nodes_sorted_by_degree)) plt.figure(figsize=(15, 15)) plt.xlabel("Number of degrees") plt.ylabel("Name of node") plt.barh(x, height) plt.savefig(str(id) + '-degs.png') plt.show()結果
シーズン1から順番に示していきます.鎧の孤島解禁以降はノイズが乗っています.
シーズン1
剣盾環境開幕.次数的にはミミッキュ・ロトム・ドラパルト・ドリュウズなどが圧倒的に環境の中心に.グラフで見ると中心に9体のポケモンが固まっていてその周りを囲むようにほかのポケモンが描画されているので,その9体が環境の中心でSランクと考えてもよいと思います.
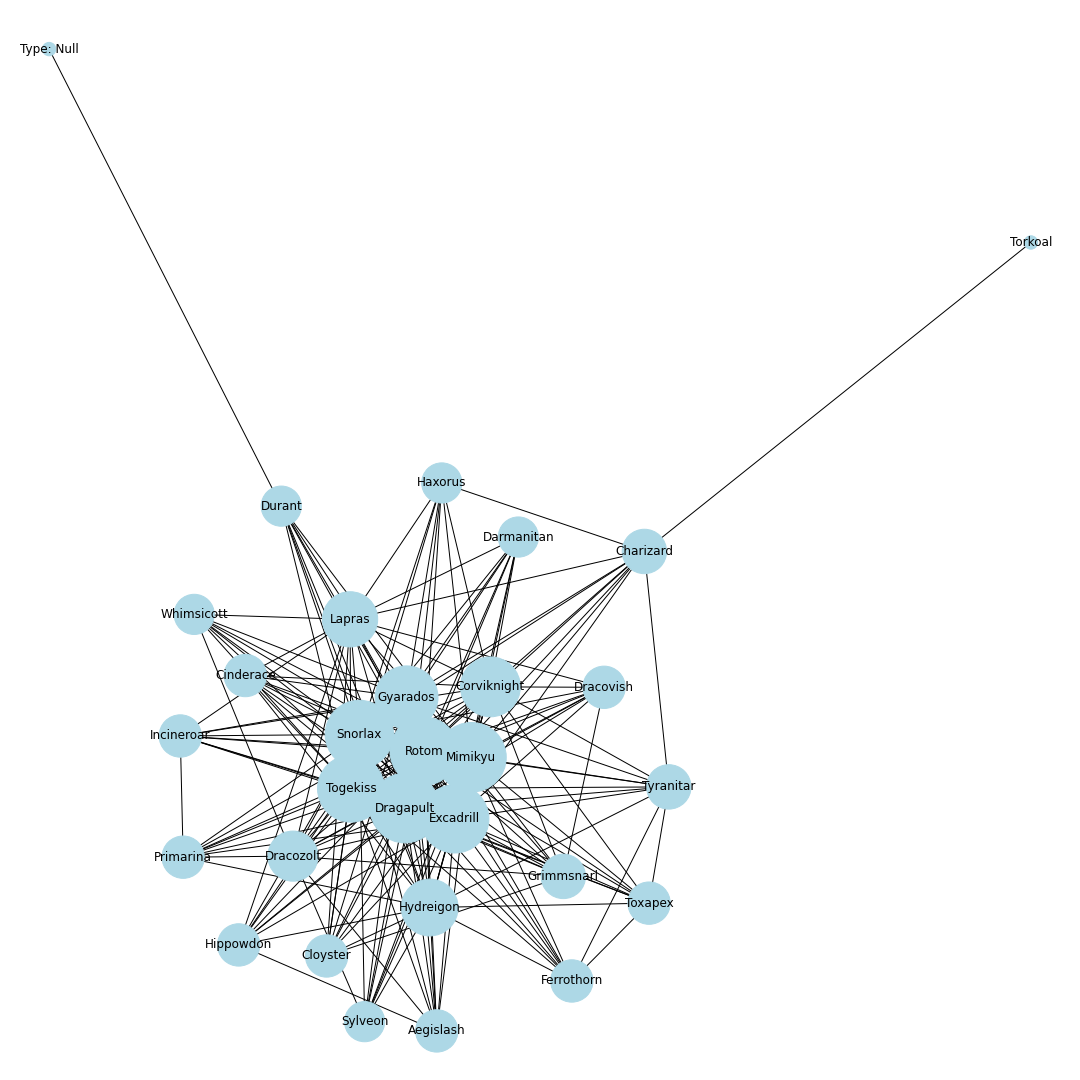
シーズン2
環境の変化が始まる.端から迫ってくる新しい並びが面白い.ヌルアント・ブリムオンドサイドン・ヌオードヒドなどが環境入りする.
シーズン3
キョダイマックスカビゴンとキョダイマックスラプラスが解禁.すぐさま環境に入ってくる.この段階だとラプラスは採用率は高いものの,環境上位のポケモンとの同時採用率はそこまで高くないため,実はこのグラフで考えるとSランクではなくAかBくらいの評価になりますね.
シーズン4
新たにキョダイマックスポケモンが何匹か解禁されるもさほど変化なし.環境にコータス・リザードンが入ってくる.
シーズン5
特に変化なし.
シーズン6
若干ナットレイが流行り始める.そのほかに変化特になし.
シーズン7
鎧の孤島解禁.エースバーン・ゴリランダーの夢特性の解禁とキョダイマックスの解禁により環境の中心に一気に食い込む.カビゴン・アーマーガアが環境の中心から外れ始める.ホルードが少し流行る.
シーズン8
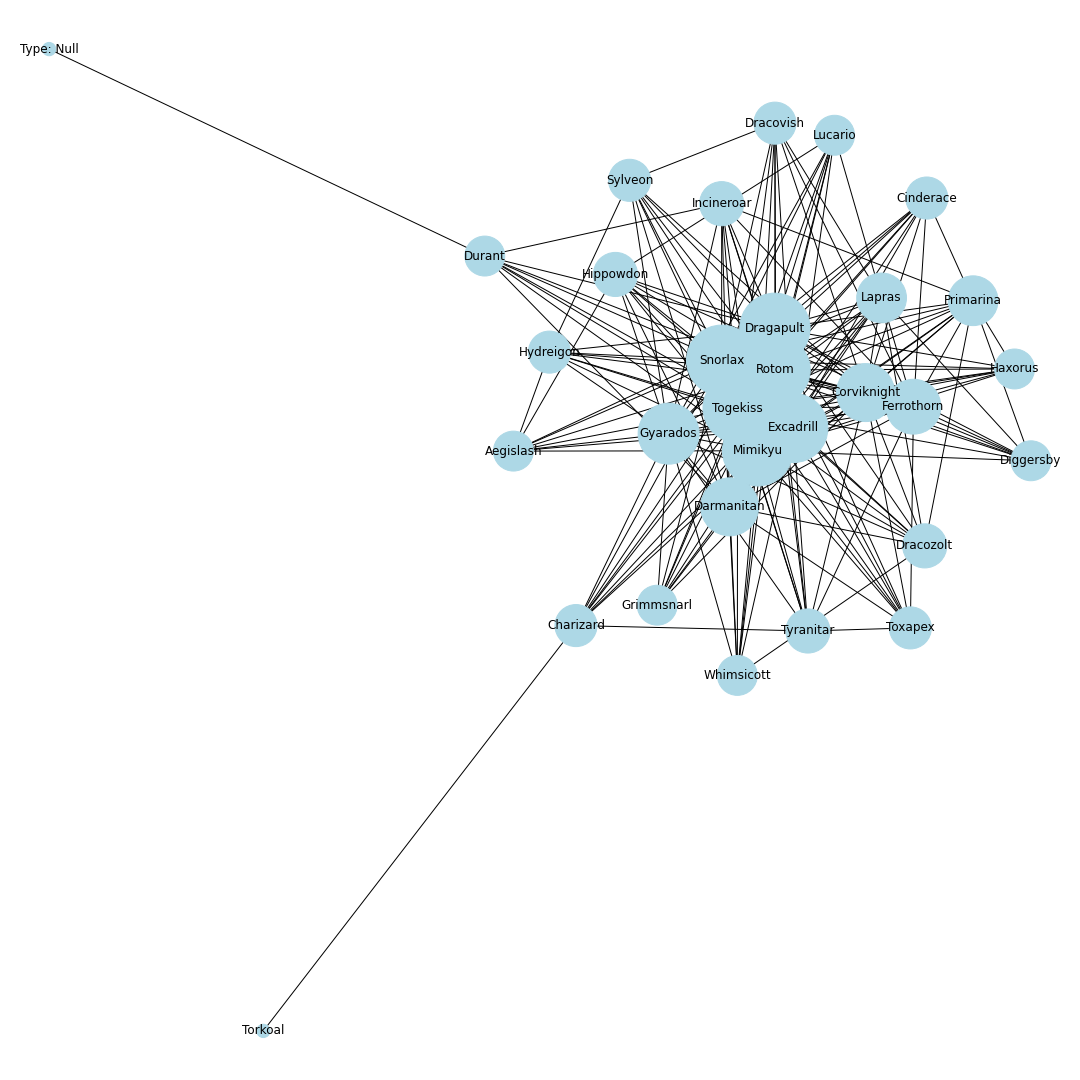
ポケモンホームが解禁.エアームド・ハピナス・ナットレイ・ドヒドイデなどの受けループ気質のポケモンが環境入り.カビゴンは受けきられてしまうため,環境から外れる.ポリゴン2が猛威を振るい環境の中心に.起点作成のためのカバルドンの価値が上がり,カバルドンが環境の中心に入る.ちなみにCharizard-1は図鑑の問題でばぐっているウーラオスです.
シーズン9
環境の中心の境界がくっきりとし始める.ギャラドスとドリュウズが少し下火になる.受けループも環境の外側に出始める.
シーズン10
上位ポケモン禁止ルールになり,環境が激変.ウーラオス・パッチラゴン・リザードン・ナットレイ・ロトム・アシレーヌが環境に中心に.カビゴンとアイアントが環境に戻ってくる.モロバレル・ピクシーが環境に顔を出すようになる.
シーズン11
ゲンガーが環境の中心入り.その他特に変化なし.
シーズン12
冠の雪原解禁.ランドロス・テッカグヤ・カプ・レヒレ・サンダー・ウツロイドなどの準伝説が環境のほとんどを占める.
シーズン12現在のデータからは,現在の環境の中心はミミッキュ・エースバーン・ポリゴン2・ゴリランダー・テッカグヤ・カプレヒレ・ランドロス・サンダー・ウツロイドあたりだといえそう.最後に
シーズン12現在のキャラランクを次数をもとに以下のように設定してみました.是非参考にしてみてください.理論に突っ込みどころ満載かもしれませんが,一つの指標として皆さんの役にたつとうれしいです.それではよきポケモンライフを!
追記: 似たようなことやってる方がいました
https://qiita.com/b_aka/items/9020e3237ff1a3e676e4
- 投稿日:2020-11-24T18:38:14+09:00
自動化プログラムを作りたい!
始め
彡(゚)(゚)「学外からアクセスする以外の方法で、Gaussianを利用したいなぁ」
彡(゚)(゚)「せや、計算機の所定のフォルダーにファイルを放り込んだら自動でかけさせたらええんや!」
彡(゚)(゚)「それならわいでもできるやろ()」
利用した物
CentOSにあるcron
python3およびそのライブラリー
実物
auto.pyimport os import subprocess path = '******' A=os.listdir(path) C=[i for i in A if '.com' in i] #計算したいファイルのリストを作成 L=[i.replace('.log', '.com') for i in A if '.log' in i] #計算の終了したファイルのリストの作成 G=[i for i in C if i not in L] #計算していないファイルを抽出 command1 = 'ps ax |grep g09 | grep -v grep | wc -l' command2 = 'cd ****** | g09 ' res = subprocess.check_output(command1, shell=True) try: if res==b'0\n': print("計算可能です") subprocess.Popen(command2 + G[0] + '&', shell=True) print("計算を実行しました") else: print("計算中です") except IndexError: print("計算するファイルがありません")*/30 * * * * /usr/bin/tcsh ./autopy.sh >>& /*******/analog-err.log色々とあって直でpythonのプログラムを動かせなかったので、迂遠な方法を利用しています
詰まった所
gaussian等々のPathがcronでは通ってなかった事に気づくのに結構な時間を使った件について
一個一個Pathを設定するのが面倒だったので、
cronに
printenvの結果を張り付けるとかいう力技を行っていますだから、実際のcronの中身はもっと長いです
あと、pythonの
ps ax |grep g09 | grep -v grep | wc -lは自力で思いついたわけじゃないですネットでジョブの実行状況を知るにはどうしたらいいか、
的なので検索して引っかかった物を自分なりに弄っただけです
このプログラムにおける一番の難題はこの部分でしたね…
終わりに
私の環境ではこれで元気に動いています
ここをこうすればもっとスマートにいくのでは?みたいなのがあったら教えてください(乞食)
ただし、python使う意味ある?全部コマンドで良くね?みたいなことに気付いた勘の良いガキは嫌いだよ
どうして私の研究室にはジョブマネージャーが無いんですか(血涙)次の目標として、ファイルの送信・転送について自動化できればいいなぁと思っています
転送は出来てるんですけど、送信がね……
最終的にはGUIを作成したいなぁと思っていたり…
それこそGo言語で何とかできないかなぁ
おしまい
- 投稿日:2020-11-24T18:35:04+09:00
異常検知入門
異常検知について勉強したのでまとめておきます。
参考文献
下記文献を大いに参考にさせていただきました:
[1] Lukas Ruff, et al.:A Unifying Review of Deep and Shallow Anomaly Detection
[2] 井手,杉山:入門 機械学習による異常検知―Rによる実践ガイド
[3] 井手,杉山:異常検知と変化検知 (機械学習プロフェッショナルシリーズ)§1 異常検知の概要
異常検知の適応例
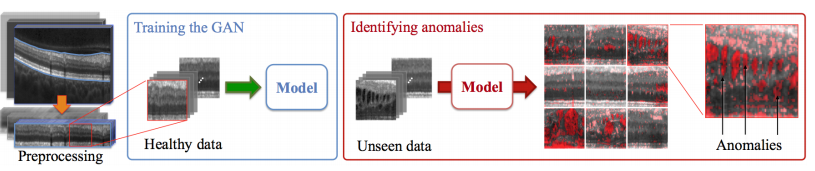
- 医療×異常検知
医療用画像からの疾患部位の特定
出典:Thomas, et al.Unsupervised Anomaly Detection with Generative Adversarial Networks to Guide Marker Discovery
- 金融×異常検知
マネーロンダリング検知
出典:Mark, et al.Anti-Money Laundering in Bitcoin: Experimenting with Graph Convolutional Networks for Financial Forensics異常は多種多様にわたる
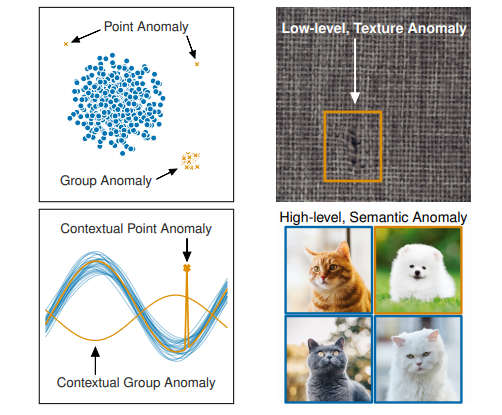
異常と一口に言っても多種多様な異常があり、大きく静的な異常と動的な異常に分けられます(私が勝手に作った切り口なのでおかしな点があればご指摘ください)。
静的な異常とは、各データ間に順序性等の関係がなく、データの値が異常の尺度になるものをいいます。
- 例えば,医療用画像において該当部位の濃淡が健康な場合のそれと大きく異なる場合に異常(疾患の存在)と判断されます。
- このような静的な異常は特に外れ値(Outlier)と呼んだりします。
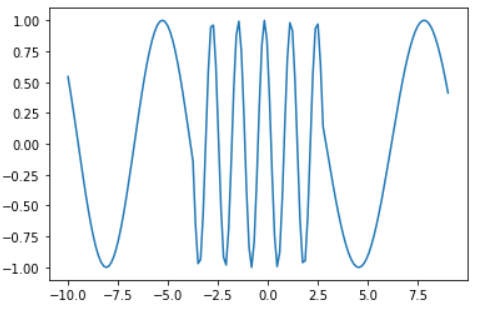
動的な異常とは、時系列データのようにデータ間に順序性等の関係があり、データの振る舞いが異常の尺度になるものをいいます。
- 振る舞いが異常であるとは,同一区間での観測値の増減が激しかったり逆に変化が著しく小さくなったりと個々の問題に依存します。
- 静的な異常との大きな違いは,動的な異常は値そのものが異常であるとは限らない点です。
- 例えば,下グラフの中央部は異常のように見えますが、値自体は取り得る値です。このケースでは、「周波数の変化」という振る舞いに異常性を見出すことになります。
import numpy as np import matplotlib.pyplot as plt x1 = np.concatenate([np.linspace(-10, -3, 50), np.zeros(50), np.linspace(3, 9, 50)]) x2 = np.concatenate([np.zeros(50), np.linspace(-3, 3, 50), np.zeros(50)]) y = np.sin(x1) + np.sin(5*x2) plt.plot(np.linspace(-10, 9, 150), y)数式で表現すると、系列データ$\{x_1, ..., x_t\}$において各点$x_j$が$p(x)=\int p(x,t){\rm d}t$の意味で正常であっても、時間の条件付き確率$p(x_j \mid t) = \int \cdots \int p(x_1,...x_t \mid t){\rm d}t_1 \cdots {\rm d}t_{j-1}{\rm d}t_{j+1}\cdots{\rm d}t_t$においては異常となり得えます。
ただしこのケースにおいても、中央部が異常ではない場合もあります。 例えば、上グラフが工場の機械動作シグナルであり中央部だけオペレーションモードが切り替わっていたため異常ではないということも考えられます。
以上のように異常には色々な側面がありますが、文献[1]ではこのように異常の種類をまとめています:
左上図のPoint Anomalyは観測エラー等による単発ノイズデータ(外れ値)に見えますが、Group Anomalyは外れ値というよりも別の分布から発生しているデータ群に見えます。両者では異なった異常検知アプローチが必要と想定されます。右下のSemantic Anomalyまで来ると古典的な異常検知手法では対応が難しく、ディープラーニングによるアプローチが必要になってきます。
このように「異常」に統一解はなく、ケースバイケースで「異常」を定義しなければいけません。
異常検知アルゴリズムは基本的に教師なし学習である
異常検知アルゴリズムは教師あり学習ではなく基本的に教師なし学習を用います。
理由は2つで:
- 大概、異常データは少ないから
- 異常データは多種多様であり網羅的にモデリングすることが難しいから
です。特に異常がクリティカルな現場(医療や工場)では、異常データを数百~数千も集められないはずです。また、理由2に関しては冒頭で説明したとおりです。「正常・異常」を当てるよりも、「正常・正常じゃない」を当てる方が簡単で筋が良い問題設定になります。後者は異常データに関して言及しておらず、正常データの構造理解のみで達成可能です。教師なし学習とは、サンプル$x_i(i=1,...,n)$から$p(x)$を推定することでデータの構造を理解する枠組みですから、異常検知は基本的に教師なし学習を用いることになります。ただし、教師なし学習を行う際のサンプルには異常データが含まれていない、もしくは圧倒的少数であると仮定できる必要があります。
余談ですが、文献[2]では以下のようなことを述べておりお気に入りです:
トルストイの『アンナ・カレーニナ』という小説に「幸せな家族は皆どこか似ているが,不幸せな家族はそれぞれ違う(Happy families are all alike; every unhappy family is unhappy in its own way)」という一節があり示唆的です.
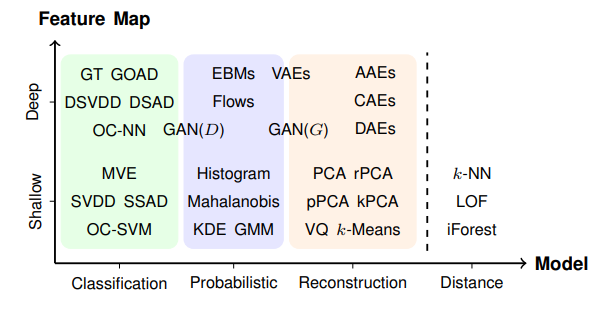
異常検知アルゴリズムは4種類に大別される
それでは具体的に異常検知アルゴリズムの紹介をしていきます。
アルゴリズムは大きく4つのアプローチにより分類できます。それぞれ、Classification Approach, Probabilistic Approach, Reconstruction Approach, Distance Approachです。この4つの分類に加えて、ディープラーニングを用いるか否かで分類をした表がこちらになります:
PCAやOC-SVM等のワードひとつひとつが過去提案されてきた異常検知アルゴリズムの名前です。各アルゴリズムの詳細は後続で紹介するので、ここでは4つのアプローチそれぞれの概要の紹介に留めます。(私自身、下記テーブルへの理解が十分ではないためご意見をいただけますと勉強になります。)
アプローチ 概要 Pros Cons Classification データの正常・異常を分ける境界を直接推定するアプローチ 分布の形状を仮定する必要がない。また、SVMを用いた手法においては比較的少数データで学習可能。 正常データが複雑な構造をしていると上手く動作しない場合がある Probabilistic 正常データの確率分布を推定し、生起確率が低い事象を異常とみなすアプローチ データが仮定したモデルに従っていれば非常に高精度を達成 分布モデル等多くの仮定が必要。また高次元への対応も困難。 Reconstruction 正常データを再構成するようwell-trainされたロジックが上手く再構成できないデータは異常である、というアプローチ 近年盛んに研究されているAEやGAN等の強力な手法が適応できる。 あくまでも次元圧縮や再構成精度を高めるために目的関数がデザインされているため、必ずしも異常検知に特化した手法ではないこと。 Distance データ間の距離に基づき異常検知を行うアプローチ。異常データは正常データから距離的に離れているという発想。 アイディアがシンプルで、出力への説明可能性も高い。 計算量が大きい($O(n^2)$など) 上記テーブルは以下資料を参考に作成しました。
1. 比戸:Jubatus Casual Talks #2 異常検知入門
2. PANG, et al. Deep Learning for Anomaly Detection: A Review§2 アルゴリズムの紹介と実験
紹介する手法まとめ
- Classification Approach
- One-Class SVM
- Probability Approach
- ホテリングのT2法
- GMM
- KDE
- Reconstruction Approach
- AE
- Distance Approach
- k-NN
- LOF
Classification Approach
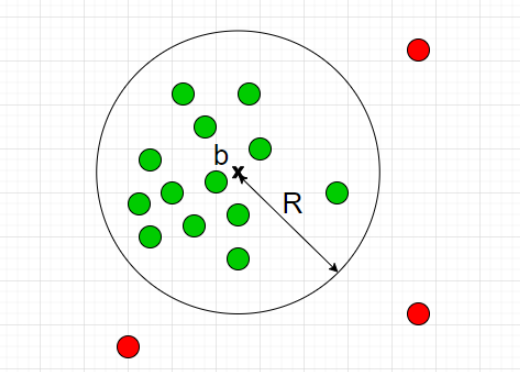
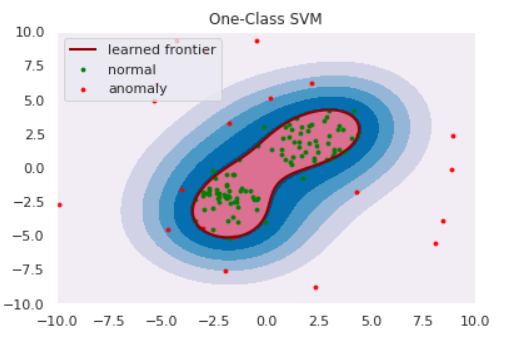
One-Class SVM
データ$D=\{x_1,...,x_N\}$が与えられているとします。ただし$D$には異常データが含まれていないか、含まれていたとしても圧倒的少数であるとします。
前章でClassification Approachは正常・異常を分ける境界を推定すると紹介しました。One-Class SVMは、正常データをすっぽり囲むような球を計算し境界とするアルゴリズムです。(球といっても三次元とは限りません。)
半径をとても大きくとれば$D$をすっぽり内包する球は作れますが、大きすぎる球ではFalse Negativeが1になってしまい嬉しくありません。そこで、
- 球の体積はできるだけ小さくする
- $D$のなかで、球からはみ出るデータがあることを許容する
の2点を許すことで汎用的な球を計算します。具体的には以下の最適化問題を解くことで最適な球の半径$R$と中心$b$が求まります:
\min_{R,b,u} \left\{R^2 + C\sum_{n=1}^{N}u_n \right\} \quad {\rm s.t.} \quad \|x_n - b\|^2 \le R^2 + u_nただし、$u$は球からはみ出した分のペナルティ項、$C$はそのペナルティをどれくらい重要視するかのパラメータです。SVMではカーネル関数によるデータの非線形変換が可能ですが、もちろんOne-Class SVMでも利用可能です。カーネル関数を適切に利用することで、適切な球が求まります。
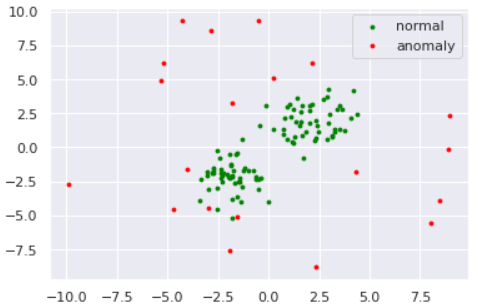
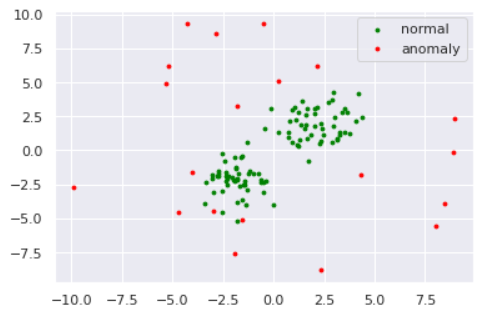
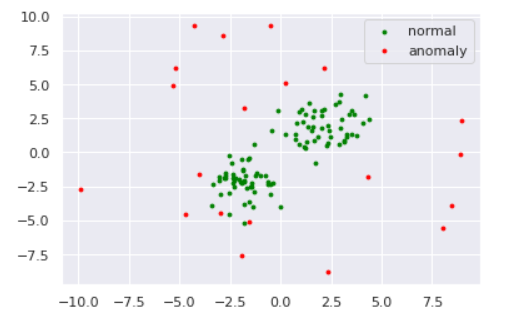
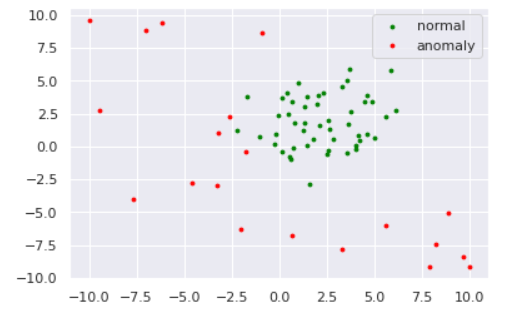
数値実験をしてみます。データは以下のように準備しました。
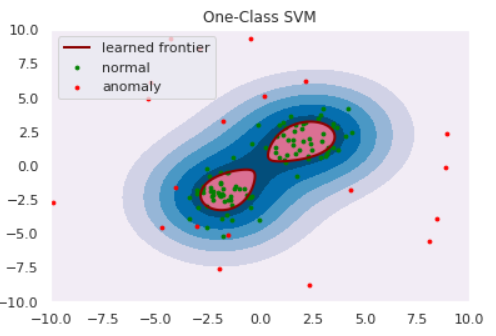
import numpy as np import matplotlib import matplotlib.pyplot as plt import seaborn as sns sns.set() np.random.seed(123) #平均と分散 mean1 = np.array([2, 2]) mean2 = np.array([-2,-2]) cov = np.array([[1, 0], [0, 1]]) # 正常データの生成(2つの正規分布から生成) norm1 = np.random.multivariate_normal(mean1, cov, size=50) norm2 = np.random.multivariate_normal(mean2, cov, size=50) norm = np.vstack([norm1, norm2]) # 異常データの生成(一様分布から生成) lower, upper = -10, 10 anom = (upper - lower)*np.random.rand(20,20) + lower s=8 plt.scatter(norm[:,0], norm[:,1], s=s, color='green', label='normal') plt.scatter(anom[:,0], anom[:,1], s=s, color='red', label='anomaly') plt.legend()from sklearn import svm xx, yy = np.meshgrid(np.linspace(-10, 10, 500), np.linspace(-10, 10, 500)) # fit the model clf = svm.OneClassSVM(nu=0.1, kernel="rbf", gamma=0.1) clf.fit(norm) def draw_boundary(norm, anom, clf): # plot the line, the points, and the nearest vectors to the plane Z = clf.decision_function(np.c_[xx.ravel(), yy.ravel()]) Z = Z.reshape(xx.shape) plt.title("One-Class SVM") plt.contourf(xx, yy, Z, levels=np.linspace(Z.min(), Z.max(), 7), cmap=plt.cm.PuBu) bd = plt.contour(xx, yy, Z, levels=[0], linewidths=2, colors='darkred') plt.contourf(xx, yy, Z, levels=[0, Z.max()], colors='palevioletred') nm = plt.scatter(norm[:, 0], norm[:, 1], c='green', s=s) am = plt.scatter(anom[:, 0], anom[:, 1], c='red', s=s) plt.axis('tight') plt.xlim((-10, 10)) plt.ylim((-10, 10)) plt.legend([bd.collections[0], nm, am], ["learned frontier", "normal", "anomaly"], loc="upper left", prop=matplotlib.font_manager.FontProperties(size=11)) plt.show() draw_boundary(norm, anom, clf)ちなみに、パラメータ$C$を小さくする、すなわち球からはみ出すペナルティを軽くみるようにするとこのように境界が変わります。(sklearnではパラメータ$\nu$で表していますが、どうやら分母にいるらしいので$\nu$を小さくする$=C$を大きくするのようです)
# fit the model clf = svm.OneClassSVM(nu=0.5, kernel="rbf", gamma=0.1) clf.fit(norm) draw_boundary(norm, anom, clf)他にもOne-Class NN等が提案されています。One-Class SVMではカーネル関数を調整して適切な非線形変換を探索していましたが、そこをNNで代用&自動化しようというアイディアです。実装は
めんどくさそうなのでやってません。Probability Approach
ホテリングのT2法
ホテリングのT2法は古典的な手法であり、理論的な解析がなされています。 結論、正規分布を仮定することで異常度(Anomaly Score)が$\chi^2$分布に従う性質を利用したアルゴリズムになります。
ここでは簡単のため1次元データで説明します。 例によってデータ$D$が与えられているとします。ここで各データ$x$が正規分布
p(x\mid \mu, \sigma) = \frac{1}{\sqrt{2\pi\sigma^2}}\exp\left\{-\frac{1}{2\sigma^2}(x-\mu)^2\right\}に従うものと仮定します。この正規分布を最尤推定法によりデータへフィッティングすることで
\begin{align*} \hat{\mu} &= \frac{1}{N}\sum_{n=1}^{N}x_n \\ \hat{\sigma}^2 &= \frac{1}{N}\sum_{n=1}^{N}(x_n - \hat{\mu})^2 \end{align*}と各パラメータが推定されます。つまり、データ$D$は正規分布$p(x\mid \hat{\mu}, \hat{\sigma}^2)$に従うと推定したわけです。
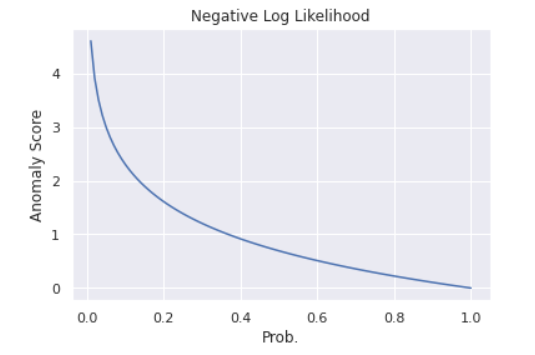
さて、ここで異常度(Anomaly Score)を導入します。異常度とは、文字通り異常の度合いを示す統計量のことで、よく負の対数尤度が用いられます。
prob = np.linspace(0.01, 1, 100) neg_log_likh = [-np.log(p) for p in prob] plt.plot(prob, neg_log_likh) plt.title('Negative Log Likelihood') plt.ylabel('Anomaly Score') plt.xlabel('Prob.')グラフのように、負の対数尤度は確率が小さいところで大きな値をとる形状をしており、異常度として機能しそうです。今推定した正規分布の異常度を計算すると
-\log p(x\mid \hat{\mu}, \hat{\sigma}^2) = \frac{1}{2\hat{\sigma}^2}(x-\hat{\mu})^2 + \frac{1}{2}\log(2\pi\hat{\sigma}^2)となります。異常度はデータ$x$に対して定義される量ですから、$x$に関係ない項は除いたうえで異常度$a(x)$は
a(x) = \left( \frac{x - \hat{\mu}}{\hat{\sigma}} \right)^2と計算されます。異常度の分子は、「データが平均から離れているほど異常度が高くなる」ということを表しています。分母は、「どれくらいデータが散らばっているか?」を表しており、散らばっているデータでは平均から遠く離れていようと大目に見る、逆に密なデータでは少しのズレも許さないというお気持ちがあります。
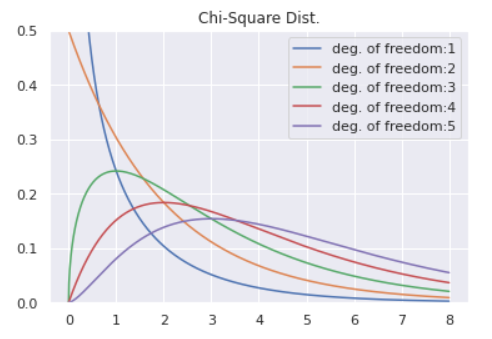
実はこの異常度$a(x)$はサンプルサイズ$N$が十分大きいときに自由度$1$のカイ二乗分布$\chi^2_1$に従うことがわかっています。
from scipy.stats import chi2 x = np.linspace(0, 8, 1000) for df in [1,2,3,4,5]: plt.plot(x, chi2.pdf(x ,df = df), label='deg. of freedom:'+str(df)) plt.legend() plt.ylim(0, 0.5) plt.title('Chi-Square Dist.')あとは有意水準$5$%で閾値$a_{th}$を決めるなどしてあげれば異常検知が可能となります。まとめると、
- データが正規分布に従うと仮定する。
- 正規分布のパラメータ$\mu$, $\sigma$を最尤推定法により推定する
- カイ二乗分布から、正常・異常を分ける点$a_{th}$を決める
- 新たなデータ$x_{new}$が、$a(x_{new})>a_{th}$だったら異常とみなす
がホテリングのT2法です。今回は1次元で説明しましたが、多次元でも同様に異常度がカイ二乗分布に従います。ただし、自由度=次元数です。
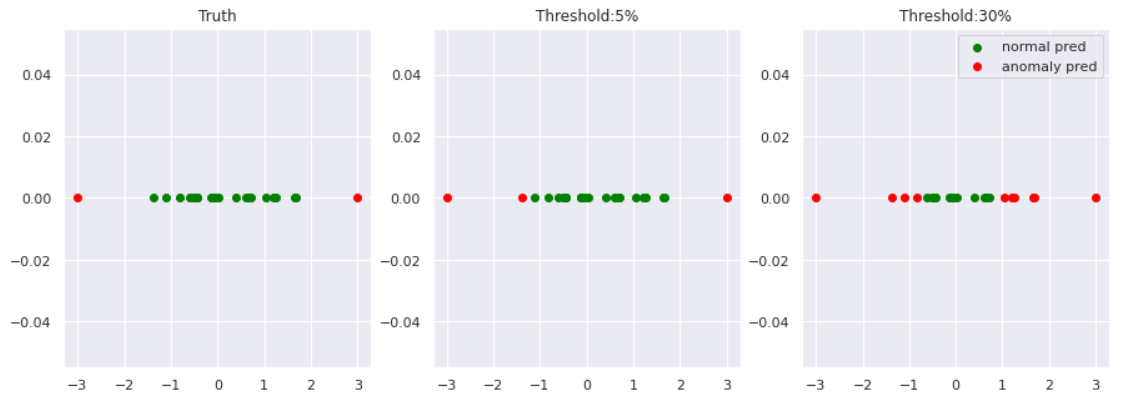
では実験してみましょう。データは以下のように作成しました。
norm = np.random.normal(0, 1, 20) # 正常データ anom = np.array([-3, 3]) # 異常データ plt.scatter(norm, [0]*len(norm), color='green', label='normal') plt.scatter(anom, [0]*len(anom), color='red', label='anomaly')# 最尤推定 N = len(norm) mu_hat = sum(norm)/N s_hat = sum([(x-mu_hat)**2 for x in norm])/N # カイ二乗分布の分位点を閾値とする a_th_5 = chi2.ppf(q=0.95, df=1) a_th_30 = chi2.ppf(q=0.7, df=1) # 各データの異常度を計算 data = np.hstack([norm, anom]) anom_score = [] for d in data: score = ((d - mu_hat)/s_hat)**2 anom_score.append(score) #描画 norm_pred_5 = [d for d,a in zip(data, anom_score) if a<=a_th_5] anom_pred_5 = [d for d,a in zip(data, anom_score) if a>a_th_5] norm_pred_30 = [d for d,a in zip(data, anom_score) if a<=a_th_30] anom_pred_30 = [d for d,a in zip(data, anom_score) if a>a_th_30] fig, axes = plt.subplots(1,3, figsize=(15,5)) axes[0].scatter(norm, [0]*len(norm), color='green', label='normal') axes[0].scatter(anom, [0]*len(anom), color='red', label='anomaly') axes[0].set_title('Truth') axes[1].scatter(norm_pred_5, [0]*len(norm_pred_5), color='green', label='normal pred') axes[1].scatter(anom_pred_5, [0]*len(anom_pred_5), color='red', label='anomaly pred') axes[1].set_title('Threshold:5%') axes[2].scatter(norm_pred_30, [0]*len(norm_pred_30), color='green', label='normal pred') axes[2].scatter(anom_pred_30, [0]*len(anom_pred_30), color='red', label='anomaly pred') axes[2].set_title('Threshold:30%') plt.legend()異常度の閾値を小さくすれば、異常と見なされる区域が広がっていくことが見て取れます。
閾値は問題に応じて決める必要があります。例えば、医療の現場など異常の見逃しがクリティカルな場合は閾値は小さく設定すべきですし、データクレンジング等の場合はもう少し大きい閾値にしてもいいかもしれません。GMM(Gaussian Mixture Model)
ホテリングのT2法ではデータに正規分布を仮定していましたが、これが不適切なケースもあるでしょう。例えば以下のようにデータが複数個所に集中している(単峰ではなく多峰)場合に正規分布でフィッティングすると
lower, upper = -3, -1 data1 = (upper - lower)*np.random.rand(20) + lower lower, upper = 1, 3 data2 = (upper - lower)*np.random.rand(30) + lower data = np.hstack([data1, data2]) # 最尤推定 N = len(data) mu_hat = sum(data)/N s_hat = sum([(x-mu_hat)**2 for x in data])/N # プロット from scipy.stats import norm X = np.linspace(-7, 7, 1000) Y = norm.pdf(X, mu_hat, s_hat) plt.scatter(data, [0]*len(data)) plt.plot(X, Y)のように、全くデータが観測されていない箇所に正規分布のピークが来るように推定されてしまいます。こういった場合は正規分布ではなく、もう少し複雑な分布を設定するのが良いでしょう。今回はその一例としてGMM(Gaussian Mixture Model, 混合正規分布)による異常検知を紹介します。
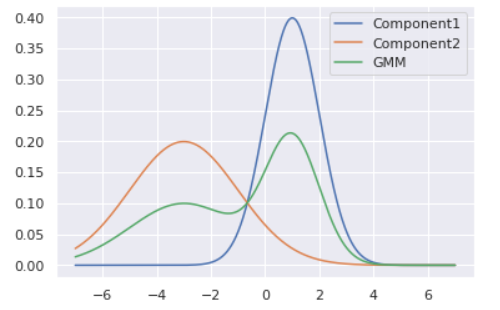
GMMとは正規分布をいくつか足し合わせることで多峰型を表現する分布です。足し合わせる個数はよく$K$で表現され、コンポーネント数と呼ばれます。GMMの数式とビジュアルは以下の通りです(1次元にしています)。
p(x\mid \pi, \mu, \sigma) = \sum_{k=1}^{K}\pi_k N(x\mid \mu_k, \sigma_k)ただし、
0 \le\pi_k\le 1,\quad \sum_{k=1}^{K}\pi_k=1,\quad N(x\mid\mu,\sigma)={\text 正規分布}です。
# プロット from scipy.stats import norm X = np.linspace(-7, 7, 1000) Y1 = norm.pdf(X, 1, 1) Y2 = norm.pdf(X, -3, 2) Y = 0.5*Y1 + 0.5*Y2 plt.plot(X, Y1, label='Component1') plt.plot(X, Y2, label='Component2') plt.plot(X, Y, label='GMM') plt.legend()各正規分布の平均($\mu$)・分散($\sigma$)と、混合率$\pi$はデータから学習されるパラメータです。しかしコンポーネント数$K$は決め打ちする必要があります。このように分析者側で決め打ちにするパラメータをハイパーパラメータと呼びます。
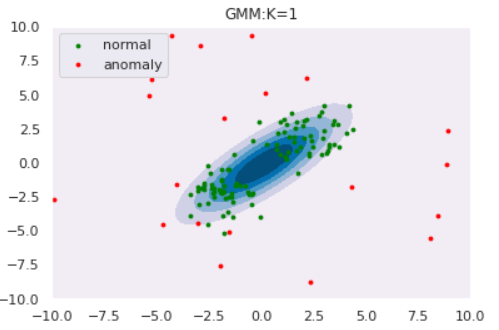
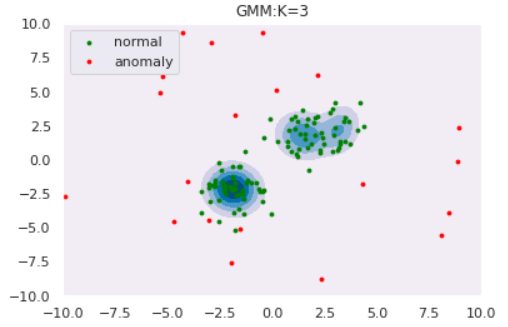
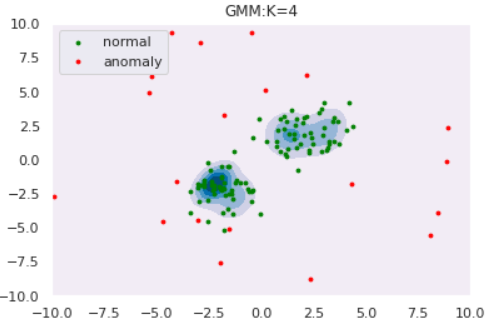
ではGMMによる異常検知を実験してみます。コンポーネント数$K$によってどのように推定分布の形状が変わるのかも観察しましょう。データはOne-Class SVMと同様以下を使用します。
def draw_GMM(norm, anom, clf, K): # plot the line, the points, and the nearest vectors to the plane xx, yy = np.meshgrid(np.linspace(-10, 10, 1000), np.linspace(-10, 10, 1000)) Z = np.exp(clf.score_samples(np.c_[xx.ravel(), yy.ravel()])) Z = Z.reshape(xx.shape) plt.title("GMM:K="+str(K)) plt.contourf(xx, yy, Z, levels=np.linspace(Z.min(), Z.max(), 7), cmap=plt.cm.PuBu) nm = plt.scatter(norm[:, 0], norm[:, 1], c='green', s=s) am = plt.scatter(anom[:, 0], anom[:, 1], c='red', s=s) plt.axis('tight') plt.xlim((-10, 10)) plt.ylim((-10, 10)) plt.legend([nm, am], ["normal", "anomaly"], loc="upper left", prop=matplotlib.font_manager.FontProperties(size=11)) plt.show()# GMMによるフィッティング from sklearn.mixture import GaussianMixture for K in [1,2,3,4]: clf = GaussianMixture(n_components=K, covariance_type='full') clf.fit(norm) draw_GMM(norm, anom, clf, K)
このようにコンポーネント数を多くすると、データの細部の傾向まで捉えていることがわかります。しかし、細部まで捉えている=汎化性能が高いわけではないので注意が必要です。コンポーネント数はWAIC等の情報量規準を用いて決定することができます(とりあえずWAICを使ってみました:GitHub)KDE(Kernel Density Estimation)
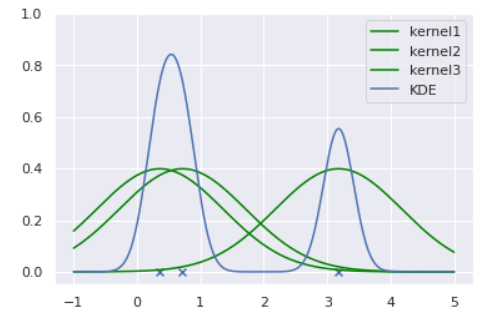
今までは正規分布や混合正規分布を仮定し、データへのフィッティングを通じて内部パラメータの推定を行ってきました。このようなアプローチをパラメトリックなアプローチといいます。しかし、パラメトリックな分布では上手く表現できないような場合にはどうしたら良いでしょうか?
ノンパラメトリックなアプローチはそのような場合に有効であり、KDEは代表的なノンパラメトリック推定手法です。KDEは、各データ点にカーネル(通常はGaussian)を重ねることで確率分布を推定する手法です。可視化すると以下のようになります。
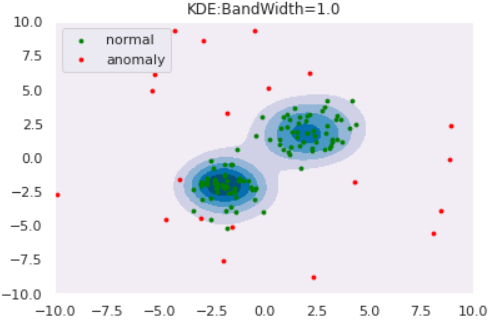
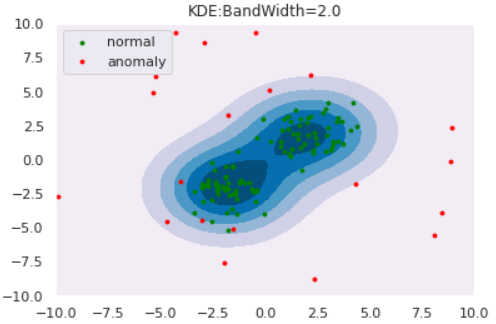
from scipy.stats import norm np.random.seed(123) lower, upper = -1, 5 data = (upper - lower)*np.random.rand(3) + lower for i, d in enumerate(data): X = np.linspace(lower,upper,500) Y = norm.pdf(X, loc=d, scale=1) knl = plt.plot(X,Y,color='green', label='kernel'+str(i+1)) # KDE n = 3 h = 0.24 Y_pdf = [] for x in X: Y = 0 for d in data: Y += norm.pdf((x-d)/h, loc=0, scale=1) Y /= n*h Y_pdf.append(Y) kde = plt.plot(X,Y_pdf, label='KDE') plt.scatter(data, [0]*len(data), marker='x') plt.ylim([-0.05, 1]) plt.legend()このようにカーネルの重ね合わせで確率分布を推定しています。カーネルに正規分布を用いる場合は分散(バンド幅と呼びます)を決め打ちする必要があります。後続の実験では、このバンド幅を変えた時の推定分布の変化も観察しましょう。
KDEの理論詳細および長所・短所は鈴木.データ解析第十回「ノンパラメトリック密度推定法」がわかりやすかったです:
推定分布
p(x) = \frac{1}{Nh}\sum_{n=1}^{N}K\left(\frac{x-X_n}{h}\right)ただし、$h>0$をバンド幅、$K()$をカーネル関数、$X_n$は各データ点とする。
長所・短所
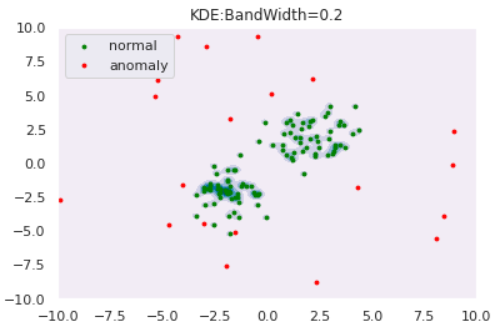
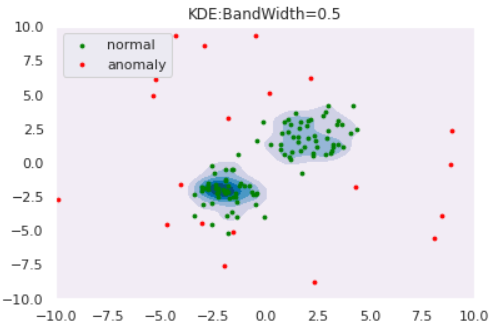
出典:鈴木.データ解析第十回「ノンパラメトリック密度推定法」ではKDEによる異常検知を実験してみましょう。データは例によって
を使用します。バンド幅を変えた時の推定分布は以下のようになりました。def draw_KDE(norm, anom, clf, K): # plot the line, the points, and the nearest vectors to the plane xx, yy = np.meshgrid(np.linspace(-10, 10, 1000), np.linspace(-10, 10, 1000)) Z = np.exp(clf.score_samples(np.c_[xx.ravel(), yy.ravel()])) Z = Z.reshape(xx.shape) plt.title("KDE:BandWidth="+str(bw)) plt.contourf(xx, yy, Z, levels=np.linspace(Z.min(), Z.max(), 7), cmap=plt.cm.PuBu) nm = plt.scatter(norm[:, 0], norm[:, 1], c='green', s=s) am = plt.scatter(anom[:, 0], anom[:, 1], c='red', s=s) plt.axis('tight') plt.xlim((-10, 10)) plt.ylim((-10, 10)) plt.legend([nm, am], ["normal", "anomaly"], loc="upper left", prop=matplotlib.font_manager.FontProperties(size=11)) plt.show()from sklearn.neighbors import KernelDensity for bw in [0.2,0.5,1.0,2.0]: clf = KernelDensity(bandwidth=bw, kernel='gaussian') clf.fit(norm) draw_KDE(norm, anom, clf, bw)
このように、バンド幅が大きくなると推定分布が滑らかになることがわかります。また、カーネル関数は正規分布に限らず他にもあるので、「バンド幅・カーネル関数」の両者を適切に設定してあげる必要があります。Reconstruction Approach
Reconstruction Approachは画像データに用いられることが多いので、ここでも画像データを使用します。正常データをMNIST、異常データをFashion-MNISTとします。
# MNISTのロード import torch from torch.utils.data import DataLoader from torchvision import datasets, transforms # 学習データの読み込み train_mnist = datasets.MNIST( root="./data", train=True, transform=transforms.Compose([ transforms.ToTensor(), transforms.Normalize((0.5,), (0.5,)) # 区間[-1,1]内で平均、標準偏差 = 0.5, 0.5に正規化 ]), download=True ) train_mnist_loader = torch.utils.data.DataLoader( train_mnist, batch_size=64, shuffle=True ) # テストデータの読み込み test_mnist = datasets.MNIST( root="./data", train=False, transform=transforms.Compose([ transforms.ToTensor(), transforms.Normalize((0.5,), (0.5,)) # 区間[-1,1]内で平均、標準偏差 = 0.5, 0.5に正規化 ]), download=True ) test_mnist_loader = torch.utils.data.DataLoader( test_mnist, batch_size=64, shuffle=True )# Fashion-MNISTのロード # 学習データの読み込み train_fashion = datasets.FashionMNIST( root="./data", train=True, transform=transforms.Compose([ transforms.ToTensor(), transforms.Normalize((0.5,), (0.5,)) # 区間[-1,1]内で平均、標準偏差 = 0.5, 0.5に正規化 ]), download=True ) train_fashion_loader = torch.utils.data.DataLoader( train_fashion, batch_size=64, shuffle=True ) # テストデータの読み込み test_fashion = datasets.FashionMNIST( root="./data", train=False, transform=transforms.Compose([ transforms.ToTensor(), transforms.Normalize((0.5,), (0.5,)) # 区間[-1,1]内で平均、標準偏差 = 0.5, 0.5に正規化 ]), download=True ) test_fashion_loader = torch.utils.data.DataLoader( test_fashion, batch_size=64, shuffle=True )import matplotlib.pyplot as plt # 画像を一気に並べて可視化する関数 def imshow(image_set, nrows=2, ncols=10): plot_num = nrows * ncols fig, ax = plt.subplots(ncols=ncols, nrows=nrows, figsize=(15, 15*nrows/ncols)) ax = ax.ravel() for i in range(plot_num): ax[i].imshow(image_set[i].reshape(28, 28), cmap='gray') ax[i].set_xticks([]) ax[i].set_yticks([])# MNISTのサンプル可視化 imshow(train_mnist.data)# Fashion-MNISTのサンプル可視化 imshow(train_fashion.data)AE(Auto Encoder)
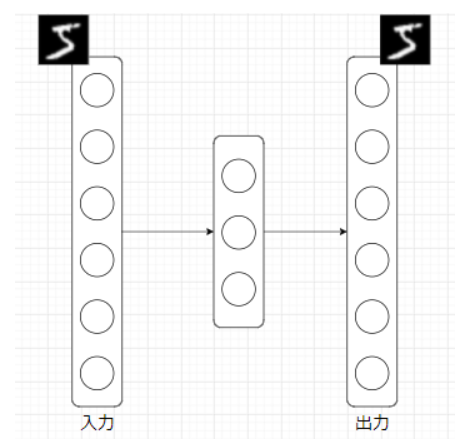
AE(Auto Encoder, オートエンコーダ)とは、NN(Neural Network, ニューラルネットワーク)の一種であり、入力と出力が等しくなるように学習するNNです。ただの恒等関数ではつまらないので、通常は入力次元よりも小さい次元の中間層を挟みます。
これは何をしているのかというと、中間層で入力データを「要約」しています。数理的には、入力データを再構成できるだけの情報を保持しつつ低次元空間へマッピングしている、ということになります。
AEを用いた異常検知は、「正常データを再構成するよう訓練されたAEは、異常データを上手く再構成できないのではないか?」という思想に基づきます。
では、AEによる異常検知を実験しましょう。AEをMNISTで訓練した後に、MNISTおよびFashion-MNISTの再構成精度を異常度として検知を試みます。
import torch.nn as nn import torch.nn.functional as F # AEの定義 class AE(nn.Module): def __init__(self): super(AE, self).__init__() self.fc1 = nn.Linear(28*28, 256) self.fc2 = nn.Linear(256, 28*28) def forward(self, x): x = torch.tanh(self.fc1(x)) x = torch.tanh(self.fc2(x)) return x ae = AE() # 損失関数と最適化手法の定義 import torch.optim as optim criterion = nn.MSELoss() optimizer = optim.Adam(ae.parameters(), lr=0.0001)# 学習ループ N_train = train_mnist.data.shape[0] N_test = test_mnist.data.shape[0] train_loss = [] test_loss = [] for epoch in range(10): # Train step running_train_loss = 0.0 for data in train_mnist_loader: inputs, _ = data inputs = inputs.view(-1, 28*28) # zero the parameter gradients optimizer.zero_grad() # forward + backward + optimize outputs = ae(inputs) loss = criterion(outputs, inputs) loss.backward() optimizer.step() running_train_loss += loss.item() running_train_loss /= N_train train_loss.append(running_train_loss) # Test step with torch.no_grad(): running_test_loss = 0 for data in test_mnist_loader: inputs, _ = data inputs = inputs.view(-1, 28*28) outputs = ae(inputs) running_test_loss += criterion(outputs, inputs).item() running_test_loss /= N_test test_loss.append(running_test_loss) if (epoch+1)%1==0: # print every 1 epoch print('epoch:{}, train_loss:{}, test_loss:{}'.format(epoch + 1, running_train_loss, running_test_loss)) plt.plot(train_loss, label='Train loss') plt.plot(test_loss, label='Test loss') plt.ylabel('Loss') plt.xlabel('epoch') plt.legend()# 再構成の様子を可視化してみる with torch.no_grad(): for data in test_mnist_loader: inputs, _ = data inputs = inputs.view(-1, 28*28) outputs = ae(inputs) n = 5 # num to viz fig, axes = plt.subplots(ncols=n, nrows=2, figsize=(15, 15*2/n)) for i in range(n): axes[0, i].imshow(inputs[i].reshape(28, 28), cmap='gray') axes[1, i].imshow(outputs[i].reshape(28, 28), cmap='gray') axes[0, i].set_title('Input') axes[1, i].set_title('Reconstruct') axes[0, i].set_xticks([]) axes[0, i].set_yticks([]) axes[1, i].set_xticks([]) axes[1, i].set_yticks([])きれいに再構成できていそうです。
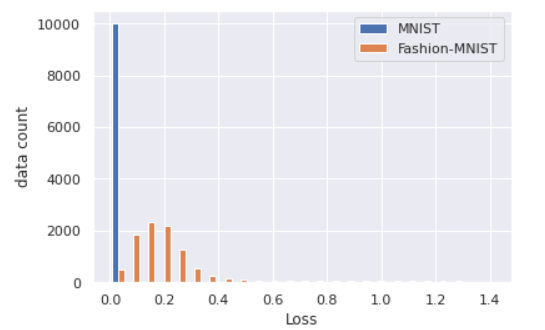
それでは、MNISTとFashion-MNISTの再構成精度(今回は二乗誤差)の分布を描いてみます。AEによる異常検知が成功していれば、Fashion-MNISTの精度は低くなるはずです。
# 再構成精度の算出 mnist_loss = [] with torch.no_grad(): for data in test_mnist_loader: inputs, _ = data for data_i in inputs: data_i = data_i.view(-1, 28*28) outputs = ae(data_i) loss = criterion(outputs, data_i) mnist_loss.append(loss.item()) fashion_loss = [] with torch.no_grad(): for data in test_fashion_loader: inputs, _ = data for data_i in inputs: data_i = data_i.view(-1, 28*28) outputs = ae(data_i) loss = criterion(outputs, data_i) fashion_loss.append(loss.item())plt.hist([mnist_loss, fashion_loss], bins=25, label=['MNIST', 'Fashion-MNIST']) plt.xlabel('Loss') plt.ylabel('data count') plt.legend() plt.show()確かにFashion-MNISTの再構成は上手くいってなさそうなので、再構成精度による異常検知は成立しそうです。
最後に、Fashion-MNISTの中でもLossが低いもの(=異常と判定し辛いもの)と高いもの(=簡単に異常と判定できるもの)をピックアップして描画してみます。
lower_loss, upper_loss = 0.1, 0.8 data_low = [] # Lossが低いデータを格納 data_up = [] # Lossが高いデータを格納 with torch.no_grad(): for data in test_fashion_loader: inputs, _ = data for data_i in inputs: data_i = data_i.view(-1, 28*28) outputs = ae(data_i) loss = criterion(outputs, data_i).item() if loss<lower_loss: data_low.append([data_i, outputs, loss]) if loss>upper_loss: data_up.append([data_i, outputs, loss])# ランダムに描画する用のインデックス np.random.seed(0) n = 3 #描画データ数 lower_indices = np.random.choice(np.arange(len(data_low)), n) upper_indices = np.random.choice(np.arange(len(data_up)), n) indices = np.hstack([lower_indices, upper_indices]) fig, axes = plt.subplots(ncols=n*2, nrows=2, figsize=(15, 15*2/(n*2))) # Lossが小さいデータの描画 for i in range(n): inputs = data_low[indices[i]][0] outputs = data_low[indices[i]][1] loss = data_low[indices[i]][2] axes[0, i].imshow(inputs.reshape(28, 28), cmap='gray') axes[1, i].imshow(outputs.reshape(28, 28), cmap='gray') axes[0, i].set_title('Input') axes[1, i].set_title('Small Loss={:.2f}'.format(loss)) axes[0, i].set_xticks([]) axes[0, i].set_yticks([]) axes[1, i].set_xticks([]) axes[1, i].set_yticks([]) # Lossが大きいデータの描画 for i in range(n, 2*n): inputs = data_up[indices[i]][0] outputs = data_up[indices[i]][1] loss = data_up[indices[i]][2] axes[0, i].imshow(inputs.reshape(28, 28), cmap='gray') axes[1, i].imshow(outputs.reshape(28, 28), cmap='gray') axes[0, i].set_title('Input') axes[1, i].set_title('Large Loss={:.2f}'.format(loss)) axes[0, i].set_xticks([]) axes[0, i].set_yticks([]) axes[1, i].set_xticks([]) axes[1, i].set_yticks([]) plt.tight_layout()Lossが低い画像は黒い面積が大きい傾向が見られる一方で、Lossが高い画像は白い面積が大きい傾向が見られます。MNISTは基本的に黒の面積が大きい画像ですので、白の面積が大きいとAEにとっては「異質」になり再構成が上手くいっていないと思われます。
Distance Approach
k-NN(k-Nearest Neighbor)
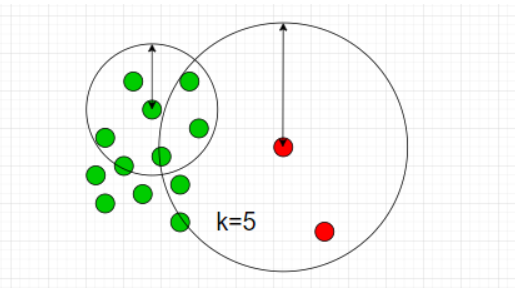
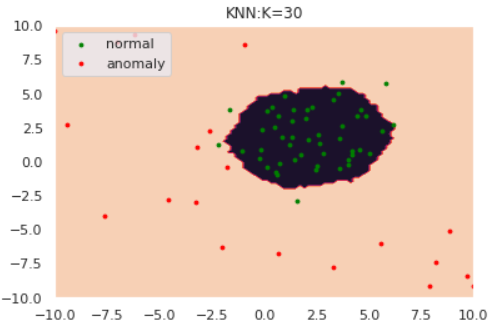
k-NNはデータ間の距離に基づき異常度を測る手法です。k-NNでは近傍のk個のデータ点を含むような円を考えます。下図は$k=5$の場合です。
図のように、「異常データが描く円は正常データよりも大きくなるはず」とう思想に基づきk-NNでは円の半径を異常度として定義します。ただし、近傍点の個数$k$と正常/異常を分ける半径の閾値は適切にチューニングする必要があります。
それでは数値実験を行います。閾値は、正常データの半径値分布における90%分位点とします。
# データの作成 np.random.seed(123) #平均と分散 mean = np.array([2, 2]) cov = np.array([[3, 0], [0, 3]]) # 正常データの生成 norm = np.random.multivariate_normal(mean1, cov, size=50) # 異常データの生成(一様分布から生成) lower, upper = -10, 10 anom = (upper - lower)*np.random.rand(20,20) + lower s=8 plt.scatter(norm[:,0], norm[:,1], s=s, color='green', label='normal') plt.scatter(anom[:,0], anom[:,1], s=s, color='red', label='anomaly') plt.legend()k-NNによる異常検知を、どのようにライブラリで完結できるか分からなかったので自作します。。。
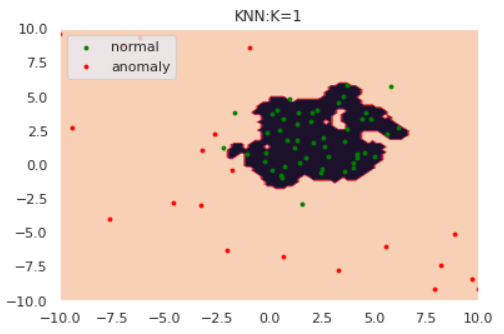
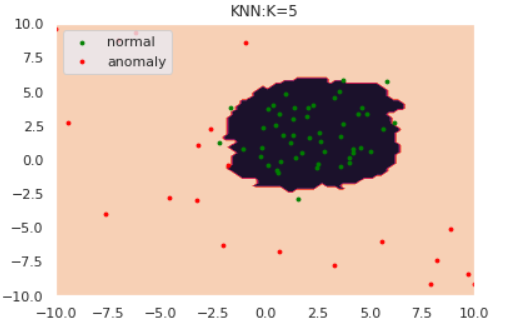
# 全データのk近傍円の半径を計算する関数 def kNN(data, k=5): """ input: data:list, k:int output: 全データのk近傍円の半径:list 距離はユークリッド距離を採用します。 """ output = [] for d in data: distances = [] for p in data: distances.append(np.linalg.norm(d - p)) output.append(sorted(distances)[k]) return output # 閾値より大きければ1, 小さければ0を返す関数 def kNN_scores(point, data, thres, K): distances = [] for p in data: distances.append(np.linalg.norm(point - p)) dist = sorted(distances)[K] if dist > thres: return 1 else: return 0def draw_KNN(norm, anom, thres, K, only_norm=False): # plot the line, the points, and the nearest vectors to the plane xx, yy = np.meshgrid(np.linspace(-10, 10, 100), np.linspace(-10, 10, 100)) Z = [kNN_scores(point, norm, thres, K) for point in np.c_[xx.ravel(), yy.ravel()]] Z = np.array(Z).reshape(xx.shape) plt.title("KNN:K="+str(K)) plt.contourf(xx, yy, Z) if not only_norm: nm = plt.scatter(norm[:, 0], norm[:, 1], c='green', s=s) am = plt.scatter(anom[:, 0], anom[:, 1], c='red', s=s) plt.axis('tight') plt.xlim((-10, 10)) plt.ylim((-10, 10)) plt.legend([nm, am], ["normal", "anomaly"], loc="upper left", prop=matplotlib.font_manager.FontProperties(size=11)) if only_norm: nm = plt.scatter(norm[:, 0], norm[:, 1], c='green', s=s) plt.axis('tight') plt.xlim((-10, 10)) plt.ylim((-10, 10)) plt.legend([nm], ["normal"], loc="upper left", prop=matplotlib.font_manager.FontProperties(size=11)) plt.show()for k in [1,5,30]: anomaly_scores = kNN(norm, k=k) thres = pd.Series(anomaly_scores).quantile(0.90) draw_KNN(norm, anom, thres, k)kが大きくなるにつれて、正常/異常の境界が滑らかになっていることがわかります。
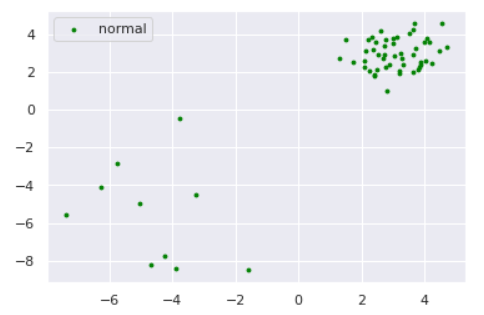
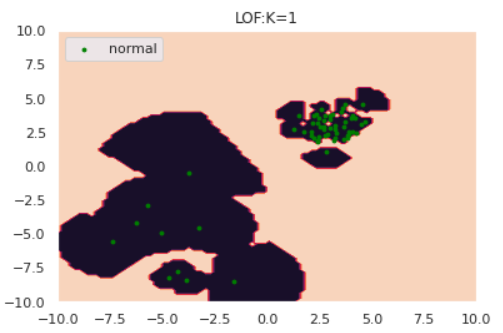
さて、ここで少しいじわるな例で実験してみましょう。以下のように、正常データが密度の違う2つの正規分布から生成されているとします。
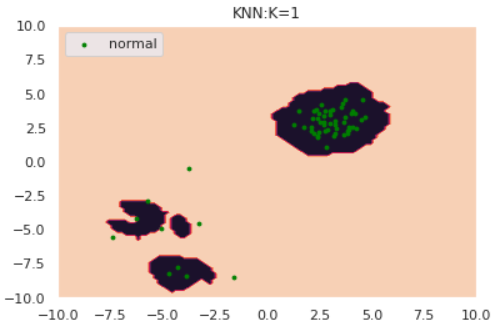
np.random.seed(123) #平均と分散 mean1 = np.array([3, 3]) mean2 = np.array([-5,-5]) cov1 = np.array([[0.5, 0], [0, 0.5]]) cov2 = np.array([[3, 0], [0, 3]]) # 正常データの生成(2つの正規分布から生成) norm1 = np.random.multivariate_normal(mean1, cov1, size=50) norm2 = np.random.multivariate_normal(mean2, cov2, size=10) norm = np.vstack([norm1, norm2]) s=8 plt.scatter(norm[:,0], norm[:,1], s=s, color='green', label='normal') plt.legend()for k in [1,5,30]: anomaly_scores = kNN(norm, k=k) thres = pd.Series(anomaly_scores).quantile(0.90) draw_KNN(norm, [], thres, k, only_norm=True)特にk=1のケースに注目すると、正常データが密度の異なる別クラスターから発生している場合kNNが上手く動作していないことがわかります。「密なクラスターからのズレは重要視して、疎なクラスターからのズレは大目に見る」という処理を加えれば対処できそうだと想像できます。
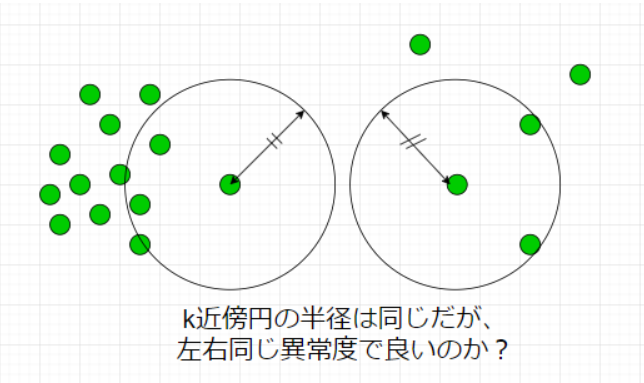
実はそれが次節で紹介するLOFという手法になります。
LOF(Local Outlier Factor, 局所外れ値因子法)
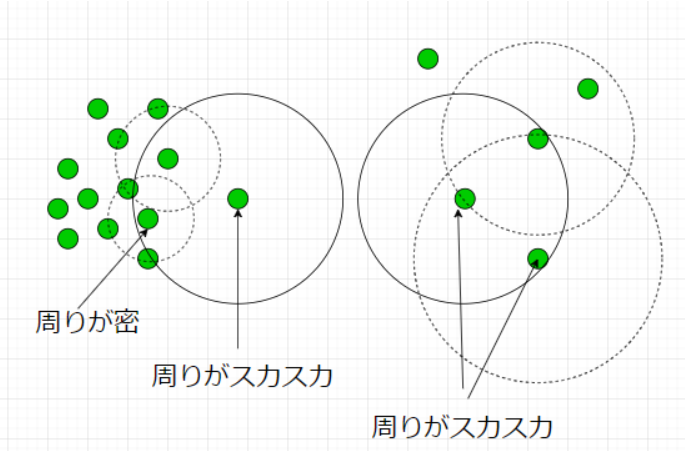
前節で紹介したk-NNが上手く動作しないケースを図で表すと
となります。もちろん、左側の異常度を高くしたいのですが、LOFではデータの密度という概念を導入しそれを実現します。数理的詳細は割愛しますが、以下のようなことをしています。
自身のスカスカ度合いと近傍点のスカスカ度合いの比で異常度を定義しています。
それではLOFの実験を行いましょう。
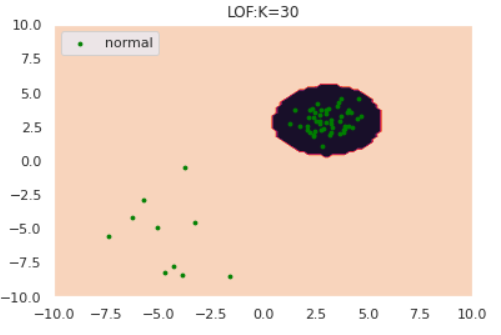
def draw_LOF(norm, anom, K, only_norm=False): # plot the line, the points, and the nearest vectors to the plane xx, yy = np.meshgrid(np.linspace(-10, 10, 100), np.linspace(-10, 10, 100)) Z = - model.predict(np.c_[xx.ravel(), yy.ravel()]) Z = np.array(Z).reshape(xx.shape) plt.title("LOF:K="+str(K)) plt.contourf(xx, yy, Z) if not only_norm: nm = plt.scatter(norm[:, 0], norm[:, 1], c='green', s=s) am = plt.scatter(anom[:, 0], anom[:, 1], c='red', s=s) plt.axis('tight') plt.xlim((-10, 10)) plt.ylim((-10, 10)) plt.legend([nm, am], ["normal", "anomaly"], loc="upper left", prop=matplotlib.font_manager.FontProperties(size=11)) if only_norm: nm = plt.scatter(norm[:, 0], norm[:, 1], c='green', s=s) plt.axis('tight') plt.xlim((-10, 10)) plt.ylim((-10, 10)) plt.legend([nm], ["normal"], loc="upper left", prop=matplotlib.font_manager.FontProperties(size=11)) plt.show()from sklearn.neighbors import LocalOutlierFactor for k in [1, 5, 30]: model = LocalOutlierFactor(n_neighbors=k, novelty=True) model.fit(norm) draw_LOF(norm, [], k, only_norm=True)このように、クラスターの密度を考慮したうえで異常検知ができていそうです。
発展的な異常検知問題
今後異常検知の勉強をしていくにあたって、以下のようなトピックを勉強してきたいと思っています:
- 時系列データの異常検知
- グラフデータの異常検知
- マネーロンダリング検知に使えそう
- 投稿日:2020-11-24T18:35:04+09:00
異常検知入門と手法まとめ
異常検知について勉強したのでまとめておきます。
参考文献
下記文献を大いに参考にさせていただきました:
[1] Lukas Ruff, et al.:A Unifying Review of Deep and Shallow Anomaly Detection
[2] 井手,杉山:入門 機械学習による異常検知―Rによる実践ガイド
[3] 井手,杉山:異常検知と変化検知 (機械学習プロフェッショナルシリーズ)§1 異常検知の概要
異常検知の適応例
- 医療×異常検知
医療用画像からの疾患部位の特定
出典:Thomas, et al.Unsupervised Anomaly Detection with Generative Adversarial Networks to Guide Marker Discovery
- 金融×異常検知
マネーロンダリング検知
出典:Mark, et al.Anti-Money Laundering in Bitcoin: Experimenting with Graph Convolutional Networks for Financial Forensics異常は多種多様にわたる
異常と一口に言っても多種多様な異常があり、大きく静的な異常と動的な異常に分けられます(私が勝手に作った切り口なのでおかしな点があればご指摘ください)。
静的な異常とは、各データ間に順序性等の関係がなく、データの値が異常の尺度になるものをいいます。
- 例えば,医療用画像において該当部位の濃淡が健康な場合のそれと大きく異なる場合に異常(疾患の存在)と判断されます。
- このような静的な異常は特に外れ値(Outlier)と呼んだりします。
動的な異常とは、時系列データのようにデータ間に順序性等の関係があり、データの振る舞いが異常の尺度になるものをいいます。
- 振る舞いが異常であるとは,同一区間での観測値の増減が激しかったり逆に変化が著しく小さくなったりと個々の問題に依存します。
- 静的な異常との大きな違いは,動的な異常は値そのものが異常であるとは限らない点です。
- 例えば,下グラフの中央部は異常のように見えますが、値自体は取り得る値です。このケースでは、「周波数の変化」という振る舞いに異常性を見出すことになります。
import numpy as np import matplotlib.pyplot as plt x1 = np.concatenate([np.linspace(-10, -3, 50), np.zeros(50), np.linspace(3, 9, 50)]) x2 = np.concatenate([np.zeros(50), np.linspace(-3, 3, 50), np.zeros(50)]) y = np.sin(x1) + np.sin(5*x2) plt.plot(np.linspace(-10, 9, 150), y)数式で表現すると、系列データ$\{x_1, ..., x_t\}$において各点$x_j$が$p(x)=\int p(x,t){\rm d}t$の意味で正常であっても、時間の条件付き確率$p(x_j \mid t) = \int \cdots \int p(x_1,...x_t \mid t){\rm d}t_1 \cdots {\rm d}t_{j-1}{\rm d}t_{j+1}\cdots{\rm d}t_t$においては異常となり得えます。
ただしこのケースにおいても、中央部が異常ではない場合もあります。 例えば、上グラフが工場の機械動作シグナルであり中央部だけオペレーションモードが切り替わっていたため異常ではないということも考えられます。
以上のように異常には色々な側面がありますが、文献[1]ではこのように異常の種類をまとめています:
左上図のPoint Anomalyは観測エラー等による単発ノイズデータ(外れ値)に見えますが、Group Anomalyは外れ値というよりも別の分布から発生しているデータ群に見えます。両者では異なった異常検知アプローチが必要と想定されます。右下のSemantic Anomalyまで来ると古典的な異常検知手法では対応が難しく、ディープラーニングによるアプローチが必要になってきます。
このように「異常」に統一解はなく、ケースバイケースで「異常」を定義しなければいけません。
異常検知アルゴリズムは基本的に教師なし学習である
異常検知アルゴリズムは教師あり学習ではなく基本的に教師なし学習を用います。
理由は2つで:
- 大概、異常データは少ないから
- 異常データは多種多様であり網羅的にモデリングすることが難しいから
です。特に異常がクリティカルな現場(医療や工場)では、異常データを数百~数千も集められないはずです。また、理由2に関しては冒頭で説明したとおりです。「正常・異常」を当てるよりも、「正常・正常じゃない」を当てる方が簡単で筋が良い問題設定になります。後者は異常データに関して言及しておらず、正常データの構造理解のみで達成可能です。教師なし学習とは、サンプル$x_i(i=1,...,n)$から$p(x)$を推定することでデータの構造を理解する枠組みですから、異常検知は基本的に教師なし学習を用いることになります。ただし、教師なし学習を行う際のサンプルには異常データが含まれていない、もしくは圧倒的少数であると仮定できる必要があります。
余談ですが、文献[2]では以下のようなことを述べておりお気に入りです:
トルストイの『アンナ・カレーニナ』という小説に「幸せな家族は皆どこか似ているが,不幸せな家族はそれぞれ違う(Happy families are all alike; every unhappy family is unhappy in its own way)」という一節があり示唆的です.
異常検知アルゴリズムは4種類に大別される
それでは具体的に異常検知アルゴリズムの紹介をしていきます。
アルゴリズムは大きく4つのアプローチにより分類できます。それぞれ、Classification Approach, Probabilistic Approach, Reconstruction Approach, Distance Approachです。この4つの分類に加えて、ディープラーニングを用いるか否かで分類をした表がこちらになります:
PCAやOC-SVM等のワードひとつひとつが過去提案されてきた異常検知アルゴリズムの名前です。各アルゴリズムの詳細は後続で紹介するので、ここでは4つのアプローチそれぞれの概要の紹介に留めます。(私自身、下記テーブルへの理解が十分ではないためご意見をいただけますと勉強になります。)
アプローチ 概要 Pros Cons Classification データの正常・異常を分ける境界を直接推定するアプローチ 分布の形状を仮定する必要がない。また、SVMを用いた手法においては比較的少数データで学習可能。 正常データが複雑な構造をしていると上手く動作しない場合がある Probabilistic 正常データの確率分布を推定し、生起確率が低い事象を異常とみなすアプローチ データが仮定したモデルに従っていれば非常に高精度を達成 分布モデル等多くの仮定が必要。また高次元への対応も困難。 Reconstruction 正常データを再構成するようwell-trainされたロジックが上手く再構成できないデータは異常である、というアプローチ 近年盛んに研究されているAEやGAN等の強力な手法が適応できる。 あくまでも次元圧縮や再構成精度を高めるために目的関数がデザインされているため、必ずしも異常検知に特化した手法ではないこと。 Distance データ間の距離に基づき異常検知を行うアプローチ。異常データは正常データから距離的に離れているという発想。 アイディアがシンプルで、出力への説明可能性も高い。 計算量が大きい($O(n^2)$など) 上記テーブルは以下資料を参考に作成しました。
1. 比戸:Jubatus Casual Talks #2 異常検知入門
2. PANG, et al. Deep Learning for Anomaly Detection: A Review§2 アルゴリズムの紹介と実験
紹介する手法まとめ
- Classification Approach
- One-Class SVM
- Probability Approach
- ホテリングのT2法
- GMM
- KDE
- Reconstruction Approach
- AE
- Distance Approach
- k-NN
- LOF
Classification Approach
One-Class SVM
データ$D=\{x_1,...,x_N\}$が与えられているとします。ただし$D$には異常データが含まれていないか、含まれていたとしても圧倒的少数であるとします。
前章でClassification Approachは正常・異常を分ける境界を推定すると紹介しました。One-Class SVMは、正常データをすっぽり囲むような球を計算し境界とするアルゴリズムです。(球といっても三次元とは限りません。)
半径をとても大きくとれば$D$をすっぽり内包する球は作れますが、大きすぎる球ではFalse Negativeが1になってしまい嬉しくありません。そこで、
- 球の体積はできるだけ小さくする
- $D$のなかで、球からはみ出るデータがあることを許容する
の2点を許すことで汎用的な球を計算します。具体的には以下の最適化問題を解くことで最適な球の半径$R$と中心$b$が求まります:
\min_{R,b,u} \left\{R^2 + C\sum_{n=1}^{N}u_n \right\} \quad {\rm s.t.} \quad \|x_n - b\|^2 \le R^2 + u_nただし、$u$は球からはみ出した分のペナルティ項、$C$はそのペナルティをどれくらい重要視するかのパラメータです。SVMではカーネル関数によるデータの非線形変換が可能ですが、もちろんOne-Class SVMでも利用可能です。カーネル関数を適切に利用することで、適切な球が求まります。
数値実験をしてみます。データは以下のように準備しました。
import numpy as np import matplotlib import matplotlib.pyplot as plt import seaborn as sns sns.set() np.random.seed(123) #平均と分散 mean1 = np.array([2, 2]) mean2 = np.array([-2,-2]) cov = np.array([[1, 0], [0, 1]]) # 正常データの生成(2つの正規分布から生成) norm1 = np.random.multivariate_normal(mean1, cov, size=50) norm2 = np.random.multivariate_normal(mean2, cov, size=50) norm = np.vstack([norm1, norm2]) # 異常データの生成(一様分布から生成) lower, upper = -10, 10 anom = (upper - lower)*np.random.rand(20,20) + lower s=8 plt.scatter(norm[:,0], norm[:,1], s=s, color='green', label='normal') plt.scatter(anom[:,0], anom[:,1], s=s, color='red', label='anomaly') plt.legend()from sklearn import svm xx, yy = np.meshgrid(np.linspace(-10, 10, 500), np.linspace(-10, 10, 500)) # fit the model clf = svm.OneClassSVM(nu=0.1, kernel="rbf", gamma=0.1) clf.fit(norm) def draw_boundary(norm, anom, clf): # plot the line, the points, and the nearest vectors to the plane Z = clf.decision_function(np.c_[xx.ravel(), yy.ravel()]) Z = Z.reshape(xx.shape) plt.title("One-Class SVM") plt.contourf(xx, yy, Z, levels=np.linspace(Z.min(), Z.max(), 7), cmap=plt.cm.PuBu) bd = plt.contour(xx, yy, Z, levels=[0], linewidths=2, colors='darkred') plt.contourf(xx, yy, Z, levels=[0, Z.max()], colors='palevioletred') nm = plt.scatter(norm[:, 0], norm[:, 1], c='green', s=s) am = plt.scatter(anom[:, 0], anom[:, 1], c='red', s=s) plt.axis('tight') plt.xlim((-10, 10)) plt.ylim((-10, 10)) plt.legend([bd.collections[0], nm, am], ["learned frontier", "normal", "anomaly"], loc="upper left", prop=matplotlib.font_manager.FontProperties(size=11)) plt.show() draw_boundary(norm, anom, clf)ちなみに、パラメータ$C$を小さくする、すなわち球からはみ出すペナルティを軽くみるようにするとこのように境界が変わります。(sklearnではパラメータ$\nu$で表していますが、どうやら分母にいるらしいので$\nu$を小さくする$=C$を大きくするのようです)
# fit the model clf = svm.OneClassSVM(nu=0.5, kernel="rbf", gamma=0.1) clf.fit(norm) draw_boundary(norm, anom, clf)他にもOne-Class NN等が提案されています。One-Class SVMではカーネル関数を調整して適切な非線形変換を探索していましたが、そこをNNで代用&自動化しようというアイディアです。実装は
めんどくさそうなのでやってません。Probability Approach
ホテリングのT2法
ホテリングのT2法は古典的な手法であり、理論的な解析がなされています。 結論、正規分布を仮定することで異常度(Anomaly Score)が$\chi^2$分布に従う性質を利用したアルゴリズムになります。
ここでは簡単のため1次元データで説明します。 例によってデータ$D$が与えられているとします。ここで各データ$x$が正規分布
p(x\mid \mu, \sigma) = \frac{1}{\sqrt{2\pi\sigma^2}}\exp\left\{-\frac{1}{2\sigma^2}(x-\mu)^2\right\}に従うものと仮定します。この正規分布を最尤推定法によりデータへフィッティングすることで
\begin{align*} \hat{\mu} &= \frac{1}{N}\sum_{n=1}^{N}x_n \\ \hat{\sigma}^2 &= \frac{1}{N}\sum_{n=1}^{N}(x_n - \hat{\mu})^2 \end{align*}と各パラメータが推定されます。つまり、データ$D$は正規分布$p(x\mid \hat{\mu}, \hat{\sigma}^2)$に従うと推定したわけです。
さて、ここで異常度(Anomaly Score)を導入します。異常度とは、文字通り異常の度合いを示す統計量のことで、よく負の対数尤度が用いられます。
prob = np.linspace(0.01, 1, 100) neg_log_likh = [-np.log(p) for p in prob] plt.plot(prob, neg_log_likh) plt.title('Negative Log Likelihood') plt.ylabel('Anomaly Score') plt.xlabel('Prob.')グラフのように、負の対数尤度は確率が小さいところで大きな値をとる形状をしており、異常度として機能しそうです。今推定した正規分布の異常度を計算すると
-\log p(x\mid \hat{\mu}, \hat{\sigma}^2) = \frac{1}{2\hat{\sigma}^2}(x-\hat{\mu})^2 + \frac{1}{2}\log(2\pi\hat{\sigma}^2)となります。異常度はデータ$x$に対して定義される量ですから、$x$に関係ない項は除いたうえで異常度$a(x)$は
a(x) = \left( \frac{x - \hat{\mu}}{\hat{\sigma}} \right)^2と計算されます。異常度の分子は、「データが平均から離れているほど異常度が高くなる」ということを表しています。分母は、「どれくらいデータが散らばっているか?」を表しており、散らばっているデータでは平均から遠く離れていようと大目に見る、逆に密なデータでは少しのズレも許さないというお気持ちがあります。
実はこの異常度$a(x)$はサンプルサイズ$N$が十分大きいときに自由度$1$のカイ二乗分布$\chi^2_1$に従うことがわかっています。
from scipy.stats import chi2 x = np.linspace(0, 8, 1000) for df in [1,2,3,4,5]: plt.plot(x, chi2.pdf(x ,df = df), label='deg. of freedom:'+str(df)) plt.legend() plt.ylim(0, 0.5) plt.title('Chi-Square Dist.')あとは有意水準$5$%で閾値$a_{th}$を決めるなどしてあげれば異常検知が可能となります。まとめると、
- データが正規分布に従うと仮定する。
- 正規分布のパラメータ$\mu$, $\sigma$を最尤推定法により推定する
- カイ二乗分布から、正常・異常を分ける点$a_{th}$を決める
- 新たなデータ$x_{new}$が、$a(x_{new})>a_{th}$だったら異常とみなす
がホテリングのT2法です。今回は1次元で説明しましたが、多次元でも同様に異常度がカイ二乗分布に従います。ただし、自由度=次元数です。
では実験してみましょう。データは以下のように作成しました。
norm = np.random.normal(0, 1, 20) # 正常データ anom = np.array([-3, 3]) # 異常データ plt.scatter(norm, [0]*len(norm), color='green', label='normal') plt.scatter(anom, [0]*len(anom), color='red', label='anomaly')# 最尤推定 N = len(norm) mu_hat = sum(norm)/N s_hat = sum([(x-mu_hat)**2 for x in norm])/N # カイ二乗分布の分位点を閾値とする a_th_5 = chi2.ppf(q=0.95, df=1) a_th_30 = chi2.ppf(q=0.7, df=1) # 各データの異常度を計算 data = np.hstack([norm, anom]) anom_score = [] for d in data: score = ((d - mu_hat)/s_hat)**2 anom_score.append(score) #描画 norm_pred_5 = [d for d,a in zip(data, anom_score) if a<=a_th_5] anom_pred_5 = [d for d,a in zip(data, anom_score) if a>a_th_5] norm_pred_30 = [d for d,a in zip(data, anom_score) if a<=a_th_30] anom_pred_30 = [d for d,a in zip(data, anom_score) if a>a_th_30] fig, axes = plt.subplots(1,3, figsize=(15,5)) axes[0].scatter(norm, [0]*len(norm), color='green', label='normal') axes[0].scatter(anom, [0]*len(anom), color='red', label='anomaly') axes[0].set_title('Truth') axes[1].scatter(norm_pred_5, [0]*len(norm_pred_5), color='green', label='normal pred') axes[1].scatter(anom_pred_5, [0]*len(anom_pred_5), color='red', label='anomaly pred') axes[1].set_title('Threshold:5%') axes[2].scatter(norm_pred_30, [0]*len(norm_pred_30), color='green', label='normal pred') axes[2].scatter(anom_pred_30, [0]*len(anom_pred_30), color='red', label='anomaly pred') axes[2].set_title('Threshold:30%') plt.legend()異常度の閾値を小さくすれば、異常と見なされる区域が広がっていくことが見て取れます。
閾値は問題に応じて決める必要があります。例えば、医療の現場など異常の見逃しがクリティカルな場合は閾値は小さく設定すべきですし、データクレンジング等の場合はもう少し大きい閾値にしてもいいかもしれません。GMM(Gaussian Mixture Model)
ホテリングのT2法ではデータに正規分布を仮定していましたが、これが不適切なケースもあるでしょう。例えば以下のようにデータが複数個所に集中している(単峰ではなく多峰)場合に正規分布でフィッティングすると
lower, upper = -3, -1 data1 = (upper - lower)*np.random.rand(20) + lower lower, upper = 1, 3 data2 = (upper - lower)*np.random.rand(30) + lower data = np.hstack([data1, data2]) # 最尤推定 N = len(data) mu_hat = sum(data)/N s_hat = sum([(x-mu_hat)**2 for x in data])/N # プロット from scipy.stats import norm X = np.linspace(-7, 7, 1000) Y = norm.pdf(X, mu_hat, s_hat) plt.scatter(data, [0]*len(data)) plt.plot(X, Y)のように、全くデータが観測されていない箇所に正規分布のピークが来るように推定されてしまいます。こういった場合は正規分布ではなく、もう少し複雑な分布を設定するのが良いでしょう。今回はその一例としてGMM(Gaussian Mixture Model, 混合正規分布)による異常検知を紹介します。
GMMとは正規分布をいくつか足し合わせることで多峰型を表現する分布です。足し合わせる個数はよく$K$で表現され、コンポーネント数と呼ばれます。GMMの数式とビジュアルは以下の通りです(1次元にしています)。
p(x\mid \pi, \mu, \sigma) = \sum_{k=1}^{K}\pi_k N(x\mid \mu_k, \sigma_k)ただし、
0 \le\pi_k\le 1,\quad \sum_{k=1}^{K}\pi_k=1,\quad N(x\mid\mu,\sigma)={\text 正規分布}です。
# プロット from scipy.stats import norm X = np.linspace(-7, 7, 1000) Y1 = norm.pdf(X, 1, 1) Y2 = norm.pdf(X, -3, 2) Y = 0.5*Y1 + 0.5*Y2 plt.plot(X, Y1, label='Component1') plt.plot(X, Y2, label='Component2') plt.plot(X, Y, label='GMM') plt.legend()各正規分布の平均($\mu$)・分散($\sigma$)と、混合率$\pi$はデータから学習されるパラメータです。しかしコンポーネント数$K$は決め打ちする必要があります。このように分析者側で決め打ちにするパラメータをハイパーパラメータと呼びます。
ではGMMによる異常検知を実験してみます。コンポーネント数$K$によってどのように推定分布の形状が変わるのかも観察しましょう。データはOne-Class SVMと同様以下を使用します。
def draw_GMM(norm, anom, clf, K): # plot the line, the points, and the nearest vectors to the plane xx, yy = np.meshgrid(np.linspace(-10, 10, 1000), np.linspace(-10, 10, 1000)) Z = np.exp(clf.score_samples(np.c_[xx.ravel(), yy.ravel()])) Z = Z.reshape(xx.shape) plt.title("GMM:K="+str(K)) plt.contourf(xx, yy, Z, levels=np.linspace(Z.min(), Z.max(), 7), cmap=plt.cm.PuBu) nm = plt.scatter(norm[:, 0], norm[:, 1], c='green', s=s) am = plt.scatter(anom[:, 0], anom[:, 1], c='red', s=s) plt.axis('tight') plt.xlim((-10, 10)) plt.ylim((-10, 10)) plt.legend([nm, am], ["normal", "anomaly"], loc="upper left", prop=matplotlib.font_manager.FontProperties(size=11)) plt.show()# GMMによるフィッティング from sklearn.mixture import GaussianMixture for K in [1,2,3,4]: clf = GaussianMixture(n_components=K, covariance_type='full') clf.fit(norm) draw_GMM(norm, anom, clf, K)
このようにコンポーネント数を多くすると、データの細部の傾向まで捉えていることがわかります。しかし、細部まで捉えている=汎化性能が高いわけではないので注意が必要です。コンポーネント数はWAIC等の情報量規準を用いて決定することができます(とりあえずWAICを使ってみました:GitHub)KDE(Kernel Density Estimation)
今までは正規分布や混合正規分布を仮定し、データへのフィッティングを通じて内部パラメータの推定を行ってきました。このようなアプローチをパラメトリックなアプローチといいます。しかし、パラメトリックな分布では上手く表現できないような場合にはどうしたら良いでしょうか?
ノンパラメトリックなアプローチはそのような場合に有効であり、KDEは代表的なノンパラメトリック推定手法です。KDEは、各データ点にカーネル(通常はGaussian)を重ねることで確率分布を推定する手法です。可視化すると以下のようになります。
from scipy.stats import norm np.random.seed(123) lower, upper = -1, 5 data = (upper - lower)*np.random.rand(3) + lower for i, d in enumerate(data): X = np.linspace(lower,upper,500) Y = norm.pdf(X, loc=d, scale=1) knl = plt.plot(X,Y,color='green', label='kernel'+str(i+1)) # KDE n = 3 h = 0.24 Y_pdf = [] for x in X: Y = 0 for d in data: Y += norm.pdf((x-d)/h, loc=0, scale=1) Y /= n*h Y_pdf.append(Y) kde = plt.plot(X,Y_pdf, label='KDE') plt.scatter(data, [0]*len(data), marker='x') plt.ylim([-0.05, 1]) plt.legend()このようにカーネルの重ね合わせで確率分布を推定しています。カーネルに正規分布を用いる場合は分散(バンド幅と呼びます)を決め打ちする必要があります。後続の実験では、このバンド幅を変えた時の推定分布の変化も観察しましょう。
KDEの理論詳細および長所・短所は鈴木.データ解析第十回「ノンパラメトリック密度推定法」がわかりやすかったです:
推定分布
p(x) = \frac{1}{Nh}\sum_{n=1}^{N}K\left(\frac{x-X_n}{h}\right)ただし、$h>0$をバンド幅、$K()$をカーネル関数、$X_n$は各データ点とする。
長所・短所
出典:鈴木.データ解析第十回「ノンパラメトリック密度推定法」ではKDEによる異常検知を実験してみましょう。データは例によって
を使用します。バンド幅を変えた時の推定分布は以下のようになりました。def draw_KDE(norm, anom, clf, K): # plot the line, the points, and the nearest vectors to the plane xx, yy = np.meshgrid(np.linspace(-10, 10, 1000), np.linspace(-10, 10, 1000)) Z = np.exp(clf.score_samples(np.c_[xx.ravel(), yy.ravel()])) Z = Z.reshape(xx.shape) plt.title("KDE:BandWidth="+str(bw)) plt.contourf(xx, yy, Z, levels=np.linspace(Z.min(), Z.max(), 7), cmap=plt.cm.PuBu) nm = plt.scatter(norm[:, 0], norm[:, 1], c='green', s=s) am = plt.scatter(anom[:, 0], anom[:, 1], c='red', s=s) plt.axis('tight') plt.xlim((-10, 10)) plt.ylim((-10, 10)) plt.legend([nm, am], ["normal", "anomaly"], loc="upper left", prop=matplotlib.font_manager.FontProperties(size=11)) plt.show()from sklearn.neighbors import KernelDensity for bw in [0.2,0.5,1.0,2.0]: clf = KernelDensity(bandwidth=bw, kernel='gaussian') clf.fit(norm) draw_KDE(norm, anom, clf, bw)
このように、バンド幅が大きくなると推定分布が滑らかになることがわかります。また、カーネル関数は正規分布に限らず他にもあるので、「バンド幅・カーネル関数」の両者を適切に設定してあげる必要があります。Reconstruction Approach
Reconstruction Approachは画像データに用いられることが多いので、ここでも画像データを使用します。正常データをMNIST、異常データをFashion-MNISTとします。
# MNISTのロード import torch from torch.utils.data import DataLoader from torchvision import datasets, transforms # 学習データの読み込み train_mnist = datasets.MNIST( root="./data", train=True, transform=transforms.Compose([ transforms.ToTensor(), transforms.Normalize((0.5,), (0.5,)) # 区間[-1,1]内で平均、標準偏差 = 0.5, 0.5に正規化 ]), download=True ) train_mnist_loader = torch.utils.data.DataLoader( train_mnist, batch_size=64, shuffle=True ) # テストデータの読み込み test_mnist = datasets.MNIST( root="./data", train=False, transform=transforms.Compose([ transforms.ToTensor(), transforms.Normalize((0.5,), (0.5,)) # 区間[-1,1]内で平均、標準偏差 = 0.5, 0.5に正規化 ]), download=True ) test_mnist_loader = torch.utils.data.DataLoader( test_mnist, batch_size=64, shuffle=True )# Fashion-MNISTのロード # 学習データの読み込み train_fashion = datasets.FashionMNIST( root="./data", train=True, transform=transforms.Compose([ transforms.ToTensor(), transforms.Normalize((0.5,), (0.5,)) # 区間[-1,1]内で平均、標準偏差 = 0.5, 0.5に正規化 ]), download=True ) train_fashion_loader = torch.utils.data.DataLoader( train_fashion, batch_size=64, shuffle=True ) # テストデータの読み込み test_fashion = datasets.FashionMNIST( root="./data", train=False, transform=transforms.Compose([ transforms.ToTensor(), transforms.Normalize((0.5,), (0.5,)) # 区間[-1,1]内で平均、標準偏差 = 0.5, 0.5に正規化 ]), download=True ) test_fashion_loader = torch.utils.data.DataLoader( test_fashion, batch_size=64, shuffle=True )import matplotlib.pyplot as plt # 画像を一気に並べて可視化する関数 def imshow(image_set, nrows=2, ncols=10): plot_num = nrows * ncols fig, ax = plt.subplots(ncols=ncols, nrows=nrows, figsize=(15, 15*nrows/ncols)) ax = ax.ravel() for i in range(plot_num): ax[i].imshow(image_set[i].reshape(28, 28), cmap='gray') ax[i].set_xticks([]) ax[i].set_yticks([])# MNISTのサンプル可視化 imshow(train_mnist.data)# Fashion-MNISTのサンプル可視化 imshow(train_fashion.data)AE(Auto Encoder)
AE(Auto Encoder, オートエンコーダ)とは、NN(Neural Network, ニューラルネットワーク)の一種であり、入力と出力が等しくなるように学習するNNです。ただの恒等関数ではつまらないので、通常は入力次元よりも小さい次元の中間層を挟みます。
これは何をしているのかというと、中間層で入力データを「要約」しています。数理的には、入力データを再構成できるだけの情報を保持しつつ低次元空間へマッピングしている、ということになります。
AEを用いた異常検知は、「正常データを再構成するよう訓練されたAEは、異常データを上手く再構成できないのではないか?」という思想に基づきます。
では、AEによる異常検知を実験しましょう。AEをMNISTで訓練した後に、MNISTおよびFashion-MNISTの再構成精度を異常度として検知を試みます。
import torch.nn as nn import torch.nn.functional as F # AEの定義 class AE(nn.Module): def __init__(self): super(AE, self).__init__() self.fc1 = nn.Linear(28*28, 256) self.fc2 = nn.Linear(256, 28*28) def forward(self, x): x = torch.tanh(self.fc1(x)) x = torch.tanh(self.fc2(x)) return x ae = AE() # 損失関数と最適化手法の定義 import torch.optim as optim criterion = nn.MSELoss() optimizer = optim.Adam(ae.parameters(), lr=0.0001)# 学習ループ N_train = train_mnist.data.shape[0] N_test = test_mnist.data.shape[0] train_loss = [] test_loss = [] for epoch in range(10): # Train step running_train_loss = 0.0 for data in train_mnist_loader: inputs, _ = data inputs = inputs.view(-1, 28*28) # zero the parameter gradients optimizer.zero_grad() # forward + backward + optimize outputs = ae(inputs) loss = criterion(outputs, inputs) loss.backward() optimizer.step() running_train_loss += loss.item() running_train_loss /= N_train train_loss.append(running_train_loss) # Test step with torch.no_grad(): running_test_loss = 0 for data in test_mnist_loader: inputs, _ = data inputs = inputs.view(-1, 28*28) outputs = ae(inputs) running_test_loss += criterion(outputs, inputs).item() running_test_loss /= N_test test_loss.append(running_test_loss) if (epoch+1)%1==0: # print every 1 epoch print('epoch:{}, train_loss:{}, test_loss:{}'.format(epoch + 1, running_train_loss, running_test_loss)) plt.plot(train_loss, label='Train loss') plt.plot(test_loss, label='Test loss') plt.ylabel('Loss') plt.xlabel('epoch') plt.legend()# 再構成の様子を可視化してみる with torch.no_grad(): for data in test_mnist_loader: inputs, _ = data inputs = inputs.view(-1, 28*28) outputs = ae(inputs) n = 5 # num to viz fig, axes = plt.subplots(ncols=n, nrows=2, figsize=(15, 15*2/n)) for i in range(n): axes[0, i].imshow(inputs[i].reshape(28, 28), cmap='gray') axes[1, i].imshow(outputs[i].reshape(28, 28), cmap='gray') axes[0, i].set_title('Input') axes[1, i].set_title('Reconstruct') axes[0, i].set_xticks([]) axes[0, i].set_yticks([]) axes[1, i].set_xticks([]) axes[1, i].set_yticks([])きれいに再構成できていそうです。
それでは、MNISTとFashion-MNISTの再構成精度(今回は二乗誤差)の分布を描いてみます。AEによる異常検知が成功していれば、Fashion-MNISTの精度は低くなるはずです。
# 再構成精度の算出 mnist_loss = [] with torch.no_grad(): for data in test_mnist_loader: inputs, _ = data for data_i in inputs: data_i = data_i.view(-1, 28*28) outputs = ae(data_i) loss = criterion(outputs, data_i) mnist_loss.append(loss.item()) fashion_loss = [] with torch.no_grad(): for data in test_fashion_loader: inputs, _ = data for data_i in inputs: data_i = data_i.view(-1, 28*28) outputs = ae(data_i) loss = criterion(outputs, data_i) fashion_loss.append(loss.item())plt.hist([mnist_loss, fashion_loss], bins=25, label=['MNIST', 'Fashion-MNIST']) plt.xlabel('Loss') plt.ylabel('data count') plt.legend() plt.show()確かにFashion-MNISTの再構成は上手くいってなさそうなので、再構成精度による異常検知は成立しそうです。
最後に、Fashion-MNISTの中でもLossが低いもの(=異常と判定し辛いもの)と高いもの(=簡単に異常と判定できるもの)をピックアップして描画してみます。
lower_loss, upper_loss = 0.1, 0.8 data_low = [] # Lossが低いデータを格納 data_up = [] # Lossが高いデータを格納 with torch.no_grad(): for data in test_fashion_loader: inputs, _ = data for data_i in inputs: data_i = data_i.view(-1, 28*28) outputs = ae(data_i) loss = criterion(outputs, data_i).item() if loss<lower_loss: data_low.append([data_i, outputs, loss]) if loss>upper_loss: data_up.append([data_i, outputs, loss])# ランダムに描画する用のインデックス np.random.seed(0) n = 3 #描画データ数 lower_indices = np.random.choice(np.arange(len(data_low)), n) upper_indices = np.random.choice(np.arange(len(data_up)), n) indices = np.hstack([lower_indices, upper_indices]) fig, axes = plt.subplots(ncols=n*2, nrows=2, figsize=(15, 15*2/(n*2))) # Lossが小さいデータの描画 for i in range(n): inputs = data_low[indices[i]][0] outputs = data_low[indices[i]][1] loss = data_low[indices[i]][2] axes[0, i].imshow(inputs.reshape(28, 28), cmap='gray') axes[1, i].imshow(outputs.reshape(28, 28), cmap='gray') axes[0, i].set_title('Input') axes[1, i].set_title('Small Loss={:.2f}'.format(loss)) axes[0, i].set_xticks([]) axes[0, i].set_yticks([]) axes[1, i].set_xticks([]) axes[1, i].set_yticks([]) # Lossが大きいデータの描画 for i in range(n, 2*n): inputs = data_up[indices[i]][0] outputs = data_up[indices[i]][1] loss = data_up[indices[i]][2] axes[0, i].imshow(inputs.reshape(28, 28), cmap='gray') axes[1, i].imshow(outputs.reshape(28, 28), cmap='gray') axes[0, i].set_title('Input') axes[1, i].set_title('Large Loss={:.2f}'.format(loss)) axes[0, i].set_xticks([]) axes[0, i].set_yticks([]) axes[1, i].set_xticks([]) axes[1, i].set_yticks([]) plt.tight_layout()Lossが低い画像は黒い面積が大きい傾向が見られる一方で、Lossが高い画像は白い面積が大きい傾向が見られます。MNISTは基本的に黒の面積が大きい画像ですので、白の面積が大きいとAEにとっては「異質」になり再構成が上手くいっていないと思われます。
Distance Approach
k-NN(k-Nearest Neighbor)
k-NNはデータ間の距離に基づき異常度を測る手法です。k-NNでは近傍のk個のデータ点を含むような円を考えます。下図は$k=5$の場合です。
図のように、「異常データが描く円は正常データよりも大きくなるはず」とう思想に基づきk-NNでは円の半径を異常度として定義します。ただし、近傍点の個数$k$と正常/異常を分ける半径の閾値は適切にチューニングする必要があります。
それでは数値実験を行います。閾値は、正常データの半径値分布における90%分位点とします。
# データの作成 np.random.seed(123) #平均と分散 mean = np.array([2, 2]) cov = np.array([[3, 0], [0, 3]]) # 正常データの生成 norm = np.random.multivariate_normal(mean1, cov, size=50) # 異常データの生成(一様分布から生成) lower, upper = -10, 10 anom = (upper - lower)*np.random.rand(20,20) + lower s=8 plt.scatter(norm[:,0], norm[:,1], s=s, color='green', label='normal') plt.scatter(anom[:,0], anom[:,1], s=s, color='red', label='anomaly') plt.legend()k-NNによる異常検知を、どのようにライブラリで完結できるか分からなかったので自作します。。。
# 全データのk近傍円の半径を計算する関数 def kNN(data, k=5): """ input: data:list, k:int output: 全データのk近傍円の半径:list 距離はユークリッド距離を採用します。 """ output = [] for d in data: distances = [] for p in data: distances.append(np.linalg.norm(d - p)) output.append(sorted(distances)[k]) return output # 閾値より大きければ1, 小さければ0を返す関数 def kNN_scores(point, data, thres, K): distances = [] for p in data: distances.append(np.linalg.norm(point - p)) dist = sorted(distances)[K] if dist > thres: return 1 else: return 0def draw_KNN(norm, anom, thres, K, only_norm=False): # plot the line, the points, and the nearest vectors to the plane xx, yy = np.meshgrid(np.linspace(-10, 10, 100), np.linspace(-10, 10, 100)) Z = [kNN_scores(point, norm, thres, K) for point in np.c_[xx.ravel(), yy.ravel()]] Z = np.array(Z).reshape(xx.shape) plt.title("KNN:K="+str(K)) plt.contourf(xx, yy, Z) if not only_norm: nm = plt.scatter(norm[:, 0], norm[:, 1], c='green', s=s) am = plt.scatter(anom[:, 0], anom[:, 1], c='red', s=s) plt.axis('tight') plt.xlim((-10, 10)) plt.ylim((-10, 10)) plt.legend([nm, am], ["normal", "anomaly"], loc="upper left", prop=matplotlib.font_manager.FontProperties(size=11)) if only_norm: nm = plt.scatter(norm[:, 0], norm[:, 1], c='green', s=s) plt.axis('tight') plt.xlim((-10, 10)) plt.ylim((-10, 10)) plt.legend([nm], ["normal"], loc="upper left", prop=matplotlib.font_manager.FontProperties(size=11)) plt.show()for k in [1,5,30]: anomaly_scores = kNN(norm, k=k) thres = pd.Series(anomaly_scores).quantile(0.90) draw_KNN(norm, anom, thres, k)kが大きくなるにつれて、正常/異常の境界が滑らかになっていることがわかります。
さて、ここで少しいじわるな例で実験してみましょう。以下のように、正常データが密度の違う2つの正規分布から生成されているとします。
np.random.seed(123) #平均と分散 mean1 = np.array([3, 3]) mean2 = np.array([-5,-5]) cov1 = np.array([[0.5, 0], [0, 0.5]]) cov2 = np.array([[3, 0], [0, 3]]) # 正常データの生成(2つの正規分布から生成) norm1 = np.random.multivariate_normal(mean1, cov1, size=50) norm2 = np.random.multivariate_normal(mean2, cov2, size=10) norm = np.vstack([norm1, norm2]) s=8 plt.scatter(norm[:,0], norm[:,1], s=s, color='green', label='normal') plt.legend()for k in [1,5,30]: anomaly_scores = kNN(norm, k=k) thres = pd.Series(anomaly_scores).quantile(0.90) draw_KNN(norm, [], thres, k, only_norm=True)特にk=1のケースに注目すると、正常データが密度の異なる別クラスターから発生している場合kNNが上手く動作していないことがわかります。「密なクラスターからのズレは重要視して、疎なクラスターからのズレは大目に見る」という処理を加えれば対処できそうだと想像できます。
実はそれが次節で紹介するLOFという手法になります。
LOF(Local Outlier Factor, 局所外れ値因子法)
前節で紹介したk-NNが上手く動作しないケースを図で表すと
となります。もちろん、左側の異常度を高くしたいのですが、LOFではデータの密度という概念を導入しそれを実現します。数理的詳細は割愛しますが、以下のようなことをしています。
自身のスカスカ度合いと近傍点のスカスカ度合いの比で異常度を定義しています。
それではLOFの実験を行いましょう。
def draw_LOF(norm, anom, K, only_norm=False): # plot the line, the points, and the nearest vectors to the plane xx, yy = np.meshgrid(np.linspace(-10, 10, 100), np.linspace(-10, 10, 100)) Z = - model.predict(np.c_[xx.ravel(), yy.ravel()]) Z = np.array(Z).reshape(xx.shape) plt.title("LOF:K="+str(K)) plt.contourf(xx, yy, Z) if not only_norm: nm = plt.scatter(norm[:, 0], norm[:, 1], c='green', s=s) am = plt.scatter(anom[:, 0], anom[:, 1], c='red', s=s) plt.axis('tight') plt.xlim((-10, 10)) plt.ylim((-10, 10)) plt.legend([nm, am], ["normal", "anomaly"], loc="upper left", prop=matplotlib.font_manager.FontProperties(size=11)) if only_norm: nm = plt.scatter(norm[:, 0], norm[:, 1], c='green', s=s) plt.axis('tight') plt.xlim((-10, 10)) plt.ylim((-10, 10)) plt.legend([nm], ["normal"], loc="upper left", prop=matplotlib.font_manager.FontProperties(size=11)) plt.show()from sklearn.neighbors import LocalOutlierFactor for k in [1, 5, 30]: model = LocalOutlierFactor(n_neighbors=k, novelty=True) model.fit(norm) draw_LOF(norm, [], k, only_norm=True)このように、クラスターの密度を考慮したうえで異常検知ができていそうです。
発展的な異常検知問題
今後異常検知の勉強をしていくにあたって、以下のようなトピックを勉強してきたいと思っています:
- 時系列データの異常検知
- グラフデータの異常検知
- マネーロンダリング検知に使えそう
- 投稿日:2020-11-24T17:12:30+09:00
MNISTをpythonでロードしてpngに出力する手順
やりたいこと
機械学習用の学習/検証データのサンプルを集めている。
- MNISTをダウンロードして、中身がどうなっているか調べたい。
- そのためにpythonでubyteからpngを作成する処理を実装したい。
前提
- pythonでのnumpyやPILの基礎知識があること
- それらの実行環境を構築済みであること
MNISTとは?
0から9までの「手書き数字」の画像データ。
「手書きの数字をAIで識別して分類する」などの機械学習に使う。
http://yann.lecun.com/exdb/mnist/
から無料でダウンロードできる。ファイル構成
- train-images-idx3-ubyte.gz : 学習用の画像データ
- train-labels-idx1-ubyte.gz : 学習用のラベルデータ
- t10k-images-idx3-ubyte.gz : 検証用の画像データ
- t10k-labels-idx1-ubyte.gz : 検証用のラベルデータ
ファイルの中身
gzファイルを解凍すると、下記のようなバイナリファイルになる。
t10k-images.idx3-ubyte画像データといっても、.jpg のようなフォーマットではないため、
そのままではプレビューできない。画像化してプレビューする方法
たとえばpythonのコードを書いて、numpyやPILでpngに出力すれば、普通の画像ファイルとして表示できる。
事前準備
wgetやunzipがまだ入っていなければインストールしておく。ubuntuの場合は下記。
apt-get install -y wgetapt-get install unzipファイルのダウンロード
まずはwgetでgzをダウンロードしておく。
wget http://yann.lecun.com/exdb/mnist/train-labels-idx1-ubyte.gzwget http://yann.lecun.com/exdb/mnist/train-images-idx3-ubyte.gzそして解凍する。
gunzip train-images-idx3-ubyte.gzgunzip train-labels-idx1-ubyte.gzすると
-rw-r--r--. 1 root root 47040016 Jul 21 2000 train-images-idx3-ubyte -rw-r--r--. 1 root root 60008 Jul 21 2000 train-labels-idx1-ubyteが出力される。
続いてpythonのコードを書く。実装(Imagesの画像化)
vi test.py
import tensorflow as tf from tensorflow.keras.models import Sequential from tensorflow.keras.layers import Dense, Conv2D, Flatten, Dropout, MaxPooling2D from tensorflow.keras.preprocessing.image import ImageDataGenerator from tensorflow.keras.datasets import mnist import os import numpy as np import matplotlib.pyplot as plt import struct from PIL import Image trainImagesFile = open('./train-images-idx3-ubyte','rb') trainLabelsFile = open('./train-labels-idx1-ubyte','rb') f = trainImagesFile magic_number = f.read( 4 ) magic_number = struct.unpack('>i', magic_number)[0] number_of_images = f.read( 4 ) number_of_images = struct.unpack('>i', number_of_images)[0] number_of_rows = f.read( 4 ) number_of_rows = struct.unpack('>i', number_of_rows)[0] number_of_columns = f.read( 4 ) number_of_columns = struct.unpack('>i', number_of_columns)[0] bytes_per_image = number_of_rows * number_of_columns raw_img = f.read(bytes_per_image) format = '%dB' % bytes_per_image lin_img = struct.unpack(format, raw_img) np_ary = np.asarray(lin_img).astype('uint8') np_ary = np.reshape(np_ary, (28,28),order='C') pil_img = Image.fromarray(np_ary) pil_img.save("output.png")実行
python test.py出力結果
output.png
解説
学習用の画像データ
train-images-idx3-ubyte
の構造は下記の通り。http://yann.lecun.com/exdb/mnist/
TRAINING SET IMAGE FILE (train-images-idx3-ubyte): [offset] [type] [value] [description] 0000 32 bit integer 0x00000803(2051) magic number 0004 32 bit integer 60000 number of images 0008 32 bit integer 28 number of rows 0012 32 bit integer 28 number of columns 0016 unsigned byte ?? pixel 0017 unsigned byte ?? pixel ........ xxxx unsigned byte ?? pixel上記に従い、offsetを4ずつズラしながら順次読み込んでいく。
magic_number = f.read( 4 )この結果は 2051 になる。
number_of_images = f.read( 4 )この結果は 60000 になる。
number_of_rows = f.read( 4 )この結果は 28 になる。
number_of_columns = f.read( 4 )この結果は 28 になる。
値を実際に確認したければ、
print('--------------------') print('magic_number'); print(magic_number); print('--------------------') print('number_of_images'); print(number_of_images); print('--------------------') print('number_of_rows'); print(number_of_rows); print('--------------------') print('number_of_columns'); print(number_of_columns);のように出力すれば確認できる。
-------------------- magic_number 2051 -------------------- number_of_images 60000 -------------------- number_of_rows 28 -------------------- number_of_columns 28そして、
[offset] [type] [value] [description] 0016 unsigned byte ?? pixelとなっているので、offset16以降には画像が入っており
bytes_per_image = number_of_rows * number_of_columns raw_img = f.read(bytes_per_image)のように読み込むことができる。
あとはnumpyに突っ込んでpng形式で保存している。ループ処理で連続してpngを出力する
下記のようにpng出力処理をループで回せば、連続して画像化できる。
あとは、出力したい枚数が10枚であれば range(10): のように、ループ回数を任意に指定すればよい。for num in range(10): raw_img = f.read(bytes_per_image) format = '%dB' % bytes_per_image lin_img = struct.unpack(format, raw_img) np_ary = np.asarray(lin_img).astype('uint8') np_ary = np.reshape(np_ary, (28,28),order='C') pil_img = Image.fromarray(np_ary) pil_img.save("output" + str(num) + ".png")出力結果は下記。
numpy配列とpngの比較
numpy配列であるnp_aryをprint()で表示させると、下記のような配列データになっている。
[ 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0] [ 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0] [ 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0] [ 0 0 0 0 0 0 0 0 0 0 0 0 3 18 18 18 126 136 175 26 166 255 247 127 0 0 0 0] [ 0 0 0 0 0 0 0 0 30 36 94 154 170 253 253 253 253 253 225 172 253 242 195 64 0 0 0 0] [ 0 0 0 0 0 0 0 49 238 253 253 253 253 253 253 253 253 251 93 82 82 56 39 0 0 0 0 0] [ 0 0 0 0 0 0 0 18 219 253 253 253 253 253 198 182 247 241 0 0 0 0 0 0 0 0 0 0] [ 0 0 0 0 0 0 0 0 80 156 107 253 253 205 11 0 43 154 0 0 0 0 0 0 0 0 0 0] [ 0 0 0 0 0 0 0 0 0 14 1 154 253 90 0 0 0 0 0 0 0 0 0 0 0 0 0 0] [ 0 0 0 0 0 0 0 0 0 0 0 139 253 190 2 0 0 0 0 0 0 0 0 0 0 0 0 0] [ 0 0 0 0 0 0 0 0 0 0 0 11 190 253 70 0 0 0 0 0 0 0 0 0 0 0 0 0] [ 0 0 0 0 0 0 0 0 0 0 0 0 35 241 225 160 108 1 0 0 0 0 0 0 0 0 0 0] [ 0 0 0 0 0 0 0 0 0 0 0 0 0 81 240 253 253 119 25 0 0 0 0 0 0 0 0 0] [ 0 0 0 0 0 0 0 0 0 0 0 0 0 0 45 186 253 253 150 27 0 0 0 0 0 0 0 0] [ 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 16 93 252 253 187 0 0 0 0 0 0 0 0] [ 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 249 253 249 64 0 0 0 0 0 0 0] [ 0 0 0 0 0 0 0 0 0 0 0 0 0 0 46 130 183 253 253 207 2 0 0 0 0 0 0 0] [ 0 0 0 0 0 0 0 0 0 0 0 0 39 148 229 253 253 253 250 182 0 0 0 0 0 0 0 0] [ 0 0 0 0 0 0 0 0 0 0 24 114 221 253 253 253 253 201 78 0 0 0 0 0 0 0 0 0] [ 0 0 0 0 0 0 0 0 23 66 213 253 253 253 253 198 81 2 0 0 0 0 0 0 0 0 0 0] [ 0 0 0 0 0 0 18 171 219 253 253 253 253 195 80 9 0 0 0 0 0 0 0 0 0 0 0 0] [ 0 0 0 0 55 172 226 253 253 253 253 244 133 11 0 0 0 0 0 0 0 0 0 0 0 0 0 0] [ 0 0 0 0 136 253 253 253 212 135 132 16 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0] [ 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0] [ 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0] [ 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0]これは画像ファイルを構成する各ピクセル位置と、その色情報であることが分かる。
ラベルデータについて
前述の実装は画像データ(Images)をpng化するものだったが、
これに加えて、ラベルデータ(Labels)も確認する必要がある。
そのための実装は下記の通りである。import tensorflow as tf from tensorflow.keras.models import Sequential from tensorflow.keras.layers import Dense, Conv2D, Flatten, Dropout, MaxPooling2D from tensorflow.keras.preprocessing.image import ImageDataGenerator from tensorflow.keras.datasets import mnist import os import numpy as np import matplotlib.pyplot as plt import struct from PIL import Image trainImagesFile = open('./train-images-idx3-ubyte','rb') trainLabelsFile = open('./train-labels-idx1-ubyte','rb') f = trainLabelsFile magic_number = f.read( 4 ) magic_number = struct.unpack('>i', magic_number)[0] number_of_images = f.read( 4 ) number_of_images = struct.unpack('>i', number_of_images)[0] print("--------------------") print("magic_number") print(magic_number) print("--------------------") print("number_of_image") print(number_of_images) print("--------------------") label_byte = f.read( 1 ) label_int = int.from_bytes(label_byte, byteorder='big') print(label_int)出力結果
-------------------- magic_number 2049 -------------------- number_of_image 60000 -------------------- 5ラベルデータの構造は下記の通り。
train-labels-idx1-ubyte
TRAINING SET LABEL FILE (train-labels-idx1-ubyte): [offset] [type] [value] [description] 0000 32 bit integer 0x00000801(2049) magic number (MSB first) 0004 32 bit integer 60000 number of items 0008 unsigned byte ?? label 0009 unsigned byte ?? label ........ xxxx unsigned byte ?? label The labels values are 0 to 9.つまり、offset 8 以降を、1ずつ読んでいけば、ラベルが読み取れる。
ループで実装するなら下記。for num in range(10): label_byte = f.read( 1 ) label_int = int.from_bytes(label_byte, byteorder='big') print(label_int)出力結果は下記。
5 0 4 1 9 2 1 3 1 4Imagesの出力結果と比較する。
それぞれのpng画像が「何の数字であるか?」を、ラベルにて正しく示している。
- 投稿日:2020-11-24T16:01:08+09:00
PythonとHerokuでLINEBOTを作ってみた
Python学習の一環でオウム返しするLINEBOTを作ってみました。
完成イメージ
環境構成
・Python
・Heroku
・LINE Developers
・Flask開発手順
1.LINE Developers 登録&設定
2.Heroku 登録&設定
3.Pythonで実装
4.再度LINE Developers 設定
5.HerokuへデプロイLINE Developers 登録&設定
下記のリンク先でLINE Developersのアカウントとプロバイダー、チャンネルを作成する。
https://developers.line.biz/ja/services/messaging-api/アカウント作成
アカウントは名前とメールアドレスで作成可能です。
プロバイダー作成
プロバイダー名(ご自分の名前や企業名)で作成
チャンネル作成
・チャンネルタイプ → Messaging API
・プロバイダー
・チャンネル名
・チャンネル説明
・カテゴリー
・サブカテゴリ
・電子メールアドレス
利用規約に同意して作成する。友達登録
チャネル基本設定→メッセージAPIのQRコードで友達登録しておく。
必要情報の確認
基本設定→チャネルシークレット確認
メッセージAPI設定→チャネルアクセストークン発行、確認Heroku 登録&設定
Herokuは簡単に言えばアプリケーションを公開するまでの準備を代行してくれるサービスです。
HerokuとはHerokuインストール
インストール設定等は下記の記事を参考にさせていただきました。
https://uepon.hatenadiary.com/entry/2018/07/27/002843Herokuの設定
GitCMDにてログイン
GitCMDheroku login heroku: Press any key to open up the browser to login or q to exit: Opening browser to https://cli-auth.heroku.com/auth/browser/XXXX Logging in... done Logged in as XXXX@XXXXアプリケーションの登録
GitCMDheroku create 自分のアプリケーション名(以下アプリ名) Creating ● アプリ名... done https://アプリ名.herokuapp.com/ | https://git.heroku.com/アプリ名.git環境変数の設定→参照:環境変数とは
先ほどLINE Developersで確認したチャネルシークレットとチャネルアクセストークンを設定する。GitCMDheroku config:set YOUR_CHANNEL_SECRET="Channel Secretの文字列" --app アプリ名 heroku config:set YOUR_CHANNEL_ACCESS_TOKEN="アクセストークンの文字列" --app アプリ名この設定により
「YOUR_CHANNEL_SECRET」はチャネルシークレット
「YOUR_CHANNEL_ACCESS_TOKEN」はチャネルアクセストークン
としてHeroku内で使用できるようになる。設定の確認
GitCMDheroku config --app アプリ名Pythonで実装
ライブラリのインストール
GitCMDにて以下の内容を入力する。
GitCMDpip3 install flask pip3 install line-bot-sdk・Flask(フラスコ/フラスク)はPythonのWebアプリケーションフレームワークで、小規模向けの簡単なWebアプリケーションを作るのに適したものです。→参考:Flaskとは
・line-bot-sdkはLINEBOTを作るために必要な機能が入っています。
→参考:line-bot-sdkとは実際のコードは以下のサイトのmain.pyを参考にさせていただきました。
https://uepon.hatenadiary.com/entry/2018/07/27/002843
また、書かれている内容の把握の為下記のサイトを参考にさせていただきました。
https://www.wantedly.com/companies/casley/post_articles/139107main.py# 必要モジュールの読み込み from flask import Flask, request, abort import os from linebot import ( LineBotApi, WebhookHandler ) from linebot.exceptions import ( InvalidSignatureError ) from linebot.models import ( MessageEvent, TextMessage, TextSendMessage, ) # 変数appにFlaskを代入。インスタンス化 app = Flask(__name__) #環境変数取得 YOUR_CHANNEL_ACCESS_TOKEN = os.environ["YOUR_CHANNEL_ACCESS_TOKEN"] YOUR_CHANNEL_SECRET = os.environ["YOUR_CHANNEL_SECRET"] line_bot_api = LineBotApi(YOUR_CHANNEL_ACCESS_TOKEN) handler = WebhookHandler(YOUR_CHANNEL_SECRET) # Herokuログイン接続確認のためのメソッド # Herokuにログインすると「hello world」とブラウザに表示される @app.route("/") def hello_world(): return "hello world!" # ユーザーからメッセージが送信された際、LINE Message APIからこちらのメソッドが呼び出される。 @app.route("/callback", methods=['POST']) def callback(): # リクエストヘッダーから署名検証のための値を取得 signature = request.headers['X-Line-Signature'] # リクエストボディを取得 body = request.get_data(as_text=True) app.logger.info("Request body: " + body) # 署名を検証し、問題なければhandleに定義されている関数を呼び出す。 try: handler.handle(body, signature) # 署名検証で失敗した場合、例外を出す。 except InvalidSignatureError: abort(400) # handleの処理を終えればOK return 'OK' # LINEでMessageEvent(普通のメッセージを送信された場合)が起こった場合に、 # def以下の関数を実行します。 # reply_messageの第一引数のevent.reply_tokenは、イベントの応答に用いるトークンです。 # 第二引数には、linebot.modelsに定義されている返信用のTextSendMessageオブジェクトを渡しています。 @handler.add(MessageEvent, message=TextMessage) def handle_message(event): line_bot_api.reply_message( event.reply_token, TextSendMessage(text=event.message.text)) # ポート番号の設定 if __name__ == "__main__": # app.run() port = int(os.getenv("PORT")) app.run(host="0.0.0.0", port=port)Webhookの設定
再度LINE Message APIへ戻り、WebhookのWebhook URLにHerokuの接続先を設定する
Webhook URL:https://アプリ名.herokuapp.com/callback
※最後にcallbackメソッドの記入を忘れない事設定ファイルの作成とデプロイ
参照:デプロイとは
Herokuに設置するファイル(先ほどのPythonソースコード「main.py」も)などを作成する。
ファイル作成の為Python等のバージョンを確認GitCMDpython --versionGitCMDpip freezeデプロイするディレクトリの作成(今回はフォルダー名をlinebotにしました)
ディレクトリ内のファイル
main.py →ソースコード
runtime.txt →Pythonのバージョンを記載
requirements.txt →インストールするライブラリの記載
Procfile →プログラムの実行方法を定義runtime.txtPython 3.9.0requirements.txtFlask==1.1.2 line-bot-sdk==1.17.0Procfile
web: python main.py
※Procfileはコマンドプロンプト等で「echo web: python main.py > Procfile」と入力し作成。Gitを用いてHerokuへ設置
GitCMDcd linebot git init git add . git commit -am "make it better" git push heroku master「cd」で作成したディレクトリに移動、それ以降の4行でHerokuに設置しています。
参照:Gitとはデプロイできたか確認
heroku openhello Worldが表示されたら正常にデプロイされています。
ログ確認
heroku logs --tail上記のコマンドでログが確認できます。
詰まったところ
LINE Message APIのチャネルアクセストークンのコピーの際、翻訳した状態でコピペをしてしまい署名検証で引っ掛かりオウム返しされなかったが翻訳を解除してコピペしたところ期待値が返ってきた。
参考文献
https://qiita.com/shimajiri/items/cf7ccf69d184fdb2fb26
https://www.casleyconsulting.co.jp/blog/engineer/3028/
https://www.sejuku.net/blog/7858
https://uepon.hatenadiary.com/entry/2018/07/27/002843
https://www.wantedly.com/companies/casley/post_articles/139107
- 投稿日:2020-11-24T15:47:07+09:00
pythonのみで2次元配列の転置を行う方法[備忘録]
やりたいこと
pythonの二次元配列を転置してさらにint をstrに変換する
pandas やnd.array は使わないで行いたいexample.txt[ [5, 0, 1, 1], [1, 0, 1, 5], [0, 1, 6, 0], [0, 4, 3, 0], [5, 2, 0, 0], [5, 0, 1, 1], [0, 6, 0, 1], [0, 1, 0, 6] ] => [ ['5', '1', '0', '0', '5', '5', '0', '0'], ['0', '0', '1', '4', '2', '0', '6', '1'], ['1', '1', '6', '3', '0', '1', '0', '0'], ['1', '5', '0', '0', '0', '1', '1', '6'] ]code
hoge.pycnt_ACGT_t = [list(map(str,i)) for i in zip(*cnt_ACGT)]転置
複数のイテラブルを受け取り、同じ場所の要素でまとめたイテレータを返す組み込み関数であるzipを用いる
zip(*cnt_ACGT)でcnt_ACGTのリストを転置したものがイテレータオブジェクトに変換された
全要素のintからstrの変換
zip(*cnt_ACGT)はイテレータなので、そのままリスト内包表記を使って各要素をstrに変換する
1行ごとのリストにたいしてmap でstrに変換する参考
https://note.nkmk.me/python-list-transpose/ おなじみnkmkさんのブログを参考にしました
- 投稿日:2020-11-24T15:29:21+09:00
普通のツイートをAWS LambdaとPythonでフリートっぽくする
フリートとは?
Fleetは、Twitterの活用や、Twitterでのコミュニケーションをこれまでにない方法ですぐに行える機能を提供します。Fleetで共有した内容は、24時間が経過すると表示されなくなるため、ふとした思いつきやうつろう気持ちを共有できます。一時的な個人的考えをフォロワーと共有でき、他の一般ユーザーからの反応が伝わることもありません。Fleetの作成者は、自分のFleetをクリックし、下部に表示される既読テキストをタップすると、ツイートを非公開にしているアカウントを含め、誰が自分のFleetを閲覧したかがわかります。
- 24時間が経過すると表示されなくなる
- つまり、24時間に1回まとめて削除すればいい
フリートの欠点
- コメント機能が無く、DMで通知される
- 絵文字を送っても、DMで通知される
- 普通のツイートと同じく、FavとRTとリプライ機能はやはり欲しい
ツイートをフリート化するには?
- 「残すツイート」と「消えるツイート」を明確化する
- 残すツイートの条件
- Favがある
- RTがある
- リプライがある
- 消すツイートの条件
- 残すツイート以外のすべて
- 上記の条件に則り、24時間に1回Lambdaを発火させ、ツイートを削除する
前提条件
- TwitterのAPIキーを発行済み
- 英作文はググればいくらでも出てくるから頑張る
- AWSのアカウントを作成済み
- AWS CLIを使えるようにしておく
構築
フォルダ構造
DeleteNoRtFavReplyTweets | lambda.yml | README.md | \---code DeleteNoRtFavReplyTweets.pyCloudformation
AAWSTemplateFormatVersion: '2010-09-09' # Twitter API Key Parameters: ConsumerKey: Type: String NoEcho: true ConsumerSecret: Type: String NoEcho: true AccessKey: Type: String NoEcho: true AccessSecret: Type: String NoEcho: true Resources: # Lambda Lambda: Type: 'AWS::Lambda::Function' Properties: Code: code Environment: Variables: ConsumerKeyVar: !Ref ConsumerKey ConsumerSecretVar: !Ref ConsumerSecret AccessKeyVar: !Ref AccessKey AccessSecretVar: !Ref AccessSecret Description: NoRtFavReplyTweetDelete FunctionName: NoRtFavReplyTweetDelete Handler: noRtFavTweetDelete.lambda_handler MemorySize: 128 Role: !Sub - arn:aws:iam::${AWS::AccountId}:role/${Domain} - { Domain: !Ref LambdaRole } Runtime: python3.8 Timeout: 180 # CloudWatchEvents Rule Rule: Type: 'AWS::Events::Rule' Properties: Description: NoRtFavReplyTweetDeleteRule Name: NoRtFavReplyTweetDeleteRule ScheduleExpression: 'cron(0 17 * * ? *)' #夜間2時 State: ENABLED Targets: - Arn: !GetAtt Lambda.Arn Id: lambda # Lambda IAM Role LambdaRole: Type: 'AWS::IAM::Role' Properties: RoleName: deleteTweetRole AssumeRolePolicyDocument: Version: '2012-10-17' Statement: - Effect: Allow Principal: Service: - lambda.amazonaws.com Action: - 'sts:AssumeRole' Path: / ManagedPolicyArns: - arn:aws:iam::aws:policy/service-role/AWSLambdaBasicExecutionRole # CloudWatchEvents Lambda excute enable LambdaEvent: Type: 'AWS::Lambda::Permission' Properties: Action: 'lambda:InvokeFunction' FunctionName: !Ref Lambda Principal: 'events.amazonaws.com' SourceArn: !GetAtt Rule.ArnPython
import tweepy import os # API Key consumer_key = os.environ['ConsumerKeyVar'] consumer_secret = os.environ['ConsumerSecretVar'] access_key = os.environ['AccessKeyVar'] access_secret = os.environ['AccessSecretVar'] # Tweepy Auth auth = tweepy.OAuthHandler(consumer_key, consumer_secret) auth.set_access_token(access_key, access_secret) api = tweepy.API(auth, wait_on_rate_limit = True) noFavRtTweet = [] mentionTweet = [] noDeleteTweet = [] def lambda_handler(event, context): # No Favorite No RT Tweet print("==========No Favorite No RT Tweet==========") for tweet in tweepy.Cursor(api.user_timeline,exclude_replies = True).items(): if tweet.favorite_count == 0 and tweet.retweet_count == 0: #Fav数に応じて変更する print(tweet.id,tweet.created_at,tweet.text.replace('\n','')) noFavRtTweet.append(tweet.id) # Reply Tweet print("==========Reply Tweet==========") for mentions in tweepy.Cursor(api.mentions_timeline).items(): print(mentions.id,mentions.created_at,mentions.text.replace('\n','')) mentionTweet.append(mentions.in_reply_to_status_id) print("==========No Favorite No RT Reply Tweet==========") # No Favorite No RT Reply Tweet for tweet in noFavRtTweet: for reptw in mentionTweet: if tweet == reptw: print(api.get_status(tweet).id,api.get_status(tweet).created_at,api.get_status(tweet).text.replace('\n','')) noDeleteTweet.append(tweet) # Extraction Delete Tweet print("==========Extraction Delete tweet==========") perfectList = set(noFavRtTweet) ^ set(noDeleteTweet) print(list(perfectList)) # Delete Tweet print("==========delete tweet==========") for deltw in perfectList: print(api.get_status(deltw).id,api.get_status(deltw).created_at,api.get_status(deltw).text) api.destroy_status(deltw)AWSへのデプロイ
- Tweepyは外部モジュールなので同梱する
pip install tweepy --target ./code
- パッケージにしてS3に転送
aws cloudformation package --s3-bucket $YourBucketName ` --template-file lambda.yml ` --output-template-file lambda-packaged.yml
- インフラ構築とデプロイ
- ここでTwitterのAPI KEYをLambdaの環境変数に指定する
aws cloudformation deploy ` --template-file lambda-packaged.yml ` --stack-name $YourStackName ` --parameter-overrides AccessSecret=$YourAccessSecret ` ConsumerKey=$YourConsumerKey ` ConsumerSecret=$YourConsumerSecret ` AccessKey=$YourAccessKey ` --capabilities CAPABILITY_NAMED_IAM
- マネコンから確認する
GitHubにあげた完全版
https://github.com/trysaildaisuki/DeleteNoReactionReply
真実
- この記事の副産物である
- ふぁぼ爆に対しての対策は自分のツイートのFav数から設定すると良い
- 投稿日:2020-11-24T14:32:53+09:00
機械学習 〜リッジ回帰〜について理解をする。
はじめに
初めまして。m0rio0818です。機械学習の勉強を最近はじました。
まだ手探り状態ですが、ここに忘備録として、残していきたいと思います。SNS全般苦手で、初めても続かなかったのですが、頑張って投稿を続けていきいです。
独学で勉強をしており、誤りなどあると思います。ご指摘していただければ、嬉しいです。最小2乗法
まず、最小2乗法について、軽く触れたいと思います。
機械学習の回帰分析では最小2乗法という理論を用いて、計算式が作られます。
最小2乗法のイメージとしては、プロットをとり、各データの中間を通る直線を引きます。
その直線式を y = ax + b とします。とりあえず、イメージをするために、適当に直線を引いてみました。
下にイメージ図を載せます。この予測と実際には、ズレが生じています。
この直線と各点との誤差を上図の e1, e2, e3, e4, e5 とします。
その誤差を2乗し、5で割ります。E = \frac{e1^2 + e2^2 + e3^2 + e4^2 + e5^2}{5}この誤差Eが小さければ小さいほど、良い回帰直線と言えます。
また、最小2乗法には、予測式(回帰式)を作る際、a,bには制約を設けていません。リッジ回帰
次にリッジ回帰についてです。リッジ回帰は先ほど述べた、最小2乗法を少し変えたものです。
具体的には、
・最小2乗法は 誤差Eのみについて取り扱った。・リッジ回帰では、誤差Eに加えて、係数の2乗を足したものFを考えます。Fのことを「正則化項」とも呼びます。
F = {a^2 + b^2}そして、 L = E + F として、Lが最小になるような係数aとbを算出します。
実際には、 L = E + 0.2 × F のように、正則化項を定数倍にすることで、Fの影響度を調整することができる。リッジ回帰は、正則化された線形回帰の一つで、線形回帰に「学習した重みの2乗を加えたもの」である。
また、リッジ回帰によって過学習を防ぐことができる。
最後に
以下の参考文献を活用しました。
リッジ回帰がどのようなものか少し理解できました。
次回はリッジ回帰の実装を行っていきたいと思います。参考文献
書籍
スッキリわかるpythonによる機械学習
- 投稿日:2020-11-24T14:29:03+09:00
pythonで文字列をdatetime型に変換する際のtimezone指定
datetimeとtimezone
文字列からdatetime型にし、それをelasticsearchに登録するときに、
timezoneを指定しないとおかしなことになりました。
その際に行ったことの備忘録です。もっと楽なやり方あったら教えていただきたいです。python3.7より前の場合
- 文字列のフォーマット:
YYYY-mm-dd HH:MM:SS+0900
+0900を末尾に追加 (UTC+9時間なので )- strptime()のフォーマット:
%Y-%m-%d %H:%M:%S%z
%zがtimezoneに対応している例from datetime import datetime as dt # 文字列 time_str = "2020-11-22 18:00:00+0900" # timezoneを含んだdatetime型 time_dt = dt.strptime(time_str, '%Y-%m-%d %H:%M:%S%z') print(time_dt.tzinfo) # UTC+09:00ptyhon3.7の場合
fromisoformat()を利用できます。
ISO 8601形式では末尾に+HH:MMまたは-HH:MMの形式でタイムゾーン情報を含んでいるので、
それに準ずる形の文字列を引数とします。# 文字列 time_str = '2020-09-12 12:22:30+09:00' # timezoneを含んだdatetime型 time_dt = dt.fromisoformat(time_str) print(time_dt.tzinfo) # UTC+09:00参考サイト
- 投稿日:2020-11-24T13:34:56+09:00
Python(matplotlib)でunixtimeの値の入った時系列データのCSVをプロットする
↓このようなCSVファイルのデータをプロットしたいとする。
$ cat 1.csv "_time",count 1604847600,0 1604848200,2 1604848800,0 1604849400,2 1604850000,0 1604850600,3 1604851200,3 1604851800,0 1604852400,3 1604853000,11604847600は,UNIXTIMEという表記法で、実際は次のような意味になる。
2020-11-09 00:00:00,0 2020-11-09 00:10:00,2 2020-11-09 00:20:00,0これを変換するプログラムを考える。。。
1from datetime import datetime 2 3import matplotlib.pyplot as plt 4import matplotlib.dates as md 5import dateutil 6 7import sys 8args = sys.argv 9 10x = [] 11y = [] 12 13f = open(args[1], 'r') 14 15line = f.readline() 16line = f.readline() 17 18while line: 19 tmp = line.split(",") 20 dt_object = datetime.fromtimestamp(int(tmp[0])) 21 22 x.append(str(dt_object)) 23 y.append(int(str(str(tmp[1]).replace('\n','')))) 24 25 line = f.readline() 26f.close() 27 28counter = 0 29for i in x: 30 print(x[counter] + "," + str(y[counter])) 31 counter = counter + 1https://github.com/RuoAndo/qiita/blob/master/python-timeseries-plot/1.py
10,11行目でタイムスタンプと値を格納するリストを作る。
10 x = [] 11 y = []20行目でUNIXTIMEを日付に変換する。
20 dt_object = datetime.fromtimestamp(int(tmp[0]))実行してみる。。。
$ python 1.py 1.csv | more 2020-11-09 00:00:00,0 2020-11-09 00:10:00,2 2020-11-09 00:20:00,0 2020-11-09 00:30:00,2 2020-11-09 00:40:00,0 2020-11-09 00:50:00,3 2020-11-09 01:00:00,3 2020-11-09 01:10:00,0 2020-11-09 01:20:00,3 2020-11-09 01:30:00,1 2020-11-09 01:40:00,0 2020-11-09 01:50:00,1 2020-11-09 02:00:00,2 2020-11-09 02:10:00,2(`ー´)b
あとはこれをプロットするプログラムを作る。
コードを見てみる。。
1from datetime import datetime 2 3import matplotlib.pyplot as plt 4import matplotlib.dates as md 5import dateutil 6 7x = [] 8y = [] 9 10f = open('1.csv', 'r') 11 12line = f.readline() 13line = f.readline() 14 15while line: 16 tmp = line.split(",") 17 dt_object = datetime.fromtimestamp(int(tmp[0])) 18 19 x.append(str(dt_object)) 20 y.append(int(str(str(tmp[1]).replace('\n','')))) 21 22 line = f.readline() 23f.close() 24 25counter = 0 26for i in x: 27 print(x[counter] + "," + str(y[counter])) 28 counter = counter + 1 29 30dates = [dateutil.parser.parse(s) for s in x] 31 32plt.subplots_adjust(bottom=0.2) 33plt.xticks( rotation= 80 ) 34ax=plt.gca() 35xfmt = md.DateFormatter('%Y-%m-%d %H:%M') 36ax.xaxis.set_major_formatter(xfmt) 37plt.plot(dates,y) 38plt.show()https://github.com/RuoAndo/qiita/blob/master/python-timeseries-plot/5.py
30行目から38行目でリストx(タイムスタンプ),y(値)をプロットする。
30dates = [dateutil.parser.parse(s) for s in x] 31 32plt.subplots_adjust(bottom=0.2) 33plt.xticks( rotation= 80 ) 34ax=plt.gca() 35xfmt = md.DateFormatter('%Y-%m-%d %H:%M') 36ax.xaxis.set_major_formatter(xfmt) 37plt.plot(dates,y) 38plt.show()(`ー´)b
- 投稿日:2020-11-24T13:14:49+09:00
「ゼロから作るDeep Learning」自習メモ(その19)Data Augmentation
「ゼロから作るDeep Learning」(斎藤 康毅 著 オライリー・ジャパン刊)を読んでいる時に、参照したサイト等をメモしていきます。 その18 ←
犬猫判定はそこそこできるようになりましたが、正解率が90%に満たないまま、というのもナンだかなあ、という感じなので、本のP245に書いてある Data Augmentation をやってみようかと思います。
Data Augmentation
データの拡張で簡単にできそうなのが
反転
回転
移動
あたりでしょうか。で、GradCAMで見た感じ、猫は背中を丸めた姿勢に反応、犬は鼻に反応しているようです。
ということは、
猫の画像を回転、反転したものを追加すれば、猫の識別精度があがるのではないか?
犬の画像の顔のあたりを拡大したら、犬の識別精度があがるのではないか?
なんてことが考えられます。そこで、拡張データを追加すると、どう学習を強化するのか、検証してみたいと思います。
まず、訓練データとテストデータを作り直します
ここまで使っていたデータは、プログラムの動作を確認できればいいからと、相当いいかげんに作っています。テストデータ件数も100件だけしかありません。
テストデータをランダムに1000件くらいはとっておきたい。
残りの訓練データについては、犬と猫の画像をマージしたあと、ランダムに並べ直す。
拡張データは、犬猫加工方法ごとに別に分けておき、学習の際に訓練データにマージして使い、効果を検証できるようにする。
検証では、全体の正解率だけでなく、犬の正解率、猫の正解率も検証する。
さらに、不正解の画像についてGradCAMでどこの特徴に反応しているかを確認する。というような方針で、訓練データを作り直しました。
def rnd_list(motoarray, toridasi): # 0~元のnp配列のデータ件数までの整数リストを作成し # ランダムに並べ替えてから、 # 指定した件数の整数のリストと残りの整数リストを返す import random import numpy as np kensuu , tate, yoko, channel = motoarray.shape moto = list(range(0, kensuu)) random.shuffle(moto) sel = moto[0:toridasi] nokori=moto[toridasi:] return sel, nokori def bunkatu(motoarray, toridasi, lblA): # np配列のデータを # 指定した件数のリストと残りのリストに # 分割する sel, nokori = rnd_list(motoarray, toridasi) tsl = [] tsi = [] trl = [] tri = [] for i in sel: imgA = dogimg[i] tsl.append(lblA) tsi.append(imgA) for i in nokori: imgA = dogimg[i] trl.append(lblA) tri.append(imgA) return tsl, tsi, trl, tri def rnd_arry(tri, trl): # 画像の配列とラベルの配列を # ランダムに並べ替えた # リストにして返す sel, nokori = rnd_list(tri, 0) wtri = [] wtrl = [] for i in nokori: imgA = tri[i] lblA = trl[i] wtri.append(imgA) wtrl.append(lblA) return wtri, wtrl # 訓練用とテスト用に分割し、犬と猫を統合する bunkatusuu = 500 ctsl, ctsi, ctrl, ctri = bunkatu(catimg, bunkatusuu, 0) dtsl, dtsi, dtrl, dtri = bunkatu(dogimg, bunkatusuu, 1) tri=np.append(ctri, dtri, axis=0) trl=np.append(ctrl, dtrl, axis=0) tsi=np.append(ctsi, dtsi, axis=0) tsl=np.append(ctsl, dtsl, axis=0) # ランダムに並べ替えて wtri, wtrl = rnd_arry(tri, trl) wtsi, wtsl = rnd_arry(tsi, tsl) # 保存する dataset = {} dataset['test_label'] = np.array(wtsl, dtype=np.uint8) dataset['test_img'] = np.array(wtsi, dtype=np.uint8) dataset['train_label'] = np.array(wtrl, dtype=np.uint8) dataset['train_img'] = np.array(wtri, dtype=np.uint8) import pickle save_file = '/content/drive/My Drive/Colab Notebooks/deep_learning/dataset/catdog.pkl' with open(save_file, 'wb') as f: pickle.dump(dataset, f, -1)訓練データ23994件(犬11997、猫11997)、テストデータ1000件(犬500、猫500)
ができました。これを入力にして、その18で作ったDeepConvNetで処理すると
Epoch 1/10
188/188 [==============================] - 373s 2s/step - loss: 0.7213 - accuracy: 0.5663
Epoch 2/10
188/188 [==============================] - 373s 2s/step - loss: 0.6378 - accuracy: 0.6290
Epoch 3/10
188/188 [==============================] - 373s 2s/step - loss: 0.5898 - accuracy: 0.6713
Epoch 4/10
188/188 [==============================] - 374s 2s/step - loss: 0.5682 - accuracy: 0.6904
Epoch 5/10
188/188 [==============================] - 373s 2s/step - loss: 0.5269 - accuracy: 0.7128
Epoch 6/10
188/188 [==============================] - 374s 2s/step - loss: 0.4972 - accuracy: 0.7300
Epoch 7/10
188/188 [==============================] - 372s 2s/step - loss: 0.4713 - accuracy: 0.7473
Epoch 8/10
188/188 [==============================] - 374s 2s/step - loss: 0.4446 - accuracy: 0.7617
Epoch 9/10
188/188 [==============================] - 373s 2s/step - loss: 0.4318 - accuracy: 0.7665
Epoch 10/10
188/188 [==============================] - 376s 2s/step - loss: 0.4149 - accuracy: 0.7755
32/32 - 4s - loss: 0.3811 - accuracy: 0.8420正解率84.2% という結果に。
結果の内容を検証してみます
predictions = model.predict(x_test) # 誤判定分の添え字をリストにする gohantei = [] kensuu, w = predictions.shape for i in range(kensuu): predictions_array = predictions[i] predicted_label = np.argmax(predictions_array) true_label = t_test[i] if predicted_label != true_label: gohantei.append(i) print(len(gohantei))158
158件の誤判定がありました。
def plot_image(i, predictions, t_label, img): class_names = ['cat', 'dog'] predictions_array = predictions[i] img = img[i].reshape((80, 80, 3)) true_label = t_label[i] plt.grid(False) plt.xticks([]) plt.yticks([]) plt.imshow(img, cmap=plt.cm.binary) predicted_label = np.argmax(predictions_array) if predicted_label == true_label: color = 'blue' else: color = 'red' plt.xlabel("{} {:2.0f}% ({})".format(class_names[predicted_label], 100*np.max(predictions_array), class_names[true_label]), color=color) num_cols = 10 num_rows = int(len(gohantei) / num_cols ) + 1 plt.figure(figsize=(2*num_cols, 2.5*num_rows)) j = 0 for i in gohantei: inuneko = t_test[i] if inuneko == 0: plt.subplot(num_rows, num_cols, j+1) plot_image(i, predictions, t_test, x_test) j +=1 plt.show() print("猫を間違えた ",j) plt.figure(figsize=(2*num_cols, 2.5*num_rows)) j = 0 for i in gohantei: inuneko = t_test[i] if inuneko == 1: plt.subplot(num_rows, num_cols, j+1) plot_image(i, predictions, t_test, x_test) j +=1 plt.show() print("犬を間違えた ",j)誤判定の内訳は 猫109件 犬49件 でした。
猫が犬の倍以上誤判定になっています。そもそも、拡張データを追加することで学習が強化されるのか?
猫のデータを拡張して追加することで、猫の誤判定が減るかを確認してみます。
#猫画像のみ取り出す catdatalist = [] kensuu = len(dataset['train_img']) for i in range(kensuu): label = dataset['train_label'][i] if label == 0: catdatalist.append(i) print(len(catdatalist))11997
#猫の左右反転画像データセットを作成する trl = [] tri = [] lbl = 0 for i in catdatalist: img = dataset['train_img'][i] img = img[:, ::-1, :] trl.append(lbl) tri.append(img) catdataset = {} catdataset['train_label'] = np.array(trl, dtype=np.uint8) catdataset['train_img'] = np.array(tri, dtype=np.uint8) tri =np.append(dataset['train_img'], catdataset['train_img'], axis=0) trl =np.append(dataset['train_label'], catdataset['train_label'], axis=0) x_train = tri / 255.0 t_train = trl反転した猫データを追加した訓練データで学習させます。
model.fit(x_train, t_train, epochs=10, batch_size=128)Epoch 1/10
282/282 [==============================] - 571s 2s/step - loss: 0.6604 - accuracy: 0.6783
Epoch 2/10
282/282 [==============================] - 569s 2s/step - loss: 0.5840 - accuracy: 0.7220
Epoch 3/10
282/282 [==============================] - 570s 2s/step - loss: 0.5407 - accuracy: 0.7511
Epoch 4/10
282/282 [==============================] - 572s 2s/step - loss: 0.5076 - accuracy: 0.7689
Epoch 5/10
282/282 [==============================] - 565s 2s/step - loss: 0.4808 - accuracy: 0.7860
Epoch 6/10
282/282 [==============================] - 566s 2s/step - loss: 0.4599 - accuracy: 0.7974
Epoch 7/10
282/282 [==============================] - 563s 2s/step - loss: 0.4337 - accuracy: 0.8115
Epoch 8/10
282/282 [==============================] - 565s 2s/step - loss: 0.4137 - accuracy: 0.8181
Epoch 9/10
282/282 [==============================] - 564s 2s/step - loss: 0.3966 - accuracy: 0.8256
Epoch 10/10
282/282 [==============================] - 565s 2s/step - loss: 0.3759 - accuracy: 0.8331test_loss, test_acc = model.evaluate(x_test, t_test, verbose=2)32/32 - 4s - loss: 0.3959 - accuracy: 0.8220
predictions = model.predict(x_test) # 誤判定分の添え字をリストにする gohantei = [] kensuu, w = predictions.shape for i in range(kensuu): predictions_array = predictions[i] predicted_label = np.argmax(predictions_array) true_label = t_test[i] if predicted_label != true_label: gohantei.append(i) print(len(gohantei))178
def plot_image(i, predictions, t_label, img): class_names = ['cat', 'dog'] predictions_array = predictions[i] img = img[i].reshape((80, 80, 3)) true_label = t_label[i] plt.grid(False) plt.xticks([]) plt.yticks([]) plt.imshow(img, cmap=plt.cm.binary) predicted_label = np.argmax(predictions_array) if predicted_label == true_label: color = 'blue' else: color = 'red' plt.xlabel("{} {:2.0f}% ({})".format(class_names[predicted_label], 100*np.max(predictions_array), class_names[true_label]), color=color) num_cols = 10 num_rows = int(len(gohantei) / num_cols ) + 1 plt.figure(figsize=(2*num_cols, 2.5*num_rows)) j = 0 for i in gohantei: inuneko = t_test[i] if inuneko == 0: plt.subplot(num_rows, num_cols, j+1) plot_image(i, predictions, t_test, x_test) j +=1 plt.show() print("猫を間違えた ",j) plt.figure(figsize=(2*num_cols, 2.5*num_rows)) j = 0 for i in gohantei: inuneko = t_test[i] if inuneko == 1: plt.subplot(num_rows, num_cols, j+1) plot_image(i, predictions, t_test, x_test) j +=1 plt.show() print("犬を間違えた ",j)猫を間違えた 28
犬を間違えた 150反転データを入れてないときは
間違い 158件 猫を間違えた109件 犬を間違えた49件 でしたから
猫についてだけは、大きく精度が上がっています。
しかし、その分、犬の精度が下がり、全体でも精度が落ちてしまいました。データを追加することで、学習は強化されるようですが、副作用も大きいということでしょうか。
間違いから正解に変わった猫の画像と、正解から間違いに変わった犬画像をしらべてみた
間違いから正解に変わった猫画像 赤いマークをしているのは、2回目も間違いだったもの
正解から間違いに変わった犬画像 黄色マークは1回目も間違いだったもの
犬っぽいお座りをしている猫画像が正しく判定されるようになったけれど、ふつうにお座りしている犬画像まで「猫」と判定されるようになった感じ?
また、1回目では鼻だけで「犬」と判定していただろう、顔の大写しの画像が「猫」に判定されています。うーむ。よーわからん。
では、犬の反転画像も追加して学習したらどうなるか? やってみました。
猫と犬の両方の反転画像を追加して学習してみた
つまり、訓練データ件数が倍になるということ。
結果は
32/32 - 4s - loss: 0.2186 - accuracy: 0.9090
正解率90%に向上。
誤判定91件 内、猫を犬と間違えた48件 犬を猫と間違えた43件反転データを入れてないときは
間違い 158件 猫を間違えた109件 犬を間違えた49件 でしたから
猫を間違えた件数が半分になっています。犬っぽいお座りをしている画像と、顔が大写しの画像が、間違いから正解に変わっているようです。
以上のことから、次のことが言えるかと。
・左右反転させた訓練データで、件数を倍に水増ししても、学習に使えるし、有効であること。
・今回の例のように2つに分類する場合、訓練データの件数は犬猫同じ件数にそろえたほうが偏りのない学習になること。ただ、訓練データ件数が倍になったら、さすがのGoogle Colab もRAMがいっぱいになってクラッシュしてしまいます。
ということで、これ以上データを増やすのは難しそうなので、犬猫データの判別は、このへんで終わりにします。その18 ←
- 投稿日:2020-11-24T11:18:08+09:00
他の人が作ってくれたCloud9でPythonのバージョンが古い時の対応方法
- 環境 : Cloud9
- 同僚がEC2を利用したCloud9を使っていたので、私も使ってみたくてそのCloud9にアカウントを作ってもらって早速使い始めた。
事象 : Pythonのバージョンが古かった
# Pythonが3じゃない! $ python --version Python 2.7.18 # pipは・・・なぜかPython3.6のもの? $ pip --version pip 20.2.4 from /home/ec2-user/.local/lib/python3.6/site-packages/pip (python 3.6) # Python3はあるようだ・・・ $ python3 --version Python 3.6.12Cloud9のPythonを3にすればよいのか?と思ったらもうなっている・・・
# 「+」がPython3にくっついている・・・けどとりあえず選択してみる $ sudo update-alternatives --config python There are 3 programs which provide 'python'. Selection Command ----------------------------------------------- * 1 /usr/bin/python2.7 + 2 /usr/bin/python3.6 3 /usr/bin/python2.6 Enter to keep the current selection[+], or type selection number: 2 # Python3にならない・・・ $ python -V Python 2.7.18原因 :
.bashrcで古いバージョンを指定しているから# 自分のアカウントのPythonが2になっている・・・ $ alias | grep python alias python='python27' # aliasでPython2.7が設定されている・・・ $ cat ~/.bashrc | grep python alias python=python27対応 :
.bashrcで指定するPythonのバージョンを変える# .bashrcじゃなくても読込順序が後のファイルならOK $ ls -la # ...省略... -rw-r--r-- 1 ec2-user ec2-user 336 Jul 24 2019 .bash_profile -rw-r--r-- 1 ec2-user ec2-user 1401 Nov 20 06:48 .bashrc -rw-rw-r-- 1 ec2-user ec2-user 118 Jul 24 2019 .zshrc # viで開いてaliasでPython3.6を指定して $ vi ~/.bashrc $ cat ~/.bashrc | grep python alias python=python36 # 反映すると $ source ~/.bashrc function # Python3.6になった・・・ $ python --version Python 3.6.12
- 投稿日:2020-11-24T10:58:45+09:00
Unable to import module 'lambda_function': No module namedとなった時の対応方法
- 環境
- Cloud9
- 同僚がEC2を利用したCloud9を使っていたので、私も使ってみたくてそのCloud9にアカウントを作ってもらって早速使い始めた。半端ない初心者。
- Python 3.6.12
事象 :
import requestsをして実行したらモジュールがなくて怒られたResponse { "errorType": "Runtime.ImportModuleError", "errorMessage": "Unable to import module 'lambda_function': No module named 'requests'" }がーん!でも
pip listはrequestsがあるとおっしゃっている。ponsuke:~/environment (asynchronous) $ pip list | grep requests requests 2.24.0原因 : 対象のLambda関数にモジュールがインストールされていないから
lambda_function.pyのディレクトリをみればモジュールがないのが分かる。
Cloud9を使ってLambda関数を作るときはpip listを見りゃいいというわけではないらしい。モジュールのインストール先が間違っていたことがエラーの原因でした.
Cloud9 は Lambda アプリケーション を利用しているため, アプリケーションディレクトリ(今回はDemoApplication/)が Lambda ファンクションの実行ディレクトリになります.
したがって, import requests で requests モジュールを読み込みたい場合はアプリケーションディレクトリ直下にモジュールをインストールする必要がありました.
Cloud9 上で Lambda を実行する際に “Unable to import module : No module named” エラーが出た場合の対処 | Developers.IO# Lambda関数のディレクトリへ移動して $ cd lambda_name/ # 中身を見ると・・・モジュールは何にもなかった・・・ ponsuke:~/environment/lambda_name (asynchronous) $ ls -la total 36 drwxr-xr-x 2 ec2-user ec2-user 4096 Nov 24 01:06 . drwxr-xr-x 32 ec2-user ec2-user 4096 Nov 20 02:35 .. -rw-r--r-- 1 ec2-user ec2-user 197 Nov 20 02:35 .application.json -rw-r--r-- 1 ec2-user ec2-user 1082 Nov 20 09:04 lambda_function.py -rw-r--r-- 1 ec2-user ec2-user 10690 Nov 24 01:06 lambda-payloads.json -rw-r--r-- 1 ec2-user ec2-user 512 Nov 20 02:35 templap対応 : 場所を指定してモジュールをインストールする
-t, --target
Install packages into . By default this will not replace existing files/folders in . Use --upgrade to replace existing packages in with new versions.
pip install — pip 9.1.0.dev0 ドキュメントponsuke:~/environment/lambda_name (asynchronous) $ pip install --target . requests Collecting requests Downloading requests-2.25.0-py2.py3-none-any.whl (61 kB) |████████████████████████████████| 61 kB 7.1 MB/s Collecting chardet<4,>=3.0.2 Using cached chardet-3.0.4-py2.py3-none-any.whl (133 kB) Collecting urllib3<1.27,>=1.21.1 Downloading urllib3-1.26.2-py2.py3-none-any.whl (136 kB) |████████████████████████████████| 136 kB 15.9 MB/s Collecting certifi>=2017.4.17 Downloading certifi-2020.11.8-py2.py3-none-any.whl (155 kB) |████████████████████████████████| 155 kB 18.3 MB/s Collecting idna<3,>=2.5 Using cached idna-2.10-py2.py3-none-any.whl (58 kB) Installing collected packages: chardet, urllib3, certifi, idna, requests Successfully installed certifi-2020.11.8 chardet-3.0.4 idna-2.10 requests-2.25.0 urllib3-1.26.2だめな対応 : 場所を指定しないでモジュールをインストールする
場所を指定しないでインストールしても、もうあるものはもうある。
# ぷ?もうある・・・権限がない? ponsuke:~/environment/lambda_name (asynchronous) $ pip install requests Defaulting to user installation because normal site-packages is not writeable Requirement already satisfied: requests in /home/ec2-user/.local/lib/python3.6/site-packages (2.24.0) Requirement already satisfied: idna<3,>=2.5 in /home/ec2-user/.local/lib/python3.6/site-packages (from requests) (2.10) Requirement already satisfied: chardet<4,>=3.0.2 in /home/ec2-user/.local/lib/python3.6/site-packages (from requests) (3.0.4) Requirement already satisfied: urllib3!=1.25.0,!=1.25.1,<1.26,>=1.21.1 in /home/ec2-user/.local/lib/python3.6/site-packages (from requests) (1.25.10) Requirement already satisfied: certifi>=2017.4.17 in /home/ec2-user/.local/lib/python3.6/site-packages (from requests) (2020.6.20) # --userオプションでDefaulting to user...は消えたけど状況変わらず ponsuke:~/environment/lambda_name (asynchronous) $ pip install --user requests Requirement already satisfied: requests in /home/ec2-user/.local/lib/python3.6/site-packages (2.24.0) Requirement already satisfied: idna<3,>=2.5 in /home/ec2-user/.local/lib/python3.6/site-packages (from requests) (2.10) Requirement already satisfied: chardet<4,>=3.0.2 in /home/ec2-user/.local/lib/python3.6/site-packages (from requests) (3.0.4) Requirement already satisfied: urllib3!=1.25.0,!=1.25.1,<1.26,>=1.21.1 in /home/ec2-user/.local/lib/python3.6/site-packages (from requests) (1.25.10) Requirement already satisfied: certifi>=2017.4.17 in /home/ec2-user/.local/lib/python3.6/site-packages (from requests) (2020.6.20) # もしかして、Python2になってるとか?と思ってみたけど何も変わらず・・ ponsuke:~/environment/lambda_name (asynchronous) $ python3 -m pip install --user requests Requirement already satisfied: requests in /home/ec2-user/.local/lib/python3.6/site-packages (2.24.0) Requirement already satisfied: idna<3,>=2.5 in /home/ec2-user/.local/lib/python3.6/site-packages (from requests) (2.10) Requirement already satisfied: chardet<4,>=3.0.2 in /home/ec2-user/.local/lib/python3.6/site-packages (from requests) (3.0.4) Requirement already satisfied: urllib3!=1.25.0,!=1.25.1,<1.26,>=1.21.1 in /home/ec2-user/.local/lib/python3.6/site-packages (from requests) (1.25.10) Requirement already satisfied: certifi>=2017.4.17 in /home/ec2-user/.local/lib/python3.6/site-packages (from requests) (2020.6.20)
- 投稿日:2020-11-24T10:20:26+09:00
決定木(load_iris)
■ はじめに
今回は、決定木の実装~プロットをまとめていきます。
【対象とする読者の方】
・決定木における、基礎のコードを学びたい方
・理論は詳しく分からないが、実装を見てイメージをつけたい方 など
1. モジュールの用意
最初に、必要なモジュールをインポートしておきます。
import numpy as np import pandas as pd import matplotlib.pyplot as plt from sklearn.datasets import load_iris from sklearn.model_selection import train_test_split from sklearn.tree import DecisionTreeClassifier from sklearn.tree import plot_tree
2. データの準備
load_iris データセットを使用します。
iris = load_iris() X, y = iris.data[:, [0, 2]], iris.target print(X.shape) print(y.shape) # (150, 2) # (150,)trainとtestデータに分割します。
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size = 0.3, random_state = 123) print(X_train.shape) print(y_train.shape) print(X_test.shape) print(y_train.shape) # (105, 2) # (105,) # (45, 2) # (45,)決定木では個々の特徴量は独立に処理され、データの分割はスケールに依存しないため
正規化や標準化は不要となります。
3. データの可視化
モデリングする前に、データをプロットして見ておきます。
fig, ax = plt.subplots() ax.scatter(X_train[y_train == 0, 0], X_train[y_train == 0, 1], marker = 'o', label = 'Setosa') ax.scatter(X_train[y_train == 1, 0], X_train[y_train == 1, 1], marker = 'x', label = 'Versicolor') ax.scatter(X_train[y_train == 2, 0], X_train[y_train == 2, 1], marker = 'x', label = 'Varginica') ax.set_xlabel('Sepal Length') ax.set_ylabel('Petal Length') ax.legend(loc = 'best') plt.show()4. モデルの作成
決定木のモデルを作成します。
tree = DecisionTreeClassifier(max_depth = 3) tree.fit(X_train, y_train) ''' DecisionTreeClassifier(ccp_alpha=0.0, class_weight=None, criterion='gini', max_depth=3, max_features=None, max_leaf_nodes=None, min_impurity_decrease=0.0, min_impurity_split=None, min_samples_leaf=1, min_samples_split=2, min_weight_fraction_leaf=0.0, presort='deprecated', random_state=None, splitter='best') '''併せて、可視化もしておきます。
fig, ax = plt.subplots(figsize=(10, 10)) plot_tree(tree, feature_names=iris.feature_names, filled=True) plt.show()5. 予測値の出力
テストデータに対する予測を行います。
y_pred = tree.predict(X_test) print(y_pred[:10]) print(y_test[:10]) # [2 2 2 1 0 1 1 0 0 1] # [1 2 2 1 0 2 1 0 0 1]0:Setosa 1:Versicolor 2:Verginica
6. 性能評価
今回の分類予測における、正解率を求めます。
print('{:.3f}'.format(tree.score(X_test, y_test))) # 0.956
■ 最後に
今回は上記1~6の手順をもとに、決定木のモデル作成・評価を行いました。
初学者の方にとって、少しでもお役に立てたようでしたら幸いです。
■ 参考文献
- 投稿日:2020-11-24T09:41:18+09:00
pythonで英単語アプリを作成
仕様
- 入力した単語を翻訳サイトからスクレイピングし、翻訳結果を表示する
- 翻訳と同時にに音声ファイルも出力する
- 音声ファイルを単語名.mp3で保存する
ライブラリ
使用したライブラリは以下の通りです。
ライブラリ 用途 os パスの確認 requests htmlの入手、mp3のダウンロード bs4 htmlの解析 tkinter GUIの作成 pygame mp3の再生 random 例外処理 コード
import os import random import tkinter import requests from bs4 import BeautifulSoup from pygame import mixer class Translation: def __init__(self): self.root = tkinter.Tk() self.root.title('Translation') self.root.geometry('500x200') self.root.attributes('-topmost', True) self.text_box = tkinter.Entry(width=20, font=('', 20), justify='center') self.text_box.focus_set() self.text_box.pack() self.root.bind('<Return>', self.scraping) self.root.bind('<space>', self.delete) self.answer = tkinter.Message(self.root, text='', font=('', 20), width=450) self.answer.pack(anchor='center', expand=1) self.root.mainloop() def scraping(self, event): #スクレイピング try: res = requests.get('https://ejje.weblio.jp/content/' + self.text_box.get()) soup = BeautifulSoup(res.content, 'html.parser') txt = soup.find('td', {'class': 'content-explanation ej'}) self.answer['text'] = txt.text self.sound(soup) except: self.answer['text'] = random.choice(('(^^;)?', '(・ω・)?', "('Д')?", '(; ・`д・´)?')) def sound(self, soup): #mp3をダウンロードし再生する mp3_directory_path = 'd:/python/Application/mp3/' #mp3の保存先 if not os.path.exists(mp3_directory_path): os.mkdir(mp3_directory_path) if os.path.exists(mp3_directory_path + self.text_box.get() + '.mp3'): pass else: audio = soup.find('audio', {'class': 'contentAudio'}) src = audio.find('source')['src'] res = requests.get(src, stream=True) with open(mp3_directory_path + self.text_box.get() + '.mp3', 'wb') as f: f.write(res.content) mixer.init() mixer.music.load(mp3_directory_path + self.text_box.get() + '.mp3') mixer.music.play() def delete(self, event): #スペースキーで入力欄の文字を全て削除する self.text_box.delete(0, tkinter.END) Translation()実行結果
感想
なんとか形にできました。他の方が作ったQiitaの記事がとても参考になりました。あとはデスクトップにbatファイルを作れば気軽に立ち上げることができると思います。また、ダウンロードしたmp3ファイルから単語のリスニングテストなんかも作れるかもしれません。
- 投稿日:2020-11-24T09:12:43+09:00
【個人開発】DjangoRESTFrameworkとVue-CliでSPAでアーティスト向けのアプリを作成した話【DRF+Vue.js】
はじめに
こんにちは。
2年目ペーペー新人エンジニアです。
今回は、個人開発したアプリをついにリリースしたので、
それを記事にしようとおもいます。作成物
アプリ:https://paintmonitor.com
今回作成したのは、『PaintMonitor』というアプリです。
名前も当初かなり迷走して、結局わかりやすく、
Paint(描いて)Monitor(見える化)にしました。
アプリの特徴としては、
描いて(Paint)、解析して(Monitor)、共有する(SNS)。の3点です。システム構成
システム構成は、一番使い慣れているUbuntuServerで、
開発言語はDjangoのDjangoRESTFrameworkを利用しました。
DjangoRESTFrameworkは、とても使いやすく、直感的に理解しやすかったです。
認証回りの構築は、『現場で使えるDjangoRESTFramework』の本を参考にしました。
このDjangoの現場で使えるシリーズは、本当に現場で使えそうな内容が豊富なのでおすすめです。フロント周りは、最近仕事でもよく使うVue.jsを利用しました。
Vue.jsは、開発コストは低く使いやすいので、
SPAの作成にはとてもよかったです。
Webpackで、利用すればhotタイミングでコードが反映されるので開発スピードを上げてくれました。
開発当初は、jQueryで開発していましたが、ネイティブ展開もしたかったので、
モダンな構築に作り変えました。DBはいつものMySQL。ストレージはAWSを利用しています。
APサーバーはGunicornを利用しています。
シングルパフォーマンスでは、uWSGIのほうが優れていますが、
システムの安定度を重視したい場合は、Gunicornがいいみたいです。
WEBサーバーはいつものNginxでリリース、それをLetsEncriptで暗号化しました。個人開発で作ろうと思ったきっかけ
ある程度WEBができるようになって、何か作りたいなぁと常に何かしらは作っていました。
アートとテクノロジーが好きなので、
それらを合わせてアーティスト向けのアプリを作ろうと思ったのがきっかけでした。
WEBで描いて、練習して、SNSにするサービスっていいなと思い、作り始めました。
最初は、tensorflowを利用して、絵をスコア化して、レート戦の
バトルをメインとするアプリを作ろうと考えたのですが、
普通に絵の練習するアプリのほうが実用的だなと思って、練習をメインとするアプリにしました。
バトルやそのほかの機能は後からのアップデートで追加していければいいかなと。個人開発で苦労したところ
全部ひとりで考えなくてはいけなくて、大変な作業量になる。
単純に作業量が暴力的なので、仕事の合間に開発するには少ししんどかったです。
会社へ行く電車の乗り降りの時間、座れる時間帯に電車にのり、Surfaceを広げてカタカタと作業してました。
でもコードを書くのは好きなので、なんだかんだ充実した日々を過ごせました。デザインや仕様を考えるのが大変。
私は考えるより手を動かす派なので、デザインに関しては、FIGMAやXD等は使わずに、簡単に画面をスケッチしてデザインしました。
私の場合は、好きなWEBサービスのPinterestを参考したりしました。
仕様は、適当に考えて、修正があればその都度修正する、みたいなチーム開発なら怒られるであろう進め方をしていました。こだわりすぎて時間がかかる
私自身よく絵を描くので、ペイント部分はかなりこだわって作りました。
WEBでありながら実用的なペイントツールを作るのに4,5回新規でペイントアプリを作り、
開発の7割はこのペイントツールに費やしたせいで、開発に半年以上かかりました。最後に
個人開発は大変でしたが、
個人のポートフォリオにもなるし、なによりめちゃくちゃ勉強になります。
開発力をつける一番の方法は、アプリを作ってリリースするのがいいのでは?
と感じました。
長々と話してしまいましたが、
作成したPaintMonitor、
昨日の夜にリリースしたばかりなので、まだ利用者が全然いません。
ぜひ触ってあそんでもらえたらうれしいです。
今後もアップデート頑張っていきたいと思います。
- 投稿日:2020-11-24T02:16:44+09:00
Pythonで医用画像解析1(SimpleITKでMRI画像を読み込む)
はじめに
DICOMを読み込むライブラリはいくつかありますが,この記事ではSimpleITKを使ってMRIやCTなどの医用画像を読み込む方法を解説します.
SimpleITKとは
SimpleITKはITKという医用画像解析のための形式をPythonやほかの言語で容易に利用するためのライブラリです.(https://en.wikipedia.org/wiki/SimpleITK)
3D SlicerやImageJ,ITK-Snapなどのソフトウェアでも利用されています.医用画像の形式
例えばMRI画像はDICOMデータとして利用することが多く,SimpleITKではDICOMを直接読み込むことができます.また,DICOM画像はしばしばNIFTI(.nii, .nii.gz)やNRRD形式(.nrrd)として保存されることがありますが同様に読み込めます.(https://simpleitk.readthedocs.io/en/master/IO.html)
ただし,出力に関してはDICOM形式では出力できないと思われます.目的
SimpleITK形式で読み込まれた画像を用いて表示やndarray形式に変換でき,処理や解析を行えます.
このライブラリを用いた目的は,PyRadiomicsという別の画像解析用ライブラリ上でSimpleITK形式で画像を読み込む必要があったためです.自分の学習記録としてもせっかくなのでまとめました.環境
- Windows 10
- Python 3.7
- Jupyter Notebook(Anaconda)
Google Collaboratoryでも同様に行えますがローカルにあるDICOMデータなどをいちいちアップロードするのは面倒なので環境構築しました.
SimpleITKのインストール
conda install -c simpleitk simpleitkpipでは普通にインストール可能です.
画像の読み込み
DICOM画像が複数枚同じフォルダーに保存されている場合を想定しています.
import SimpleITK as sitk import numpy as np import matplotlib.pyplot as plt #Dicomの読み込み imgdir = "DICOMが存在するディレクトリ名" sys.argv[1] = imgdir reader = sitk.ImageSeriesReader() dicom_names = reader.GetGDCMSeriesFileNames(sys.argv[1]) reader.SetFileNames(dicom_names) image = reader.Execute() #ITK形式で読み込まれた画像 #NIFTIやNRRDを読み込む場合 image = sitk.ReadImage("画像のディレクトリ") #画像のサイズ取得 size = pre.GetSize() print("Image size:", size[0], size[1], size[2]) #ITK形式をndarray形式に変換 ndImage = sitk.GetArrayFromImage(image) #画像を表示 plt.imshow(ndImage[n], cmap='gray') #nは任意のスライスについて表示まとめ&読み込んだ画像の使い道
上記のコードではサイズのチェックと簡単な表示を行いました.
画像を読み込んだあとの使い道は様々です.
例えば,3次元のndarray形式になっているのでprintで任意のボクセルの信号値を見ることもできます.
画像の出力やどんな解析ができるかどうかは別の記事でまとめたいと思います.
- 投稿日:2020-11-24T01:58:47+09:00
macOS に最適化された TensorFlow を Apple M1 チップ 環境で import するまでの Tips
概要
- "Mac-optimized TensorFlow and TensorFlow Addons" (https://github.com/apple/tensorflow_macos) を MacBook Air with M1 で動かすための試行錯誤
- Intel アーキテクチャの MacBook Pro (Early 2015) から環境を移行した上で、 ARM ベース アーキテクチャに合わせるための方法の模索
- (Mac-optimized) TensorFlow の実行結果は含まず、ひとまず import するところまで
背景
日本時間の 2020/11/11 03:00 から行われた Apple Event で詳細が公開された、Apple の新型チップ「M1」。
発表の中で、具体的なソフトウェアの名称として「TensorFlow」の名前が挙がっていたので気になっていたのですが、その後、Apple の GitHub Organization から "Mac-optimized TensorFlow and TensorFlow Addons" (https://github.com/apple/tensorflow_macos) が公開されました。プレリリースのドライバーで、Apple のML Computeframework を使っている、とのことです。ちょうど MacBook Pro (Early 2015) からのリプレースを考えていたため、M1 チップの載った MacBook Air を購入し、いろいろと動作を試してみることにしました。1
tensorflow_macosを動かす上で、Intel アーキテクチャの Mac から ARM ベースのアーキテクチャの Mac への移行で引っかかるところがいくつかありましたので、記録として残せればと思います。2申し遅れましたが、私は @forest1988 と申します。
Qiita への投稿は今回が初めてとなります。至らない部分が多々あるかと思いますが、ご笑覧いただき、その上で少しでもお役に立つ部分があれば嬉しく思います。試行錯誤の過程を書いているために無駄に遠回りになっている部分や、私の勘違いから誤謬が含まれている可能性があります。
お気付きの点がありましたら、ご指摘をいただけますと大変幸いです。環境
- MacBook Air
- Apple M1 チップ (8コアCPU, 8コアGPU)
- macOS 11.0.1 (Big Sur)
- MacBook Pro (Early 2015) から「移行アシスタント」によりデータ移行済み
環境を移行したことで、初期状態の macOS 11 (Big Sur) とは変わっているところがあります(たとえば、terminal の shell が zsh ではなく、macOS Catalina 以前から使っている bash のままになっているなど)。
そのため、データの移行などをせずにまっさらな状態で Big Sur with M1 を使われる方や、Windows マシンなどから移行される方、そもそも作っていた環境がまるで違う方とは、
同様に実行しても異なる結果になる可能性が十分にあることを予めお断りしておきます。まず試して失敗したこと
MacBook Pro で作っていた python の環境がそのままコピーできていたため、まずはそれで動かすことを試みました。
具体的には、pyenv で anaconda3-2020.07 を入れていた状態です。 Python の version は 3.8.3 でした。リンクを辿り、 https://github.com/apple/tensorflow_macos/releases にある
tensorflow_macos-0.1alpha0.tar.gzをダウンロードしてきます。GitHub のリポジトリにあるのはダウンロードとインストール用のスクリプトですが、このプレリリースの圧縮ファイルの中には(スクリプトでダウンロードできる)最適化された TensorFlow などが含まれているため、331.6MBほどのサイズになっています。
まずは、Python 3.8 の venv を使って
tensorflow_macos用の仮想環境を作ります。pyenv の anaconda にさらに venv を重ねるとおかしくならないだろうか……という不安はあったのですが、とりあえず試してみることに(後述するように、それ以前の問題だったわけですが)。
$ /bin/bash install_venv.sh --prompt --python /path/to/anaconda3-2020.7/bin/pythonを実行し、途中のプロンプトはデフォルト設定で進めました。3
プロンプトで指定した場所にtensorflow_macos_venvディレクトリが作成され、ここにある/bin/activateを使うことで作られた仮想環境に入ることができます。4$ . /path/to/tensorflow_macos_venv/bin/activate (tensorflow_macos_venv) (base) primary_prompt_string$ python Python 3.8.3 (default, Jul 2 2020, 11:26:31) [Clang 10.0.0 ] :: Anaconda, Inc. on darwin Type "help", "copyright", "credits" or "license" for more information.tensorflow_macos のリポジトリ の README.md には、CPU と GPU を選択するためのコードスニペットが書かれているので、まずはこれを試してみることにしました。
# Import mlcompute module to use the optional set_mlc_device API for device selection with ML Compute. from tensorflow.python.compiler.mlcompute import mlcomputeこれを、先ほど対話モードで立ち上げた python で試してみます。
>>> # Import mlcompute module to use the optional set_mlc_device API for device selection with ML Compute. >>> from tensorflow.python.compiler.mlcompute import mlcompute Traceback (most recent call last): File "/path/to/tensorflow_macos_venv/lib/python3.8/site-packages/tensorflow/python/pywrap_tensorflow.py", line 64, in <module> from tensorflow.python._pywrap_tensorflow_internal import * ImportError: dlopen(/path/to/tensorflow_macos_venv/lib/python3.8/site-packages/tensorflow/python/_pywrap_tensorflow_internal.so, 6): no suitable image found. Did find: /path/to/tensorflow_macos_venv/lib/python3.8/site-packages/tensorflow/python/_pywrap_tensorflow_internal.so: mach-o, but wrong architecture /path/to/tensorflow_macos_venv/lib/python3.8/site-packages/tensorflow/python/_pywrap_tensorflow_internal.so: mach-o, but wrong architecture During handling of the above exception, another exception occurred: Traceback (most recent call last): File "<stdin>", line 1, in <module> File "/path/to/tensorflow_macos_venv/lib/python3.8/site-packages/tensorflow/__init__.py", line 41, in <module> from tensorflow.python.tools import module_util as _module_util File "/path/to/tensorflow_macos_venv/lib/python3.8/site-packages/tensorflow/python/__init__.py", line 39, in <module> from tensorflow.python import pywrap_tensorflow as _pywrap_tensorflow File "/path/to/tensorflow_macos_venv/lib/python3.8/site-packages/tensorflow/python/pywrap_tensorflow.py", line 83, in <module> raise ImportError(msg) ImportError: Traceback (most recent call last): File "/path/to/tensorflow_macos_venv/lib/python3.8/site-packages/tensorflow/python/pywrap_tensorflow.py", line 64, in <module> from tensorflow.python._pywrap_tensorflow_internal import * ImportError: dlopen(/path/to/tensorflow_macos_venv/lib/python3.8/site-packages/tensorflow/python/_pywrap_tensorflow_internal.so, 6): no suitable image found. Did find: /path/to/tensorflow_macos_venv/lib/python3.8/site-packages/tensorflow/python/_pywrap_tensorflow_internal.so: mach-o, but wrong architecture /path/to/tensorflow_macos_venv/lib/python3.8/site-packages/tensorflow/python/_pywrap_tensorflow_internal.so: mach-o, but wrong architecture Failed to load the native TensorFlow runtime. See https://www.tensorflow.org/install/errors for some common reasons and solutions. Include the entire stack trace above this error message when asking for help.それならばと思い、もう一方のインストール方法を試してみることにしました。
リポジトリの README.md の INSTALLATION の項目 を見て、
次のスクリプトを動かしました。/bin/bash -c "$(curl -fsSL https://raw.githubusercontent.com/apple/tensorflow_macos/master/scripts/download_and_install.sh)"やはり同様のエラーが起き、ここで改めてエラーメッセージをしっかり確認して、
wrong architectureというところに目が留まりました。使っていた anaconda が Intel のアーキテクチャである x86-64 対応のものであったのが原因なのではないか、と考え、 Python の環境を整え直すことにしました。
Homebrew での Python のインストール(結果的に回り道でした)
まずは、
pyenvやanacondaに関する PATH の設定を~/.bash_profileから削除し、 terminal を再起動しました。これで、移行元のマシンからコピーした pyenv, anaconda を参照しなくなるはずです。その上で、既に入っていた Homebrew で Python の入れ直しを試したところ、以下のエラーに遭遇しました。
Error: Cannot install in Homebrew on ARM processor in Intel default prefix (/usr/local)! Please create a new installation in /opt/homebrew using one of the "Alternative Installs" from: https://docs.brew.sh/Installation You can migrate your previously installed formula list with: brew bundle dump"Alternative Installs" を読み、以下のように Homebrew を入れ直します。
$ mkdir homebrew && curl -L https://github.com/Homebrew/brew/tarball/master | tar xz --strip 1 -C homebrew $ mv homebrew/ /opt/
~/.bashrcに以下を追記して、export PATH=/opt/homebrew/bin/:$PATH
sourceを使って反映します。$ source ~/.bashrc次に、
brew bundle dumpで install 済みの package の list を作ります。5$ /usr/local/Homebrew/bin/brew bundle dumpこの結果、
Brewfileとして以下のようなファイルが生成されました。tap "cartr/qt4" tap "homebrew/bundle" ... <略> ... tap "sanemat/font" brew "automake" brew "python@3.9" brew "python@3.8" ... <略> ... brew "sanemat/font/ricty"これを以下のように反映します。
$ brew tap Homebrew/bundle $ touch Brewfile $ brew bundle --file Brewfileしかし、ここで以下のような warning が発生し、いくつかの package の install に失敗しました。
Warning: You are running macOS on a arm64 CPU architecture. We do not provide support for this (yet). Reinstall Homebrew under Rosetta 2 until we support it. You will encounter build failures with some formulae. Please create pull requests instead of asking for help on Homebrew's GitHub, Twitter or any other official channels. You are responsible for resolving any issues you experience while you are running this unsupported configuration.Python を入れることはできていましたが、先と同様の問題を生じそうだったため、これはアンインストールして、次に進みます。
System の Python を試す
REQUIREMENTS に Xcode Command Line Tools の使用について書かれていたので、
$ xcode-select --installで Xcode Command Line Tools を入れました。 6
これで Python の問題が解決する……かと思いきや、環境を移行した関係か、なぜか Python 3.9 が動いてしまいます。
これでは、Python 3.8 を要件とするtensorflow_macosは動かない……と焦ったのですが、これには二つの原因がありました。
- Homebrew でのアンインストールに失敗していた。
- 環境移行のためか、Python 3.9 が
/usr/local/bin/python3に残っていた。どうやら、原因の一つは、Homebrew で install した Python がうまくアンインストールできなかったことのようです。
which pythonで確認したところ、/opt/homebrew/bin//python3が参照されていました。 7
実行した際に表示される日付などから見ても、上述のインストール時のものと思われます。再度アンインストールを試みると、
$ brew uninstall python3 Error: Refusing to uninstall /opt/homebrew/Cellar/python@3.9/3.9.0_2 because it is required by itstool, libxml2 and sphinx-doc, which are currently installed. You can override this and force removal with: brew uninstall --ignore-dependencies python3と Error が出るので、この指示に従ってアンインストールを行います。
$ brew uninstall --ignore-dependencies python3 Uninstalling /opt/homebrew/Cellar/python@3.9/3.9.0_2... (9,224 files, 137.5MB)これで、
which python3が/usr/local/bin/python3を指してくれるようになりました。しかし、これも Python 3.9.0 になっています。
$ /usr/local/bin/python3 Python 3.9.0 (default, Nov 4 2020, 22:18:28) [Clang 12.0.0 (clang-1200.0.32.21)] on darwin Type "help", "copyright", "credits" or "license" for more information. >>>ここで、 Homebrew の warning を思い返してみると、
Error: Cannot install in Homebrew on ARM processor in Intel default prefix (/usr/local)!と書いてありました。
これまでは特に詳しく考えずに使っていたのですが、/usr/localが Intel の default prefix であり、 ARM processor に対応していないということは、
この/usr/local/binにある python はどこから来たものなのでしょうか?おそらく、環境移行をする前に Homebrew で入れていた Python が残っているのではないか、と思われます。
そこで、移行前に入れてあった Homebrew を使ってアンインストールを試みます。$ /usr/local/Homebrew/bin/brew uninstall python3先と同様の依存関係のエラーが出るので、
--ignore-dependenciesを付けて再度実行します。$ /usr/local/Homebrew/bin/brew uninstall --ignore-dependencies python3今回はアンインストールに成功し、
which python3が/usr/bin/python3を指してくれるようになりました。$ which python3 /usr/bin/python3 $ python3 Python 3.8.2 (default, Oct 2 2020, 10:45:41) [Clang 12.0.0 (clang-1200.0.32.27)] on darwin Type "help", "copyright", "credits" or "license" for more information. >>>これで、ようやく Python 環境の準備が整ったはずです!
再度、README に従って実行
$ /bin/bash install_venv.sh --prompt --python=/usr/bin/python3何の問題もなく、すっきりとインストールできました。
$ . "/path/to/tensorflow_macos_venv/bin/activate" (tensorflow_macos_venv) primary_prompt_string$ which python /path/to/tensorflow_macos_venv/bin/python (tensorflow_macos_venv) primary_prompt_string$ python Python 3.8.2 (default, Oct 2 2020, 10:45:41) [Clang 12.0.0 (clang-1200.0.32.27)] on darwin Type "help", "copyright", "credits" or "license" for more information. >>> # Import mlcompute module to use the optional set_mlc_device API for device selection with ML Compute. >>> from tensorflow.python.compiler.mlcompute import mlcompute >>>今回はエラーなく import ができました!
では、 CPU と GPU の使用についてはどうでしょうか。>>> # Select CPU device. >>> mlcompute.set_mlc_device(device_name='cpu') >>> # Select GPU device. >>> mlcompute.set_mlc_device(device_name='gpu') WARNING:tensorflow:Eager mode on GPU is extremely slow. Consider to use CPU insteadGPU については Warning が出ていますが、ひとまず CPU と GPU の指定は可能だと考えて良さそうです。
Eager mode on GPU is extremely slowとのことで、 TensorFlow 2.0 で default になっている Eager mode (Define-by-Run) ではなく、TensorFlow 1系の Define-and-Run での実行が前提になっているのでしょうか。改めてリポジトリの README.md を見ると
Please note that in eager mode, ML Compute will use the CPU.と書かれていますので、上記では Warning と共に強制的に CPU に切り替わっている可能性があります。"Mac-optimized TensorFlow and TensorFlow Addons" の真価を試すには、 Eager mode ではないコードを用意する必要がありそうです。
まとめ
"Mac-optimized TensorFlow and TensorFlow Addons" (https://github.com/apple/tensorflow_macos) を MacBook Air with M1 で動かすことを目標として、
Intel プロセッサ の Mac から M1 搭載の Mac にデータ移行をした場合の Python 環境構築で引っ掛かった部分についてまとめてみました。詰まりどころとしては、移行元の環境で作っていた Python の環境を消去、ないしは参照しないように設定し直す必要があるところでしょうか。
アーキテクチャが違うので、考えてみれば当たり前のことなのですが、移行元でせっかく作っていた環境をなるべく利用できないか……と考えてしまったこともあり、思いのほか手こずってしまいました。Homebrew が現時点で ARM ベース のアーキテクチャに対応していないということもありましたが、
We do not provide support for this (yet).という書き方をしていることからしても、これについては将来的に解消されるものではないかと思います。
その他のソフトウェアについても M1 対応が進んでいくものと思われますので、Intel プロセッサ搭載 Mac から M1 搭載 Mac への移行はどんどん楽になっていくものと期待されます。
この記事については、あくまで2020年11月24日現在の状況での試みを記録したものであり、近いうちに(もしかしたら今日のうちにでも)、状況の変化する可能性があります。そもそも
tensorflow_macosもプレリリースのものですし、正式リリース版がどのようなものになるのかを楽しみにしたいと思います。(以上)
2020/11/11 (JST) に発表・予約開始された、というのが印象的で、購入に踏み切ったというのもあります。ゲーム・アニメ等が苦手ではない方で、お手すきのお時間がありましたら「アイドルマスター 趣味 プログラミング 誕生日」でご検索ください。今回は「開発」というほどのことはしておりませんが、こうした点で「アイマス駆動開発」(参考: https://www.slideshare.net/treby/imas-driven-developemnt ) の影響を受けたものと言えるかもしれません。 ↩
なお、 Intel アーキテクチャの Mac での動作については Qiita 記事「appleがmac用tensorflowを出したようなのでintelのMacBookairにインストールしてみる」(https://qiita.com/notfolder/items/b27cc00bd77eb1587832) があり、参考にさせていただきました。お礼申し上げます。 ↩
macOS Catalina からは zsh がデフォルトになっているはずなので、敢えて bash を使っているのに何か理由があるのかは興味があるところです。 ↩
$PS1の設定で表示できるホスト名やユーザー名などはprimary_prompt_stringに、 tensorflow_macos_venv を置いた場所は/path/to/で置き換えています。ご自身の環境に合わせて読み替えていただければと存じます。特に前者について、以下の記事を参考にさせていただきました。お礼申し上げます:「[Mac] ターミナルの$前の出力内容をカスタマイズする」(https://www.yoheim.net/blog.php?q=20140309) , 「プロンプトの確認や設定 - Pocketstudio.jp Linux Wiki」(http://pocketstudio.jp/linux/?%A5%D7%A5%ED%A5%F3%A5%D7%A5%C8%A4%CE%B3%CE%C7%A7%A4%E4%C0%DF%C4%EA) ↩既に入っていた Homebrew で行う必要があるので、新しい Homebrew を入れる前に実行したほうが、フルパス指定をする面倒がなくて良かったかと思います。 ↩
Xcode Command Line Tools のインストールについては Qiita 記事:「MacでXcodeと一緒にpython3をインストールする」(https://qiita.com/todotani/items/73877deea8773c316694) を参考にさせていただきました。お礼申し上げます。 ↩
"//" になっているのは誤記ではなく、実際にそのように表示されていました。
export PATH=/opt/homebrew/bin/:$PATHの際に、binの後に/を付けたのが良くなかったのだと思われます。 ↩
- 投稿日:2020-11-24T00:48:42+09:00
カスタムオブジェクトの JSON エンコード/デコード
概要
カスタムクラスのインスタンス (オブジェクト) を含む複雑な構造を JSON エンコード/デコードする例について説明します。
そういう複雑なデータをシリアライズするには
pickleを使うのが楽で良いのですが、Python 以外で読み書きしたい場合や、シリアライズしてもある程度の可読性がほしい場合はJSONを選ぶことが多いです。ここで説明する方法以外にもやり方はあると思いますが、以下の点において、この方法が気に入っています。
- Python 標準の json モジュールを使う。
- エンコード/デコードのロジックを、クラスごとに分けられる。
カスタムで複雑なデータの例
説明に使うものなので、そこまで複雑にはしませんが、以下の条件を満たすデータでやってみます。
- カスタムクラスが複数入っていること。
- カスタムオブジェクトの属性にもカスタムオブジェクトが入っていること。
class Person: def __init__(self, name): self.name = name class Robot: def __init__(self, name, creator=None): self.name = name self.creator = creator alan = Person('Alan') beetle = Robot('Beetle', creator=alan) chappy = Robot('Chappy', creator=beetle)
alanは人間、beetleとchappyはロボットです。
以下、ロボットのデータをリストにして、そのリストをエンコード/デコードしてみたいと思います。robots = [beetle, chappy]エンコード
このリストには
PersonクラスとRobotクラスのオブジェクトが含まれているので、それらについてエンコードをする必要があります。シンプルなエンコード
まずはシンプルな
Personクラスのエンコードをしてみます。エンコードの仕様を決める
カスタムのオブジェクトをエンコードするには、どのようにエンコードするか決めなければなりません (仕様というやつです)。
ここでは、クラス名 と 属性の内容 をそれぞれ name-value ペアにして出力することにします。
上記のalanの場合ですと、以下のような JSON 文字列になる想定です。{"class": "Person", "name": "Alan"}カスタムのエンコーダを作る
標準の json.dumps 関数に
clsパラメタを指定することで、カスタムのエンコーダを使うことができます。
カスタムのエンコーダは、json.JSONEncoder を継承して、defaultメソッドをオーバーライドして作ります。
defaultメソッドの引数にオブジェクトが入ってくるので、json.JSONEncoderで扱える形 (ここではstrのみを含むdict) にして返せば OK です。import json class PersonEncoder(json.JSONEncoder): def default(self, obj): if isinstance(obj, Person): return {'class': 'Person', 'name': obj.name} else: return super().default(obj) print(json.dumps(alan, cls=PersonEncoder)) # 結果: {"class": "Person", "name": "Alan"}複雑なエンコード
次に
Robotクラスのエンコーダを作りますが、これが複雑という訳ではありません。
「概要」で書いたように、エンコードのロジックはクラスごとに分ける からです。class RobotEncoder(json.JSONEncoder): def default(self, obj): if isinstance(obj, Robot): return {'class': 'Robot', 'name': obj.name, 'creator': obj.creator} else: return super().default(obj)ほぼ
PersonEncoderと同じです。
ただし、先ほどPersonEncoderでやったようには行きません。返り値の中のcreatorをjson.JSONEncoderで扱える形にしていないからです。
敢えてそのようにしてロジックを分けて、実際にエンコードする時には2つのエンコーダを合体させて使います。エンコーダを合体させる
エンコーダを合体させるには、多重継承を使って新しいクラスを作ります。
class XEncoder(PersonEncoder, RobotEncoder): pass print(json.dumps(robots, cls=XEncoder)) # 結果: [{"class": "Robot", "name": "Beetle", "creator": {"class": "Person", "name": "Alan"}}, {"class": "Robot", "name": "Chappy", "creator": {"class": "Robot", "name": "Beetle", "creator": {"class": "Person", "name": "Alan"}}}]
結果を整形した場合 (クリックで表示)。
print(json.dumps(robots, cls=XEncoder, indent=4)) # 結果: [ { "class": "Robot", "name": "Beetle", "creator": { "class": "Person", "name": "Alan" } }, { "class": "Robot", "name": "Chappy", "creator": { "class": "Robot", "name": "Beetle", "creator": { "class": "Person", "name": "Alan" } } } ]
json.dumps関数にエンコーダのクラスを1個しか指定できないのでこのような方法になりますが、これでオブジェクトの種類が増えた場合でも拡張が可能です。(補足) 多重継承による動作について
上記の
XEncoderを作ることでなぜ上手く動作するのか、簡単に説明します。Python のクラスの多重継承では、継承順に属性を参照に行きます。
XEncoderのdefaultメソッドを呼び出すと、まず先に継承しているPersonEncoderのdefaultメソッドを見に行きます。
PersonEncoder.defaultメソッドでは、objがPersonオブジェクトなら自分でdictを返し、そうでないならスーパーメソッドを呼ぶようになっています。この場合のスーパーメソッドは、
json.JSONEncoder.defaultではなくRobotEncoder.defaultになります。
これが Python の多重継承の動きです。
RobotEncoder.defaultがスーパーメソッドを呼べば、それ以上は多重継承していないので、本来のスーパークラスであるjson.JSONEncoderへと処理が委ねられます。
defaultメソッドがどのように再帰的に呼ばれるのかは調べていませんが、if文でクラス判定をしている限りは、継承順が逆になっても同じ結果が得られるようです。デコード
json.loads メソッドに
object_hookパラメタを渡すことで、デコード後のオブジェクトにカスタムの処理を加えることができます。シンプルな object_hook の例
まずは
Personクラスのオブジェクトだけをエンコードし、それをデコードする例を見てみます。
object_hookとして渡す関数はデコード済みのオブジェクト (dict等) を受け取るので、'class' の値が 'Person' であるdictだった場合の処理を書きます。def person_hook(obj): if type(obj) == dict and obj.get('class') == 'Person': return Person(obj['name']) else: return obj # JSON 文字列にエンコードする alan_encoded = json.dumps(alan, cls=PersonEncoder) # JSON 文字列からデコードする alan_decoded = json.loads(alan_encoded, object_hook=person_hook) print(alan_decoded.__class__.__name__, vars(alan_decoded)) # 結果: Person {'name': 'Alan'}object_hook を合体させる
次に、
Robotクラスのためのobject_hookを作って、2つを合体させた新しい関数を作ります。def robot_hook(obj): if type(obj) == dict and obj.get('class') == 'Robot': return Robot(obj['name'], creator=obj['creator']) else: return obj def x_hook(obj): return person_hook(robot_hook(obj))合体させた関数
x_hookは以下のようにも書けます。少し長くなりますが、hook を増やしやすいです (上の例とは hook の適用順が変わるが、問題ない)。def x_hook(obj): hooks = [person_hook, robot_hook] for hook in hooks: obj = hook(obj) return objこれを使って、上の方で作ったロボットのリストをエンコード/デコードしてみます。
# JSON 文字列にエンコードする robots_encoded = json.dumps(robots, cls=XEncoder) # JSON 文字列からデコードする robots_decoded = json.loads(robots_encoded, object_hook=x_hook) for robot in robots_decoded: print(robot.__class__.__name__, vars(robot)) # 結果: Robot {'name': 'Beetle', 'creator': <__main__.Person object at 0x0000029A48D34CA0>} Robot {'name': 'Chappy', 'creator': <__main__.Robot object at 0x0000029A48D38100>}エンコードの時と同様に (おそらく内側から再帰的にデコードされるので)、hook を適用する順番を変えても結果は変わりませんでした。
(補足) エンコードも同じ方法でカスタマイズできる
実は、エンコード側も同じように関数を与える形でカスタマイズする事ができます。
逆に、デコード側をデコーダのサブクラスを作る形にしようとすると、もっとややこしくなります。カスタムのエンコードのロジックを合体させる時に、シンプルに多重継承のみで書きたい場合はサブクラスを作る方法を、デコード側のスタイルに合わせたい場合は関数を与える方法を選ぶと良いでしょう。
def person_default(obj): if isinstance(obj, Person): return {'class': 'Person', 'name': obj.name} else: return obj def robot_default(obj): if isinstance(obj, Robot): return {'class': 'Robot', 'name': obj.name, 'creator': obj.creator} else: return obj def x_default(obj): defaults = [person_default, robot_default] for default in defaults: obj = default(obj) return obj print(json.dumps(robots, default=x_default)) # 結果: [{"class": "Robot", "name": "Beetle", "creator": {"class": "Person", "name": "Alan"}}, {"class": "Robot", "name": "Chappy", "creator": {"class": "Robot", "name": "Beetle", "creator": {"class": "Person", "name": "Alan"}}}]課題
デコードの方に少し課題があります。
上記の例でいうと、最初にデコードしたロボット 'Beetle' と、'Chappy' のcreatorである 'Beetle' は、元々は同じオブジェクトでした。
また、それらの 'Beelte' のcreatorである 'Alan' も同じオブジェクトでした。上記のデコード方法では、「名前が同じだからすでに作ったオブジェクトを使い回す」という事はしていないので、完全にはエンコード前の状況を再現していません。
それをしたい場合は、PersonクラスやRobotクラスの方にその仕組みを作って、object_hookからは名前を指定するだけで適切なオブジェクトを受け取れるようにすれば良いでしょう。
- 投稿日:2020-11-24T00:42:27+09:00
[Python] フォームで送信
[Python]フォームで送信
<!DOCTYPE html> <html lang="ja"> <html> <head> <meta charset="utf-8"> </head> <body> <form action="{% url 'index' %}" method="post"> {% csrf_token %} {{form}} <input type="submit" value="click"> </form> </body> </html>action="{% url 'index' %}" method="post"でデータの送信先を指定している
{% csrf_token %}でCSRF対策をしている
{{form}}はフォームの具体的な内容が作成される
- 投稿日:2020-11-24T00:32:18+09:00
LeetCodeに毎日挑戦してみた 21. Merge Two Sorted Lists(Python、Go)
はじめに
無料英単語サイトE-tanを運営中の@ishishowです。
プログラマとしての能力を上げるために毎日leetcodeに取り組み、自分なりの解き方を挙げていきたいと思います。
Leetcodeとは
leetcode.com
ソフトウェア開発職のコーディング面接の練習といえばこれらしいです。
合計1500問以上のコーデイング問題が投稿されていて、実際の面接でも同じ問題が出されることは多いらしいとのことです。Go言語入門+アルゴリズム脳の強化のためにGolangとPythonで解いていこうと思います。(Pythonは弱弱だが経験あり)
7問目(問題21)
21. Merge Two Sorted Lists
- 問題内容(日本語訳)
2つのソートされたリンクリストをマージし、新しいソートされたリストとして返します。新しいリストは、最初の2つのリストのノードをつなぎ合わせて作成する必要があります。
Example 1:
Input: l1 = [1,2,4], l2 = [1,3,4] Output: [1,1,2,3,4,4]Example 2:
Input: l1 = [], l2 = [] Output: []Example 3:
Input: l1 = [], l2 = [0] Output: [0]考え方
- 新規ListNodeを作成
- 作成したListNodeの参照をresという変数に渡す
- ループを回して、l1、l2のvalが小さいほうを進めてcurに代入する
- どちらかが終点まで行くとループが終わるので残りも代入する。
- returnはresにする 最初は0のためその次から
- 解答コード
class Solution(object): def mergeTwoLists(self, l1, l2): cur = ListNode(0) res = cur while l1 and l2: if l1.val < l2.val: cur.next = l1 l1 = l1.next else: cur.next = l2 l2 = l2.next cur = cur.next cur.next = l1 or l2 return res.next
- Goでも書いてみます!
func mergeTwoLists(l1 *ListNode, l2 *ListNode) *ListNode { cur := &ListNode{} res := cur for l1 != nil && l2 != nil { if l1.Val < l2.Val { cur.Next = l1 l1 = l1.Next } else { cur.Next = l2 l2 = l2.Next } cur = cur.Next } if l1 != nil { cur.Next = l1 } else if l2 != nil { cur.Next = l2 } return res.Next }
- 投稿日:2020-11-24T00:16:40+09:00
【Lambda】pythonの外部モジュールをS3経由で取り入れてみた
概要
lambdaを使ってdisny監視botを作る際にlambdaを使って定期実行したかったのでawsのlambdaを使うことにした。
この際に、line-sdk-botをlambdaに取り入れる必要があったのでその方法を記す。※lambdaを初めて触るため間違っているところがあったら指摘ください。
ローカルの設定
まずはdocker-lambdaディレクトリを作成する。
mkdir docker-lambda cd docker-lambdaディレクトリの構造は以下。
docker-lambda
├── Dockerfile
├── deploy.sh
└── requirements.txtそれぞれのファイルは以下のように構成する。
Dockerfileは以下。
DockerfileFROM lambci/lambda:build-python3.7 ENV AWS_DEFAULT_REGION ap-northeast-1 ADD . . CMD pip install -r requirements.txt -t python/lib/python3.7/site-packages/ && \ zip -r linetest.zip ./pythonlambdaではline-bot-sdk、pandas、numpy、requests、boto3を入れたかったので以下のようにrequirements.txtを作成。
requirements.txtline-bot-sdk pandas numpy requests boto3最後に実行用にshファイルは以下のように構成する。
docker build -t lambda_test .
に関しては一度imageを作成してしまったらコメントアウトシてしまって構わない。deploy.sh# 初回のみ コンテナの立ち上げ docker build -t lambda_test . # コンテナの実行 docker run -v "$PWD":/var/task lambda_test # 権限の付与(lambdaで読み込ができるようにする) chmod -R 755 ./*それぞれのファイルが作成し終わったら、ターミナルで以下を実行する。
terminalsh deploy.shoutputadding: python/lib/python3.7/site-packages/urllib3-1.26.2.dist-info/RECORD (deflated 62%) adding: python/lib/python3.7/site-packages/urllib3-1.26.2.dist-info/WHEEL (deflated 14%) adding: python/lib/python3.7/site-packages/urllib3-1.26.2.dist-info/top_level.txt (stored 0%)このように実行が完了すると、pythonというフォルダとlinetest.zipというzipファイルが完成する。
ここでlinetest.zipはpythonをzip化したものである。
作成されたzipファイルをlambdaのレイヤーとして取り入れることで外部モジュールを使用可能になる。lsDockerfile lambda_function.py python deploy.sh linetest.zip requirements.txtレイヤーの作成
awsのコンソールに行き、lambda→レイヤー→レイヤーの作成 で選択する。
名前:好きなレイヤー名(後で何のためのレイヤーかわかるように)
説明:どんなモジュールが入っているかなどの説明を書く
upload:<=10MB→zipファイルをアップロード
>10MB→s3経由でアップロード
ランタイム:今回はpython3.7を選択
ライセンス:何も書かなくて良い今回はzipファイルのサイズが10MBを超えるため、S3を経由してzipファイルをアップロードする。
サービス→s3→バケットを作成
バケット名:何でも良い
リージョン:lambdaにあわせる(今回は東京で統一)
パケット設定:全てブロック
パケットのバージョニング:無効にする
デフォルトの暗号化:無効にする
詳細設定:変更なし↓
バケットの作成バケットが作成できたら、作成したバケットを選択。(今回はpypy-testというパケットを作成)
アップロード→ファイルを追加→先程作成したzipファイルをアップロードアップロードしたzipファイルを選択するとオブジェクトURLを見れるのでこれをコピーしておく。
再びlambdaに戻り、レイヤーを選択。
アップロードのところにオブジェクトURLを貼り付ける。関数の作成
lambda→関数→関数の追加
作り方:一から作成
関数名:適当
ランタイム:python3.7
↓
関数の作成ここでハマったこと
リージョンが東京じゃないとs3が使えないので注意。
初めは北アフリカとかになってたはず。。。関数を選択し、レイヤーの追加を選択。
カスタムレイヤーを選択し、作成したレイヤーを選択し、追加をクリックする。
(ここで作成したレイヤーが表示されない場合、ランタイムがあっていない可能性があるので確認する。)最後にテスト用にAPI Gatewayの設定をする。
トリガーを選択→API Gateway→APIを作成する→HTTP API→セキュリティ:オープン→追加今回はテスト用にnumpyが使用できるか確認する。
以下のようにlambdaのコードを書き換える。テストを選択して、実行が成功することを確認する。
API Gatewayを選択し、APIエンドポイントからurlにジャンプすると、以下のようにsin(1)の結果が帰ってきているのが確認できる。
次回以降これを応用してlinebotを作成する。
参考記事