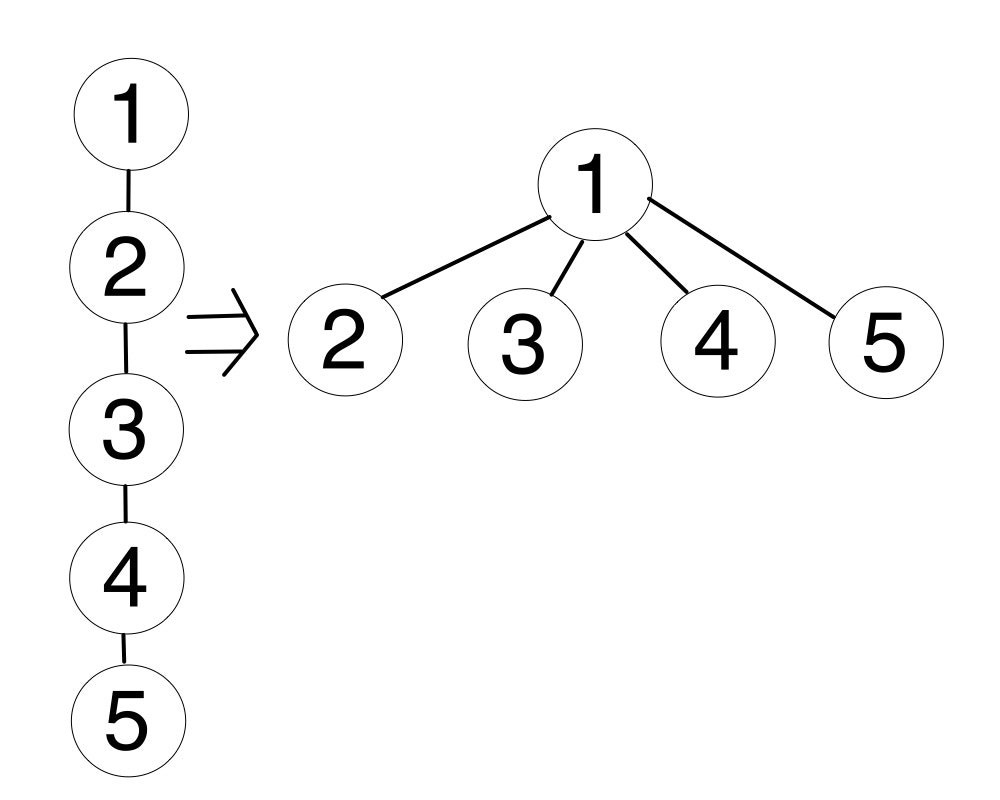
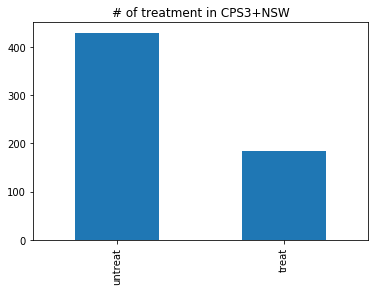- 投稿日:2020-02-27T23:36:26+09:00
エラー解決 python バージョン確認
単純にpythonのバージョン確認しようとしたら
変な内容が出たので書きます。python -V
通常のバージョン確認これは問題なし
最後のVを大文字にしなかっただけでした。
python -v
これだとなぜか
下記のような内容が出てきます。import _frozen_importlib # frozen import _imp # builtin import '_thread' # <class '_frozen_importlib.BuiltinImporter'> import '_warnings' # <class '_frozen_importlib.BuiltinImporter'> import '_weakref' # <class '_frozen_importlib.BuiltinImporter'> # installing zipimport hook import 'zipimport' # <class '_frozen_importlib.BuiltinImporter'> # installed zipimport hook import '_frozen_importlib_external' # <class '_frozen_importlib.FrozenImporter'> import '_io' # <class '_frozen_importlib.BuiltinImporter'> import 'marshal' # <class '_frozen_importlib.BuiltinImporter'> import 'posix' # <class '_frozen_importlib.BuiltinImporter'> import _thread # previously loaded ('_thread') import '_thread' # <class '_frozen_importlib.BuiltinImporter'> import _weakref # previously loaded ('_weakref') import '_weakref' # <class '_frozen_importlib.BuiltinImporter'> # 以下略
- 投稿日:2020-02-27T22:53:36+09:00
Java言語で学ぶデザインパターン入門の2章のまとめ
お久しぶりです。白々です。
前回は、「Iteratorパターン」に関しての記事を作成しました。
前回の記事は、後述のappendixに記載しています。
今回は、「Adapterパターン」に関して記載したいと思います。
また、「Java言語で学ぶデザインパターン入門」には、サンプルプログラムも有りますが、著作権の都合上省かせて頂きます。御了承ください。第2章 Adapter -皮かぶせて再利用
日本で使っている電化製品があると思います。
この電気製品を海外でコンセントを利用する時に、電圧が違うので、コンセントの変換器を持っていくと思います。
このように、すでに供給されている物をそのまま利用できない場合に、使うことができる形に変換することで利用できるようになります。
このような、変換を行うパターンをAdapterパターンと言うそうです。
「Java言語で学ぶデザインパターン入門」には、「すでに提供されているもの」と「必要なもの」との「ずれ」を埋めるためのデザインパターン
と言う記載がありました。
このAdapterパターンは、Wrapperパターンとも呼ぶそうです。なんでこんなパターンが必要なの?
「Java言語で学ぶデザインパターン入門」にも、記載がありましたが、必要なメソッドがあるなら追加で作ればいいという意見もあると思います。
ただ、全てのメソッドを一から作ると、テストの手間がものすごく増えてしまいます。例えば、unixtimeを取得しint型で返すメソッドがあるとし、新しく時間を文字列型で返すメソッドを欲しいとします。
こんな簡単な例なら、メソッドの返り値を文字列型でキャストすれば問題ないですが、ここでは新しく時間を文字列型で返すメソッドを作成したとします。
この新しく作成したメソッドでエラーが発生した場合、時間を取得している部分で失敗しているのか、文字列型でキャストしている部分で失敗したかエラーが起きたという部分だけではわからないですよね?
ですが、既存のunixtimeを取得しint型で返すメソッドを使用し、新しく時間を文字列型で返すメソッドを作ってエラーが出た場合は、時間を取得する部分のエラーに関しては、既存のメソッドでないことを確認済みなので、int型から文字列型でキャストする時に失敗しているということがすぐに分かります。
また、メソッドを作成する時に、既存のメソッドを呼ぶだけで済むので簡単に実装することができます。
このように、Adapterパターンを使用することで、実装工数の短縮及び実装時に発生したエラー原因を減らすことができます。既存のunixtimeを取得しint型で返すメソッドの返り値を文字列型でキャストするように修正する場合も考えてみましょう。
「Java言語で学ぶデザインパターン入門」にも、記載がありましたが、この場合でもAdapterパターンを使用した方が良いです。
理由としては、既存の実装を変更してしまうので、再度このメソッドを使用している部分をテストしなけばならないため、少しの改変でもテストの工数がかかってしまい無駄になります。「Java言語で学ぶデザインパターン入門」にも、記載がありましたが、機能がかなり離れていたら新規でクラスを作るしかないです。
イメージとしては以下のような場合です。交流100ボルトの電源を元にして水道の水を出すわけにはいかないですからね
Adapterパターンの種類
このAdapterパターンは、以下の2種類あるそうです。
・クラスによるAdapterパターン(継承を使用)
・インスタンスによるAdapterパターン(移譲を使用)
例に対してそれぞれのパターンで記載していきたいと思います。例
インスタンスを作成した時間のunixtimeを取得しint型で返すメソッドを持つクラスを使用して、string型で返すAdapterクラスを作成することにします。
クラスによるAdapterパターン
クラス図
このクラス図は、PlantUMLというもので記載しています。
私が、書いたPlantUMLのコードは以下のGitHubに記載がありますのでReadMeを読んでお使いください。
該当ファイル名は、class.txtです。
https://github.com/sirajirasajiki/design_pattern_uml/blob/master/adapter/class.txtPlantUMLのインストール方法と使い方に関しては、後述のappendixに記載しています。
クラス図を元にpythonで実装
以下に実装したコードを公開しています。python 3.7で実装しました。
https://github.com/sirajirasajiki/design_pattern_python/tree/master/Adapter/class_caseインスタンスによるAdapterパターン
クラス図
クラスによるAdapterパターンと異なるのは、GetTimeStrにてGetTimeの使い方が違います。
このクラス図は、PlantUMLというもので記載しています。
私が、書いたPlantUMLのコードは以下のGitHubに記載がありますのでReadMeを読んでお使いください。
https://github.com/sirajirasajiki/design_pattern_uml/blob/master/adapter/instance.txtクラス図を元にpythonで実装
以下に実装したコードを公開しています。python 3.7で実装しました。
https://github.com/sirajirasajiki/design_pattern_python/tree/master/Adapter/instance_caseまとめ
既存クラスと使用したい形式が違った場合のズレを吸収する物がAdapterパターンということが分かりました。
第2章感想
既存実装を使い新しいクラスを作ることによって、新規作成するよりも工数を減らすことができ、新規実装分でエラーが発生した場合、エラーの原因の範囲が少なくて済むので簡単に実装できると思いました。
また、返り値の型を変えるような簡単な修正でも既存実装に修正を加えてしまうとテストをやり直しになってしまうので、そのような場合にこのAdapterパターンは使うんだなということがわかりました。最後に
何か間違っているところがあれば、ご指摘していただけると嬉しいです!
appendix
前回の記事
https://qiita.com/sirajirasajiki/items/55269e5d6c6e158de16e
PlantUMLに関するサイト
PlantUMLのインストールの時にお世話になったサイトは以下です。
https://qiita.com/kohashi/items/1d2c6e859eeac72ed926
PlantUMLを書くときにお世話になっているサイトは以下です。
https://qiita.com/ogomr/items/0b5c4de7f38fd1482a48
- 投稿日:2020-02-27T22:28:23+09:00
discord.pyをasyncからrewrite版にバージョンアップしたときのメモ
いやぁ・・・w
せっかくdiscordの板とかで質問とかして回答いただいても、
「あ・・・ワイの古かった・・・」
ってなると回答してくれた方にも申し訳ないし、なによりずっと古いバージョンのものを使ってる事自体が情けないというか。きっと僕以外にも、
「バージョンアップしないといけないのはわかってはいるんだけどどんな壁が待っているのかわからないし・・・とりあえず来月やろう」
って思っている方もいると信じて、そういった方に少しでも勇気を分けてあげられればと。基本的には、
1.最新のdiscord.pyにする。
↓
2.とりあえず今まで通り動かしてみる
↓
3.エラーがでる
↓
4.https://discordpy.readthedocs.io/ja/latest/migrating.html でエラーがでてるメソッドを検索かけてみて修正するをひたすら繰り返せばなんとかなります。
また、pyenvのvirtualenvとかで既存の環境とは分けてやったほうが安全です。
また、gitとかで管理しているのであればブランチは切りましょう(当たり前や)それでは書いていきます。
idがstr型からint型になった。
idをとって何か処理したり分岐させてるところでもしシングルコートで囲って判別してるところがあったら外してあげる。
どうしても今まで通りでやりたい場合は、str(client.user.id)という形でくくってあげる。
メッセージの投稿
async
await client.send_message(message.channel, "メッセージ")rewrite
await message.channel.send(res)画像の添付
async
await client.send_file(message.channel, res)rewrite
await message.channel.send(file=discord.File(res,))メッセージを送るときと同じメソッドを使うんですね。
resには、添付したい画像へのパスが入っている想定です。
またタプルで渡しているところにも注意です。(複数貼りたいときは普通にカンマ区切りで大丈夫)リアクションの付与
async
await client.add_reaction(message, e)rewrite
await message.add_reaction(e)あれ・・・以上・・・?
えっと・・・
終わっちゃいました!!w
本当はもっといっぱいあると思ってたんですけどやってみたらこんなもんでした。(僕の場合は)
多分修正行数としては5行くらいだったんじゃないでしょうか。(idを直書きでstrで判定してた部分が2行あった)
この記事で一番大事なのは、やってみれば以外と大したことないのでとりあえずやり始めてみよう!!ってことになると思います!
もしかしたら僕が見落としてるだけで別のところも修正しないといけないかもしれませんが、
そういうのがあったらまた適宜追記していきたいと思います。
- 投稿日:2020-02-27T22:10:41+09:00
【初心者】Heroku、Flask、Python、Gitでアップロードする方法(その④)
Heroku、Flask、Pythonについて、Progateやドットインストール、Qiita等でひととおり学習したので、まとめてみる
(1)作業ディレクトリを作成
(2)仮想環境を設定
(3)必要なフレームワークとWEBサーバーをインストール
(4)FlaskファイルをPythonで作成
(5)Flaskファイルを実行してみる
(6)Herokuにデプロイする
①Herokuにアカウント登録する
②HomebrewでHerokuをインストールする
③Gitを用意する
④Herokuにアプリを作成する
⑤ローカルアプリとHerokuアプリを紐つける
⑥Herokuへのデプロイに必要なファイルを作成する
⑦Herokuへデプロイする
(7)エラー対応
①heroku ps:scale web=1で正常になるケース
②heroku ps:scale web=1で正常にならないケース
(8)ファイルを更新する(7)エラー対応
以下を入力し、デプロイすると、ブラウザに以下のようなエラーメッセージが表示された。
heroku openターミナルに以下を入力し、ログを確認する。
heroku logs --tailすると、
heroku[router]: at=error code=H14 desc="No web processes running"というメッセージ。
①heroku ps:scale web=1で正常になるケース
code=14をググると、以下のサイトに詳細が書かれている
https://github.com/herokaijp/devcenter/wiki/error-codesheroku ps:scale web=1として、
heroku open無事、正常にデプロイできた。
②heroku ps:scale web=1で正常にならないケース
code=14をググると、以下のサイトに詳細が書かれているが、改善しなかった。
もう一度、デプロイに必要なファイルを確認すると、Procfileにタイポを発見。
(誤)web:gunicorn hello:app --log-file -
(正)web: gunicorn hello:app --log-file -
:の後ろに空白がなかった。修正し、再度以下の通り進める。
git status以下のように更新したファイルが表示された。
modified: Procfile今度は、'the-second'として入力。
git commit -m'the-second'git push heroku masterheroku open無事、ブラウザに以下のように表示され、正常にデプロイできた。
(8)ファイルを更新する
基本的に(7)の②の要領でファイルを追加、または修正したものをHerokuにデプロイすればOK。
- 投稿日:2020-02-27T22:09:55+09:00
【初心者】Heroku、Flask、Python、Gitでアップロードする方法(その③)
Heroku、Flask、Pythonについて、Progateやドットインストール、Qiita等でひととおり学習したので、まとめてみる
(1)作業ディレクトリを作成
(2)仮想環境を設定
(3)必要なフレームワークとWEBサーバーをインストール
(4)FlaskファイルをPythonで作成
(5)Flaskファイルを実行してみる
(6)Herokuにデプロイする
①Herokuにアカウント登録する
②HomebrewでHerokuをインストールする
③Gitを用意する
④Herokuにアプリを作成する
⑤ローカルアプリとHerokuアプリを紐つける
⑥Herokuへのデプロイに必要なファイルを作成する
⑦Herokuへデプロイする
(7)エラー対応
①heroku ps:scale web=1で正常になるケース
②heroku ps:scale web=1で正常にならないケース
(8)ファイルを更新する⑥Herokuへのデプロイに必要なファイルを作成する
デプロイに必要なファイルを2つ作成する。
まず、Herokuでアプリを動かすために、どんなライブラリが必要かを伝えるファイルとして、requirements.txtを作成する。
my-projet内で以下を入力する。pip freeze > requirements.txtrequirements.txtには、今までインストールしたライブラリ一覧が表示されたファイルが作成される。
次に、Herokuでアプリを起動した際に最初に実行されるコマンドを記載したファイルとして、Procfileを作成する(拡張子の指定はしない)。
touch Profile作成したProcfileを開き、以下を入力して保存する。
web: gunicorn hello:app --log-file -hello.pyの中の、appというサーバーを開くという意味になる。
webの後ろは、必ずスペースを付けないと、後々Herokuにデプロイした際にエラーとなるので注意が必要。⑦Herokuへデプロイする
まず、以下をターミナルに入力し、今のmy-project内のファイルの状態を表します。
git status次に、どのファイルをHerokuにデプロイするのかを指定します。
全てのファイルを指定することとし、以下を入力。git add .その後に以下のように入力し、再度ステータスを確認すると、ファイルをデプロイするための準備が整った状態を確認できる。
git status次に、これらのファイルはどういう内容が更新されたのか、何が変更されたのかを説明する必要を記載する必要がある。今回は'the-first'として、以下のように入力。
git commit -m'the-first'再度ステータスを確認すると、
git status全てのファイルをデプロイできる状態にした(コミットした)と表示される。
On branch master nothing to commit, working tree cleanこれでHerokuにファイルをpushする(デプロイする)準備が整った。
以下を入力し、Herokuにpush(デプロイ)する。git push heroku master最後に以下を入力してデプロイを確認する。
heroku open
- 投稿日:2020-02-27T22:09:15+09:00
【初心者】Heroku、Flask、Python、Gitでアップロードする方法(その②)
Heroku、Flask、Pythonについて、Progateやドットインストール、Qiita等でひととおり学習したので、まとめてみる
(1)作業ディレクトリを作成
(2)仮想環境を設定
(3)必要なフレームワークとWEBサーバーをインストール
(4)FlaskファイルをPythonで作成
(5)Flaskファイルを実行してみる
(6)Herokuにデプロイする
①Herokuにアカウント登録する
②HomebrewでHerokuをインストールする
③Gitを用意する
④Herokuにアプリを作成する
⑤ローカルアプリとHerokuアプリを紐つける
⑥Herokuへのデプロイに必要なファイルを作成する
⑦Herokuへデプロイする
(7)エラー対応
①heroku ps:scale web=1で正常になるケース
②heroku ps:scale web=1で正常にならないケース
(8)ファイルを更新する(6)Herokuにデプロイする
①Herokuにアカウント登録する
以下のHerokuページにアクセスして、Herokuのアカウント登録をする。
https://www.heroku.com/②HomebrewでHerokuをインストールする
まずはHomebrewというパッケージマネージャーをインストールする。
以下のHomebrewページにアクセスして、表示されているスクリプトをターミナルに貼り付けて実行する。
https://brew.sh/index_ja/usr/bin/ruby -e "$(curl -fsSL https://raw.githubusercontent.com/Homebrew/install/master/install)"その後、以下をターミナルに入力してHerokuをインストールする。
brew install heroku/brew/heroku③Gitを用意する
Gitについての詳細は以下のページを参考にした。
https://backlog.com/ja/git-tutorial/
https://prog-8.com/languages/gitGitのバージョンを確認してみる。
git versionGitがインストールされていれば、以下の通りとなる。
git version 2.20.1 (Apple Git-117)初期設定の確認として、以下をそれぞれ入力する。
git config user.namegit config user.email何も反応なければ、以下の例のように名前やemailを登録する。
(登録しなければ、Gitが正常に動かない)git config user.name "hogehoge"git config user.email "example@gmial.com"④Herokuにアプリを作成する
ローカルで作成したhello.pyアプリを、Heroku(リモート)を結びつける必要がある。
まずは、以下のようにHerokuにログインする。heroku login以下のように表示されたら、q以外の任意の文字を入力するとHerokuページが表示される。
Press any key to open up the browser to login or q to exit:その後ターミナルで、Herokuに、例として、sample-noonnoon というアプリを作成することとし、以下のように入力する。
heroku create sample-noonnoonこれでHerokuにアプリが作成された。Herokuページにログインし、ダッシュボードを確認すると、以下のようにsample-noonnoonが確認できる。
⑤ローカルアプリとHerokuアプリを紐つける
ローカルで作成したアプリと、Herokuで作成したアプリを紐つける。
まずは、my-project内で、Gitを初期化する。git init本当に初期化されたかどうか以下を入力し確認する。(.gitが表示されていればOK)
ls -aGitを初期化することで、ディレクトリmy-projedt内の変更履歴を管理できるようになった。
次にGitとHerokuを紐つける。heroku git:remote -a sample-noonnoonこうすることでローカル上のアプリとHerokuのアプリが紐つく。
set git remote heroku to https://git.heroku.com/sample-noonnoon.git
- 投稿日:2020-02-27T22:02:41+09:00
【初心者】Heroku、Flask、Python、Gitでアップロードする方法(その①)
Heroku、Flask、Pythonについて、Progateやドットインストール、Qiita等でひととおり学習したので、まとめてみる
(1)作業ディレクトリを作成
(2)仮想環境を設定
(3)必要なフレームワークとWEBサーバーをインストール
(4)FlaskファイルをPythonで作成
(5)Flaskファイルを実行してみる
(6)Herokuにデプロイする
①Herokuにアカウント登録する
②HomebrewでHerokuをインストールする
③Gitを用意する
④Herokuにアプリを作成する
⑤ローカルアプリとHerokuアプリを紐つける
⑥Herokuへのデプロイに必要なファイルを作成する
⑦Herokuへデプロイする
(7)エラー対応
①heroku ps:scale web=1で正常になるケース
②heroku ps:scale web=1で正常にならないケース
(8)ファイルを更新する(1)作業ディレクトリを作成
デスクトップ上でいつも作業してるので、今回もデスクトップ上にディレクトリを設置した。
まず、Macのターミナルを起動して、cd desktop と入力、次にmkdir myprojectと入力、これで、デスクトップ上に、ディレクトリmy-projectを作成。(2)仮想環境を設定
次に ターミナルで、cd myproject とし、ディレクトリmy-project内に移動。
Pythonのvenvという仮想環境を提供する機能を使ってローカルに開発環境を設定する。
仮想環境は、python -m venv ●●●と設定。●●●は任意の名称で良いが、任意のサブディレクトリが作成され、サブディレクトリ内に仮想環境ファイルが作成される。ただし今回は、python -m venv . としてmyproject直下に仮想環境ファイルを作成した。サブディレクトリ下に仮想環境ファイルを作成すると、後述するHerokuへのデプロイ時になぜかエラーが発生したため、直下に作成した。python3 -m venv .その後、仮想環境を有効化するために以下のように入力する。
source bin/activateちなみに仮想環境を無効化する場合は、以下のように入力する。
deactivate(3)必要なフレームワークとWEBサーバーをインストール
次に、フレームワークをインストールする。
pip install flaskそれと、WEBサーバーをインストールする。
pip install gnicorn(4)FlaskファイルをPythonで作成
有効化した仮想環境上で、以下のように入力してmy-project内に実行ファイルを作成。今回はhello.pyというファイルを作成。
touch hello.py次にhello.pyを開き、以下のように入力する。
なお、app.run()はこのままだとapp.run(debug=false)となり、デバックモードはオフの状態。本番環境ではdebug=trueとすると危険ぽいのでデフォルトのままとした。
詳しくは以下を参考にした。
https://www.subarunari.com/entry/2018/03/10/いまさらながら_Flask_についてまとめる_〜Debugger〜hello.py# -*- coding: utf-8 -*- # 日本語を使う場合にコメントアウトでも必要 # flaskなどの必要なライブラリをインポート from flask import Flask # 自分の名称を app という名前でインスタンス化 app = Flask(__name__) @app.route('/') def index(): return 'Hello World!' #app.run()はデフォルトではfalseが設定されている if __name__ == '__main__': app.run()(5)Flaskファイルを実行してみる
再びターミナルで操作する。
作成したhello.pyがちゃんと動くかどうかローカル環境で試してみる。
ターミナルで以下を入力。python3 hello.py flask runすると、以下のように表示される。
* Serving Flask app "hello" (lazy loading) * Environment: production WARNING: This is a development server. Do not use it in a production deployment. Use a production WSGI server instead. * Debug mode: off * Running on http://127.0.0.1:5000/ (Press CTRL+C to quit) 127.0.0.1 - -ブラウザで、上記の部分の http://127.0.0.1:5000/ にアクセスすると、以下のように表示される。
これでローカル環境では正常に動くことが確認できた。
control + c でターミナル入力モードに戻る。
- 投稿日:2020-02-27T21:37:14+09:00
[Python]turtleによる渦巻模様の描画
今回は、Pythonにて渦巻模様(曲線ではありませんが)を描画する方法を紹介していきたいと思います。
タートルグラフィクス
直線や曲線を描画する時に非常に便利なツールの一つに、「タートルグラフィクス」があります。
これは子供向けの教材として開発されたものなので、非常に扱いやすく、感覚的に理解もし易く設計されています。Pythonには標準でこのタートルグラフィクスを扱うためのturtleライブラリが用意されています。
ソースコード
import turtle t = turtle.Turtle() l = 200 #辺の長さの初期値 angle = 90 #直線を引き終わった後に方向転換する角度 step = 10 #辺の長さの減少値 while l > 10: #lが10より大きい間は繰り返す t.forward(l) #lだけ直進 t.left(angle) #左に90度回転 l -= step #lを10だけ減らすこのコードを実行すると、以下のような渦巻模様が描かれます。
まとめ
今回はタートルグラフィクスを用いて渦巻模様を描くコードを紹介しました。
他にもタートルグラフィクスを用いると様々な図形を描画することができます。
是非試してみてください。
- 投稿日:2020-02-27T21:30:24+09:00
将来の売上予測への挑戦:②PyFluxを用いた時系列分析
はじめに
前回、将来の売上予測への挑戦:①時系列分析って何?では、将来の売上予測を実現する手段として、時系列分析を紹介しました。
紹介と言うより、(数学が全くわからない)自分なりの解釈でまとめてみたという感じで、しっかりと数式で理解されている方にとっては、「勘違いしているな~」という点もあったかもしれません。何かあれば、ぜひ、ご指摘いただけると幸いです。さて、今回は実際に時系列分析のモデルを作っていきたいと思います。
モデルは、時系列データの予測ライブラリ--PyFlux--で紹介されていた、PyFluxを使ってARIMAモデルとARIMAXモデルを検証していきます。
(本当は、季節性を考慮したSARIMAもやりたかったのですが、PyFluxだとやり方が分からなかったので、今回は断念)分析環境
Google Colaboratory
対象とするデータ
データは超シンプルに、日別の売上と説明変数として気温(平均・最高・最低)を用います。
日付 売上金額 平均気温 最高気温 最低気温 2018-01-01 7,400,000 4.9 7.3 2.2 2018-01-02 6,800,000 4.0 8.0 0.0 2018-01-03 5,000,000 3.6 4.5 2.7 2018-01-04 7,800,000 5.6 10.0 2.6 1.元データ作成
いつもの通り、BigQueryにある対象データをColaboratoryのPython環境にダウンロードします。
例のごとく、項目名は、和名ではつけられませんが、わかりやすくするためにここでは和名にしています。import pandas as pd query = """ SELECT * FROM `myproject.mydataset.mytable` WHERE CAST(日付 AS TIMESTAMP) between CAST("2018-01-01" AS TIMESTAMP) AND CAST("2018-12-31" AS TIMESTAMP) ORDER BY p_date' """ df = pd.io.gbq.read_gbq(query, project_id="myproject", dialect="standard") # 欠損値はゼロに df.fillna(0, inplace=True) # 日付をDatetime型にして、dfのIndexに df = df[1:].set_index('日付') df.index=pd.to_datetime(df.index, utc=True).tz_convert('Asia/Tokyo') df.index = df.index.tz_localize(None) df= df.sort_index()後々の処理のために、日付をDatetime型にしてIndex化することが必要です。
BQ→Pythonの連携には、なぜかBigQuery Storage APIを使ってみたで検証した際に、一番遅かったPandasのread_gbqを使ってますね。
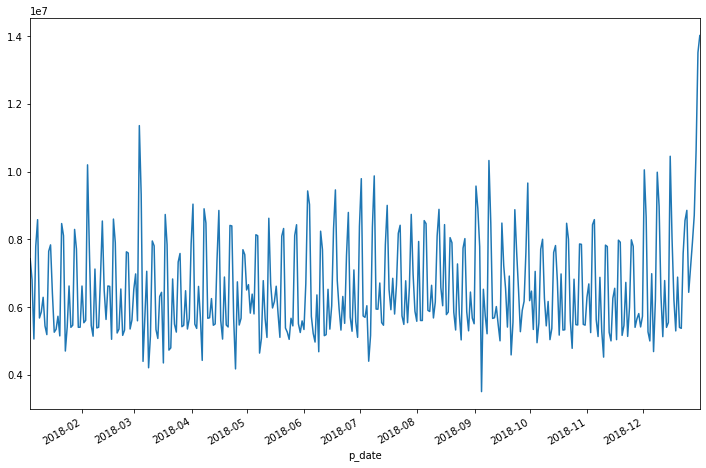
元データがそんなに重くないときは、シンプルに書けて楽ですし。。。(言い訳)ひとまず売上データを見てみましょう。
%matplotlib inline import matplotlib.pyplot as plt plt.figure() df["売上金額"].plot(figsize=(12, 8))日別なのでかなり上がったり・下がったりしています。3月始めや年末は急に上がっていますね。
2.ARIMAモデルの構築
ここから、PyFluxを用いて時系列分析をしていきます。
とはいえ、ColaboratoryにはPyFluxが標準インストールされていないので、まずはインストールしましょう。pip install pyfluxモデルのプログラミング自体は、超シンプル。ARIMAに渡される引数は次の5つです。
- ar : 自己回帰の次数
- ma : 移動平均の次数
- integ : 差分の階数
- target : 目的変数
- family : 確率分布
そして、MLE(最尤推定)でモデルを予測します。
import pyflux as pf model = pf.ARIMA(data=df, ar=5, ma=5, integ=1, target='売上金額', family=pf.Normal()) x = model.fit('MLE')とはいえ、arやma, integの値をいくつにするべきなのかは、予想もつかないので、まずは動かしてみるしかないですね。
x.summary()そうすると出てくるサマリがこちら。
モデル自体の評価は、AIC(赤池情報量基準)やBIC(ベイズ情報量規準)を使えば、それが低いモデルが良いと言えそうです。
変数の評価は、P値(P>|z|)を使えばいいでしょうか?Constant(固定値)や、AR(3)・MA(5)はP値が高くて有意でなさそうですね。AR・MAの1つが有意でないというのは、それぞれの次数を1つ減らせばいいということでしょうかね。
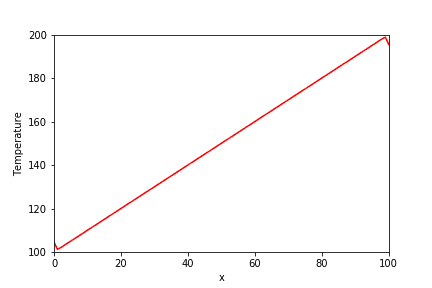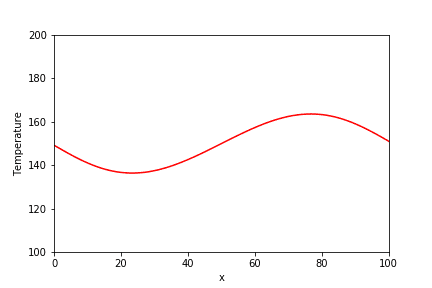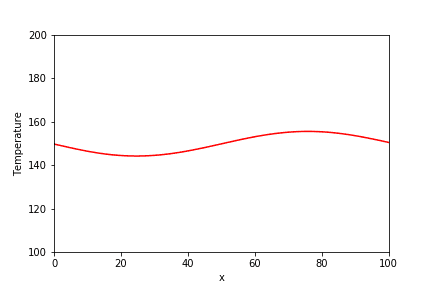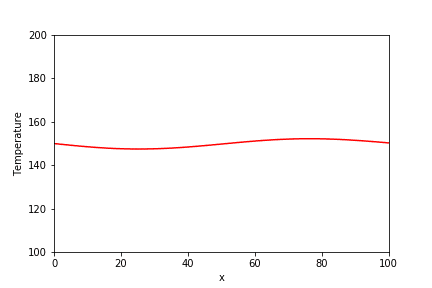
ただ、固定値が有意でないといわれてしまうと、そもそもこのデータはARIMAモデルに使うのは厳しいってことかな?Normal ARIMA(5,1,5) ======================================================= ================================================== Dependent Variable: Differenced kingaku Method: MLE Start Date: 2018-01-09 03:00:00 Log Likelihood: -5401.3927 End Date: 2019-01-01 03:00:00 AIC: 10826.7854 Number of observations: 357 BIC: 10873.3182 ========================================================================================================== Latent Variable Estimate Std Error z P>|z| 95% C.I. ======================================== ========== ========== ======== ======== ========================= Constant 18212.1745 51000.329 0.3571 0.721 (-81748.4703 | 118172.819 AR(1) 0.2046 0.0583 3.507 0.0005 (0.0902 | 0.3189) AR(2) -0.9284 0.0476 -19.4981 0.0 (-1.0217 | -0.8351) AR(3) -0.0762 0.0807 -0.9438 0.3453 (-0.2343 | 0.082) AR(4) -0.4864 0.0465 -10.4663 0.0 (-0.5774 | -0.3953) AR(5) -0.5857 0.0555 -10.5508 0.0 (-0.6945 | -0.4769) MA(1) -0.8716 0.0787 -11.0703 0.0 (-1.0259 | -0.7173) MA(2) 0.9898 0.0905 10.9326 0.0 (0.8123 | 1.1672) MA(3) -0.5321 0.1217 -4.3708 0.0 (-0.7707 | -0.2935) MA(4) 0.4706 0.0945 4.9784 0.0 (0.2853 | 0.6558) MA(5) 0.007 0.0725 0.0973 0.9225 (-0.135 | 0.1491) Normal Scale 900768.835 ==========================================================================================================統計量的には、あまり良くない感じでしたが、グラフではどうなっているでしょう。
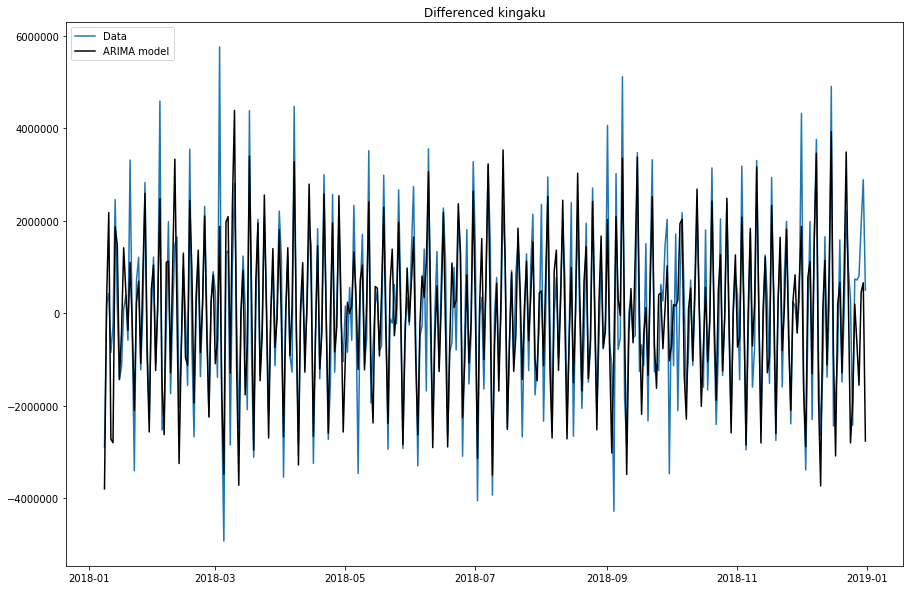
model.plot_fit(figsize=(15, 10))青が実数、黒がモデル値ですね。
上がったり、下がったりするタイミングは似ていますが、実数の振れ幅の大きさを予測しきれていないタイミングが多く見られますね。3.ARIMAXモデルの構築
続いて、ARIMA+変数を使えるARIMAXモデルを構築してみましょう。
こちらもプログラムは超シンプルです。formulaという引数に、「目的変数~1+説明変数」と書くようです。(ちょっと直感的ではない書き方ですが)import pyflux as pf model = pf.ARIMA(data=df, formula='売上金額~1+平均気温+最高気温+最低気温', ar=5, ma=5, integ=1, target='売上金額', family=pf.Normal()) x = model.fit('MLE')その後、ARIMAと同じ様にモデルを評価します。
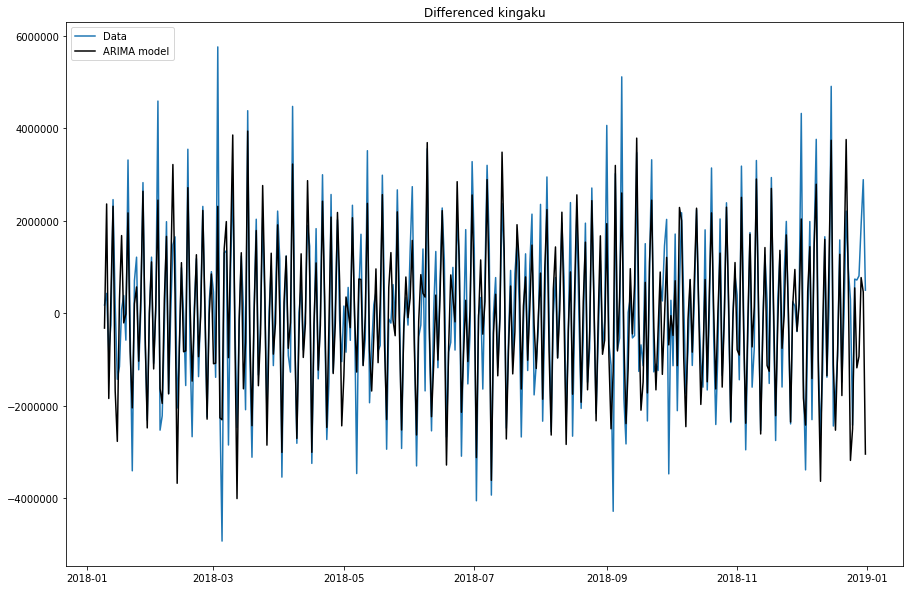
x.summary()AIC・BICともほぼ同じですね。説明変数として追加した気温の変数は、P値が1.0と全く駄目ですね。。。
Normal ARIMAX(5,1,5) ======================================================= ================================================== Dependent Variable: Differenced kingaku Method: MLE Start Date: 2018-01-09 03:00:00 Log Likelihood: -5401.6313 End Date: 2019-01-01 03:00:00 AIC: 10829.2627 Number of observations: 357 BIC: 10879.6732 ========================================================================================================== Latent Variable Estimate Std Error z P>|z| 95% C.I. ======================================== ========== ========== ======== ======== ========================= AR(1) 0.2036 0.0581 3.5023 0.0005 (0.0897 | 0.3175) AR(2) -0.9277 0.0475 -19.5352 0.0 (-1.0208 | -0.8346) AR(3) -0.0777 0.0804 -0.9658 0.3342 (-0.2353 | 0.08) AR(4) -0.4857 0.0463 -10.4841 0.0 (-0.5765 | -0.3949) AR(5) -0.5869 0.0552 -10.6292 0.0 (-0.6952 | -0.4787) MA(1) -0.8687 0.0775 -11.2101 0.0 (-1.0205 | -0.7168) MA(2) 0.989 0.0902 10.9702 0.0 (0.8123 | 1.1657) MA(3) -0.5284 0.1211 -4.3651 0.0 (-0.7657 | -0.2912) MA(4) 0.47 0.0942 4.9874 0.0 (0.2853 | 0.6547) MA(5) 0.0097 0.0715 0.1353 0.8924 (-0.1305 | 0.1499) Beta 1 0.0 59845.8347 0.0 1.0 (-117297.836 | 117297.836 Beta kion_min -0.0 755.0035 -0.0 1.0 (-1479.8069 | 1479.8068) Normal Scale 901399.389 ==========================================================================================================model.plot_fit(figsize=(15, 10))ん~。ARIMAモデルと変わらない。
おわりに
PyFluxで時系列分析ぼプログラム自体は、非常に簡単につくることができました。
ただ、ARIMA、ARIMAXともに上がる・下がるの方向性はいいのですが、その幅が小さくてモデルの精度も上がらないですね。1つは季節性があるのかもしれません。(PyFluxだと使えなかった、SARIMAか?)
また、今回は気温を使った説明変数が全く役立っていなかったので、ここも改善余地がありそうです。
- 投稿日:2020-02-27T21:22:47+09:00
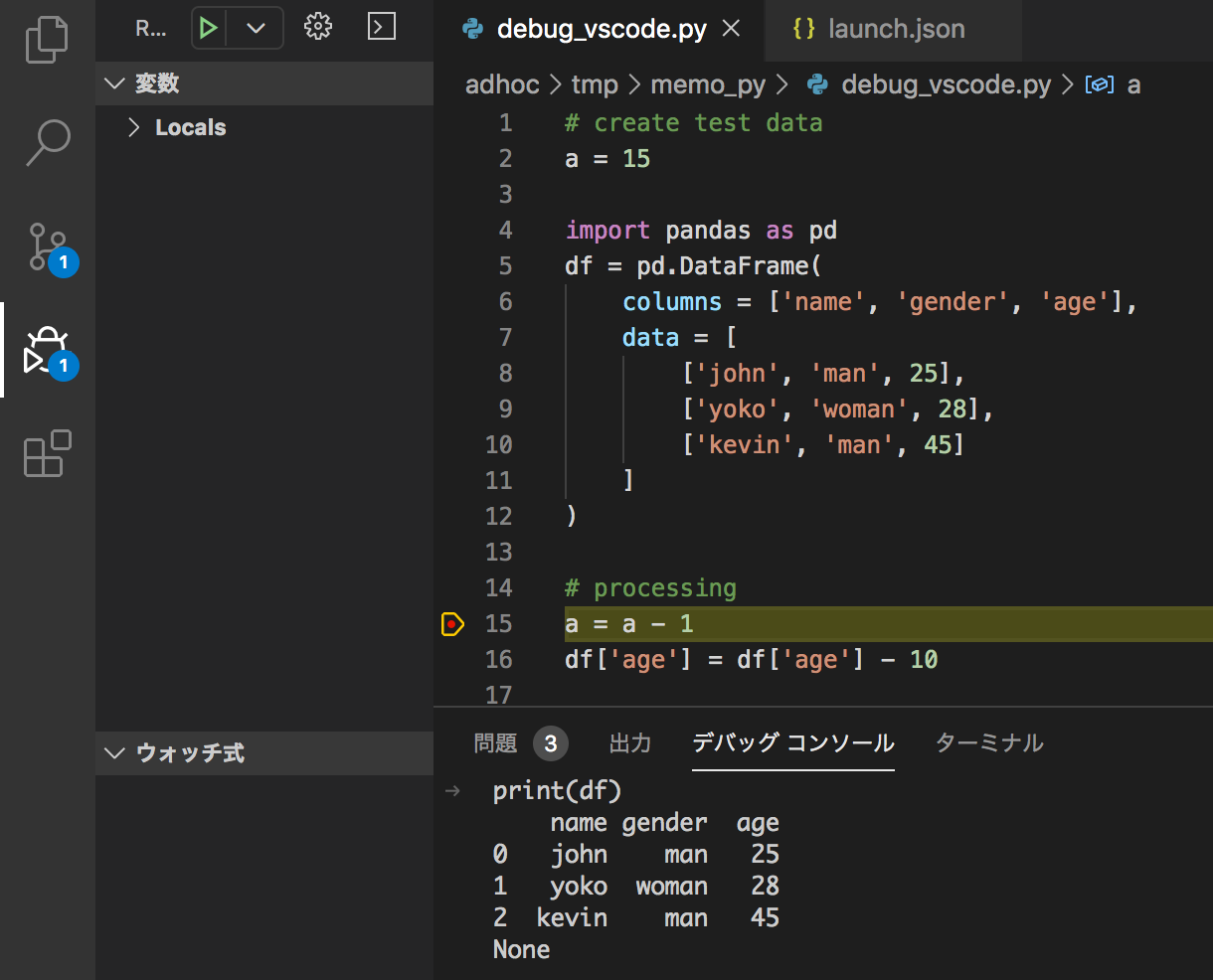
[Python] VScodeのデバッグコンソールでデータフレームを表示
課題
- データフレームをいじくり回す処理のデバッグをサクサクやりたい
- pdbデバッグもいいけど、ソースを編集するのが嫌
前提
VScodeが好き
解決方法概要(目次)
VScodeにて
1. ブレークポイントを置いて
2. デバッグモードで実行し
3. デバッグコンソールを使う
4. データフレームを表示させるために、launch.jsonに一行追記解決方法詳細
ブレークポイントを置く
行番号の左にマウスカーソルを持っていき、クリックして赤丸をつける
デバッグモードで実行
デバッグコンソールを使う
デバッグコンソール画面に移動し、変数名を入れてエンターキー押したりして、色々確認する
データフレームが上手く表示されなくて困るデータフレームを表示させるために、launch.jsonに一行追記
cmd + shift + Pを押してコマンドパレットを開き、
「launch」と入力し、
launch.jsonを開くをクリック
「"redirectOutput": true」を追記して上書き保存(直前行の最後に「,」も忘れず)
一度デバッグを終了して再度デバッグを開始。
デバッグコンソールでprint(df)をやってみる
データフレームが見やすく整列して表示された※なぜこれで上手くいくのかは分かってない。ググって↓に辿り着いて真似た。
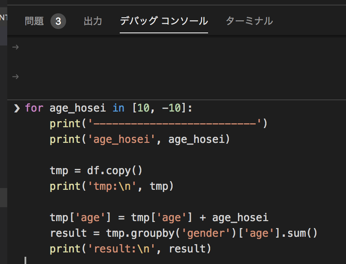
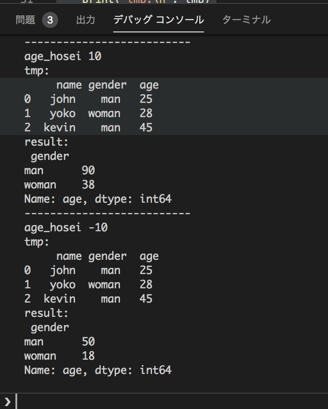
https://github.com/microsoft/ptvsd/issues/2036#issuecomment-573343490おまけ)デバッグコンソールで複数行の処理
補足
動作環境: Mac、Python3.7
デバッグ対象としたサンプルコード:
# create test data a = 15 import pandas as pd df = pd.DataFrame( columns = ['name', 'gender', 'age'], data = [ ['john', 'man', 25], ['yoko', 'woman', 28], ['kevin', 'man', 45] ] ) # processing a = a - 1 df['age'] = df['age'] - 10おまけのデバッグコンソールで使ったコード:
for age_hosei in [10, -10]: print('--------------------------') print('age_hosei', age_hosei) tmp = df.copy() print('tmp:\n', tmp) tmp['age'] = tmp['age'] + age_hosei result = tmp.groupby('gender')['age'].sum() print('result:\n', result)
- 投稿日:2020-02-27T21:00:48+09:00
UnionFindをPythonでやるための最低限の実装
こんにちは!しいです。
たまにUnionFindの問題が出ても他人のライブラリ使いがちだったので復習しとこうとおもって書きます。忘れっぽい自分用のノートみたいなものだとおもってもらえれば
重要例題 AtCoder Typical Contest 001 B - Union Find
https://atcoder.jp/contests/atc001/tasks/unionfind_aUnionFindの純粋な問題ですね
解説スライドもついているのでこれに沿って理解していきます。https://www.slideshare.net/chokudai/union-find-49066733
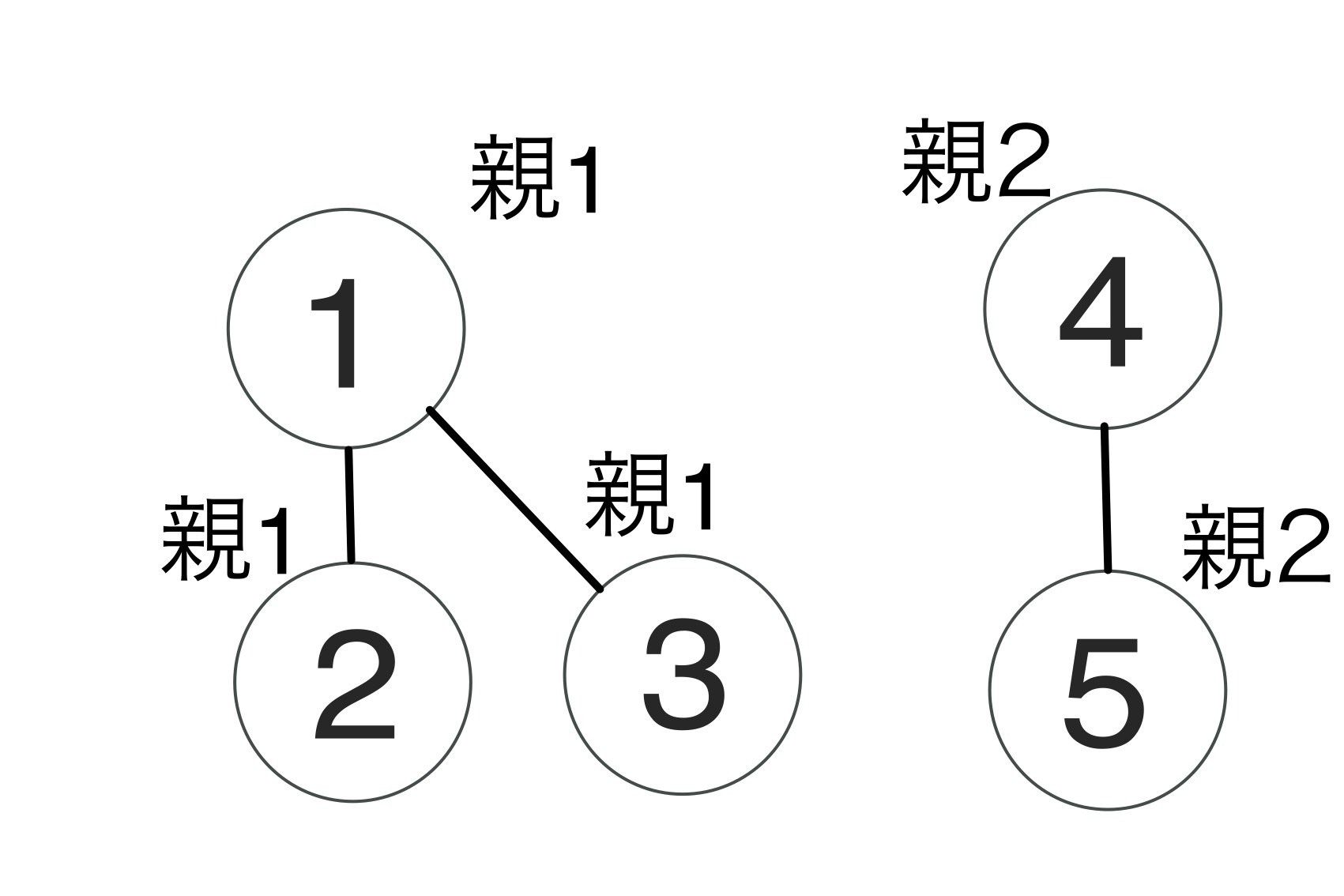
Union find(素集合データ構造) from AtCoder IncUnionFindの機能
1. グループ作り (Union) 2. グループに属しているかどうか (Find)この2つだけです。グループ作成の具体的なイメージはこんな感じ↓
input で与えられた者同士をつなげて木として扱います。グループの作成
グループに属しているか
グループに属しているかどうかは同一の親を持つかどうかで判断します。
高速化のtip1:経路圧縮
縦に長くなると毎回親を探すのに時間がかかってしまいます。しかし今回はどのグループに属しているかが大事なので誰と誰がつながっているかはあまり重要ではないためfindするたびに親に直接つなぎ合わせてあげることで高速化します。
高速化のtip2 : ランク
木の高さを保持しておき低いほうを高いほうにつなげることで経路圧縮の計算量を減らし高速化する
実装(rankなし)
まず各要素の親をpar(parentsの略)で管理します。
最初は各要素はどこのグループにも属していないので自分自身が親になります。要素数をNとするとpar = [i for i in range(N+1)]となります。
次に自分の親を見つける関数をfind、要素x,yが同じグループかを確認する関数をsameとすると
find
自分が親であるときは自分の番号を返しそれ以外の時はもう一度findを行うことで親を探すと同時につなぎなおしています(経路圧縮)
def find(x): if par[x] == x: return x else: par[x] = find(par[x]) #経路圧縮 return par[x]same
お互いの親が一緒であるかを確認して同じグループかどうか判断しています。
def same(x,y): return find(x) == find(y)unite
それぞれの親を確認して異なる場合のみ親を統一します。
def unite(x,y): x = find(x) y = find(y) if x == y: return 0 par[x] = y以上をまとめ、今回の例題を解いてみると
N,Q = map(int,input().split()) par = [i for i in range(N+1)] def find(x): if par[x] == x: return x else: par[x] = find(par[x]) #経路圧縮 return par[x] def same(x,y): return find(x) == find(y) def unite(x,y): x = find(x) y = find(y) if x == y: return 0 par[x] = y for i in range(Q): p,a,b = map(int,input().split()) if p == 0: unite(a,b) else: if same(a,b): print('Yes') else: print('No')https://atcoder.jp/contests/atc001/submissions/10365710
rankを実装するパターン
rankを実装する場合は各頂点の高さを管理してあげる必要があります
N,Q = map(int,input().split()) par = [i for i in range(N+1)] rank = [0]*(N+1) def find(x): if par[x] == x: return x else: par[x] = find(par[x]) #経路圧縮 return par[x] def same(x,y): return find(x) == find(y) def unite(x,y): x = find(x) y = find(y) if x == y: return 0 if rank[x] < rank[y]: par[x] = y else: par[y] = x if rank[x]==rank[y]:rank[x]+=1 for i in range(Q): p,a,b = map(int,input().split()) if p == 0: unite(a,b) else: if same(a,b): print('Yes') else: print('No')最後に
以上がPythonによる最低限の実装でした。
もし何か間違ったとこがあればどんどんいってください!参考にさせてもらったサイト様
- 投稿日:2020-02-27T20:04:34+09:00
コンテナ上のpython/pyodbcでホストのSQL Serverにアクセスする
概要
先日の以下の記事でmysqlにアクセスする方法をご紹介しましたが、
dockerコンテナ内からホストのmysqlにアクセスする
SQL serverバージョンをご紹介します。前提
ホストSQL Server
ODBC Driver 17 for SQL Server
SQLServer認証でポート1443で動作している手順
ODBCが動くDockerコンテナの作り方の記事内にある以下のリポジトリをcloneします。
https://github.com/Microsoft/mssql-docker/tree/master/oss-drivers/pyodbc> git clone https://github.com/microsoft/mssql-docker.git > cd mssql-docker/oss-drivers/pyodbc以下のツリーになっていると思います。
pyodbc ├── Dockerfile ├── README.md ├── docker-compose.yml *docker-composeで立ち上げるために追加 ├── entrypoint.sh └── sample.py *編集します。もしくは別ファイルを作ってもよいです。touch入ってませんが...Dockerfile# mssql-python-pyodbc # Python runtime with pyodbc to connect to SQL Server FROM ubuntu:16.04 # apt-get and system utilities RUN apt-get update && apt-get install -y \ curl apt-utils apt-transport-https debconf-utils gcc build-essential g++-5\ && rm -rf /var/lib/apt/lists/* # adding custom MS repository RUN curl https://packages.microsoft.com/keys/microsoft.asc | apt-key add - RUN curl https://packages.microsoft.com/config/ubuntu/16.04/prod.list > /etc/apt/sources.list.d/mssql-release.list # install SQL Server drivers RUN apt-get update && ACCEPT_EULA=Y apt-get install -y msodbcsql unixodbc-dev # install SQL Server tools RUN apt-get update && ACCEPT_EULA=Y apt-get install -y mssql-tools RUN echo 'export PATH="$PATH:/opt/mssql-tools/bin"' >> ~/.bashrc RUN /bin/bash -c "source ~/.bashrc" # python libraries RUN apt-get update && apt-get install -y \ python-pip python-dev python-setuptools \ --no-install-recommends \ && rm -rf /var/lib/apt/lists/* # install necessary locales RUN apt-get update && apt-get install -y locales \ && echo "en_US.UTF-8 UTF-8" > /etc/locale.gen \ && locale-gen RUN pip install --upgrade pip # install SQL Server Python SQL Server connector module - pyodbc RUN pip install pyodbc # install additional utilities RUN apt-get update && apt-get install gettext nano vim -y # add sample code RUN mkdir /sample ADD . /sample WORKDIR /sample CMD /bin/bash ./entrypoint.shdocker-compose.yamlversion: '3' services: pyodbc: restart: always build: . container_name: 'pyodbc' working_dir: '/sample' tty: true volumes: - .:/sample extra_hosts: - "(ホストのCOMPUTERNAME):(ホストのIPアドレス)" ports: - 1443:1443sample.pyimport pyodbc server = '(ホストのIPアドレス),(ホストのSQLServer動作ポート)' username = 'sa' password = 'password' cnxn = pyodbc.connect('DRIVER={ODBC Driver 17 for SQL Server};SERVER='+server+';database=(データベース名);UID='+username+';PWD='+password) cursor = cnxn.cursor() print ('Using the following SQL Server version:') tsql = "SELECT @@version;" with cursor.execute(tsql): rows = cursor.fetchall() for row in rows: print(str(row))> docker-compose up -d --build ... > docker container ps CONTAINER ID IMAGE COMMAND CREATED STATUS PORTS NAMES 2c51574bfd36 pyodbc_pyodbc "/bin/sh -c '/bin/ba…" 51 minutes ago Up 51 minutes 0.0.0.0:1443->1443/tcp pyodbc > docker exec -it 2c51574bfd36 /bin/bash $ python --version Python 2.7.12 $ python sample.py Using the following SQL Server version: (u'Microsoft SQL Server 2017 (RTM) - 14.0.1000.169 (X64) \n\tAug 22 2017 17:04:49 \n\tCopyright (C) 2017 Microsoft Corporation\n\tExpress Edition (64-bit) on Windows 10 Pro 10.0 <X64> (Build 18362: ) (Hypervisor)\n', )結果
無事SQL ServerにアクセスしDB参照できました。
- 投稿日:2020-02-27T19:40:50+09:00
競プロ私的テンプレート(Python3)
- 投稿日:2020-02-27T19:30:10+09:00
CrossEntropyLossを数式入りでちょっと理解する
はじめに
Pytorchの損失関数の基準によく
criterion=torch.nn.CrossEntropyLoss()を使用しているため,
詳細を理解するためにアウトプットしてます.
間違ってたらそっと教えてください.CrossEntropyLoss
Pytorchのサンプル(1)を参考にして,
torch.manual_seed(42) #再現性を保つためseed固定 loss = nn.CrossEntropyLoss() input_num = torch.randn(1, 5, requires_grad=True) target = torch.empty(1, dtype=torch.long).random_(5) print('input_num:',input_num) print('target:',target) output = loss(input_num, target) print('output:',output)input_num: tensor([[ 0.3367, 0.1288, 0.2345, 0.2303, -1.1229]], requires_grad=True) target: tensor([0]) output: tensor(1.3472, grad_fn=<NllLossBackward>)正解クラスを $class$,クラス数を $n$ とするとCrossEntropyLossの誤差 $loss$ は以下の式で表すことができます.
loss=-\log(\frac{\exp(x[class])}{\sum_{j=0}^{n} \exp(x[j])}) \\ =-(\log(\exp(x[class])- \log(\sum_{j=0}^{n} \exp(x[j])) \\ =-\log(\exp(x[class]))+\log(\sum_{j=0}^{n} \exp(x[j])) \\ =-x[class]+\log(\sum_{j=0}^{n} \exp(x[j])) \\ソースコードのサンプルより正解クラスは $class=0$,クラス数は $n=5$ なので,確かめてみると
loss=-x[0]+\log(\sum_{j=0}^{5} \exp(x[j]))\\ =-x[0]+\log(\exp(x[0])+\exp(x[1])+\exp(x[2])+\exp(x[3])+\exp(x[4])) \\ = -0.3367 + \log(\exp(0.3367)+\exp(0.1288)+\exp(0.2345)+\exp(0.2303)+\exp(-1.1229)) \\ = 1.34717 \cdots \\ \fallingdotseq 1.34712プログラムの結果と無事合いましたね!
ちなみに計算は以下のコードでしてます(手計算の算数は無理・・・)from math import exp, log x_sum = exp(0.3367)+exp( 0.1288)+exp(0.2345)+exp(0.2303)+exp(-1.1229) x = 0.3367 ans = -x + log(x_sum) print(ans) # 1.3471717976017477ごり押しです.
さいごに
丸め込み誤差(小数点の二進数表示による循環小数の発生)は今はあまり考えなくてもよさそう
普段はrandom.seed(42)なのですが,Pytorchだとtorch.manual_seed(42)なのではえ~って感じです.参考文献
(1)TORCH.NN
- 投稿日:2020-02-27T19:10:49+09:00
「効果検証入門 3章 傾向スコアを用いた分析」をやってみる
この記事は、「効果検証入門 ~正しい比較のための因果推論/計量経済学の基礎」の「3章 傾向スコアを用いた分析」を読んで、学んだことや感じたことを整理したものです。
基礎編
傾向スコアとは
- 観測された共変量$X$が与えられた条件下で介入群に割り当てられる確率$e(X)=P(Z=1|X)$1
- 全ての共変量が観測されることはない
回帰分析 v.s. 傾向スコア2
- 回帰分析の仮定:
- CIA(Conditional Independence Assumption): $\{Y^{(1)}, Y^{(0)}\}\perp Z|X$
- アウトカムと共変量の線形性(本書では省略?)
- 傾向スコアの仮定
CIA v.s. 強く無視できる割り当て
- 本書ではCIAが使われているが、厳密には強く無視できる割り当てが正しい7
- 強く無視できる割り当てはCIAに次の2つの仮定が加わる
- 共変量$X$は観測されたもののみ
- $0<P(Z=1|X)<1$
- 要するにどのサンプルも、介入群/非介入群どちらにも割り当てられる可能性がないとNGということで、結構重要だと思う
- 例えば男性が全員介入群に割り当てられるとすると、$P(Z=1|男性)=1$となるのでNG
傾向スコアの推定
- 傾向スコアの推定には多くの場合でロジスティック回帰が使われるが、機械学習でもOK
- モデルには共変量だけでなく、アウトカムに影響を及ぼすその他の変数も含める8
- 機械学習を使う場合はprobability calibrationが必要9
- ランダムフォレストとSVMくらい?
- 一方、損失関数がloglossなら確率に収束するのでcalibration不要10
傾向スコアを使った介入効果の推定
ATT v.s. ATE
- ATT(Average Treatment effect on Treated):
- $E[Y^{(1)}-Y^{(0)}|Z=1]$
- 介入を受けたサンプルにおける介入効果の期待値
- 本書ではマッチングによって求める
- ATE(Average Treatment Effect):
- $E[Y^{(1)}-Y^{(0)}]$
- サンプル全体の介入効果の期待値
- 本書ではIPWによって求める
マッチング v.s. IPW11
- マッチング:
- 介入群と非介入群から傾向スコアの値が近いサンプルのペアを取り出し、一方の欠測(反事実)をもう一方で補完する
- IPW(Inverse Probability Weighting):
- 傾向スコアの逆数でサンプルを重み付けし、母集団の分布に近づける
- IPWはコモンサポート(後述)を考慮しないため、使うべきではない?8
- コモンサポートを考慮すれば使ってもよいと思う
ATT in マッチング
- 介入群のサンプルに対し傾向スコアの値が近いサンプルを非介入群からマッチング、ペアの差の平均をATTとする
ATE in マッチング
- 非介入群のサンプルに対しても同様にマッチング、ペアの差の平均をATEとする
ATT in IPW
- 次式で求める
\frac{\sum_{i=1}^{N}Z_{i}Y_{i}}{\sum_{i=1}^{N}Z_{i}} -\frac{\sum_{i=1}^{N}\frac{(1-Z_{i})\hat{e}(X_{i})}{1-\hat{e}(X_{i})}Y_{i}}{\sum_{i=1}^{N}\frac{(1-Z_{i})\hat{e}(X_{i})}{1-\hat{e}(X_{i})}}ATE in IPW
- 次式で求める
\frac{\sum_{i=1}^{N}\frac{Z_{i}}{\hat{e}(X_{i})}Y_{i}}{\sum_{i=1}^{N}\frac{Z_{i}}{\hat{e}(X_{i})}} -\frac{\sum_{i=1}^{N}\frac{1-Z_{i}}{1-\hat{e}(X_{i})}Y_{i}}{\sum_{i=1}^{N}\frac{1-Z_{i}}{1-\hat{e}(X_{i})}}コモンサポートについて
- コモンサポートとは、介入群と非介入群の傾向スコアの分布が重なっている範囲
- IPWの計算においてコモンサポート外のサンプルを使うと、傾向スコアの逆数(重み)が大きくなりすぎてうまく推定できない12
- 「コモンサポート外であること」と「重みが大きくなりすぎること」は等価か?
- コモンサポート外のサンプルは除外するのが一般的?
- 本書ではpropensity score trimmig/clippingを紹介
- コモンサポート外を除外することで新たなバイアスを生むのでは?
- だから除外しなくてもいいように、比較可能な2群をちゃんと観測できるように実験計画をデザインすることが重要
- その上で生じたバイアスを傾向スコア等で調整すべき
- コモンサポート外では反事実を補完できるサンプルがないため、そもそも「強く無視できる割り当て」を満たしていないのでは?
- $P(Z=1|X)=0$または$P(Z=1|X)=1$でないのはロジスティック回帰を使ってるから
傾向スコアの評価
- 傾向スコアによって介入群と非介入群の共変量の分布が2群間でバランシングされているかどうかが重要
- つまり、仮定を$\{Y^{(1)}, Y^{(0)}\}\perp Z|e(X)$から$\{Y^{(1)}, Y^{(0)}\}\perp Z|X$に戻すということだと思う
- 本書では標準化平均差(ASAM)で評価している
- ASAMが0.1以下ならOK(なぜ?)
- 旧来のAUCが0.8以上ならOK説はナンセンス?13
- AUCは0.8以上あればOKとされている14
- AUCが小さい(0.5に近い)とはどういうことか:
- 割り当てが共変量に依存しない($Z\perp X$) → 傾向スコアで調整する必要がない → AUCはある程度大きくないと意味がない?
- AUCが大きい(1に近い)とはどういうことか:
- コモンサポートが小さい → 介入効果を推定できない → AUCは大きければ良いというものでもない?
実践編
MineThatData E-Mail Analytics And Data Mining Challenge dataset15
- 後日追記予定
LaLonde(1986)
- データの説明については本書参照
- 論文の説明についてはこちら参照
データ読み込み
import numpy as np import pandas as pd import matplotlib.pyplot as plt import warnings warnings.filterwarnings('ignore')df_cps1 = pd.read_stata('https://users.nber.org/~rdehejia/data/cps_controls.dta') df_cps3 = pd.read_stata('https://users.nber.org/~rdehejia/data/cps_controls3.dta') df_nsw = pd.read_stata('https://users.nber.org/~rdehejia/data/nsw_dw.dta')データセット作成
- NSW: RCTのデータセット
- CPS1+NSW: NSWの非介入群をCPS1で置き換えたデータセット
- CPS3+NSW: NSWの非介入群をCPS3で置き換えたデータセット
treat_label = {0: 'untreat', 1: 'treat'} X_columns = ['age', 'education', 'black', 'hispanic', 'married', 'nodegree', 're74', 're75'] y_column = 're78' z_column = 'treat'CPS1+NSW
df_cps1_nsw = pd.concat([df_nsw[df_nsw[z_column] == 1], df_cps1], ignore_index=True)CPS3+NSW
df_cps3_nsw = pd.concat([df_nsw[df_nsw[z_column] == 1], df_cps3], ignore_index=True)介入群と非介入群の分布を確認
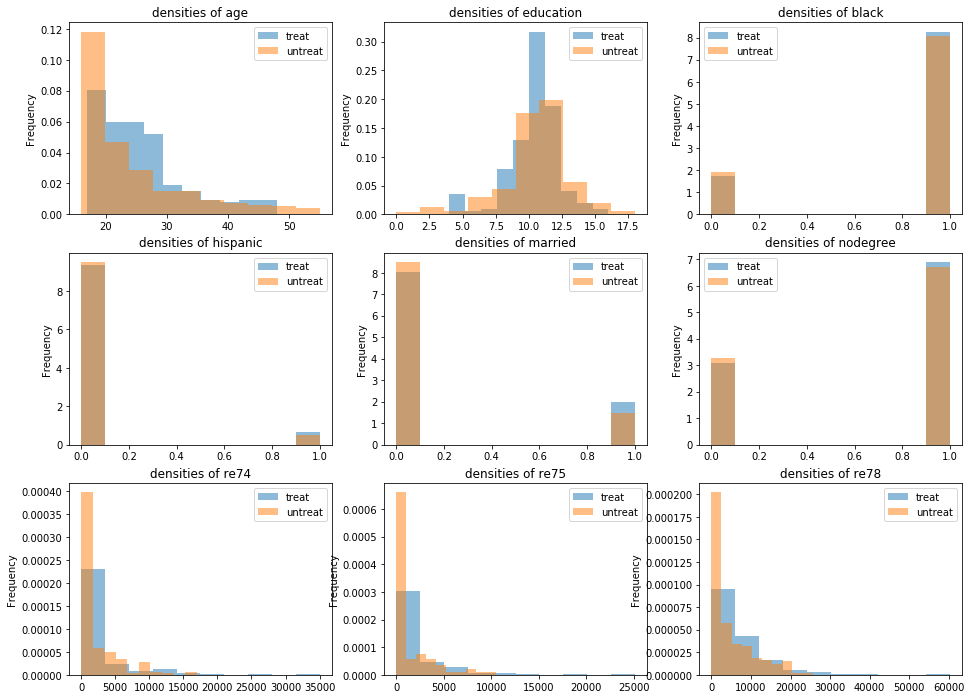
NSW
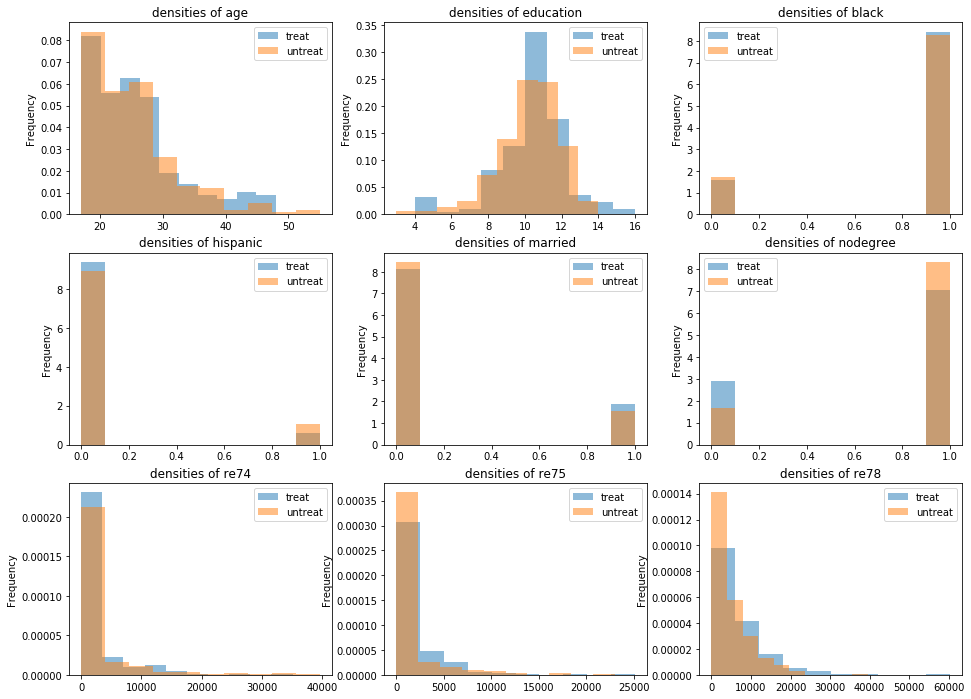
- RCTを謳うだけあって均衡の取れたデータである
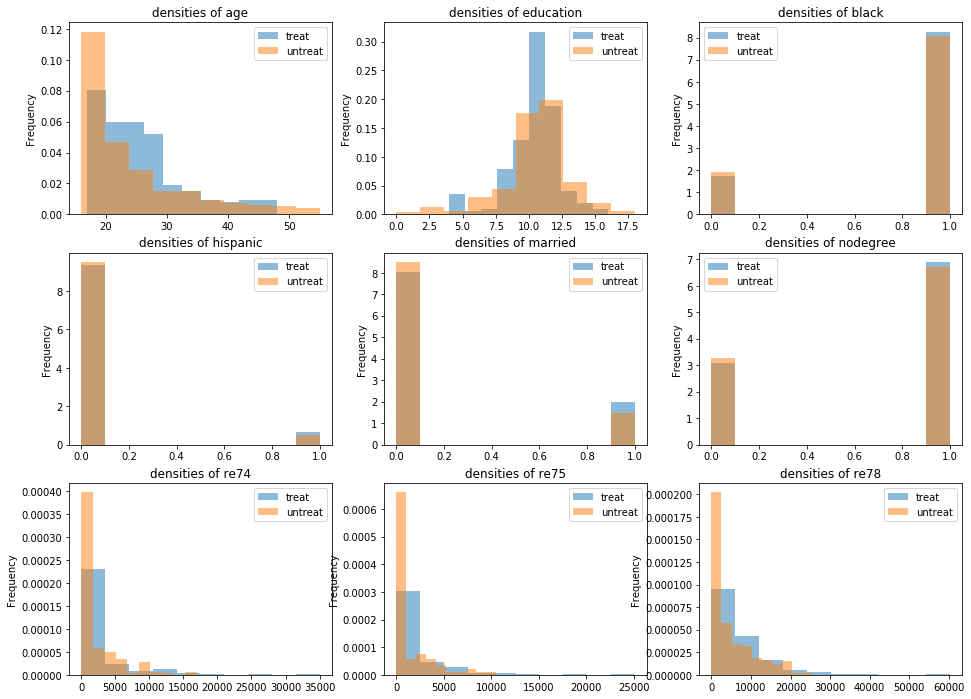
df_nsw[z_column].map(treat_label).value_counts().plot.bar(title='# of treatment in NSW')_, ax = plt.subplots(3, 3, figsize=(16, 12)) for i, c in enumerate(X_columns + [y_column]): (df_nsw[c].groupby(df_nsw[z_column].map(treat_label)) .plot.hist(density=True, alpha=0.5, title=f'densities of {c}', legend=True, ax=ax[i // 3, i % 3]))CPS1+NSW
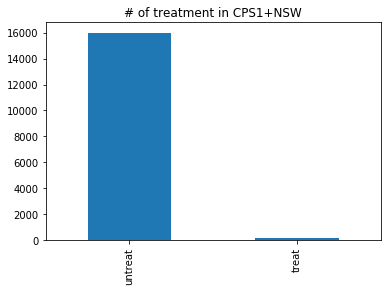
- なかなか不均衡なデータである
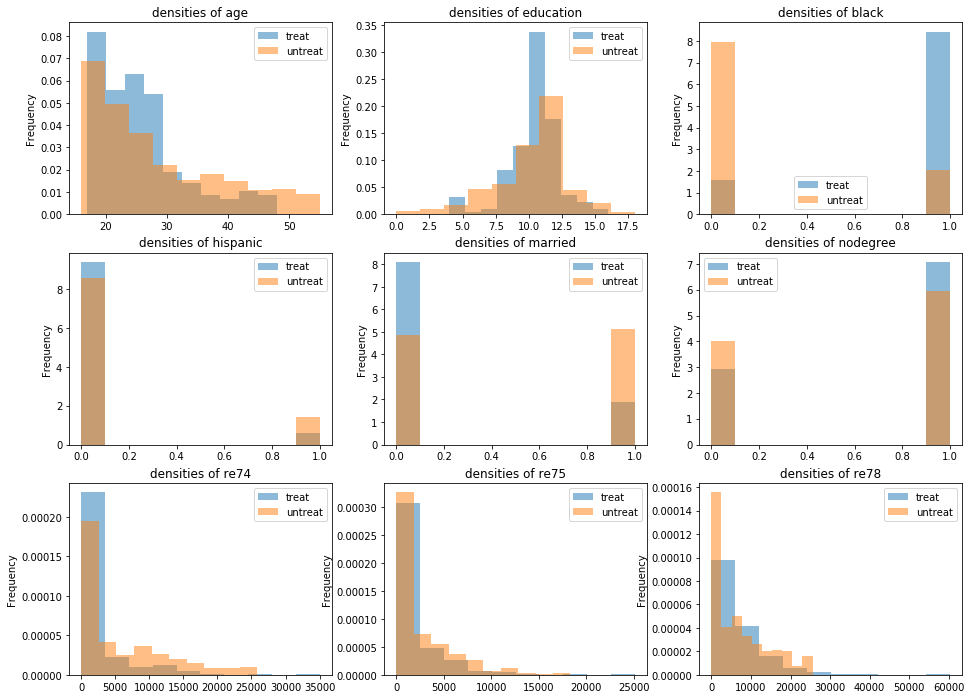
df_cps1_nsw[z_column].map(treat_label).value_counts().plot.bar(title='# of treatment in CPS1+NSW')_, ax = plt.subplots(3, 3, figsize=(16, 12)) for i, c in enumerate(X_columns + [y_column]): (df_cps1_nsw[c].groupby(df_cps1_nsw[z_column].map(treat_label)) .plot.hist(density=True, alpha=0.5, title=f'densities of {c}', legend=True, ax=ax[i // 3, i % 3]))CPS3+NSW
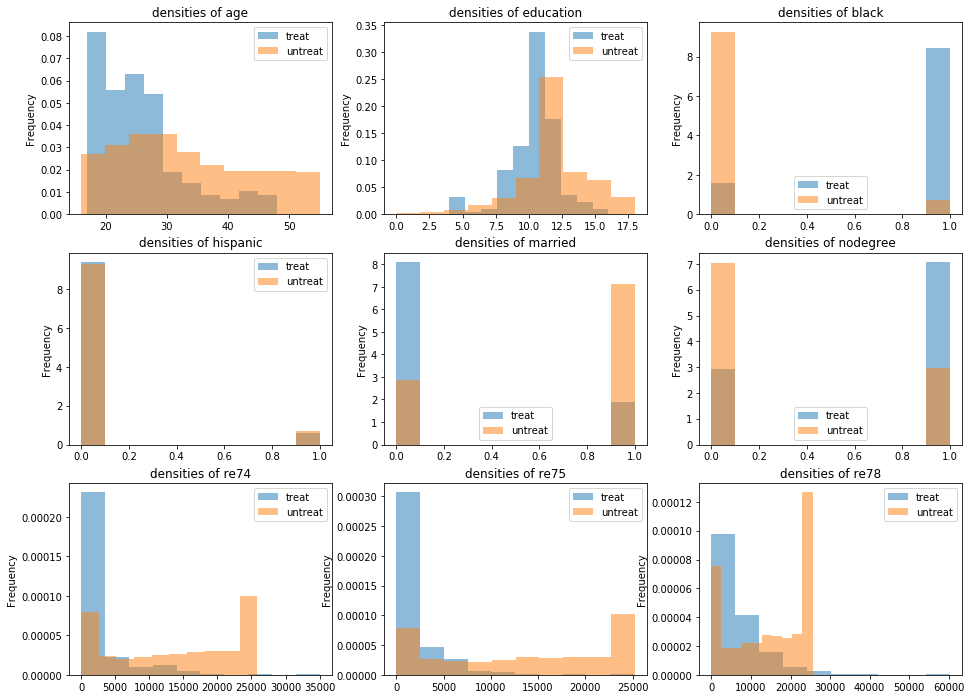
- NSWほどではないが、CPS1+NSWよりは均衡の取れたデータである
df_cps3_nsw[z_column].map(treat_label).value_counts().plot.bar(title='# of treatment in CPS3+NSW')_, ax = plt.subplots(3, 3, figsize=(16, 12)) for i, c in enumerate(X_columns + [y_column]): (df_cps3_nsw[c].groupby(df_cps3_nsw[z_column].map(treat_label)) .plot.hist(density=True, alpha=0.5, title=f'densities of {c}', legend=True, ax=ax[i // 3, i % 3]))回帰分析
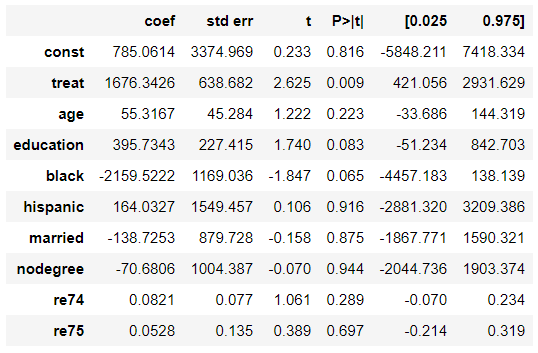
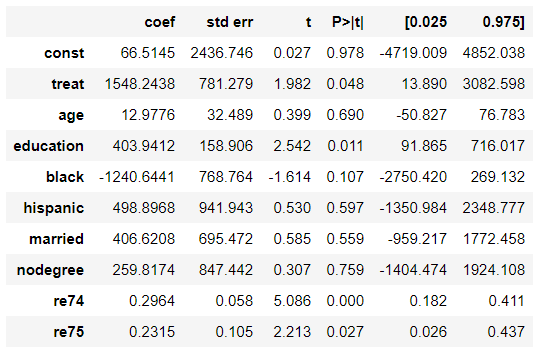
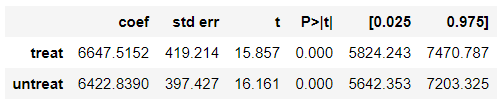
データセットごとの介入効果
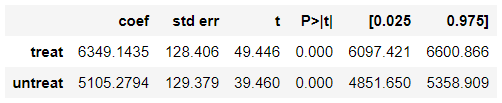
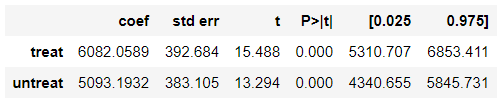
データセット 回帰分析 NSW 1676.3 CPS1+NSW 699.1 CPS3+NSW 1548.2 import statsmodels.api as smNSW
results = sm.OLS(df_nsw[y_column], sm.add_constant(df_nsw[[z_column] + X_columns])).fit() results.summary().tables[1]CPS1+NSW
results = sm.OLS(df_cps1_nsw[y_column], sm.add_constant(df_cps1_nsw[[z_column] + X_columns])).fit() results.summary().tables[1]CPS3+NSW
results = sm.OLS(df_cps3_nsw[y_column], sm.add_constant(df_cps3_nsw[[z_column] + X_columns])).fit() results.summary().tables[1]ゲームスタート
- NSWの介入効果は約$1600
- バイアスを含んだ状態から傾向スコアを用いてどれだけ$1600に近づけるかチャレンジ
傾向スコアの推定
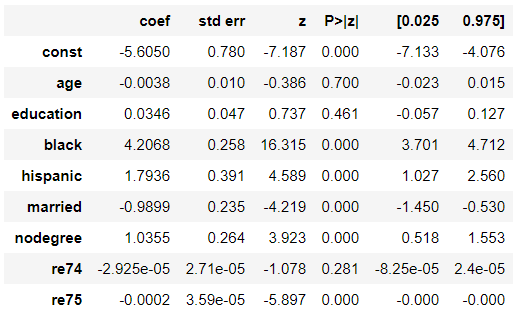
- 本書の
I(re74^2)とI(re75^2)はどこから出てきた?CPS1+NSW
- 傾向スコアをロジスティック回帰で推定
ps_model = sm.Logit(df_cps1_nsw[z_column], sm.add_constant(df_cps1_nsw[X_columns])).fit() ps_model.summary().tables[1]df_cps1_nsw['ps'] = 1 / (1 + np.exp(-ps_model.fittedvalues))
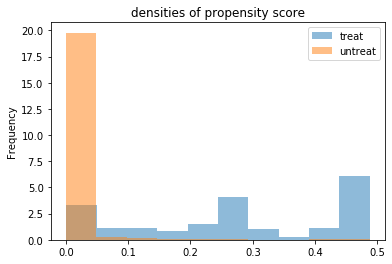
- 傾向スコアの分布を確認
(df_cps1_nsw.groupby(df_cps1_nsw[z_column].map(treat_label))['ps'] .plot.hist(density=True, alpha=0.5, title=f'densities of propensity score', legend=True))
- AUCを計算
from sklearn.metrics import roc_auc_score roc_auc_score(df_cps1_nsw[z_column], df_cps1_nsw['ps'])0.9708918648513447CPS3+NSW
- 傾向スコアをロジスティック回帰で推定
ps_model = sm.Logit(df_cps3_nsw[z_column], sm.add_constant(df_cps3_nsw[X_columns])).fit() ps_model.summary().tables[1]df_cps3_nsw['ps'] = 1 / (1 + np.exp(-ps_model.fittedvalues))
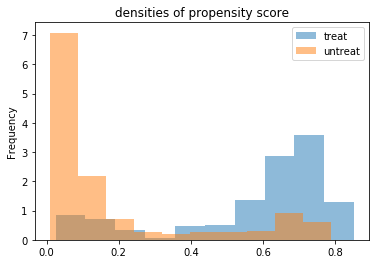
- 傾向スコアの分布を確認
(df_cps3_nsw.groupby(df_cps3_nsw[z_column].map(treat_label))['ps'] .plot.hist(density=True, alpha=0.5, title=f'densities of propensity score', legend=True))
- AUCを計算
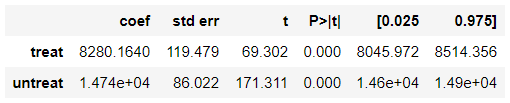
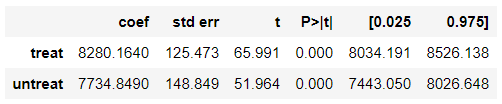
from sklearn.metrics import roc_auc_score roc_auc_score(df_cps3_nsw[z_column], df_cps3_nsw['ps'])0.8742266742266742ATTの推定
データセット・推定手法ごとのATT
データセット マッチング IPW IPW(コモンサポート) CPS1+NSW 1929.1 1180.4 1243.9 CPS3+NSW 1463.1 1214.1 988.9 CPS1+NSW
マッチング
- ペアを作る関数(再帰ジェネレータ)を定義
- こちらを参考にさせていただいた
from sklearn.neighbors import NearestNeighbors def pairs_generator(s0: pd.Series, s1: pd.Series, threshold: np.float) -> pd.DataFrame: """ K近傍法を使ってペアを作る再帰ジェネレータ。 Parameters ---------- s0, s1 : pd.Series threshold : np.float ペアを作る距離の閾値。最近傍点でも閾値を超えた場合はペアとならない。 Returns ------- pairs : pd.DataFrame s0のサンプルと、最も近いs1のサンプルで作られたペアのインデックス。 列lがs0のインデックス、列rがs1のインデックスとなる。 Notes ----- 複数のペアに同じサンプルが使われることはない。 Examples -------- s0 = df[df[z_column] == 0]['ps'] s1 = df[df[z_column] == 1]['ps'] threshold = df['ps'].std() * 0.1 pairs = pd.concat(pairs_generator(s1, s0, threshold), ignore_index=True) """ neigh_dist, neigh_ind = NearestNeighbors(1).fit(s0.values.reshape(-1, 1)).kneighbors(s1.values.reshape(-1, 1)) pairs = pd.DataFrame( {'d': neigh_dist[:, 0], 'l': s0.iloc[neigh_ind[:, 0]].index}, index=s1.index, ).query(f'd < {threshold}').groupby('l')['d'].idxmin().rename('r').reset_index() if len(pairs) > 0: yield pairs yield from pairs_generator(s0.drop(pairs['l']), s1.drop(pairs['r']), threshold)
- ペアを作る
s1 = df_cps1_nsw[df_cps1_nsw[z_column] == 1]['ps'] s0 = df_cps1_nsw[df_cps1_nsw[z_column] == 0]['ps'] threshold = df_cps1_nsw['ps'].std() * 0.1 pairs1_cps1_nsw = pd.concat(pairs_generator(s1, s0, threshold), ignore_index=True) pairs0_cps1_nsw = pd.concat(pairs_generator(s0, s1, threshold), ignore_index=True) # ATEの計算で使う
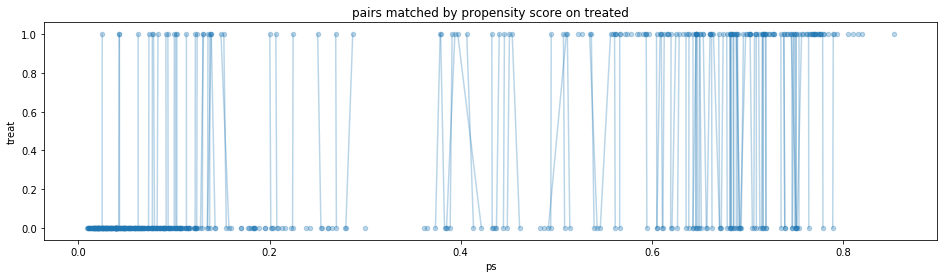
- 介入群のペアを確認
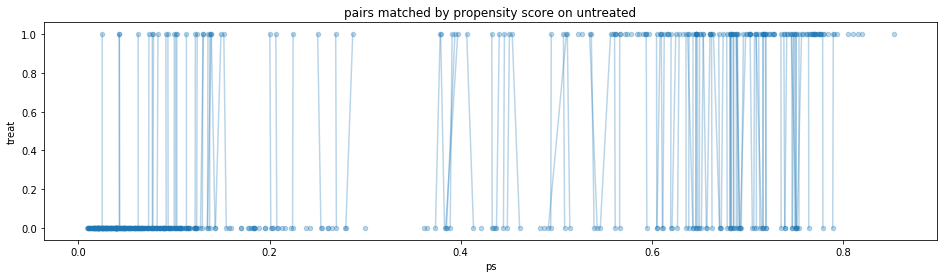
- 本来は図のような交差が発生しないようにペアを作るのが望ましい(と思う)
df_cps1_nsw.plot.scatter(x='ps', y=z_column, alpha=0.3, figsize=(16, 4), title='pairs matched by propensity score on treated') for x in zip(df_cps1_nsw['ps'].iloc[pairs1_cps1_nsw['l']].values, df_cps1_nsw['ps'].iloc[pairs1_cps1_nsw['r']].values): plt.plot(x, [1, 0], c='tab:blue', alpha=0.3)
- 非介入群のペアを確認
- これらのペアはATEの計算で使う
df_cps1_nsw.plot.scatter(x='ps', y=z_column, alpha=0.3, figsize=(16, 4), title='pairs matched by propensity score on untreated') for x in zip(df_cps1_nsw['ps'].iloc[pairs0_cps1_nsw['l']].values, df_cps1_nsw['ps'].iloc[pairs0_cps1_nsw['r']].values): plt.plot(x, [0, 1], c='tab:blue', alpha=0.3)
- ATTを計算
(pairs1_cps1_nsw['l'].map(df_cps1_nsw[y_column]) - pairs1_cps1_nsw['r'].map(df_cps1_nsw[y_column])).agg(['mean', 'std'])mean 1929.083008 std 9618.346680 dtype: float64
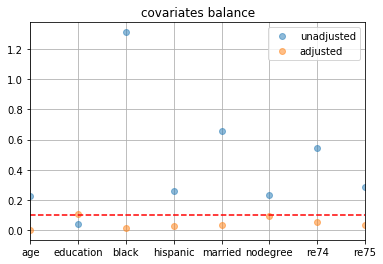
- 共変量の2群間の標準化平均差を確認
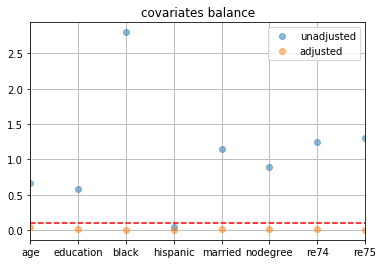
- 本書のグラフと一致しない?(特に調整前のblack)
pd.DataFrame({ 'Unadjusted': [np.subtract(*df_cps1_nsw.groupby(z_column)[c].mean()) for c in X_columns], 'Adjusted': [pairs1_cps1_nsw['l'].map(df_cps1_nsw[c]).mean() - pairs1_cps1_nsw['r'].map(df_cps1_nsw[c]).mean() for c in X_columns], }, index=X_columns).abs().divide(df_cps1_nsw[X_columns].std(), axis=0).plot(linestyle='', marker='o', alpha=0.5, grid=True) plt.axhline(y=0.1, linestyle='--', c='r')
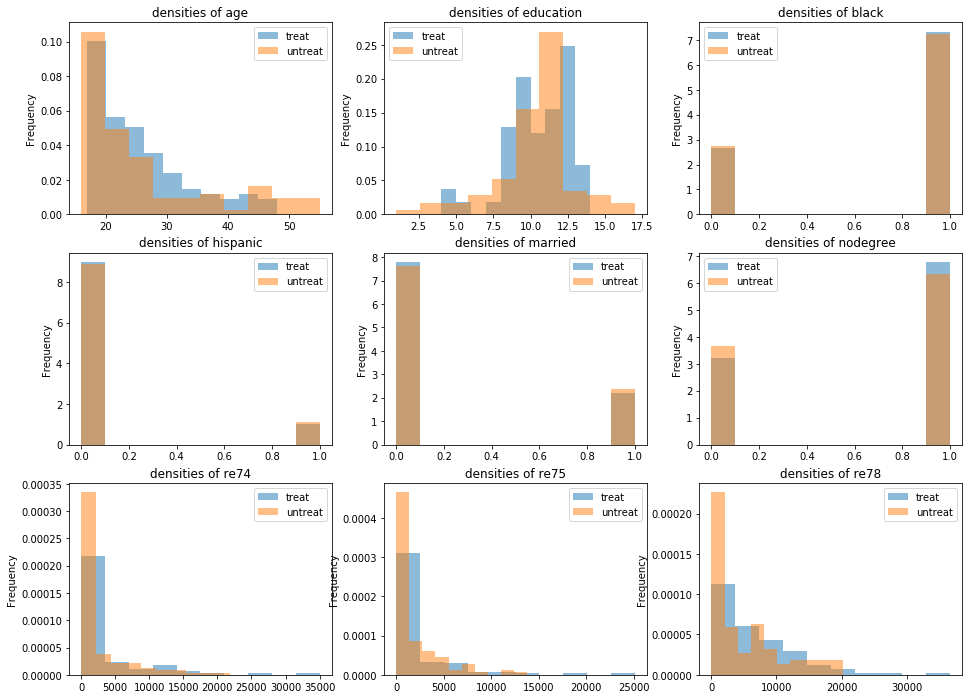
- 共変量の分布を確認
_, ax = plt.subplots(3, 3, figsize=(16, 12)) for i, c in enumerate(X_columns + [y_column]): (df_cps1_nsw.loc[pd.concat([pairs1_cps1_nsw['l'], pairs1_cps1_nsw['r']])][c].groupby(df_cps1_nsw[z_column].map(treat_label)) .plot.hist(density=True, alpha=0.5, title=f'densities of {c}', legend=True, ax=ax[i // 3, i % 3]))IPW
- 重みを計算
df_cps1_nsw['w'] = df_cps1_nsw[z_column] / df_cps1_nsw['ps'] + (1 - df_cps1_nsw[z_column]) / (1 - df_cps1_nsw['ps'])
- 重み付き線形回帰でATTを推定
att_model = sm.WLS(df_cps1_nsw[y_column], df_cps1_nsw[[z_column]].assign(untreat=1-df_cps1_nsw[z_column]), weights=df_cps1_nsw['w'] * df_cps1_nsw['ps']).fit() att_model.summary().tables[1]att_model.params['treat'] - att_model.params['untreat']1180.4078220960955
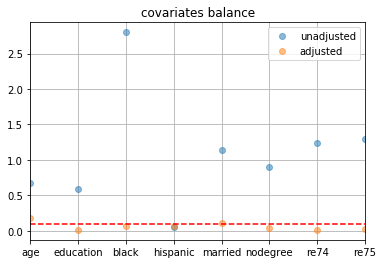
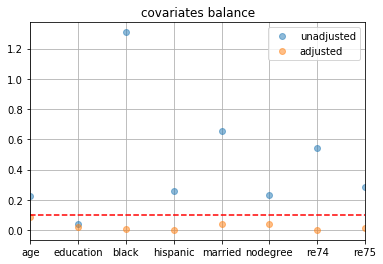
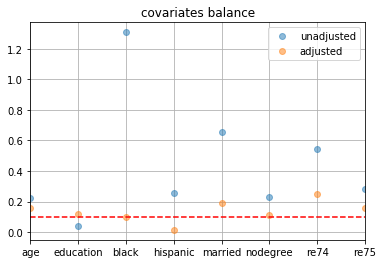
- 共変量の2群間の標準化平均差を確認
pd.DataFrame({ 'unadjusted': [np.subtract(*df_cps1_nsw.groupby(z_column)[c].mean()) for c in X_columns], 'adjusted': [np.subtract(*df_cps1_nsw.groupby(z_column).apply(lambda x: np.average(x[c], weights=x['w'] * x['ps']))) for c in X_columns], }, index=X_columns).abs().divide(df_cps1_nsw[X_columns].std(), axis=0).plot(linestyle='', marker='o', alpha=0.5, grid=True, title='covariates balance') plt.axhline(y=0.1, linestyle='--', c='r')
- 共変量の分布を確認
- 後日追記予定
IPW(コモンサポート)
- 傾向スコアの分布が重なっている範囲のサンプルを抽出
df_cps1_nsw_cs = df_cps1_nsw[df_cps1_nsw['ps'].between(*df_cps1_nsw.groupby(z_column)['ps'].agg(['min', 'max']).agg({'min': 'max', 'max': 'min'}))].copy()
- 重み付き線形回帰でATTを推定
att_model = sm.WLS(df_cps1_nsw_cs[y_column], df_cps1_nsw_cs[[z_column]].assign(untreat=1-df_cps1_nsw_cs[z_column]), weights=df_cps1_nsw_cs['w'] * df_cps1_nsw_cs['ps']).fit() att_model.summary().tables[1]att_model.params['treat'] - att_model.params['untreat']1243.8640545001808
- 共変量の2群間の標準化平均差を確認
pd.DataFrame({ 'unadjusted': [np.subtract(*df_cps1_nsw_cs.groupby(z_column)[c].mean()) for c in X_columns], 'adjusted': [np.subtract(*df_cps1_nsw_cs.groupby(z_column).apply(lambda x: np.average(x[c], weights=x['w'] * x['ps']))) for c in X_columns], }, index=X_columns).abs().divide(df_cps1_nsw_cs[X_columns].std(), axis=0).plot(linestyle='', marker='o', alpha=0.5, grid=True, title='covariates balance') plt.axhline(y=0.1, linestyle='--', c='r')
- 共変量の分布を確認
- 後日追記予定
CPS3+NSW
マッチング
- ペアを作る
s1 = df_cps3_nsw[df_cps3_nsw[z_column] == 1]['ps'] s0 = df_cps3_nsw[df_cps3_nsw[z_column] == 0]['ps'] threshold = df_cps3_nsw['ps'].std() * 0.1 pairs1_cps3_nsw = pd.concat(pairs_generator(s1, s0, threshold), ignore_index=True) pairs0_cps3_nsw = pd.concat(pairs_generator(s0, s1, threshold), ignore_index=True) # ATEの計算で使う
- 介入群のペアを確認
df_cps3_nsw.plot.scatter(x='ps', y=z_column, alpha=0.3, figsize=(16, 4), title='pairs matched by propensity score on treated') for x in zip(df_cps3_nsw['ps'].iloc[pairs1_cps3_nsw['l']].values, df_cps3_nsw['ps'].iloc[pairs1_cps3_nsw['r']].values): plt.plot(x, [1, 0], c='tab:blue', alpha=0.3)
- 非介入群のペアを確認
- これらのペアはATEの計算で使う
df_cps3_nsw.plot.scatter(x='ps', y=z_column, alpha=0.3, figsize=(16, 4), title='pairs matched by propensity score on untreated') for x in zip(df_cps3_nsw['ps'].iloc[pairs0_cps3_nsw['l']].values, df_cps3_nsw['ps'].iloc[pairs0_cps3_nsw['r']].values): plt.plot(x, [0, 1], c='tab:blue', alpha=0.3)
- ATTを計算
(pairs1_cps3_nsw['l'].map(df_cps3_nsw[y_column]) - pairs1_cps3_nsw['r'].map(df_cps3_nsw[y_column])).agg(['mean', 'std'])mean 1463.115845 std 8779.598633 dtype: float64
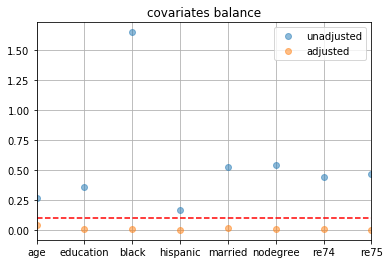
- 共変量の2群間の標準化平均差を確認
pd.DataFrame({ 'unadjusted': [np.subtract(*df_cps3_nsw.groupby(z_column)[c].mean()) for c in X_columns], 'adjusted': [pairs1_cps3_nsw['l'].map(df_cps3_nsw[c]).mean() - pairs1_cps3_nsw['r'].map(df_cps3_nsw[c]).mean() for c in X_columns], }, index=X_columns).abs().divide(df_cps3_nsw[X_columns].std(), axis=0).plot(linestyle='', marker='o', alpha=0.5, grid=True, title='covariates balance') plt.axhline(y=0.1, linestyle='--', c='r')
- 共変量の分布を確認
_, ax = plt.subplots(3, 3, figsize=(16, 12)) for i, c in enumerate(X_columns + [y_column]): (df_cps3_nsw.loc[pd.concat([pairs1_cps3_nsw['l'], pairs1_cps3_nsw['r']])][c].groupby(df_cps3_nsw[z_column].map(treat_label)) .plot.hist(density=True, alpha=0.5, title=f'densities of {c}', legend=True, ax=ax[i // 3, i % 3]))IPW
- 重みを計算
df_cps3_nsw['w'] = df_cps3_nsw[z_column] / df_cps3_nsw['ps'] + (1 - df_cps3_nsw[z_column]) / (1 - df_cps3_nsw['ps'])
- 重み付き線形回帰でATTを推定
att_model = sm.WLS(df_cps3_nsw[y_column], df_cps3_nsw[[z_column]].assign(untreat=1-df_cps3_nsw[z_column]), weights=df_cps3_nsw['w'] * df_cps3_nsw['ps']).fit() att_model.summary().tables[1]att_model.params['treat'] - att_model.params['untreat']1214.0711733143507
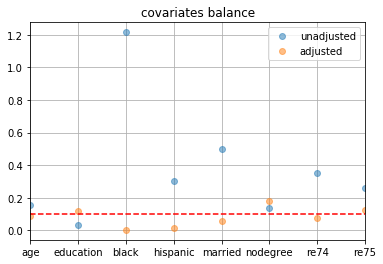
- 共変量の2群間の標準化平均差を確認
pd.DataFrame({ 'unadjusted': [np.subtract(*df_cps3_nsw.groupby(z_column)[c].mean()) for c in X_columns], 'adjusted': [np.subtract(*df_cps3_nsw.groupby(z_column).apply(lambda x: np.average(x[c], weights=x['w'] * x['ps']))) for c in X_columns], }, index=X_columns).abs().divide(df_cps3_nsw[X_columns].std(), axis=0).plot(linestyle='', marker='o', alpha=0.5, grid=True, title='covariates balance') plt.axhline(y=0.1, linestyle='--', c='r')
- 共変量の分布を確認
- 後日追記予定
IPW(コモンサポート)
- 傾向スコアの分布が重なっている範囲のサンプルを抽出
df_cps3_nsw_cs = df_cps3_nsw[df_cps3_nsw['ps'].between(*df_cps3_nsw.groupby(z_column)['ps'].agg(['min', 'max']).agg({'min': 'max', 'max': 'min'}))].copy()
- 重み付き線形回帰でATTを推定
att_model = sm.WLS(df_cps3_nsw_cs[y_column], df_cps3_nsw_cs[[z_column]].assign(untreat=1-df_cps3_nsw_cs[z_column]), weights=df_cps3_nsw_cs['w'] * df_cps3_nsw_cs['ps']).fit() att_model.summary().tables[1]att_model.params['treat'] - att_model.params['untreat']988.8657151185471
- 共変量の2群間の標準化平均差を確認
pd.DataFrame({ 'unadjusted': [np.subtract(*df_cps1_nsw_cs.groupby(z_column)[c].mean()) for c in X_columns], 'adjusted': [np.subtract(*df_cps1_nsw_cs.groupby(z_column).apply(lambda x: np.average(x[c], weights=x['w'] * x['ps']))) for c in X_columns], }, index=X_columns).abs().divide(df_cps1_nsw_cs[X_columns].std(), axis=0).plot(linestyle='', marker='o', alpha=0.5, grid=True, title='covariates balance') plt.axhline(y=0.1, linestyle='--', c='r')
- 共変量の分布を確認
- 後日追記予定
ATEの推定
データセット・推定手法ごとのATE
データセット マッチング IPW IPW(コモンサポート) CPS1+NSW 1836.5 -6456.3 545.3 CPS3+NSW 1486.6 224.7 801.0 CPS1+NSW
マッチング
- ATEを計算
(pd.concat([pairs1_cps1_nsw['l'], pairs0_cps1_nsw['r']], ignore_index=True).map(df_cps1_nsw[y_column]) - pd.concat([pairs1_cps1_nsw['r'], pairs0_cps1_nsw['l']], ignore_index=True).map(df_cps1_nsw[y_column])).agg(['mean', 'std'])mean 1836.516968 std 9634.636719 dtype: float64
- 共変量の2群間の標準化平均差を確認
pd.DataFrame({ 'unadjusted': [np.subtract(*df_cps1_nsw.groupby(z_column)[c].mean()) for c in X_columns], 'adjusted': [pd.concat([pairs1_cps1_nsw['l'], pairs0_cps1_nsw['r']], ignore_index=True).map(df_cps1_nsw[c]).mean() - pd.concat([pairs1_cps1_nsw['r'], pairs0_cps1_nsw['l']], ignore_index=True).map(df_cps1_nsw[c]).mean() for c in X_columns], }, index=X_columns).abs().divide(df_cps1_nsw[X_columns].std(), axis=0).plot(linestyle='', marker='o', alpha=0.5, grid=True, title='covariates balance') plt.axhline(y=0.1, linestyle='--', c='r')
- 共変量の分布を確認
_, ax = plt.subplots(3, 3, figsize=(16, 12)) for i, c in enumerate(X_columns + [y_column]): (df_cps1_nsw.loc[pd.concat([pairs1_cps1_nsw['l'], pairs0_cps1_nsw['r'], pairs1_cps1_nsw['r'], pairs0_cps1_nsw['l']])][c].groupby(df_cps1_nsw[z_column].map(treat_label)) .plot.hist(density=True, alpha=0.5, title=f'densities of {c}', legend=True, ax=ax[i // 3, i % 3]))IPW
- 重み付き線形回帰でATEを推定
ate_model = sm.WLS(df_cps1_nsw[y_column], df_cps1_nsw[[z_column]].assign(untreat=1-df_cps1_nsw[z_column]), weights=df_cps1_nsw['w']).fit() ate_model.summary().tables[1]ate_model.params['treat'] - ate_model.params['untreat']-6456.300804768925
- 共変量の2群間の標準化平均差を確認
pd.DataFrame({ 'unadjusted': [np.subtract(*df_cps1_nsw.groupby(z_column)[c].mean()) for c in X_columns], 'adjusted': [np.subtract(*df_cps1_nsw.groupby(z_column).apply(lambda x: np.average(x[c], weights=x['w']))) for c in X_columns], }, index=X_columns).abs().divide(df_cps1_nsw[X_columns].std(), axis=0).plot(linestyle='', marker='o', alpha=0.5, grid=True, title='covariates balance') plt.axhline(y=0.1, linestyle='--', c='r')
- 共変量の分布を確認
- 後日追記予定
IPW(コモンサポート)
- 重み付き線形回帰でATEを推定
ate_model = sm.WLS(df_cps1_nsw_cs[y_column], df_cps1_nsw_cs[[z_column]].assign(untreat=1-df_cps1_nsw_cs[z_column]), weights=df_cps1_nsw_cs['w']).fit() ate_model.summary().tables[1]ate_model.params['treat'] - ate_model.params['untreat']545.3149727568589
- 共変量の2群間の標準化平均差を確認
pd.DataFrame({ 'unadjusted': [np.subtract(*df_cps1_nsw_cs.groupby(z_column)[c].mean()) for c in X_columns], 'adjusted': [np.subtract(*df_cps1_nsw_cs.groupby(z_column).apply(lambda x: np.average(x[c], weights=x['w']))) for c in X_columns], }, index=X_columns).abs().divide(df_cps1_nsw_cs[X_columns].std(), axis=0).plot(linestyle='', marker='o', alpha=0.5, grid=True, title='covariates balance') plt.axhline(y=0.1, linestyle='--', c='r')
- 共変量の分布を確認
- 後日追記予定
CPS3+NSW
マッチング
- ATEを計算
(pd.concat([pairs1_cps3_nsw['l'], pairs0_cps3_nsw['r']], ignore_index=True).map(df_cps3_nsw[y_column]) - pd.concat([pairs1_cps3_nsw['r'], pairs0_cps3_nsw['l']], ignore_index=True).map(df_cps3_nsw[y_column])).agg(['mean', 'std'])mean 1486.608276 std 8629.639648 dtype: float64
- 共変量の2群間の標準化平均差を確認
pd.DataFrame({ 'unadjusted': [np.subtract(*df_cps3_nsw.groupby(z_column)[c].mean()) for c in X_columns], 'adjusted': [pd.concat([pairs1_cps3_nsw['l'], pairs0_cps3_nsw['r']], ignore_index=True).map(df_cps3_nsw[c]).mean() - pd.concat([pairs1_cps3_nsw['r'], pairs0_cps3_nsw['l']], ignore_index=True).map(df_cps3_nsw[c]).mean() for c in X_columns], }, index=X_columns).abs().divide(df_cps3_nsw[X_columns].std(), axis=0).plot(linestyle='', marker='o', alpha=0.5, grid=True, title='covariates balance') plt.axhline(y=0.1, linestyle='--', c='r')
- 共変量の分布を確認
_, ax = plt.subplots(3, 3, figsize=(16, 12)) for i, c in enumerate(X_columns + [y_column]): (df_cps1_nsw.loc[pd.concat([pairs1_cps1_nsw['l'], pairs0_cps1_nsw['r'], pairs1_cps1_nsw['r'], pairs0_cps1_nsw['l']])][c].groupby(df_cps1_nsw[z_column].map(treat_label)) .plot.hist(density=True, alpha=0.5, title=f'densities of {c}', legend=True, ax=ax[i // 3, i % 3]))IPW
- 重み付き線形回帰でATEを推定
ate_model = sm.WLS(df_cps3_nsw[y_column], df_cps3_nsw[[z_column]].assign(untreat=1-df_cps3_nsw[z_column]), weights=df_cps3_nsw['w']).fit() ate_model.summary().tables[1]ate_model.params['treat'] - ate_model.params['untreat']224.6762634665365
- 共変量の2群間の標準化平均差を確認
pd.DataFrame({ 'unadjusted': [np.subtract(*df_cps3_nsw.groupby(z_column)[c].mean()) for c in X_columns], 'adjusted': [np.subtract(*df_cps3_nsw.groupby(z_column).apply(lambda x: np.average(x[c], weights=x['w']))) for c in X_columns], }, index=X_columns).abs().divide(df_cps3_nsw[X_columns].std(), axis=0).plot(linestyle='', marker='o', alpha=0.5, grid=True, title='covariates balance') plt.axhline(y=0.1, linestyle='--', c='r')
- 共変量の分布を確認
- 後日追記予定
IPW(コモンサポート)
- 重み付き線形回帰でATEを推定
ate_model = sm.WLS(df_cps3_nsw_cs[y_column], df_cps3_nsw_cs[[z_column]].assign(untreat=1-df_cps3_nsw_cs[z_column]), weights=df_cps3_nsw_cs['w']).fit() ate_model.summary().tables[1]ate_model.params['treat'] - ate_model.params['untreat']801.0736196669177
- 共変量の2群間の標準化平均差を確認
pd.DataFrame({ 'unadjusted': [np.subtract(*df_cps3_nsw_cs.groupby(z_column)[c].mean()) for c in X_columns], 'adjusted': [np.subtract(*df_cps3_nsw_cs.groupby(z_column).apply(lambda x: np.average(x[c], weights=x['w']))) for c in X_columns], }, index=X_columns).abs().divide(df_cps3_nsw_cs[X_columns].std(), axis=0).plot(linestyle='', marker='o', alpha=0.5, grid=True, title='covariates balance') plt.axhline(y=0.1, linestyle='--', c='r')
- 共変量の分布を確認
- 後日追記予定
所感
- 全然「バイアス」を取り除けてない…
本書では傾向スコアは$P(X)$と表記されているが、便宜上$e(X)$とする ↩
少し変な比較だが、本書からはこのように読み取れる ↩
星野崇宏(2009),調査観察データの統計科学,岩波書店. ↩
傾向スコアの文脈でしか見たことがないが、回帰分析でもSUTVAを仮定する? ↩
Paul R. Rosenbaum and Donald B. Rubin. "The Central Role of the Propensity Score in Observational Studies for Causal Effects." Biometrika, Vol. 70, pp. 41-55. ↩
https://healthpolicyhealthecon.com/2015/05/07/propensity-score-2/ ↩
門脇大輔,阪田隆司,保坂桂佑,平松雄司(2019),Kaggleで勝つデータ分析の技術,技術評論社. ↩
傾向スコアを使った介入効果の推定手法はマッチングとIPW以外にもあるが、本書ではこの2つを取り扱っている ↩
https://healthpolicyhealthecon.com/2015/05/04/propensity-score-1/ ↩
https://speakerdeck.com/tomoshige_n/causal-inference-and-data-analysis ↩
岩波データサイエンス刊行委員会(2016),岩波データサイエンス Vol.3,岩波書店. ↩
https://blog.minethatdata.com/2008/03/minethatdata-e-mail-analytics-and-data.html ↩
- 投稿日:2020-02-27T18:22:41+09:00
Google Edge TPU推論の概要
Edge TPUはTensorFlow Liteモデルのみと互換性があります。 そのため、TensorFlowモデルをトレーニングし、TensorFlow Liteに変換し、Edge TPU用にコンパイルする必要があります。 次に、このページで説明されているオプションのいずれかを使用して、Edge TPUでモデルを実行できます。(Edge TPUと互換性のあるモデルの作成の詳細については、Edge TPUのTensorFlowモデルをご覧ください。https://coral.ai/docs/edgetpu/models-intro/)
Pythonで推論を実行する
Pythonを使用して推論を実行している場合、2つのオプションがあります。
TensorFlow Lite APIを使用します:
これは、TensorFlow Liteモデルを実行する従来のアプローチです。 データの入出力を完全に制御できるため、さまざまなモデルアーキテクチャで推論を実行できます。
TensorFlow Liteを以前に使用したことがある場合、Interpreterコードはごくわずかな変更を加えるだけでエッジTPUでモデルを実行できます。(詳細については、PythonでTensorFlow Liteを使用して推論を実行するをご覧ください。https://coral.ai/docs/edgetpu/tflite-python/)Edge TPU APIを使用します:
これは、
TensorFlow Lite C ++ APIの上に構築されたPythonライブラリであるため、画像分類モデルとオブジェクト検出モデルを使用して、より簡単に推論を実行できます。
このAPIは、TensorFlow Lite APIの経験がなく、単に画像分類またはオブジェクト検出を実行する場合に役立ちます。入力テンソルの準備と結果の解析に必要なコードを抽象化するためです。 また、Edge TPUで分類モデルの高速転送学習を実行する独自のAPIも提供します。(詳細については、Edge TPU Python APIの概要をご覧ください。https://coral.ai/docs/edgetpu/api-intro/)C ++で推論を実行する
C ++でコードを記述したい場合は、他のプラットフォームでTensorFlow Liteを実行するのと同じように、
TensorFlow Lite C ++ APIを使用する必要があります。 ただし、edgetpu.hまたはedgetpu_c.hファイルのAPIを使用して、コードにいくつかの変更を加える必要があります。 基本的に、Interpreterオブジェクトの外部コンテキストとしてEdge TPUデバイスを登録するだけです。(詳細については、「C ++でTensorFlow Liteで推論を実行する」を参照してください。https://coral.ai/docs/edgetpu/tflite-cpp/)Coral Acceleratorモジュールは、2020年初頭にCoral Webサイトで販売されます。Google Coral製品、および大量販売または大量販売(ボリュームディスカウント)の詳細に興味がある場合は、ようこそCoral 海外代理店 Gravitylink : https://store.gravitylink.com/global
- 投稿日:2020-02-27T18:20:44+09:00
FlaskでHelloWorld【適当メモ】
- 投稿日:2020-02-27T17:06:04+09:00
AtCoder Beginner Contest 045 過去問復習
所要時間
感想
D問題は(実装が下手ですが)うまくいきました。
しかし、B,C問題でコーナーケースを間違えるなど3WAを出したので反省して次に生かしたいと思います。A問題
面積求めるだけ。
answerA.pya=input() b=input() h=input() print((int(a)+int(b))*int(h)//2)B問題
シミュレーションをしていけば良い。
while文の中でどのような状態になっているかを正確に捉える。
ここでは、そのターンがどの人であるか、その人がどのアルファベットのカードを持っているか、を考える。answerB.pys=[input() for i in range(3)] l=[len(s[i]) for i in range(3)] abc=[0,0,0] now=0 while abc[now]!=l[now]: s_sub=s[now][abc[now]] abc[now]+=1 now=(0 if s_sub=="a" else 1 if s_sub=="b" else 2) print("A" if now==0 else "B" if now==1 else "C")C問題
文字列の長さをlとした時+が入りうる箇所はl-1箇所であり、lが十分に小さいことから$2^{l-1}$のパターンを全て考えることができます(bit全探索)。したがって、あとは+の入る場所がわかっている状態での足し算を考えます。ここで、数字の文字列を前から順に見ると、+がi文字目とi+1文字目の間に入る時はその左側の数と右側の数の足し算を考え、+がi文字目とi+1文字目の間に入らない時はその左側の数と右側の数字の文字列としての和を考えれば良いです。
したがって、bit全探索を書いて、数字の間を順に見ていき、j番目(j=0→l-2)に+が入る場合はその時点で保存されている数字の列(subst)を整数に直しans_subに加え(j+1番目の数をsubstにするのも忘れずに)、j番目(j=0→l-2)に+が入らない場合はsubstに文字列として足し算を行えば、以下のようなコードになります。(bit全探索のコードで1かどうかの判定を(i>>j)&1にするか(1<<j)&iにするかで迷うので統一したいです…。)追記
後から気づいたのですが、+を挟んで行って+を基準に最後にsplitすればコード書く量は減りますね…。
answerC.pys=input() l=len(s) ans=0 for i in range(2**(l-1)): ans_sub=0 subst=s[0] for j in range(l-1): if (i>>j)&1: ans_sub+=int(subst) subst=s[j+1] else: subst+=s[j+1] if j==l-2: ans_sub+=int(subst) ans+=ans_sub if l==1: print(int(s)) else: print(ans)D問題
初めは全てのマス目について調べていけば良いと感じましたが、O(HW)なので間に合うわけがありません。ここで、塗りつぶすマス目の数に注目すると10^5以下なので塗りつぶすことを中心に考えればプログラムを書けそうなことがわかります。ここで、あるマス目を塗りつぶした時、隣り合うマス目を中心とする$3 \times 3$の盤面にそのマス目は含まれることが分かります。
したがって、その隣り合うマス目(のうち$3 \times 3$の盤面を作るマス目)を保存し、そのマス目を中心とする$3 \times 3$の盤面に含まれるようなマス目が出てくるたびに更新を行えば良いです。また、中心の座標を全て保存するのは空間計算量的に無理なので、出てきた座標のみを辞書に保存するようにしています(一回も出てこないマス目は全ての(中心となりうる)マス目の数$(h-2) \times (w-2)$から出てきた座標を引けば良いです。)。answerD.pyh,w,n=map(int,input().split()) d=dict() def change(a,b): global h,w,d if 1<=a<h-1 and 1<=b<w-1: if (a,b) in d: d[(a,b)]+=1 else: d[(a,b)]=1 for i in range(n): a,b=map(int,input().split()) a-=1 b-=1 for j in range(3): for k in range(3): change(a+j-1,b+k-1) ans=[0]*10 ans[0]=(h-2)*(w-2) for i in d: ans[d[i]]+=1 ans[0]-=1 for i in range(10): print(ans[i])
- 投稿日:2020-02-27T16:42:50+09:00
AtCoder Beginner Contest 046 過去問復習
所要時間
感想
D問題は発想ゲーでそこまで難しくなかったので良かったのですが、C問題でceil関数により誤差が出てしまいかなり時間を食ってしまいました。
A問題
a,b,cが全て同じ場合を先に除くと場合分けが楽。
answerA.pya,b,c=map(int,input().split()) if a==b and b==c: print(1) elif a==b or b==c or c==a: print(2) else: print(3)B問題
前から順に色を決めていけば良い。
一番初めのみk通りで、それ以降は直前の色以外を用いればよくk-1通りになる。answerB.pyn,k=map(int,input().split()) print(k*((k-1)**(n-1)))C問題
i回目の得票数の比から、実際の得票数はTi*x,Ai*x(xは1以上の整数)であり、直前の投票数をT,Aとした時、Ti*x>=T,Ai*x>=A、が成り立てば良いです。したがって、xはceil(T/Ti),ceil(A/Ai)の大きい方になります。
しかし、Pythonでは割り算の結果がintではなくfloat型になるので大きい数字の際に誤差が生じることになります。そのため、ここではceil関数を用いてはなりません。
したがって、ceil(X/Y)の代わりに-((-X)//Y)を用いることで誤差を出さずに計算を行うことができます。answerC.pyn=int(input()) nt,na=map(int,input().split()) for i in range(n-1): t,a=map(int,input().split()) #xを求める部分で誤差が出る #x=max(ceil(nt/t),ceil(na/a)) x=-min((-nt)//t,(-na)//a) nt,na=x*t,x*a print(nt+na)D問題
まず、パーをできるだけ多く出したい(n-n//2回)ので、そのような方法を考えるとグーパーグーパーグーパー…と交互に出す方法がもっとも多く出す方法としてあげられます。僕は野暮用があってあまり時間が取れず、バチャコン中はこの適当な考察で提出しました。
グーとパーの出す回数は決まったので、グーとパーの出す順番が得点に関係ないことを考察します。
手の出し方を考えれば、グーを相手が出している際にパーを出した場合は+1グーを出した場合は0、パーを相手が出している際にパーを出した場合は0グーを出した場合は-1となります。つまり、いずれの場合でも、自分がグーを出す場合はパーを出す場合と比べて-1の影響を全体に与えることになります。
よって、グーとパーの出す回数が決まった時、得点はグーとパーの出す順番に関係ないと言えます。(感覚的に書いているので詳しくは解答を参照してください。)以上を実装すると以下のようになります(グーパーグーパーグーパー…と交互に出す際の得点を数え上げるだけです。)。
answerD.pys=input() n=len(s) ans=0 for i in range(n): if i%2==0: if s[i]=="p": ans-=1 else: if s[i]=="g": ans+=1 print(ans)
- 投稿日:2020-02-27T15:33:37+09:00
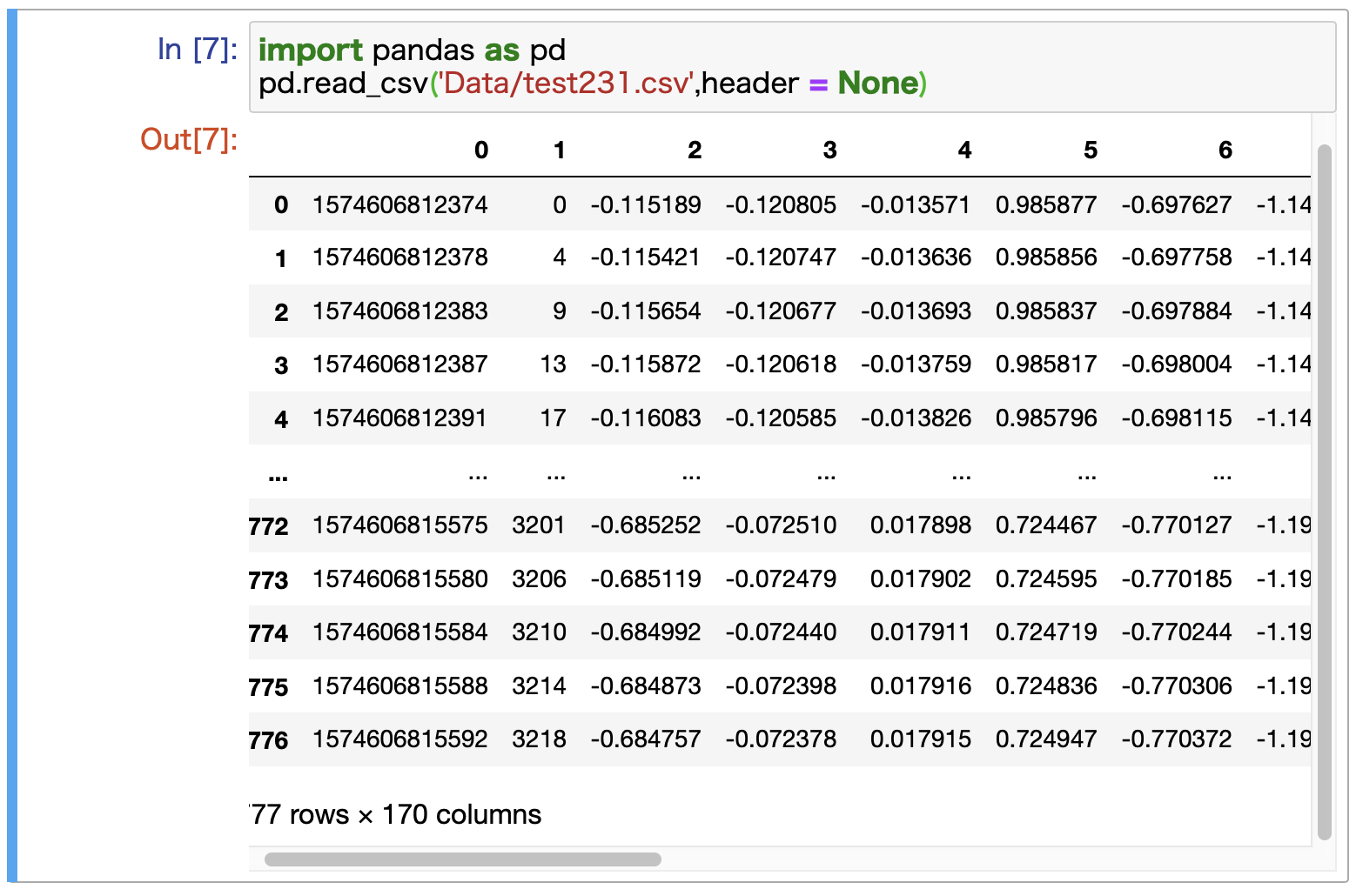
pandasでcsvの読み込みをしてIndexをいじってみる
はじめに
構造化データを扱うのにpandasのDataFrameが良い!と風の噂で聞きました(本当は書店で立ち読みしたデータサイエンスの本で読みました).
超初心者の覚書としてメモを残しておきたいと思います.環境
Python3.6.10
pandas-1.0.1
Jupyter notebookまずはインストール
難しいことは何もなかった...
pip install pandascsvファイルの読み込み
今回扱いたいデータがcsv形式だったので,csvファイルを読み込んでいきます.
少々調べたところ,pandasにはSeries,DataFrameという二つのデータ構造があり,Seriesが一次元データ,DataFrameが二次元データに対応するらしいということがわかりました(SeriesとDataFrameについては理解が甘々なので勉強し直して別の記事にできたらいいな)とりあえず今回は,csvデータをDataFrameとして読んでいきたいと思います.
import pandas as pd pd.read_csv('データパス',header = None)区切り文字がカンマのcsvファイルを読みたかったので,read_csv()を使いました.
区切り文字がタブ(\t)の場合はread_table()を代わりに使用できるそうです.また,読みたいcsvファイルには見出し行が存在しないので,headerにNoneを指定しています.
実際に読み込んだデータは,jupyter上では下図のように表示されました↓
データの整形
読み込んだデータを次のように整形していきます
・0列目は不要なので削除
・1列目をインデクス(見出し列)にする列の削除
スライスで0列目だけ切り落としていきます.
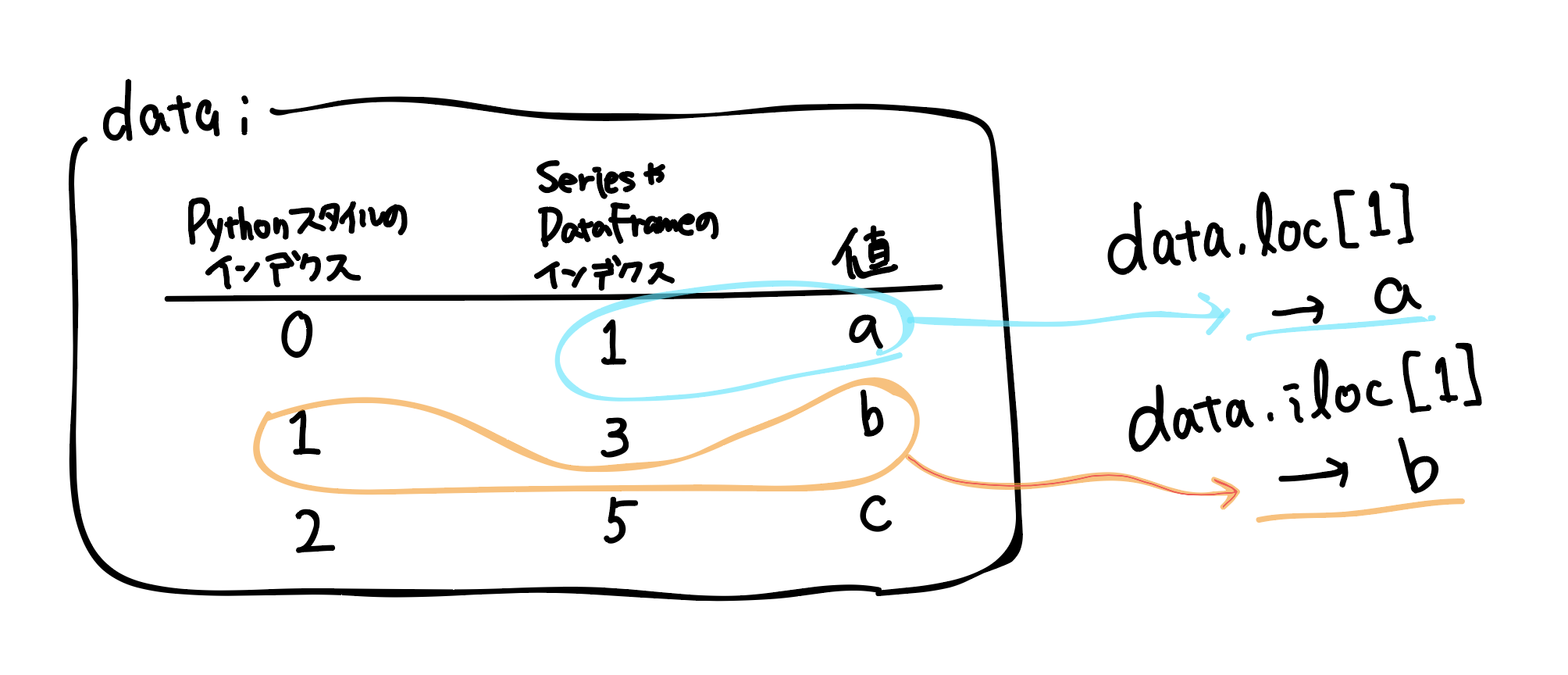
SeriesやDataFrameではインデクスに文字列を指定することもできますし,(任意の)数値を指定することもできます.特に,インデクスに数値を指定した際の混乱を避けるため,ilocやlocといったインデクス属性を用いてデータにアクセスします.
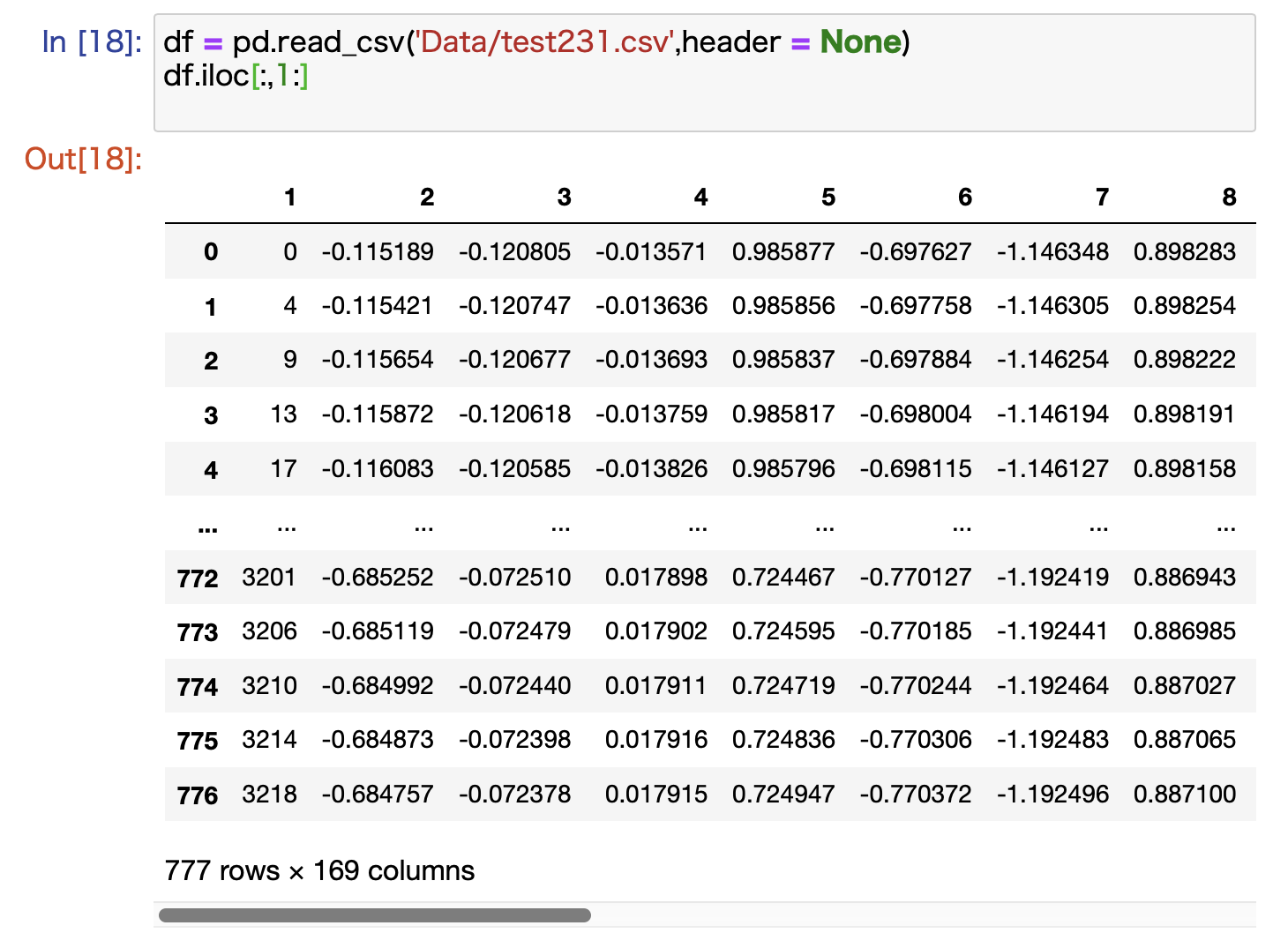
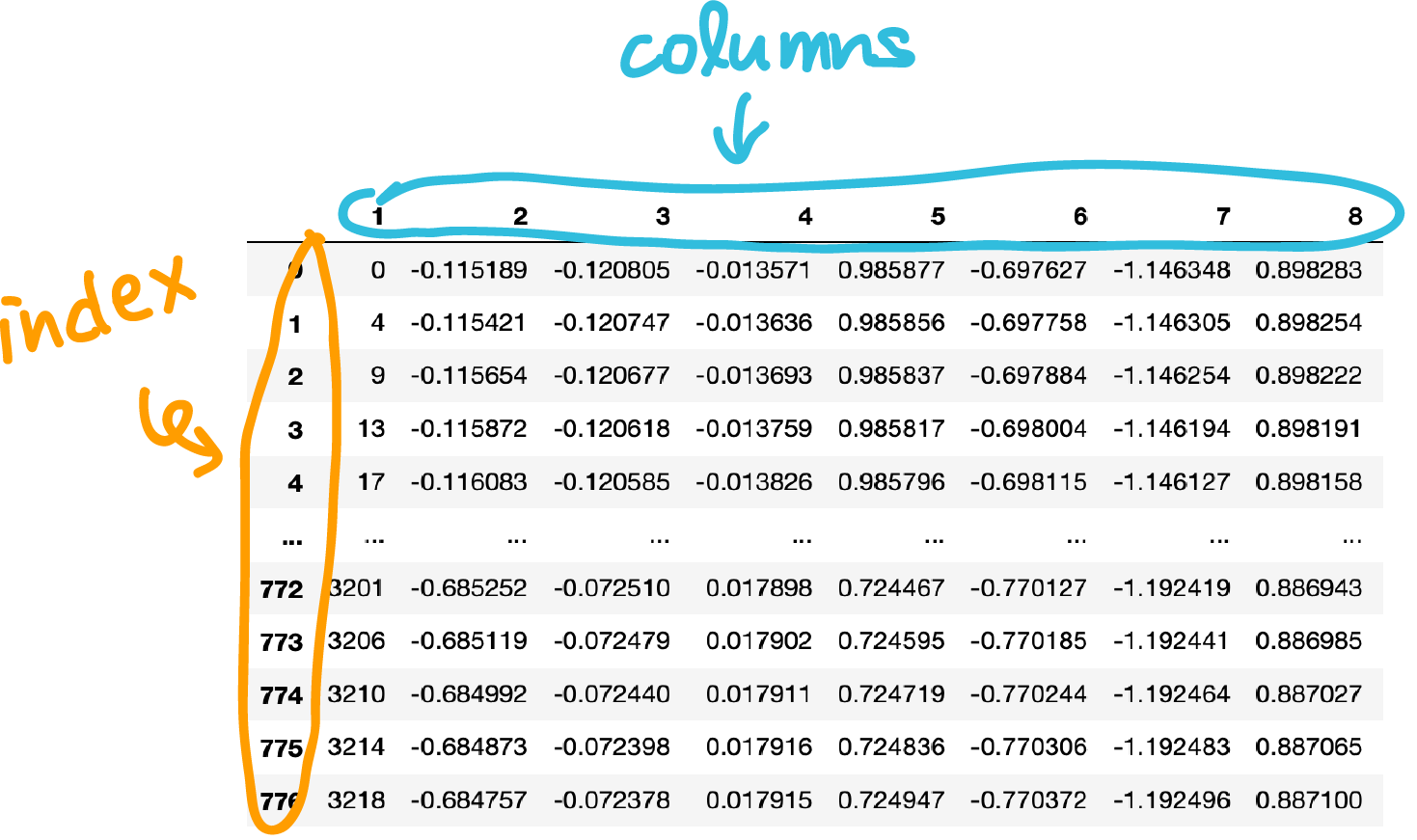
今回は(今のところ),DataFrameのインデクスは行・列の両方とも,Pythonスタイルのインデクスと一致しているので,ilocとlocのどちらを用いても同じ結果が得られます.次のようにしてデータの0行目を切り落としました(1行目以降のみ取り出しました)
#csvを読み込む df = pd.read_csv('Data/test231.csv',header = None) #スライスする df.iloc[:,1:]前の画像と見比べて列(columns)数が170から169になっていて,先頭一列のみが減っていると確認できました.
1列目をインデクスにする
pandasのDataFrameでは,行見出しのことをindex,列見出しのことをcolumnsと呼んでいるらしい.
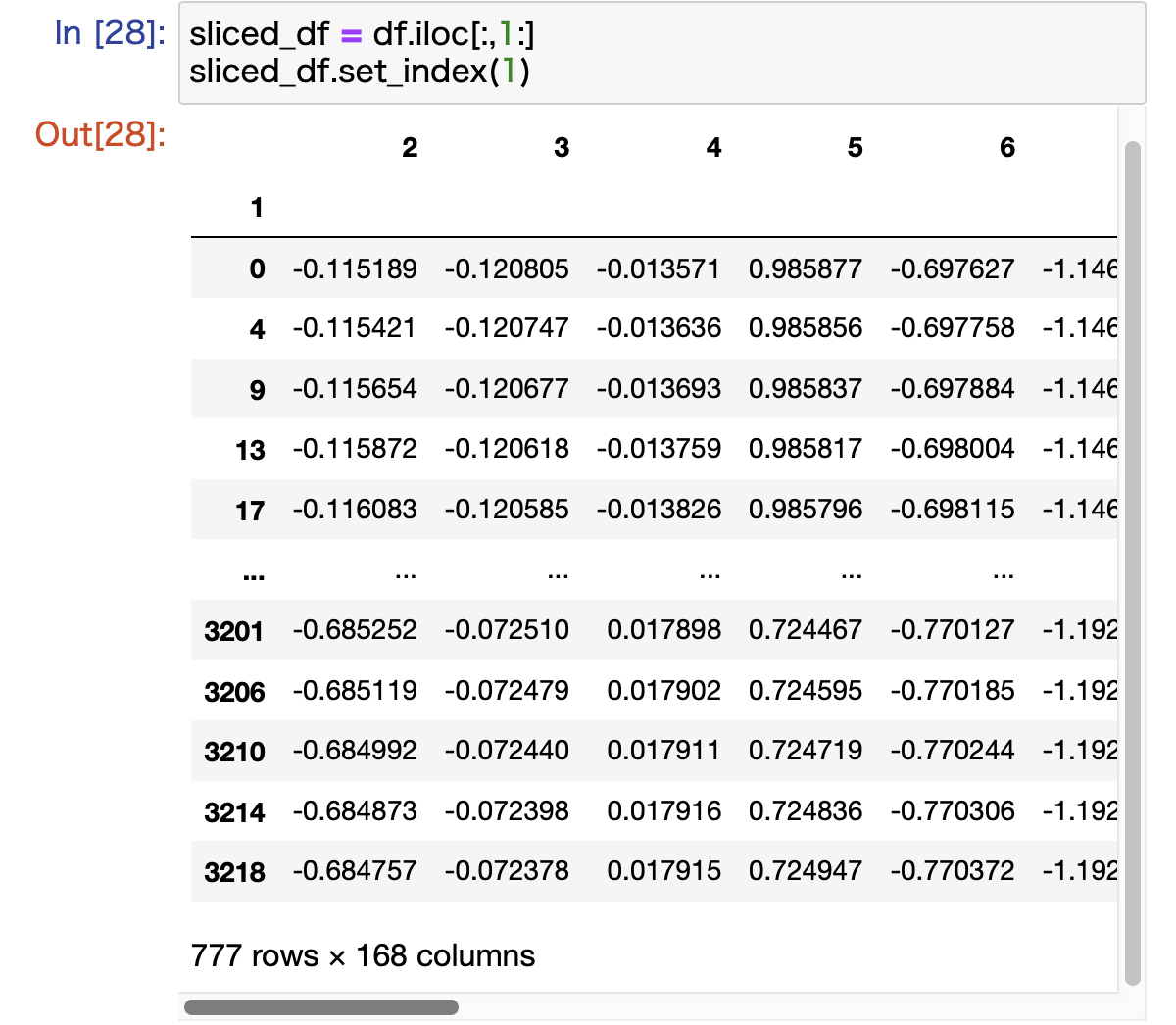
次のようにすることで,上の図で"1"という名前のついているcolumnをindexに指定することができました.
#スライスする sliced_df = df.iloc[:,1:] #名前が"1"のcolumnをindexに指定する sliced_df.set_index(1)下のように実行できました
おわりに
次はcolumnnの名前を変えたり,このテーブルを階層データとして処理していく記録を覚書として記事にしたいと考えています.
- 投稿日:2020-02-27T14:59:00+09:00
【boto3】EC2インスタンスをタグでフィルタリング
boto3のresourceでEC2をNameタグでフィルタリングしたい場合、
tag:(タグキー)という形式で指定する。
Nameタグがweb-server01のインスタンスは下記のように取得。ec2_r = boto3.resource('ec2') inst_resources = ec2_r.instances.filter( Filters=[ {'Name': 'tag:Name', 'Values': 'web-server01' } ])
Systemタグがwebの場合は下記のように。inst_resources = ec2_r.instances.filter( Filters=[ {'Name': 'tag:System', 'Values': 'web' } ])
- 投稿日:2020-02-27T13:44:57+09:00
windowsでanaconda環境をコマンドプロンプトからでも使用可能にする
まずはAnacondaのインストール
- Anacondaからpython3.7の方のディストリビューションをダウンロードする.
- ダウンロードしたものを実行するとGUIインストーラーが出てくるが,特にこだわりがなければデフォルトのままポチポチしていってインストール完了
PyCharmとかJupyter使うならそれらで個別にanacondaインタプリタにpathを通すのでここまでで終わりの話.
でもcmdでpython -vってしてもエラーが出るはず.pathが通ってないから.Pathを通す
コントロールパネルから以下のpathを通す
C:\Users\hoge\Anaconda3 C:\Users\hoge\Anaconda3\Scripts C:\Users\hoge\Anaconda3\Library\binおしまい
多分これで,cmdからでも
import numpy`とかできるようになってると思います.
追加で使いたいモジュールがあれば,conda(pipより便利)でインストールしてください.
Anaconda でよく使うコマンド一覧最後に
(PyCharmなど便利なIDEがあるこのご時世に)cmdでanaconda環境が使用できて何が嬉しいか説明します.
超小さなローカルアプリを作るとします.
ホスティングもしないし,DBも構うつもりもないとするとき,データはcsvファイルなどでローカルで扱うと楽です.
そのときに,javascriptやphpよりpythonやcでロジックを書いた方が幸せという人は少なくないと思います(フロントのことを考えなくて良いから).フロントとのつなぎ方としてはjs上でサブルーチン走らせてそこでpythonプログラムを実行すれば良い感じになります.
そのpythonを走らせるときにプログラムでnumpyやpandasなどのモジュールを使用する際,cmdでanaconda環境が使えないとエラーが出ます.
サブルーチンはcmdだからです.
って感じで,「サービス・アプリ」なんて荘厳な名前をつけるのは憚られるような超内輪ローカルおもちゃを開発する際に重宝されるtipsの紹介でした.
- 投稿日:2020-02-27T12:27:56+09:00
PythonでWindowsのGUIを操作するためのツールまとめ
WebであればSeleniumなど有名所がありますが、WindowsのGUIを操作するためのツールのデファクトはまだない印象です。
実際には選択肢はたくさんあるので、ここでまとめておきます。
PythonでWindowsのGUIを操作するためのツール
ahk
Python向けの、AutoHotkeyラッパーです。
上記PyPIに載っているサンプルが以下。
from ahk import AHK from ahk.window import Window ahk = AHK() win = ahk.active_window # Get the active window win = ahk.win_get(title='Untitled - Notepad') # by title win = list(ahk.windows()) # list of all windows win = Window(ahk, ahk_id='0xabc123') # by ahk_id win = Window.from_mouse_position(ahk) # the window under the mouse cursor win = Window.from_pid('20366') # by process IDウィンドウタイトルやidで対象を指定して操作できます。
PyAutoIt
こちらはPython用のAutoItのラッパーです。
AutoHotKeyと似たような書き方で操作できます。
(※AutoHotKey自体が元はAutoItから分かれたツールです)autoit
AutoItと関係あるのか正直不明ですが、中身的には自前でがんばって実装しているようです。
SikuliX
わりと有名なほうだと思いますSikuliX。IDEがついていてSikuli単体でコードを書くこともできますが、Pythonのモジュールとして読み込んで使うパターンもあります。
pynput
moses-palmer/pynput: Sends virtual input commands
マウス操作とキーボード操作を自動化できるツールで、画面上のElementを取得して云々、はできない模様。
pyautogui
こちらもマウス操作やキーボード操作が自動化できますが、マルチプラットフォーム=Windows, Mac, Linuxが操作できます。
Pywinauto
同種の他のモジュールに比べても、コミット数などが多いため活発な印象です。
公式にあるサンプルが以下です。
from pywinauto import Desktop, Application Application().start('explorer.exe "C:\\Program Files"') # connect to another process spawned by explorer.exe # Note: make sure the script is running as Administrator! app = Application(backend="uia").connect(path="explorer.exe", title="Program Files") app.ProgramFiles.set_focus() common_files = app.ProgramFiles.ItemsView.get_item('Common Files') common_files.right_click_input() app.ContextMenu.Properties.invoke() # this dialog is open in another process (Desktop object doesn't rely on any process id) Properties = Desktop(backend='uia').Common_Files_Properties Properties.print_control_identifiers() Properties.Cancel.click() Properties.wait_not('visible') # make sure the dialog is closedProgramFilesやItemsViewなどの部分、可読性も高く良い感じです。
WinAppDriver
microsoft/WinAppDriver: Windows Application Driver
SeleniumのようにWindowsのGUIを操作できる、Microsoft製のツールです。
サンプルコードみても、「Seleniumっぽさ」がわかります。
Windowsの電卓の操作です。def test_combination(self): self.driver.find_element_by_name("Seven").click() self.driver.find_element_by_name("Multiply by").click() self.driver.find_element_by_name("Nine").click() self.driver.find_element_by_name("Plus").click() self.driver.find_element_by_name("One").click() self.driver.find_element_by_name("Equals").click() self.driver.find_element_by_name("Divide by").click() self.driver.find_element_by_name("Eight").click() self.driver.find_element_by_name("Equals").click() self.assertEqual(self.getresults(),"8")ただ、日本語環境での動作がいまいちな印象で、なかなか解消されません。。
その他
パット見でいまひとつかも・・・?と思ったものの、念の為リストアップ
- 投稿日:2020-02-27T11:52:32+09:00
【自分用メモ】python OpenCVでターミナル再起動のたびにimport cv2が"No module named cv2"になる時の対策
症状
MacのCatalinaでpython OpenCVの環境構築したはずが、ターミナル再起動のたびに
import cv2するとImportError: No module named cv2エラーの原因
pythonのバージョンが2.7.16になってる
python -Vした時に2.7.16って出たらアウト対策
ターミナルで
source ~/.bashrc
python -Vした時に3.8.1って出たらOK治る理由
多分~
~/.bash_profileか~/.bashrcか~/etc/pathsあたりの
記述内容が間違ってて、インストールした最新バージョンじゃなくて
デフォルトでインストールしてある方を指定しちゃってる。
source ~/.bashrcで最新のバージョンを指定し直してくれるから治る恒久対策
上記の3ファイルのうちいずれかを正しく記述すれば多分治る
記述方法を知ることができれば。。
- 投稿日:2020-02-27T11:35:05+09:00
Python学習1秒の初学者が作る「Twitterで特定のアカウントをRTした人を探すやつ」
MS Accessを持っている人向けのアンチョコ的な記事です。
持ってない人はDB操作部分を消してCSVファイルをいじってみて下さい。まずはコード
# -*- coding:utf-8 -*- #インポートするライブラリ。 この辺りでエラーが出る場合にはコンソール上で「pip install ライブラリ名」を打ち込む。(例:pip install tweepy) import csv import tweepy import pyodbc import time import datetime # APIの秘密鍵 CK = '****' # コンシューマーキー CKS = '****' # コンシューマーシークレット AT = '****' # アクセストークン ATS = '****' # アクセストークンシークレット #メインルーチン def main(total): conn_str = 'Driver={{Microsoft Access Driver (*.mdb, *.accdb)}};Dbq={0};'.format("E:\\ex\\RTers\\RTers.accdb") #format()内にMS Accessデータベースファイルのフルパスを指定(相対パスはNG)。Pythonの文字列仕様上「\」は要エスケープ。「\\」と書く。 #ここが動かない場合、アクセスODBCドライバを別途インストールする。 #参考:https://yottagin.com/?p=2909 #DB操作のための定型文的なやつ。あまり気にしなくていい。 conn = pyodbc.connect(conn_str) cur = conn.cursor() #「このIDのアカウントのツイートをRTしてる人を検索する」という指定。@の後ろの文字列を入れる。 target_id = "dappi2019" #twitter APIに接続するための定型文的な奴。あまり気にしなくていい。 auth = tweepy.OAuthHandler(CK, CKS) auth.set_access_token(AT, ATS) api = tweepy.API(auth, wait_on_rate_limit=True) useruser = api.user_timeline(id=target_id) #resulut.csv(検索結果を一時的に書き出すファイル)を空にする f = open('result.csv', 'w') f.write('') f.close() try: #ここのtry内でtwitter APIから引っ張ってきたRT奴データをCSVに書き出している。「dbquery=~」のごちゃごちゃした文字列操作はCSVにした時に邪魔になる文字を全角文字等に置換している。 for rt in useruser: retweeter = api.retweets(id=rt.id_str) for rter in retweeter: with open('result.csv', 'a',encoding='utf-8') as f: dbquery=str(rter.user.id)+","+str(rter.user.name).replace(",",",").replace("\"","”").replace("\r"," ").replace("\n"," ")+",@"+ str(rter.user.screen_name)+","+str(rter.user.followers_count)+","+str(rter.user.description).replace(",",",").replace("\"","”").replace("\r"," ").replace("\n"," ") print(dbquery, file=f) #ファイルに書き出し except ConnectionError as e: print(e.reason) pass #上でCSVに書き出したデータをDBに流し込む。 with open("result.csv",'r',encoding='utf-8') as csvfile: read = csv.reader(csvfile) next(read) #CSVから一行読み出し count1=0 #CSV内に入っている件数(行数) count2=0 #CSVからDBに流し込んだ件数 for row in read: #今読み出したユーザと一致する人は既にDBにいる? sql = "SELECT * FROM RTers WHERE ID =\'" + str(row[0]) + "\';" cur.execute(sql) count1=count1+1 row2=cur.fetchone() #既にDBにいる場合にはこのfetchoneでデータが返る。なので結果がない場合(None)のみDBに追加 if row2 is None : count2=count2+1 #CSVから読み出したデータをDBにインサート sql = "INSERT INTO RTers VALUES(\ '{}','{}','{}','{}','{}')" #DBへのインサート時に一部不具合の出る文字がCSVに残ってしまっているため、このタイミングで邪魔な文字を置換している sql = sql.format(row[0],row[1].translate(str.maketrans({chr(0x0021 + i): chr(0xFF01 + i) for i in range(94)})),row[2],row[3],row[4].translate(str.maketrans({chr(0x0021 + i): chr(0xFF01 + i) for i in range(94)}))) cur.execute(sql) #print("新顔":" +str(row[2])) #else: #print("もういる:" +str(row[2])) total=total+count2 print(str(count1)+" 件中 " +str(count2)+" 件をDBに新規追加。今回のプログラム実行で合計 "+str(total)+ "件を追加しています。") #commitしないと処理が反映されない conn.commit() #DBをクローズ cur.close() conn.close() return(total) if __name__ == '__main__': i=0 total=0 while i==0 : #このプログラムは手動ストップしない限り無限ループで走り続ける。 dt_now = datetime.datetime.now() print(dt_now) #時刻をprint。 total=main(total) #ここでmain関数をコールしている time.sleep(210) # API制限に引っかからず動かし続けるために明示的にスリープを挟む。(API制限は15分間に一定回数らしい) i=iコード以外に必要なもの
・プログラムが参照するMS Accessデータベースファイル。
テーブル「RTers」を作成し、中に「ID」「名前」「screen_name」「フォロワー数」「プロフィール」の5つのフィールドを作っておきます。
・Twitter APIを使えるようにしておく
コード内の「コンシューマーキー」等は各自で用意し、書き込んでください。実行するとどうなるか
注意点
あまり効率のいい探し方ではない上、漏れなく正確にリストアップできるわけでもありません。
1ヶ月くらい動かしてどの程度のデータが取れるか様子を見てみる予定です。
- 投稿日:2020-02-27T11:24:35+09:00
Pythonを用いてCSVファイルを読み込んでみた
実行環境
・Mac OS X
PythonからCSVファイルを閲覧しようと思い、実際にやってみました。
まず、Python公式ドキュメントのCSVモジュールの使用例のコードをそのまま利用してみました。
read.pyimport csv with open('hoge.csv', newline='') as f: reader = csv.reader(f) for row in reader: print(row)早速、ターミナルで実行してみましょう。
$ python3 read.pyすると、次のようなエラーが出てしまいました。
UnicodeDecodeError: 'utf-8' codec can't decode byte 0x8e in position 1: invalid start byte何やら文字エンコードに問題があるようです。そこで、次のようにエンコードをcp932に指定してみました。
read.pyimport csv with open('hoge.csv', newline='', encoding='cp932') as f: reader = csv.reader(f) for row in reader: print(row)こうしてエラーは解決し、無事にPythonスクリプトからcsvファイルを開くことができました。
- 投稿日:2020-02-27T11:11:45+09:00
【ROS2】python形式のlaunchでbagファイルを再生する方法
- 投稿日:2020-02-27T11:10:56+09:00
【解説付き】PythonでFizz Buzz (paizaランク C 相当)を解く
初めに
paizaのレベルアップ問題集を解いていたのですが、模範解答がなかったので解説付きで自分で作ってみました。
言語はPython3です。問題
Paizaのスキルチェック見本問題 Fizz Buzz (paizaランク C 相当)
https://paiza.jp/works/mondai/skillcheck_sample/fizz-buzz?language_uid=python3
paizaは登録が無料ですぐにできるので、とりあえず登録してみることをおススメします。下記の問題をプログラミングしてみよう!
整数 N が入力として与えられます。
1からNまでの整数を1から順に表示してください。
ただし、表示しようとしている数値が、
・3の倍数かつ5の倍数のときには、"Fizz Buzz"
・3の倍数のときには、"Fizz"
・5の倍数のときには、"Buzz"を数値の代わりに表示してください。
入力される値
入力は以下のフォーマットで与えられます。
N
N は1以上N以下の整数です。
入力値最終行の末尾に改行が1つ入ります。
文字列は標準入力から渡されます。期待する出力
最後は改行し、余計な文字、空行を含んではいけません。
条件
すべてのテストケースにおいて、以下の条件をみたします。
・1 ≦ N ≦ 100
・N は整数入力例1
5
出力例1
1
2
Fizz
4
Buzz入力例2
20
出力例2
1
2
Fizz
4
Buzz
Fizz
7
8
Fizz
Buzz
11
Fizz
13
14
Fizz Buzz
16
17
Fizz
19
Buzz解説
入力値を受け取る
入力値は0-100の整数値です。
input関数を用いて入力値を受け取った後、int関数を用いて、str型からint型に型変換しておきます。fizz-buzz_1.pyn = int(input())数値を順に表示する
次に、代わりに表示しなければいけない数値を探します。今回は、1からnまでの整数のを順番に探していけばよいので、range関数を用います。
この時注意しなければならないのが、range関数の範囲の指定の仕方です。range関数で引数を二つ入力した場合は、第一引数は
beginstart、第二引数はendstopとなります。
この時、endstop引数は、その数自身を含まず、一つ前の整数を出力するように指定します。つまり、
range(1,**5**)とした場合の出力は、1,2,3,4となり、5は含まれません。
1,2,3,4,5 と出力したい場合は、range(1, 5+1)と指定する必要があります。range_sample.pyprint(list(range(1,5))) # [1,2,3,4] print(list(range(1,6))) # [1,2,3,4,5]今回の場合、1からnまでの整数値が欲しいので、range関数には 1, n+1 と引数を渡します。
for文と組み合わせるとこのようになります。fizz-buzz_2.pyn = int(input()) for i in range(1, n+1): print(i) """ n = 5の時、 1 2 3 4 5 """FizzBuzzを見つける
次に、fizzBuzzに変換しなければならない数値を判定します。
そのために % (余りを求める代数演算子)を使用します。もし、n を 3で割った余りがゼロであれば、nは 3の倍数です。
もし、n を 5で割った余りがゼロであれば、nは 5の倍数です。
もし、n を15で割った余りがゼロであれば、nは 3の倍数であり、かつ5の倍数です。3の倍数であり、かつ5の倍数ということは、15の倍数であるということです。
今回はif-elif文を用いて判定します。
この時、判定する順番は注意が必要です。
n が3、または5の倍数であるかどうかの判定を、
15の倍数であるかどうかの判定よりも先に書いてしまうとうまくいきません。nが3、または5の倍数であるかどうかの判定を、
15の倍数であるかどうかの判定よりも先に書く場合には少し工夫が要ります。
@shiracamus様が指摘された方法があり、下のコメント欄で確認してみてください。
今回は判定する順番により解決したいと思います。if_sample.py# ダメなコード n = 15 if n%3 == 0: print("nは3の倍数です。") elif n%5 == 0: print("nは5の倍数です。") elif n%15 == 0: print("nは3の倍数かつ5の倍数です。") else: print("nは3の倍数でも5の倍数でもありません。") # nは3の倍数です。もしnが15の倍数であった場合、15の倍数ではなくて、3、または5の倍数と判定されてしまいます。
上から順番に処理されていくことを意識しておきましょう。したがって、まず最初に、15の倍数であるかどうかを判定します。
その後に、5の倍数であるか、3の倍数であるかを判定するとうまくいきます。fizz-buzz_3.pyn = 15 if n%15 == 0: print("nは3の倍数かつ5の倍数です。") elif n%5 == 0: print("nは5の倍数です。") elif n%3 == 0: print("nは3の倍数です。") else: print("nは3の倍数でも5の倍数でもありません。") # nは3の倍数かつ5の倍数です。一般的に、if-elif文を書くときは狭い条件の方から先に書くとうまくいくことが多いようです。
解答コード
fizz-buzz.pyn = int(input()) for i in range(1, n+1): if i%15==0: print("Fizz Buzz") elif i%5==0: print("Buzz") elif i%3==0: print("Fizz") else: print(i)参考
https://qiita.com/KoyanagiHitoshi/items/3286fbc65d56dd67737c
最後に
過去の自分が引っかかったところを詳しく説明したつもりです。
まわりくどくて分かりにくいところもあったかもしれませんが、
なにか気になる点があれば気軽にコメントしてくださいね。
- 投稿日:2020-02-27T10:47:23+09:00
【Python】疎行列計算ができるようになる記事
はじめに
疎行列とは、行列の要素の大部分が0である行列のことを言います。数値計算や機械学習、グラフ理論など幅広い分野で扱われている(と思います)。行列内のほとんどが0という特徴から、0を無視する工夫をすれば、高速に行列計算を実施することが可能になります。例えば、9割近くが0の大規模疎行列(100万行×100万列とか)で非ゼロ部分だけ用いて計算すれば、計算量が約1/10になり計算が早く終わらせることが可能になることが容易に想像できます。
本記事では、Python・Numpy・Scipyを用いて、こうした疎行列計算について解説していきます。追加したいことが出てきたら適宜更新していきます。間違い等ございましたら、コメントにてご連絡して頂けたら幸いです。対象読者
- Pythonを扱える人
- 行列計算に興味がある人
概要
疎行列計算をPythonで扱う上で必要な概念をまとめた後、直接法・反復法のライブラリについて言及します。最終的に前回記事で作成した問題を非定常反復法と呼ばれる手法で解きます。
使用するライブラリ:Scipy・Numpy・matplotlib
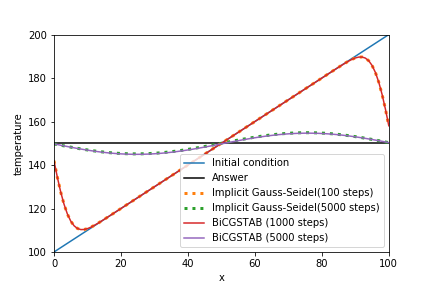
計算結果
記事内目次
1. 疎行列
1-1. 密行列と疎行列
密行列
密な行列。行列内に0がほとんどない行列。数学の問題とかで、おそらくこちらの方が見慣れている人が多いかも。
\left( \begin{array}{cccc} 2 & 1 & 1 & \ldots & \ldots & 1 \\ 1 & 2 & -1 & 1 & \ldots & 1 \\ 1 & 1 & 2 & 1 & 1 & \vdots \\ \vdots & \ddots & \ddots & \ddots & \ddots & \vdots\\ 1 & 1 & \ddots & 1 & 2 & 1 \\ 1 & 1 & \ldots & 1 & 1 & 2 \end{array} \right)
疎行列
疎(まば)らな行列。単に行列内に0がたくさんあるってだけ。0が関係する計算の部分を工夫すれば、ほとんど計算しなくて済むため、特殊な手法で計算する。本記事で扱う。
\left( \begin{array}{cccc} 2 & -1 & 0 & \ldots & \ldots & 0 \\ -1 & 2 & -1 & 0 & \ldots & 0 \\ 0 &-1 & 2 & -1 & \ddots & \vdots \\ \vdots & \ddots & \ddots & \ddots & \ddots & 0\\ 0 & 0 & \ddots & -1 & 2 & -1 \\ 0 & 0 & \ldots & 0 & -1 & 2 \end{array} \right)1-2. 疎行列の可視化
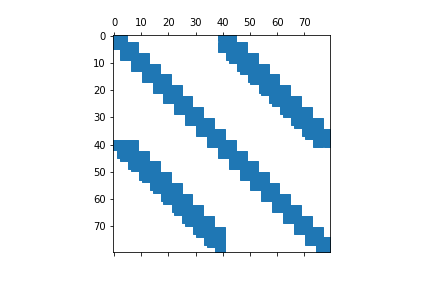
疎行列の可視化には、matplotlibのspy関数を用いるのが一番だと思います。これは行列で0の部分は白色、何か数が入っている場合は色付きで表示してくれます。
上の疎行列が$100 \times 100$の行列だとすると、以下のようになります。2. 疎行列の格納形式
0を無視して疎行列を扱うために、特殊な方式を用いて疎行列を表現します。こうすることにより、使用するメモリや計算量などを大幅に削減することが可能になります。よくわからないかもしれないので、下に示す行列を用いながら説明します。ただし、a=1, b=2, c=3, d=4, e=5, f=6とします。
\left( \begin{array}{cccc} a & 0 & 0 & 0 & 0 & 0 \\ b & 0 & 0 & 0 & 0 & 0 \\ 0 & 0 & 0 & c & 0 & d \\ 0 & 0 & 0 & 0 & 0 & 0 \\ 0 & 0 & 0 & 0 & e & 0 \\ 0 & 0 & 0 & 0 & 0 & f \end{array} \right)2-1. リストのリスト(List of Lists : LIL)
行ごとに、(列,要素)のタプルを格納します。
最初に行列にデータを格納する際は、LIL方式を用いるのが楽です。その後、下で示すCRSやCCS方式に変換するのがわかりやすくて良いと思います。PythonのlistやNumpyのarrayの操作に慣れてたら、そんなに難しくないです。上の例だと、
行 (列, 要素) 0 (0, a) 1 (0, b) 2 (3, c), (5, d) 4 (4, e) 5 (5, f) という形で格納され、少ないメモリで疎行列を記述できることがわかります。大規模な疎行列になればなるほど、こうした0を無視する格納方式の良さが出てきます。
scipy.sparse.lil_matrixscipyで用意されているLIL形式を扱うための関数lil_matrix。行列の非ゼロ要素の入力が楽に行えるため、疎行列を生成する際によく用いられることが多いです。
from scipy.sparse import lil_matrix # Create sparse matrix a=lil_matrix((6,6)) # Set the non-zero values a[0,0]=1.; a[1,0]=2.; a[2, 3]=3.; a[2, 5]=4.; a[4, 4]=5.; a[5, 5]=6. print("(row, column) value") print(a, "\n") print("Normal type: ") print(a.todense()) # 普通の行列の形に戻す print("LIL type: ") print(a.rows) # 各行で何列目に非ゼロ要素が入っているか print(a.data) # 各行での非ゼロ要素実行結果は以下の通り。
(row, column) value (0, 0) 1.0 (1, 0) 2.0 (2, 3) 3.0 (2, 5) 4.0 (4, 4) 5.0 (5, 5) 6.0 Normal type: [[1. 0. 0. 0. 0. 0.] [2. 0. 0. 0. 0. 0.] [0. 0. 0. 3. 0. 4.] [0. 0. 0. 0. 0. 0.] [0. 0. 0. 0. 5. 0.] [0. 0. 0. 0. 0. 6.]] LIL type: [list([0]) list([0]) list([3, 5]) list([]) list([4]) list([5])] [list([1.0]) list([2.0]) list([3.0, 4.0]) list([]) list([5.0]) list([6.0])]2-2. 圧縮行格納方式(Compressed Sparse Row: CSR)
CRS(Compressed Row Storage)と呼ぶ人もいる。行方向に探索し、非ゼロの行列の要素を格納していく。わかりにくい場合は、東京大学の中島先生の資料をお読みください。対角成分を除外してCSR方式を実行する例が示されております。
CSR方式を用いると、
\left( \begin{array}{cccc} a & 0 & 0 & 0 & 0 & 0 \\ b & 0 & 0 & 0 & 0 & 0 \\ 0 & 0 & 0 & c & 0 & d \\ 0 & 0 & 0 & 0 & 0 & 0 \\ 0 & 0 & 0 & 0 & e & 0 \\ 0 & 0 & 0 & 0 & 0 & f \end{array} \right)は、
- indices:
IA = [0, 0, 3, 5, 4, 5]
- 上の行から順に要素が何列目にあるかを表示している。indicesのサイズは非ゼロ要素の数となります。
- data:
A = [a, b, c, d, e, f]
- indicesに合わせて、その非ゼロの要素を示す。dataはindicesと同じサイズになります。
- indptr:
IN = [0, 1, 2, 4, 4, 5, 6]
- 各行で要素が何個あるかを数えて、それを積算した値を示す。0から始まるため、行数+1のサイズになります。
の3つのリストで表現することが可能です。こちらも、少ないメモリで疎行列を記述できることがわかります。疎行列計算を行う際は、このCSR形式を用いることが多いと思います。如何に早く計算が終わるのかは、3章の四則演算のところで述べてます。
これらのリストが何を表すのかを示すために、indicesを元の行列の要素の下付き文字、indptrを右側に示すと以下のようになります。
\left( \begin{array}{cccccc|c} col0 & col1 & col2 & col3 & col4 & col5 & indptr \\ & & & & & & 0 \\ a_0 & 0 & 0 & 0 & 0 & 0 & 1 \\ b_0 & 0 & 0 & 0 & 0 & 0 & 2 \\ 0 & 0 & 0 & c_3 & 0 & d_5& 4 \\ 0 & 0 & 0 & 0 & 0 & 0 & 4 \\ 0 & 0 & 0 & 0 & e_4 & 0 & 5 \\ 0 & 0 & 0 & 0 & 0 & f_5 & 6 \end{array} \right)考え方をこの例を用いながら説明すると、
- 初期状態
indices: IA = []data: A = []indptr: IN = [0, 0, 0, 0, 0, 0, 0](行数+1のサイズ)とします。- 0行目
- 0列目から順に非ゼロ要素がないかを探索します。
- aを発見!Aに要素aを追加。
- aは0列目にあるため、IAに0を追加。
- 0行目では要素を1つ見つけたため、
IN[1]に1を追加します。IA = [0], A = [a], IN = [0, 1, 0, 0, 0, 0, 0]- 1行目
- 0列目から順に非ゼロ要素がないかを探索します。
- bを発見!Aに要素bを追加。
- bは0列目にあるため、IAに0を追加。
- 1行目では要素を1つ見つけたため、
IN[2]に1+IN[1]を追加します。IA = [0, 0], A = [a, b], IN = [0, 1, 2, 0, 0, 0, 0]- 2行目以降、これを繰り返します。
IA = [0, 0, 3, 5, 4, 5], A = [a, b, c, d, e, f], IN = [0, 1, 2, 4, 4, 5, 6]
scipy.sparse.csr_matrixscipyで用意されているCSR形式を扱うための関数csr_matrix。
scipy.sparse.csr_matrix(mtx)のように任意の行列mtxを入れれば、CSR形式に直してくれます。mtxの形式は、list、numpy.ndarray、numpy.matrixなどです。LIL形式からCSR形式にする際は、tocsr()を用いれば良いです。a_csr = a.tocsr() print("Normal type: ") print(a_csr.todense()) print("csr type:") print(a_csr.indices) print(a_csr.data) print(a_csr.indptr)Normal type: [[1. 0. 0. 0. 0. 0.] [2. 0. 0. 0. 0. 0.] [0. 0. 0. 3. 0. 4.] [0. 0. 0. 0. 0. 0.] [0. 0. 0. 0. 5. 0.] [0. 0. 0. 0. 0. 6.]] csr type: [0 0 3 5 4 5] # indices: 上の行から順に要素が何列目にあるかを表示している。 [1. 2. 3. 4. 5. 6.] # data: indicesに合わせて、その非ゼロの要素を示す。 [0 1 2 4 4 5 6] # indptr: 各行で要素が何個あるかを数えて、それを積算する。0から始まるため、行数+1のサイズになる。2-3. 圧縮列格納方式(Compressed Sparse Column: CSC)
CCSと呼ぶ人もいる。列方向に探索し、非ゼロの行列の要素を格納していく。CSR方式の列バージョン。
scipy.sparse.csc_matrixscipyで用意されているCSC形式を扱うための関数csc_matrix。
scipy.sparse.csc_matrix(mtx)のように任意の行列mtxを入れれば、CSC形式に直してくれます。mtxの形式は、list、numpy.ndarray、numpy.matrixなどです。LIL形式からCSC形式にする際は、tocsc()を用いれば良いです。a_csc = a.tocsc() print("Normal type: ") print(a_csc.todense()) print("csc type:") print(a_csc.indices) print(a_csc.data) print(a_csc.indptr)Normal type: [[1. 0. 0. 0. 0. 0.] [2. 0. 0. 0. 0. 0.] [0. 0. 0. 3. 0. 4.] [0. 0. 0. 0. 0. 0.] [0. 0. 0. 0. 5. 0.] [0. 0. 0. 0. 0. 6.]] csc type: [0 1 2 4 2 5] # indices: 左の列から順に要素が何行目にあるかを表示している。 [1. 2. 3. 5. 4. 6.] # data: indicesに合わせて、その非ゼロの要素を示す。 [0 2 2 2 3 4 6] # indptr: 各列で要素が何個あるかを数えて、それを積算する。0から始まるため、列数+1のサイズになる。2-4. その他
他にも色々な行列格納方式があるので、詳しく知りたい方はWikipedia等をご覧ください。
3. 四則演算など
疎行列の計算をする際は、CSR方式かCSC方式で格納した後に計算を行うのが、最も一般的です。慣れ親しんでいるnumpyの四則演算と同じように実行することが可能です。
# csr example # Create sparse matrix a = scipy.sparse.lil_matrix((6,6)) # Set the non-zero values a[0,0]=1.; a[1,0]=2.; a[2, 3]=3.; a[2, 5]=4.; a[4, 4]=5.; a[5, 5]=6. a_csr = a.tocsr() a_matrix = a.todense() b = np.arange(6).reshape((6,1)) print(b) print(a_matrix * b) print(a_csr * b) print(a_matrix.dot(b)) print(a_csr.dot(b))CSR形式で格納した疎行列同士の掛け算もこんな感じで計算可能。
b_sparse = scipy.sparse.lil_matrix((6,6)) b_sparse[0,1]=1.; b_sparse[3,0]=2.; b_sparse[3, 3]=3.; b_sparse[5, 4]=4. b_csr = b_sparse.tocsr() print(b_csr.todense()) print(a_matrix * b_csr) print((a_csr * b_csr).todense())これらを踏まえ、CSR(圧縮行格納)方式の有効性を確認してみましょう。以下のコードをJupyter notebook上で実行してみてください。普通の行列を用いて解くよりCSR方式の方が相当(1000倍くらい)早く計算が終わるのがわかると思います。
import scipy.sparse import numpy as np import matplotlib.pyplot as plt num = 10**4 # Creat LIL type sparse matrix a_large = scipy.sparse.lil_matrix((num,num)) # Set the non-zero values a_large.setdiag(np.ones(num)*2) a_large.setdiag(np.ones(num-1)*-1, k=1) a_large.setdiag(np.ones(num-1)*-1, k=-1) a_large_csr = a_large.tocsr() a_large_dense = a_large.todense() plt.spy(a_large_csr) b = np.ones(num) %timeit a_large_csr.dot(b) # CSR方式 %timeit a_large_dense.dot(b) # 普通の行列注意点
np.dot(a_csr, b)のようにnumpyのdot関数の引数にcsr形式の行列を入れてしまうと、[[<6x6 sparse matrix of type '<class 'numpy.float64'>' with 6 stored elements in Compressed Sparse Row format>] [<6x6 sparse matrix of type '<class 'numpy.float64'>' with 6 stored elements in Compressed Sparse Row format>] [<6x6 sparse matrix of type '<class 'numpy.float64'>' with 6 stored elements in Compressed Sparse Row format>] [<6x6 sparse matrix of type '<class 'numpy.float64'>' with 6 stored elements in Compressed Sparse Row format>] [<6x6 sparse matrix of type '<class 'numpy.float64'>' with 6 stored elements in Compressed Sparse Row format>] [<6x6 sparse matrix of type '<class 'numpy.float64'>' with 6 stored elements in Compressed Sparse Row format>]]のようになりうまく計算できません。
4. Matrix Market 形式
本記事では使用しませんが、疎行列の表現の一つであるMatrix Market形式についても言及しておきます。疎行列のデータは、この形式かRutherford Boeingという形式で基本的には配布されており、疎行列計算に関する論文や実装などを参照しようとすると必要な知識になります。
疎行列問題のデータセットとしては、The SuiteSparse Matrix Collection formerly the University of Florida Sparse Matrix Collectionが有名かと思います。\left( \begin{array}{cccc} a & 0 & 0 & 0 & 0 & 0 \\ b & 0 & 0 & 0 & 0 & 0 \\ 0 & 0 & 0 & c & 0 & d \\ 0 & 0 & 0 & 0 & 0 & 0 \\ 0 & 0 & 0 & 0 & e & 0 \\ 0 & 0 & 0 & 0 & 0 & f \end{array} \right)をMatrix Market(MM)形式で表すと
%%MatrixMarket matrix coordinate real general %================================================================================= % % This ASCII file represents a sparse MxN matrix with L % nonzeros in the following Matrix Market format: % % +----------------------------------------------+ % |%%MatrixMarket matrix coordinate real general | <--- header line % |% | <--+ % |% comments | |-- 0 or more comment lines % |% | <--+ % | M N L | <--- rows, columns, entries % | I1 J1 A(I1, J1) | <--+ % | I2 J2 A(I2, J2) | | % | I3 J3 A(I3, J3) | |-- L lines % | . . . | | % | IL JL A(IL, JL) | <--+ % +----------------------------------------------+ % % Indices are 1-based, i.e. A(1,1) is the first element. % %================================================================================= 6 6 6 1 1 a 2 1 b 3 4 c 5 5 e 3 6 d 6 6 fとなります。上のコメントを見ればわかると思います。
この記事がわかりやすいです。とりあえず適当に選んだsteam3という疎行列で色々遊んでみます。
Matrix market形式を読み込む際にはscipy.ioを用います。
scipy.io.mminfoを実行すると、何行何列の行列で、非ゼロの要素が何個あるかなどを教えてくれます。scipy.io.mmreadでMatrix Market形式の行列を読み込むことが可能です。返り値は、numpy.arrayかcoo_matrix形式(csr格納形式と似たようなものです)です。import scipy.io as io print(io.mminfo("steam3/steam3.mtx")) steam3 = io.mmread("steam3/steam3.mtx").tocsr() print(steam3.todense()) plt.spy(steam3)(80, 80, 928, 'coordinate', 'real', 'general') matrix([[-3.8253876e+05, 0.0000000e+00, 0.0000000e+00, ..., 0.0000000e+00, 0.0000000e+00, 0.0000000e+00], [ 9.1147205e-01, -9.5328382e+03, 0.0000000e+00, ..., 0.0000000e+00, 0.0000000e+00, 0.0000000e+00], [ 0.0000000e+00, 0.0000000e+00, -2.7754141e+02, ..., 0.0000000e+00, 0.0000000e+00, 0.0000000e+00], ..., [ 0.0000000e+00, 0.0000000e+00, 0.0000000e+00, ..., -4.6917325e+08, 0.0000000e+00, -1.2118734e+05], [ 0.0000000e+00, 0.0000000e+00, 0.0000000e+00, ..., 0.0000000e+00, -1.3659626e+07, 0.0000000e+00], [ 0.0000000e+00, 0.0000000e+00, 0.0000000e+00, ..., 2.1014810e+07, -3.6673643e+07, -1.0110894e+04]])5. 直接法ライブラリ
scipy.sparse.linalg.dsolveの中に入っています。前の記事で解いたAx=bという問題(詳しくは下の式参照)を例に実装しました(実装はほぼ変わっていません)。\left( \begin{array}{cccc} 2 \left(\frac{1}{d} + 1 \right) & -1 & 0 & \ldots & \ldots & 0 \\ -1 & 2 \left(\frac{1}{d} + 1 \right) & -1 & 0 & \ldots & 0 \\ 0 &-1 & 2 \left(\frac{1}{d} + 1 \right) & -1 & 0 & \ldots \\ \vdots & \ddots & \ddots & \ddots & \ddots & \ddots\\ 0 & 0 & \ddots & -1 & 2 \left(\frac{1}{d} + 1 \right) & -1 \\ 0 & 0 & \ldots & 0 & -1 & 2 \left(\frac{1}{d} + 1 \right) \end{array} \right) \left( \begin{array}{c} T_1^{n+1} \\ T_2^{n+1} \\ T_3^{n+1} \\ \vdots \\ T_{M-1}^{n+1} \\ T_M^{n+1} \end{array} \right) = \left( \begin{array}{c} T_2^{n} + 2 \left(\frac{1}{d} - 1 \right) T_1^{n} + \left(T_0^n + T_0^{n+1}\right) \\ T_3^{n} + 2 \left(\frac{1}{d} - 1 \right) T_2^{n} + T_1^n \\ T_4^{n} + 2 \left(\frac{1}{d} - 1 \right) T_3^{n} + T_2^n \\ \vdots \\ T_M^{n} + 2 \left(\frac{1}{d} - 1 \right) T_{M-1}^{n} + T_{M-2}^n \\ \left(T_{M+1}^n + T_{M+1}^{n+1}\right) + 2 \left(\frac{1}{d} - 1 \right) T_{M}^{n} + T_{M-1}^n \end{array} \right)デフォルトの設定では、
scipy.sparse.linalg.dsolve(csr形式のA行列, bベクトル)でSuperLU分解が行われるはずです。定常反復法のガウス・ザイデル法と比較結果が下の画像です。
import numpy as np import matplotlib.pyplot as plt import scipy.sparse # Make stencils # Creat square wave Num_stencil_x = 101 x_array = np.float64(np.arange(Num_stencil_x)) temperature_array = x_array + 100 temperature_lower_boundary = 150 temperature_upper_boundary = 150 Time_step = 100 Delta_x = max(x_array) / (Num_stencil_x-1) C = 1 Delta_t = 0.2 kappa = 0.5 d = kappa * Delta_t / Delta_x**2 total_movement = C * Delta_t * (Time_step+1) exact_temperature_array = (temperature_upper_boundary - temperature_lower_boundary) / (x_array[-1] - x_array[0]) * x_array + temperature_lower_boundary plt.plot(x_array, temperature_array, label="Initial condition") print("Δx:", Delta_x, "Δt:", Delta_t, "d:", d) temperature_sp = temperature_array.copy() for n in range(Time_step): a_matrix = np.identity(len(temperature_sp)) * 2 *(1/d+1) \ - np.eye(len(temperature_sp), k=1) \ - np.eye(len(temperature_sp), k=-1) temp_temperature_array = np.append(np.append( temperature_lower_boundary, temperature_sp), temperature_upper_boundary) b_array = 2 * (1/d - 1) * temperature_sp + temp_temperature_array[2:] + temp_temperature_array[:-2] b_array[0] += temperature_lower_boundary b_array[-1] += temperature_upper_boundary a_csr = scipy.sparse.csr_matrix(a_matrix) temperature_sp = spla.dsolve.spsolve(a_csr, b_array) plt.plot(x_array, temperature_sp, label="SuperLU") plt.legend(loc="lower right") plt.xlabel("x") plt.ylabel("temperature") plt.xlim(0, max(x_array)) plt.ylim(100, 200)6. 反復法ライブラリ
非定常反復法のライブラリがscipyでは実装されております。
scipy.sparse.linalg.isolveの中に入っております。非定常反復法がよくわからない人は前の記事をご覧ください。使用できるライブラリの一部を以下に列挙しておきます。これら以外のライブラリを使いたい場合は、Scipyのマニュアルから探してみてください。
- 対称行列
cg共役勾配法(Conjugate Gradient method : CG法) - 正定値対称行列のみminres最小残差法(MINimum RESidual: MINRES法)- 非対称行列
bicg双共役勾配法(Bi-Conjugate Gradient: BiCG法)cgs自乗共役勾配法(Conjugate Residual Squared: CGS法)bicgstab双共役勾配安定化法(BiCG Stabilization: BiCGSTAB法)gmres一般化最小残差法(Generalized Minimal Residual: GMRES法)qmr準最小残差法(Quasi-Minimal Residual: QMR法)直接法で使用した同じ問題に対して、BiCGSTAB法での実装例とその実行結果を以下に示します。
temperature_sp = temperature_array.copy() for n in range(Time_step): a_matrix = np.identity(len(temperature_sp)) * 2 *(1/d+1) \ - np.eye(len(temperature_sp), k=1) \ - np.eye(len(temperature_sp), k=-1) temp_temperature_array = np.append(np.append( temperature_lower_boundary, temperature_sp), temperature_upper_boundary) b_array = 2.0 * (1/d - 1.0) * temperature_sp + temp_temperature_array[2:] + temp_temperature_array[:-2] b_array[0] += temperature_lower_boundary b_array[-1] += temperature_upper_boundary a_csr = scipy.sparse.csr_matrix(a_matrix) temperature_sp = spla.isolve.bicgstab(a_csr, b_array)[0] plt.plot(x_array, temperature_sp, label="Scipy.isolve (1000 steps)") plt.legend(loc="lower right") plt.xlabel("x") plt.ylabel("temperature") plt.xlim(0, max(x_array)) plt.ylim(100, 200)まとめ
- 疎行列には0を無視する格納方式がある
- LIL方式は疎行列を作成するときに用いることが多い。
- CSR方式とCSC方式は疎行列計算をするときに用いることが多い。
- CSR方式で計算すると、普通の行列の形で計算するより、圧倒的に早く計算が終わることが確認できた。
- Matrix Market方式で疎行列は配布されることが多い。
- 直接法と非定常反復法の関数がScipyに実装されており、簡単に使用することができる。
という感じかと思います。
参考文献
- 投稿日:2020-02-27T10:47:23+09:00
【Python】疎行列計算が高速にできるようになる記事
はじめに
疎行列とは、行列の要素の大部分が0である行列のことを言います。数値計算や機械学習、グラフ理論など幅広い分野で扱われている(と思います)。行列内のほとんどが0という特徴から、0を無視する工夫をすれば、高速に行列計算を実施することが可能になります。例えば、9割近くが0の大規模疎行列(100万行×100万列とか)で非ゼロ部分だけ用いて計算すれば、計算量が約1/10になり計算が早く終わらせることが可能になることが容易に想像できます。
本記事では、Python・Numpy・Scipyを用いて、こうした疎行列計算について解説していきます。追加したいことが出てきたら適宜更新していきます。間違い等ございましたら、コメントにてご連絡して頂けたら幸いです。対象読者
- Pythonを扱える人
- 行列計算に興味がある人
概要
疎行列計算をPythonで扱う上で必要な概念をまとめた後、直接法・反復法のライブラリについて言及します。最終的に前回記事で作成した問題を非定常反復法と呼ばれる手法で解きます。
使用するライブラリ:Scipy・Numpy・matplotlib
計算結果
記事内目次
1. 疎行列
1-1. 密行列と疎行列
密行列
密な行列。行列内に0がほとんどない行列。数学の問題とかで、おそらくこちらの方が見慣れている人が多いかも。
\left( \begin{array}{cccc} 2 & 1 & 1 & \ldots & \ldots & 1 \\ 1 & 2 & -1 & 1 & \ldots & 1 \\ 1 & 1 & 2 & 1 & 1 & \vdots \\ \vdots & \ddots & \ddots & \ddots & \ddots & \vdots\\ 1 & 1 & \ddots & 1 & 2 & 1 \\ 1 & 1 & \ldots & 1 & 1 & 2 \end{array} \right)
疎行列
疎(まば)らな行列。単に行列内に0がたくさんあるってだけ。0が関係する計算の部分を工夫すれば、ほとんど計算しなくて済むため、特殊な手法で計算する。本記事で扱う。
\left( \begin{array}{cccc} 2 & -1 & 0 & \ldots & \ldots & 0 \\ -1 & 2 & -1 & 0 & \ldots & 0 \\ 0 &-1 & 2 & -1 & \ddots & \vdots \\ \vdots & \ddots & \ddots & \ddots & \ddots & 0\\ 0 & 0 & \ddots & -1 & 2 & -1 \\ 0 & 0 & \ldots & 0 & -1 & 2 \end{array} \right)1-2. 疎行列の可視化
疎行列の可視化には、matplotlibのspy関数を用いるのが一番だと思います。これは行列で0の部分は白色、何か数が入っている場合は色付きで表示してくれます。
上の疎行列が$100 \times 100$の行列だとすると、以下のようになります。2. 疎行列の格納形式
0を無視して疎行列を扱うために、特殊な方式を用いて疎行列を表現します。こうすることにより、使用するメモリや計算量などを大幅に削減することが可能になります。よくわからないかもしれないので、下に示す行列を用いながら説明します。ただし、a=1, b=2, c=3, d=4, e=5, f=6とします。
\left( \begin{array}{cccc} a & 0 & 0 & 0 & 0 & 0 \\ b & 0 & 0 & 0 & 0 & 0 \\ 0 & 0 & 0 & c & 0 & d \\ 0 & 0 & 0 & 0 & 0 & 0 \\ 0 & 0 & 0 & 0 & e & 0 \\ 0 & 0 & 0 & 0 & 0 & f \end{array} \right)2-1. リストのリスト(List of Lists : LIL)
行ごとに、(列,要素)のタプルを格納します。
最初に行列にデータを格納する際は、LIL方式を用いるのが楽です。その後、下で示すCRSやCCS方式に変換するのがわかりやすくて良いと思います。PythonのlistやNumpyのarrayの操作に慣れてたら、そんなに難しくないです。上の例だと、
行 (列, 要素) 0 (0, a) 1 (0, b) 2 (3, c), (5, d) 4 (4, e) 5 (5, f) という形で格納され、少ないメモリで疎行列を記述できることがわかります。大規模な疎行列になればなるほど、こうした0を無視する格納方式の良さが出てきます。
scipy.sparse.lil_matrixscipyで用意されているLIL形式を扱うための関数lil_matrix。行列の非ゼロ要素の入力が楽に行えるため、疎行列を生成する際によく用いられることが多いです。
from scipy.sparse import lil_matrix # Create sparse matrix a=lil_matrix((6,6)) # Set the non-zero values a[0,0]=1.; a[1,0]=2.; a[2, 3]=3.; a[2, 5]=4.; a[4, 4]=5.; a[5, 5]=6. print("(row, column) value") print(a, "\n") print("Normal type: ") print(a.todense()) # 普通の行列の形に戻す print("LIL type: ") print(a.rows) # 各行で何列目に非ゼロ要素が入っているか print(a.data) # 各行での非ゼロ要素実行結果は以下の通り。
(row, column) value (0, 0) 1.0 (1, 0) 2.0 (2, 3) 3.0 (2, 5) 4.0 (4, 4) 5.0 (5, 5) 6.0 Normal type: [[1. 0. 0. 0. 0. 0.] [2. 0. 0. 0. 0. 0.] [0. 0. 0. 3. 0. 4.] [0. 0. 0. 0. 0. 0.] [0. 0. 0. 0. 5. 0.] [0. 0. 0. 0. 0. 6.]] LIL type: [list([0]) list([0]) list([3, 5]) list([]) list([4]) list([5])] [list([1.0]) list([2.0]) list([3.0, 4.0]) list([]) list([5.0]) list([6.0])]2-2. 圧縮行格納方式(Compressed Sparse Row: CSR)
CRS(Compressed Row Storage)と呼ぶ人もいる。行方向に探索し、非ゼロの行列の要素を格納していく。わかりにくい場合は、東京大学の中島先生の資料をお読みください。対角成分を除外してCSR方式を実行する例が示されております。
CSR方式を用いると、
\left( \begin{array}{cccc} a & 0 & 0 & 0 & 0 & 0 \\ b & 0 & 0 & 0 & 0 & 0 \\ 0 & 0 & 0 & c & 0 & d \\ 0 & 0 & 0 & 0 & 0 & 0 \\ 0 & 0 & 0 & 0 & e & 0 \\ 0 & 0 & 0 & 0 & 0 & f \end{array} \right)は、
- indices:
IA = [0, 0, 3, 5, 4, 5]
- 上の行から順に要素が何列目にあるかを表示している。indicesのサイズは非ゼロ要素の数となります。
- data:
A = [a, b, c, d, e, f]
- indicesに合わせて、その非ゼロの要素を示す。dataはindicesと同じサイズになります。
- indptr:
IN = [0, 1, 2, 4, 4, 5, 6]
- 各行で要素が何個あるかを数えて、それを積算した値を示す。0から始まるため、行数+1のサイズになります。
の3つのリストで表現することが可能です。こちらも、少ないメモリで疎行列を記述できることがわかります。疎行列計算を行う際は、このCSR形式を用いることが多いと思います。如何に早く計算が終わるのかは、3章の四則演算のところで述べてます。
これらのリストが何を表すのかを示すために、indicesを元の行列の要素の下付き文字、indptrを右側に示すと以下のようになります。
\left( \begin{array}{cccccc|c} col0 & col1 & col2 & col3 & col4 & col5 & indptr \\ & & & & & & 0 \\ a_0 & 0 & 0 & 0 & 0 & 0 & 1 \\ b_0 & 0 & 0 & 0 & 0 & 0 & 2 \\ 0 & 0 & 0 & c_3 & 0 & d_5& 4 \\ 0 & 0 & 0 & 0 & 0 & 0 & 4 \\ 0 & 0 & 0 & 0 & e_4 & 0 & 5 \\ 0 & 0 & 0 & 0 & 0 & f_5 & 6 \end{array} \right)考え方をこの例を用いながら説明すると、
- 初期状態
indices: IA = []data: A = []indptr: IN = [0, 0, 0, 0, 0, 0, 0](行数+1のサイズ)とします。- 0行目
- 0列目から順に非ゼロ要素がないかを探索します。
- aを発見!Aに要素aを追加。
- aは0列目にあるため、IAに0を追加。
- 0行目では要素を1つ見つけたため、
IN[1]に1を追加します。IA = [0], A = [a], IN = [0, 1, 0, 0, 0, 0, 0]- 1行目
- 0列目から順に非ゼロ要素がないかを探索します。
- bを発見!Aに要素bを追加。
- bは0列目にあるため、IAに0を追加。
- 1行目では要素を1つ見つけたため、
IN[2]に1+IN[1]を追加します。IA = [0, 0], A = [a, b], IN = [0, 1, 2, 0, 0, 0, 0]- 2行目以降、これを繰り返します。
IA = [0, 0, 3, 5, 4, 5], A = [a, b, c, d, e, f], IN = [0, 1, 2, 4, 4, 5, 6]
scipy.sparse.csr_matrixscipyで用意されているCSR形式を扱うための関数csr_matrix。
scipy.sparse.csr_matrix(mtx)のように任意の行列mtxを入れれば、CSR形式に直してくれます。mtxの形式は、list、numpy.ndarray、numpy.matrixなどです。LIL形式からCSR形式にする際は、tocsr()を用いれば良いです。a_csr = a.tocsr() print("Normal type: ") print(a_csr.todense()) print("csr type:") print(a_csr.indices) print(a_csr.data) print(a_csr.indptr)Normal type: [[1. 0. 0. 0. 0. 0.] [2. 0. 0. 0. 0. 0.] [0. 0. 0. 3. 0. 4.] [0. 0. 0. 0. 0. 0.] [0. 0. 0. 0. 5. 0.] [0. 0. 0. 0. 0. 6.]] csr type: [0 0 3 5 4 5] # indices: 上の行から順に要素が何列目にあるかを表示している。 [1. 2. 3. 4. 5. 6.] # data: indicesに合わせて、その非ゼロの要素を示す。 [0 1 2 4 4 5 6] # indptr: 各行で要素が何個あるかを数えて、それを積算する。0から始まるため、行数+1のサイズになる。2-3. 圧縮列格納方式(Compressed Sparse Column: CSC)
CCSと呼ぶ人もいる。列方向に探索し、非ゼロの行列の要素を格納していく。CSR方式の列バージョン。
scipy.sparse.csc_matrixscipyで用意されているCSC形式を扱うための関数csc_matrix。
scipy.sparse.csc_matrix(mtx)のように任意の行列mtxを入れれば、CSC形式に直してくれます。mtxの形式は、list、numpy.ndarray、numpy.matrixなどです。LIL形式からCSC形式にする際は、tocsc()を用いれば良いです。a_csc = a.tocsc() print("Normal type: ") print(a_csc.todense()) print("csc type:") print(a_csc.indices) print(a_csc.data) print(a_csc.indptr)Normal type: [[1. 0. 0. 0. 0. 0.] [2. 0. 0. 0. 0. 0.] [0. 0. 0. 3. 0. 4.] [0. 0. 0. 0. 0. 0.] [0. 0. 0. 0. 5. 0.] [0. 0. 0. 0. 0. 6.]] csc type: [0 1 2 4 2 5] # indices: 左の列から順に要素が何行目にあるかを表示している。 [1. 2. 3. 5. 4. 6.] # data: indicesに合わせて、その非ゼロの要素を示す。 [0 2 2 2 3 4 6] # indptr: 各列で要素が何個あるかを数えて、それを積算する。0から始まるため、列数+1のサイズになる。2-4. その他
他にも色々な行列格納方式があるので、詳しく知りたい方はWikipedia等をご覧ください。
3. 四則演算など
疎行列の計算をする際は、CSR方式かCSC方式で格納した後に計算を行うのが、最も一般的です。慣れ親しんでいるnumpyの四則演算と同じように実行することが可能です。
# csr example # Create sparse matrix a = scipy.sparse.lil_matrix((6,6)) # Set the non-zero values a[0,0]=1.; a[1,0]=2.; a[2, 3]=3.; a[2, 5]=4.; a[4, 4]=5.; a[5, 5]=6. a_csr = a.tocsr() a_matrix = a.todense() b = np.arange(6).reshape((6,1)) print(b) print(a_matrix * b) print(a_csr * b) print(a_matrix.dot(b)) print(a_csr.dot(b))CSR形式で格納した疎行列同士の掛け算もこんな感じで計算可能。
b_sparse = scipy.sparse.lil_matrix((6,6)) b_sparse[0,1]=1.; b_sparse[3,0]=2.; b_sparse[3, 3]=3.; b_sparse[5, 4]=4. b_csr = b_sparse.tocsr() print(b_csr.todense()) print(a_matrix * b_csr) print((a_csr * b_csr).todense())これらを踏まえ、CSR(圧縮行格納)方式の有効性を確認してみましょう。以下のコードをJupyter notebook上で実行してみてください。普通の行列を用いて解くよりCSR方式の方が相当(1000倍くらい)早く計算が終わるのがわかると思います。
import scipy.sparse import numpy as np import matplotlib.pyplot as plt num = 10**4 # Creat LIL type sparse matrix a_large = scipy.sparse.lil_matrix((num,num)) # Set the non-zero values a_large.setdiag(np.ones(num)*2) a_large.setdiag(np.ones(num-1)*-1, k=1) a_large.setdiag(np.ones(num-1)*-1, k=-1) a_large_csr = a_large.tocsr() a_large_dense = a_large.todense() plt.spy(a_large_csr) b = np.ones(num) %timeit a_large_csr.dot(b) # CSR方式 %timeit a_large_dense.dot(b) # 普通の行列注意点
np.dot(a_csr, b)のようにnumpyのdot関数の引数にcsr形式の行列を入れてしまうと、[[<6x6 sparse matrix of type '<class 'numpy.float64'>' with 6 stored elements in Compressed Sparse Row format>] [<6x6 sparse matrix of type '<class 'numpy.float64'>' with 6 stored elements in Compressed Sparse Row format>] [<6x6 sparse matrix of type '<class 'numpy.float64'>' with 6 stored elements in Compressed Sparse Row format>] [<6x6 sparse matrix of type '<class 'numpy.float64'>' with 6 stored elements in Compressed Sparse Row format>] [<6x6 sparse matrix of type '<class 'numpy.float64'>' with 6 stored elements in Compressed Sparse Row format>] [<6x6 sparse matrix of type '<class 'numpy.float64'>' with 6 stored elements in Compressed Sparse Row format>]]のようになりうまく計算できません。
4. Matrix Market 形式
本記事では使用しませんが、疎行列の表現の一つであるMatrix Market形式についても言及しておきます。疎行列のデータは、この形式かRutherford Boeingという形式で基本的には配布されており、疎行列計算に関する論文や実装などを参照しようとすると必要な知識になります。
疎行列問題のデータセットとしては、The SuiteSparse Matrix Collection formerly the University of Florida Sparse Matrix Collectionが有名かと思います。\left( \begin{array}{cccc} a & 0 & 0 & 0 & 0 & 0 \\ b & 0 & 0 & 0 & 0 & 0 \\ 0 & 0 & 0 & c & 0 & d \\ 0 & 0 & 0 & 0 & 0 & 0 \\ 0 & 0 & 0 & 0 & e & 0 \\ 0 & 0 & 0 & 0 & 0 & f \end{array} \right)をMatrix Market(MM)形式で表すと
%%MatrixMarket matrix coordinate real general %================================================================================= % % This ASCII file represents a sparse MxN matrix with L % nonzeros in the following Matrix Market format: % % +----------------------------------------------+ % |%%MatrixMarket matrix coordinate real general | <--- header line % |% | <--+ % |% comments | |-- 0 or more comment lines % |% | <--+ % | M N L | <--- rows, columns, entries % | I1 J1 A(I1, J1) | <--+ % | I2 J2 A(I2, J2) | | % | I3 J3 A(I3, J3) | |-- L lines % | . . . | | % | IL JL A(IL, JL) | <--+ % +----------------------------------------------+ % % Indices are 1-based, i.e. A(1,1) is the first element. % %================================================================================= 6 6 6 1 1 a 2 1 b 3 4 c 5 5 e 3 6 d 6 6 fとなります。上のコメントを見ればわかると思います。
この記事がわかりやすいです。とりあえず適当に選んだsteam3という疎行列で色々遊んでみます。
Matrix market形式を読み込む際にはscipy.ioを用います。
scipy.io.mminfoを実行すると、何行何列の行列で、非ゼロの要素が何個あるかなどを教えてくれます。scipy.io.mmreadでMatrix Market形式の行列を読み込むことが可能です。返り値は、numpy.arrayかcoo_matrix形式(csr格納形式と似たようなものです)です。import scipy.io as io print(io.mminfo("steam3/steam3.mtx")) steam3 = io.mmread("steam3/steam3.mtx").tocsr() print(steam3.todense()) plt.spy(steam3)(80, 80, 928, 'coordinate', 'real', 'general') matrix([[-3.8253876e+05, 0.0000000e+00, 0.0000000e+00, ..., 0.0000000e+00, 0.0000000e+00, 0.0000000e+00], [ 9.1147205e-01, -9.5328382e+03, 0.0000000e+00, ..., 0.0000000e+00, 0.0000000e+00, 0.0000000e+00], [ 0.0000000e+00, 0.0000000e+00, -2.7754141e+02, ..., 0.0000000e+00, 0.0000000e+00, 0.0000000e+00], ..., [ 0.0000000e+00, 0.0000000e+00, 0.0000000e+00, ..., -4.6917325e+08, 0.0000000e+00, -1.2118734e+05], [ 0.0000000e+00, 0.0000000e+00, 0.0000000e+00, ..., 0.0000000e+00, -1.3659626e+07, 0.0000000e+00], [ 0.0000000e+00, 0.0000000e+00, 0.0000000e+00, ..., 2.1014810e+07, -3.6673643e+07, -1.0110894e+04]])5. 直接法ライブラリ
scipy.sparse.linalg.dsolveの中に入っています。前の記事で解いたAx=bという問題(詳しくは下の式参照)を例に実装しました(実装はほぼ変わっていません)。\left( \begin{array}{cccc} 2 \left(\frac{1}{d} + 1 \right) & -1 & 0 & \ldots & \ldots & 0 \\ -1 & 2 \left(\frac{1}{d} + 1 \right) & -1 & 0 & \ldots & 0 \\ 0 &-1 & 2 \left(\frac{1}{d} + 1 \right) & -1 & 0 & \ldots \\ \vdots & \ddots & \ddots & \ddots & \ddots & \ddots\\ 0 & 0 & \ddots & -1 & 2 \left(\frac{1}{d} + 1 \right) & -1 \\ 0 & 0 & \ldots & 0 & -1 & 2 \left(\frac{1}{d} + 1 \right) \end{array} \right) \left( \begin{array}{c} T_1^{n+1} \\ T_2^{n+1} \\ T_3^{n+1} \\ \vdots \\ T_{M-1}^{n+1} \\ T_M^{n+1} \end{array} \right) = \left( \begin{array}{c} T_2^{n} + 2 \left(\frac{1}{d} - 1 \right) T_1^{n} + \left(T_0^n + T_0^{n+1}\right) \\ T_3^{n} + 2 \left(\frac{1}{d} - 1 \right) T_2^{n} + T_1^n \\ T_4^{n} + 2 \left(\frac{1}{d} - 1 \right) T_3^{n} + T_2^n \\ \vdots \\ T_M^{n} + 2 \left(\frac{1}{d} - 1 \right) T_{M-1}^{n} + T_{M-2}^n \\ \left(T_{M+1}^n + T_{M+1}^{n+1}\right) + 2 \left(\frac{1}{d} - 1 \right) T_{M}^{n} + T_{M-1}^n \end{array} \right)デフォルトの設定では、
scipy.sparse.linalg.dsolve(csr形式のA行列, bベクトル)でSuperLU分解が行われるはずです。定常反復法のガウス・ザイデル法と比較結果が下の画像です。
import numpy as np import matplotlib.pyplot as plt import scipy.sparse # Make stencils # Creat square wave Num_stencil_x = 101 x_array = np.float64(np.arange(Num_stencil_x)) temperature_array = x_array + 100 temperature_lower_boundary = 150 temperature_upper_boundary = 150 Time_step = 100 Delta_x = max(x_array) / (Num_stencil_x-1) C = 1 Delta_t = 0.2 kappa = 0.5 d = kappa * Delta_t / Delta_x**2 total_movement = C * Delta_t * (Time_step+1) exact_temperature_array = (temperature_upper_boundary - temperature_lower_boundary) / (x_array[-1] - x_array[0]) * x_array + temperature_lower_boundary plt.plot(x_array, temperature_array, label="Initial condition") print("Δx:", Delta_x, "Δt:", Delta_t, "d:", d) temperature_sp = temperature_array.copy() for n in range(Time_step): a_matrix = np.identity(len(temperature_sp)) * 2 *(1/d+1) \ - np.eye(len(temperature_sp), k=1) \ - np.eye(len(temperature_sp), k=-1) temp_temperature_array = np.append(np.append( temperature_lower_boundary, temperature_sp), temperature_upper_boundary) b_array = 2 * (1/d - 1) * temperature_sp + temp_temperature_array[2:] + temp_temperature_array[:-2] b_array[0] += temperature_lower_boundary b_array[-1] += temperature_upper_boundary a_csr = scipy.sparse.csr_matrix(a_matrix) temperature_sp = spla.dsolve.spsolve(a_csr, b_array) plt.plot(x_array, temperature_sp, label="SuperLU") plt.legend(loc="lower right") plt.xlabel("x") plt.ylabel("temperature") plt.xlim(0, max(x_array)) plt.ylim(100, 200)6. 反復法ライブラリ
非定常反復法のライブラリがscipyでは実装されております。
scipy.sparse.linalg.isolveの中に入っております。非定常反復法がよくわからない人は前の記事をご覧ください。使用できるライブラリの一部を以下に列挙しておきます。これら以外のライブラリを使いたい場合は、Scipyのマニュアルから探してみてください。
- 対称行列
cg共役勾配法(Conjugate Gradient method : CG法) - 正定値対称行列のみminres最小残差法(MINimum RESidual: MINRES法)- 非対称行列
bicg双共役勾配法(Bi-Conjugate Gradient: BiCG法)cgs自乗共役勾配法(Conjugate Residual Squared: CGS法)bicgstab双共役勾配安定化法(BiCG Stabilization: BiCGSTAB法)gmres一般化最小残差法(Generalized Minimal Residual: GMRES法)qmr準最小残差法(Quasi-Minimal Residual: QMR法)直接法で使用した同じ問題に対して、BiCGSTAB法での実装例とその実行結果を以下に示します。
temperature_sp = temperature_array.copy() for n in range(Time_step): a_matrix = np.identity(len(temperature_sp)) * 2 *(1/d+1) \ - np.eye(len(temperature_sp), k=1) \ - np.eye(len(temperature_sp), k=-1) temp_temperature_array = np.append(np.append( temperature_lower_boundary, temperature_sp), temperature_upper_boundary) b_array = 2.0 * (1/d - 1.0) * temperature_sp + temp_temperature_array[2:] + temp_temperature_array[:-2] b_array[0] += temperature_lower_boundary b_array[-1] += temperature_upper_boundary a_csr = scipy.sparse.csr_matrix(a_matrix) temperature_sp = spla.isolve.bicgstab(a_csr, b_array)[0] plt.plot(x_array, temperature_sp, label="Scipy.isolve (1000 steps)") plt.legend(loc="lower right") plt.xlabel("x") plt.ylabel("temperature") plt.xlim(0, max(x_array)) plt.ylim(100, 200)まとめ
- 疎行列には0を無視する格納方式がある
- LIL方式は疎行列を作成するときに用いることが多い。
- CSR方式とCSC方式は疎行列計算をするときに用いることが多い。
- CSR方式で計算すると、普通の行列の形で計算するより、圧倒的に早く計算が終わることが確認できた。
- Matrix Market方式で疎行列は配布されることが多い。
- 直接法と非定常反復法の関数がScipyに実装されており、簡単に使用することができる。
という感じかと思います。
参考文献