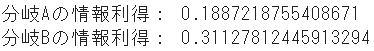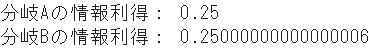- 投稿日:2020-08-08T22:56:14+09:00
JupyterにGTFS Realtimeの位置情報をプロットしたい!(バルーン付き)
はじめに
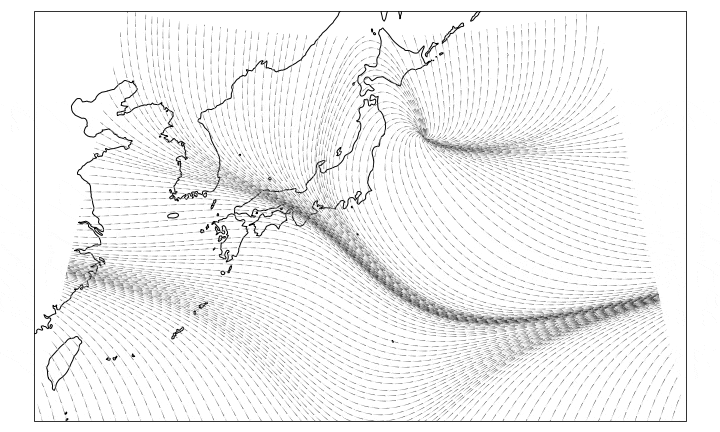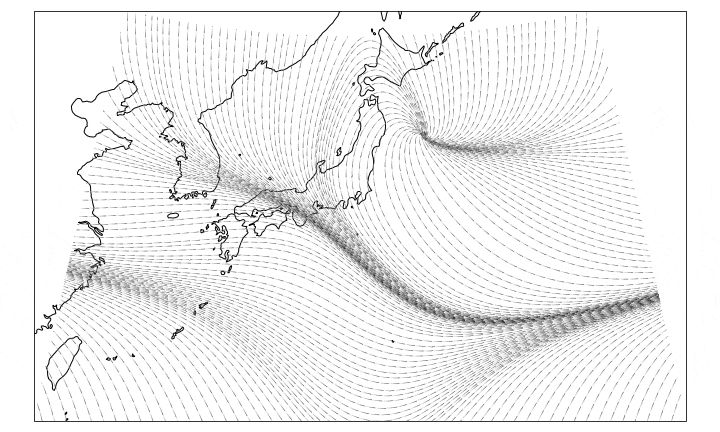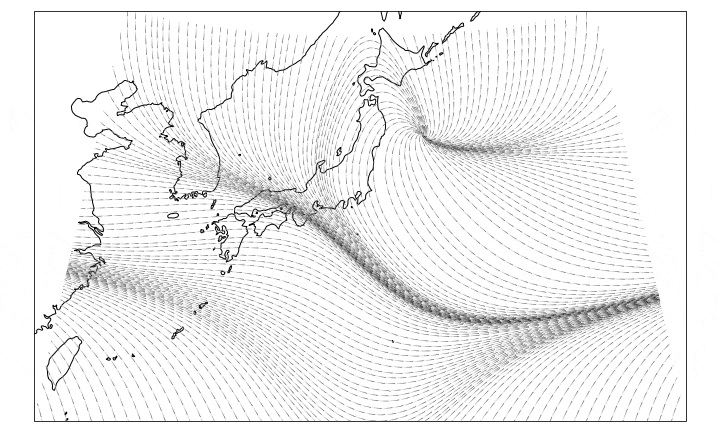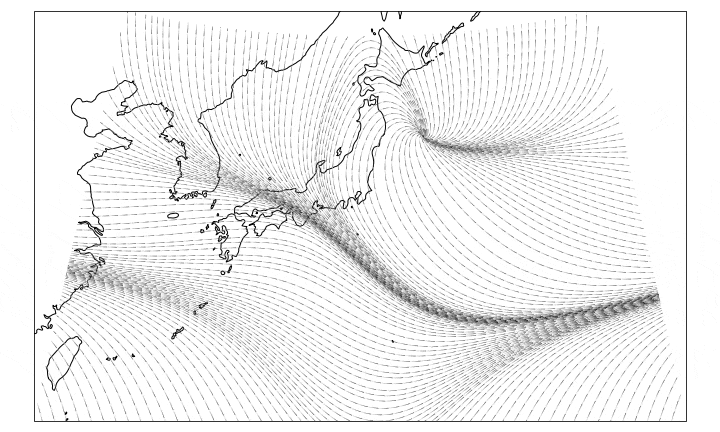
バスの現在位置は、Google Mapからでも見られる時代ですが、自分の手でマッピングしてみましょう。pythonにfoliumというWeb地図表示プラグインを用いて表示してみます。
地図のアイコンはクリッカブルで、詳細な情報を表示することも可能です。実装
realtimebus.py3from google.transit import gtfs_realtime_pb2 import urllib.request, urllib.error import folium import pandas as pd #GTFS-RTの公開されているURL(宇野バスさん) url ='http://www3.unobus.co.jp/GTFS/GTFS_RT-VP.bin' #カラム名の宣言 list_df = pd.DataFrame(columns=['id' , 'vehicle_id', 'trip_id','vehicle_timestamp','longitude','latitude','occupancy_status']) feed = gtfs_realtime_pb2.FeedMessage() #データダウンロードとフォーマット変換 with urllib.request.urlopen(url) as res: #データダウンロード feed.ParseFromString(res.read()) #プロトコルバッファをDeserialize for entity in feed.entity: tmp_se = pd.Series( [ entity.id, #車両ID entity.vehicle.vehicle.id, #車両ナンバー entity.vehicle.trip.trip_id, #路線番号? entity.vehicle.timestamp, #車両時刻 entity.vehicle.position.longitude, #車両緯度 entity.vehicle.position.latitude, #車両経度 entity.vehicle.occupancy_status #混雑度 ], index=list_df.columns ) list_df = list_df.append( tmp_se, ignore_index=True ) #地図の中心地点を算出 average_pos = list_df.mean() # 中心座標付近の地図を作成 m = folium.Map(location=[average_pos['latitude'], average_pos['longitude']], zoom_start=11) # 各バスをプロット(色は青) list_df.apply(lambda row: folium.Marker( location=[row['latitude'], row['longitude']], popup='<table border="1"><tr><th>occupancy_status</th></tr><tr><td>' +str(row['occupancy_status']) +'</td></tr></table>', icon=folium.Icon(color='blue',icon='bus', prefix='fa') ).add_to(m), axis=1) m結果
ダウンロードされたリアルタイム位置情報が地図上にプロットされました
実行したタイミングは22時半過ぎてますが、まだバス動いているのですね。。。
- 投稿日:2020-08-08T22:45:27+09:00
【Python】データサイエンス100本ノック(構造化データ加工編) 022 解説
Youtube
動画解説もしています。
問題
P-022: レシート明細データフレーム(df_receipt)の顧客ID(customer_id)に対し、ユニーク件数をカウントせよ。
解答
コードlen(df_receipt['customer_id'].unique())出力8307解説
・PandasのDataFrame/Seriesにて、ユニークな行数をカウントする方法です。
・ユニークな行数が全部で何件あるのかを確認したい時に使用します。※ちなみに、以下のコードもユニークな件数をカウントするコードであり、同じ結果を出力します
コードlen(df_receipt['customer_id'].value_counts())
- 投稿日:2020-08-08T21:54:36+09:00
【Django】adminページをカスタマイズしたいって?
はじめに
「Djangoの管理者ページのカスタマイズなんて、今までしたことないよ〜」という方向け。
初級編です。カスタマイズしなくても十分使えそうだが、エンジニア以外の人が触るようになると、デフォルトだと少し寂しい?
詳細な解説というより、ググると良い用語をパパッと上げていくスタイルです。
ちゃんとまとめると大変ですからね。。。Djangoのドキュメント
https://docs.djangoproject.com/en/3.0/ref/contrib/admin/ModelAdminクラス
基本的には、
ModelAdminクラスのサブクラスを作成して、カスタマイズ内容を記述する。
例えばSampleModelという名前のモデルをカスタマイズしようと思ったら、以下のようにする。admin.pyfrom django.contrib import admin # 通常は「モデル名+Admin」という名前にする class SampleModelAdmin(admin.ModelAdmin): ''' このクラスの中にカスタマイズ内容を記述 ''' # SampleModelにSampleModelAdminを適用 admin.site.register(SampleModel, SampleModelAdmin)これ以降は、ModelAdminクラスの中の記述のみを書く。
例えばdef hoge(): returnと書いたら、実際は
admin.pyclass SampleModelAdmin(admin.ModelAdmin): def hoge(): returnと読み替えてください。
どんなカラムを表示するか
レコード一覧の一番上にカラム名が表示されるが、どんなカラムを表示したいか設定しよう。
以下はモデルがtext,updated_atというフィールドを持っていた場合の例。list_display = ('__str__', 'text', 'updated_at') # この3つのカラムを表示する次に、フィールドにはない値を表示する場合。
list_display = ('__str__', 'text', 'updated_at', 'get_owner_name') def get_owner_name(self, obj): # 第二引数のobjはレコード自身を表す return "I am owner" get_owner_name.short_description = 'オーナー名'こうすると「オーナー名」というカラムが追加され、この場合は全てのレコードに「I am owner」と表示される。
検索対象を設定する
上部に検索ボックスが存在するが、その検索対象を絞ることができる。
# SampleModel.text, SampleModel.updated_at, SampleModel.owner.username を検索対象にする search_fields = ('text', 'updated_at', 'owner__username`)操作を追加する
デフォルトでは「選択された<モデル名>の削除」という操作が存在するはず。
自作のアクションは以下のようにactionsにリスト形式で記述するが、ここに明示的に含めなくても「選択された<モデル名>の削除」は存在し続ける。actions = ['my_custom_action'] def my_custom_action(self, request, queryset): # なんか処理 return None my_custom_action.short_description = '私だけのアクション'
return Noneとすれば操作完了時に何もしないが、例えばテンプレートを返してページ遷移なんかもできる。モデルの表示名を変更する
これは
ModelAdminではなく元のModelクラスに書いておく。class SampleModel(models.Model): class Meta: verbose_name = 'サンプルモデル' verbose_name_plural = 'サンプルモデル一覧'フィールドにリンクを貼るか
list_display_links = ('text',)応用編:検索処理のカスタマイズ
検索対象のカスタマイズを紹介しましたが、検索ワード(クエリ)が与えられた時に、実際にどのような検索を行うかもカスタマイズできます。
これは長くなるので概要だけにしますが、以下の関数を定義して検索処理を上書きします。def get_changelist(self, request, **kwargs):最後に
以上のカスタマイズができれば、はじめはそこそこ満足ではないでしょうか?
adminページのテンプレート自体のカスタマイズや、admi内でのページ遷移については別の記事で書こうかと思います。
- 投稿日:2020-08-08T21:44:40+09:00
2. Pythonで綴る多変量解析 7-2. 決定木(scikit-learn)[分割基準の違い]
- 決定木における分類の分割基準はいくつかあります。
- 一般的に使用されているものに、ジニ不純度(gini impurity)、エントロピー(entropy)という2つの不純物測定と、それから分類誤差(misclassification error)という分割基準があります。
- これらの違いを見ていきます。
import numpy as np import matplotlib.pyplot as plt %matplotlib inline2クラス分類での各指標の比較
- 2クラス分類で、一方のクラスの割合を p とすると、各指標はそれぞれ次のように求められます。
- ジニ(gini): $2p * (1-p)$
- エントロピー(entropy): $-p * \log p-(1-p) * log(1-p)$
- 誤分類率(misclassification rate): $1-max(p, 1-p)$
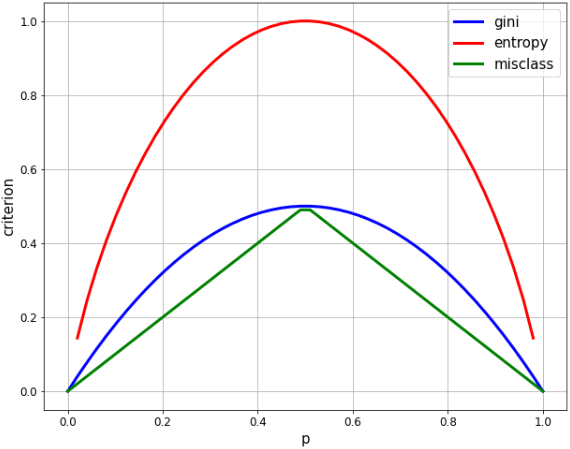
# p に相当する等差数列を生成 xx = np.linspace(0, 1, 50) # 開始値0、終了値1、要素数50 plt.figure(figsize=(10, 8)) # 各指標を計算 gini = [2 * x * (1-x) for x in xx] entropy = [-x * np.log2(x) - (1-x) * np.log2(1-x) for x in xx] misclass = [1 - max(x, 1-x) for x in xx] # グラフを表示 plt.plot(xx, gini, label='gini', lw=3, color='b') plt.plot(xx, entropy, label='entropy', lw=3, color='r') plt.plot(xx, misclass, label='misclass', lw=3, color='g') plt.xlabel('p', fontsize=15) plt.ylabel('criterion', fontsize=15) plt.legend(fontsize=15) plt.xticks(fontsize=12) plt.yticks(fontsize=12) plt.grid()
- 2クラスの割合が均等のとき(p=0.5)、いずれも最大値となりますが、
- エントロピーの最大値は 1.0、ジニは 0.5 なので、比較しやすいようにエントロピーの方を1/2の係数でスケーリングします。
# p に相当する等差数列を生成 xx = np.linspace(0, 1, 50) # 開始値0、終了値1、要素数50 plt.figure(figsize=(10, 8)) # 各指標を計算 gini = [2 * x * (1-x) for x in xx] entropy = [(x * np.log((1-x)/x) - np.log(1-x)) / (np.log(2)) for x in xx] entropy_scaled = [(x * np.log((1-x)/x) - np.log(1-x)) / (2*np.log(2)) for x in xx] misclass = [1 - max(x, 1-x) for x in xx] # グラフを表示 plt.plot(xx, gini, label='gini', lw=3, color='b') plt.plot(xx, entropy, label='entropy', lw=3, color='r', linestyle='dashed') plt.plot(xx, entropy_scaled, label='entropy(scaled)', lw=3, color='r') plt.plot(xx, misclass, label='misclass', lw=3, color='g') plt.xlabel('p', fontsize=15) plt.ylabel('criterion', fontsize=15) plt.legend(fontsize=15) plt.xticks(fontsize=12) plt.yticks(fontsize=12) plt.grid()
- ジニ(gini)とエントロピー(entropy)はよく似ていて、どちらも二次曲線を描いて p = 1/2 で最大化しています。
- 誤分類率(misclass)は、線形であるという点で明らかに異なりますが、多くの同じ特性を共有しています。p = 1/2 で最大化し、p = 0, 1 でゼロに等しく、山なりに変化しています。
情報利得にみる各指標の違い
1. 情報利得とは
- 情報利得(information gain)とは、ある変数を使ってデータを分割したとき、分割の前後でどれだけ不純度が減少したかを表す指標です。
- 不純度が減れば減るほど、その変数は分類条件として有益だということになります。
- その意味で、いかに不純度を低減できるか、ということは各分岐における情報利得の最大化と同義といえます。
- 2クラス分類の場合、情報利得(IG)は次の式に定義されます。
- $\displaystyle IG(D_{p}, a) = I(D_{p}) - \frac{N_{left}}{N} I(D_{left}) - \frac{N_{right}}{N} I(D_{right})$
- $I$は不純度を意味し、$D_{p}$は親(parent)ノードのデータ、子ノードのデータの左右が$D_{left}$と$D_{right}$で、また親ノードのサンプル数$N$を分母として子ノードの左右それぞれの割合となっています。
2. 各指標での情報利得を計算する
➀ジニ不純度による情報利得
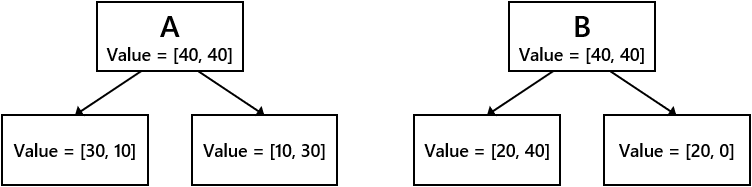
# 親ノードのジニ不純度 IGg_p = 2 * 1/2 * (1-(1/2)) # 子ノードAのジニ不純度 IGg_A_l = 2 * 3/4 * (1-(3/4)) # 左 IGg_A_r = 2 * 1/4 * (1-(1/4)) # 右 # 子ノードBのジニ不純度 IGg_B_l = 2 * 2/6 * (1-(2/6)) # 左 IGg_B_r = 2 * 2/2 * (1-(2/2)) # 右 # 各分岐の情報利得 IG_gini_A = IGg_p - 4/8 * IGg_A_l - 4/8 * IGg_A_r IG_gini_B = IGg_p - 6/8 * IGg_B_l - 2/8 * IGg_B_r print("分岐Aの情報利得:", IG_gini_A) print("分岐Bの情報利得:", IG_gini_B)➁エントロピーによる情報利得
# 親ノードのエントロピー IGe_p = (4/8 * np.log((1-4/8)/(4/8)) - np.log(1-4/8)) / (np.log(2)) # 子ノードAのエントロピー IGe_A_l = (3/4 * np.log((1-3/4)/(3/4)) - np.log(1-3/4)) / (np.log(2)) # 左 IGe_A_r = (1/4 * np.log((1-1/4)/(1/4)) - np.log(1-1/4)) / (np.log(2)) # 右 # 子ノードBのエントロピー IGe_B_l = (2/6 * np.log((1-2/6)/(2/6)) - np.log(1-2/6)) / (np.log(2)) # 左 IGe_B_r = (2/2 * np.log((1-2/2+1e-7)/(2/2)) - np.log(1-2/2+1e-7)) / (np.log(2)) # 右、+1e-7は0除算回避のために微小値を加算 # 各分岐の情報利得 IG_entropy_A = IGe_p - 4/8 * IGe_A_l - 4/8 * IGe_A_r IG_entropy_B = IGe_p - 6/8 * IGe_B_l - 2/8 * IGe_B_r print("分岐Aの情報利得:", IG_entropy_A) print("分岐Bの情報利得:", IG_entropy_B)➂誤分類率による情報利得
# 親ノードの誤分類率 IGm_p = 1 - np.maximum(4/8, 1-4/8) # 子ノードAの誤分類率 IGm_A_l = 1 - np.maximum(3/4, 1-3/4) # 左 IGm_A_r = 1 - np.maximum(1/4, 1-1/4) # 右 # 子ノードBの誤分類率 IGm_B_l = 1 - np.maximum(2/6, 1-2/6) # 左 IGm_B_r = 1 - np.maximum(2/2, 1-2/2) # 右 # 各分岐の情報利得 IG_misclass_A = IGm_p - 4/8 * IGm_A_l - 4/8 * IGm_A_r IG_misclass_B = IGm_p - 6/8 * IGm_B_l - 2/8 * IGm_B_r print("分岐Aの情報利得:", IG_misclass_A) print("分岐Bの情報利得:", IG_misclass_B)まとめ
分類条件A 分類条件B ジニ不純度 0.125 0.167 エントロピー 0.189 0.311 誤分類率 0.250 0.250
- ジニとエントロピーでは共に、分類条件Bの方が優先されるのは明白です。実際、結果も非常に似たものになります。
- 一方、誤分類率では、分類条件AとBがほぼ同率となっています。
- このように、誤分類率は変数間で明らかな差分をとらない傾向があることから、sklearnの決定木モデルでは、分割基準をジニとエントロピーの2つとしている(らしいです)。
- なお、ジニの方が、エントロピーのような対数の計算がない分だけ少し処理が速いかも知れませんね。
- 投稿日:2020-08-08T21:44:40+09:00
2. Pythonで綴る多変量解析 7-2. 決定木[分割基準の違い]
- 決定木における分類の分割基準はいくつかあります。
- 一般的に使用されているものに、ジニ不純度(gini impurity)、エントロピー(entropy)という2つの不純物測定と、それから分類誤差(misclassification error)という分割基準があります。
- これらの違いを見ていきます。
import numpy as np import matplotlib.pyplot as plt %matplotlib inline2クラス分類での各指標の比較
- 2クラス分類で、一方のクラスの割合を p とすると、各指標はそれぞれ次のように求められます。
- ジニ(gini): $2p * (1-p)$
- エントロピー(entropy): $-p * \log p-(1-p) * log(1-p)$
- 誤分類率(misclassification rate): $1-max(p, 1-p)$
# p に相当する等差数列を生成 xx = np.linspace(0, 1, 50) # 開始値0、終了値1、要素数50 plt.figure(figsize=(10, 8)) # 各指標を計算 gini = [2 * x * (1-x) for x in xx] entropy = [-x * np.log2(x) - (1-x) * np.log2(1-x) for x in xx] misclass = [1 - max(x, 1-x) for x in xx] # グラフを表示 plt.plot(xx, gini, label='gini', lw=3, color='b') plt.plot(xx, entropy, label='entropy', lw=3, color='r') plt.plot(xx, misclass, label='misclass', lw=3, color='g') plt.xlabel('p', fontsize=15) plt.ylabel('criterion', fontsize=15) plt.legend(fontsize=15) plt.xticks(fontsize=12) plt.yticks(fontsize=12) plt.grid()
- 2クラスの割合が均等のとき(p=0.5)、いずれも最大値となりますが、
- エントロピーの最大値は 1.0、ジニは 0.5 なので、比較しやすいようにエントロピーの方を1/2の係数でスケーリングします。
# p に相当する等差数列を生成 xx = np.linspace(0, 1, 50) # 開始値0、終了値1、要素数50 plt.figure(figsize=(10, 8)) # 各指標を計算 gini = [2 * x * (1-x) for x in xx] entropy = [(x * np.log((1-x)/x) - np.log(1-x)) / (np.log(2)) for x in xx] entropy_scaled = [(x * np.log((1-x)/x) - np.log(1-x)) / (2*np.log(2)) for x in xx] misclass = [1 - max(x, 1-x) for x in xx] # グラフを表示 plt.plot(xx, gini, label='gini', lw=3, color='b') plt.plot(xx, entropy, label='entropy', lw=3, color='r', linestyle='dashed') plt.plot(xx, entropy_scaled, label='entropy(scaled)', lw=3, color='r') plt.plot(xx, misclass, label='misclass', lw=3, color='g') plt.xlabel('p', fontsize=15) plt.ylabel('criterion', fontsize=15) plt.legend(fontsize=15) plt.xticks(fontsize=12) plt.yticks(fontsize=12) plt.grid()
- ジニ(gini)とエントロピー(entropy)はよく似ていて、どちらも二次曲線を描いて p = 1/2 で最大化しています。
- 誤分類率(misclass)は、線形であるという点で明らかに異なりますが、多くの同じ特性を共有しています。p = 1/2 で最大化し、p = 0, 1 でゼロに等しく、山なりに変化しています。
情報利得にみる各指標の違い
1. 情報利得とは
- 情報利得(information gain)とは、ある変数を使ってデータを分割したとき、分割の前後でどれだけ不純度が減少したかを表す指標です。
- 不純度が減れば減るほど、その変数は分類条件として有益だということになります。
- その意味で、いかに不純度を低減できるか、ということは各分岐における情報利得の最大化と同義といえます。
- 2クラス分類の場合、情報利得(IG)は次の式に定義されます。
- $\displaystyle IG(D_{p}, a) = I(D_{p}) - \frac{N_{left}}{N} I(D_{left}) - \frac{N_{right}}{N} I(D_{right})$
- $I$は不純度を意味し、$D_{p}$は親(parent)ノードのデータ、子ノードのデータの左右が$D_{left}$と$D_{right}$で、また親ノードのサンプル数$N$を分母として子ノードの左右それぞれの割合となっています。
2. 各指標での情報利得を計算する
➀ジニ不純度による情報利得
# 親ノードのジニ不純度 IGg_p = 2 * 1/2 * (1-(1/2)) # 子ノードAのジニ不純度 IGg_A_l = 2 * 3/4 * (1-(3/4)) # 左 IGg_A_r = 2 * 1/4 * (1-(1/4)) # 右 # 子ノードBのジニ不純度 IGg_B_l = 2 * 2/6 * (1-(2/6)) # 左 IGg_B_r = 2 * 2/2 * (1-(2/2)) # 右 # 各分岐の情報利得 IG_gini_A = IGg_p - 4/8 * IGg_A_l - 4/8 * IGg_A_r IG_gini_B = IGg_p - 6/8 * IGg_B_l - 2/8 * IGg_B_r print("分岐Aの情報利得:", IG_gini_A) print("分岐Bの情報利得:", IG_gini_B)➁エントロピーによる情報利得
# 親ノードのエントロピー IGe_p = (4/8 * np.log((1-4/8)/(4/8)) - np.log(1-4/8)) / (np.log(2)) # 子ノードAのエントロピー IGe_A_l = (3/4 * np.log((1-3/4)/(3/4)) - np.log(1-3/4)) / (np.log(2)) # 左 IGe_A_r = (1/4 * np.log((1-1/4)/(1/4)) - np.log(1-1/4)) / (np.log(2)) # 右 # 子ノードBのエントロピー IGe_B_l = (2/6 * np.log((1-2/6)/(2/6)) - np.log(1-2/6)) / (np.log(2)) # 左 IGe_B_r = (2/2 * np.log((1-2/2+1e-7)/(2/2)) - np.log(1-2/2+1e-7)) / (np.log(2)) # 右、+1e-7は0除算回避のために微小値を加算 # 各分岐の情報利得 IG_entropy_A = IGe_p - 4/8 * IGe_A_l - 4/8 * IGe_A_r IG_entropy_B = IGe_p - 6/8 * IGe_B_l - 2/8 * IGe_B_r print("分岐Aの情報利得:", IG_entropy_A) print("分岐Bの情報利得:", IG_entropy_B)➂誤分類率による情報利得
# 親ノードの誤分類率 IGm_p = 1 - np.maximum(4/8, 1-4/8) # 子ノードAの誤分類率 IGm_A_l = 1 - np.maximum(3/4, 1-3/4) # 左 IGm_A_r = 1 - np.maximum(1/4, 1-1/4) # 右 # 子ノードBの誤分類率 IGm_B_l = 1 - np.maximum(2/6, 1-2/6) # 左 IGm_B_r = 1 - np.maximum(2/2, 1-2/2) # 右 # 各分岐の情報利得 IG_misclass_A = IGm_p - 4/8 * IGm_A_l - 4/8 * IGm_A_r IG_misclass_B = IGm_p - 6/8 * IGm_B_l - 2/8 * IGm_B_r print("分岐Aの情報利得:", IG_misclass_A) print("分岐Bの情報利得:", IG_misclass_B)まとめ
分類条件A 分類条件B ジニ不純度 0.125 0.167 エントロピー 0.189 0.311 誤分類率 0.250 0.250
- ジニとエントロピーでは共に、分類条件Bの方が優先されるのは明白です。実際、結果も非常に似たものになります。
- 一方、誤分類率では、分類条件AとBがほぼ同率となっています。
- このように、誤分類率は変数間で明らかな差分をとらない傾向があることから、sklearnの決定木モデルでは、分割基準をジニとエントロピーの2つとしている(らしいです)。
- なお、ジニの方が、エントロピーのような対数の計算がない分だけ少し処理が速いかも知れませんね。
- 投稿日:2020-08-08T21:37:12+09:00
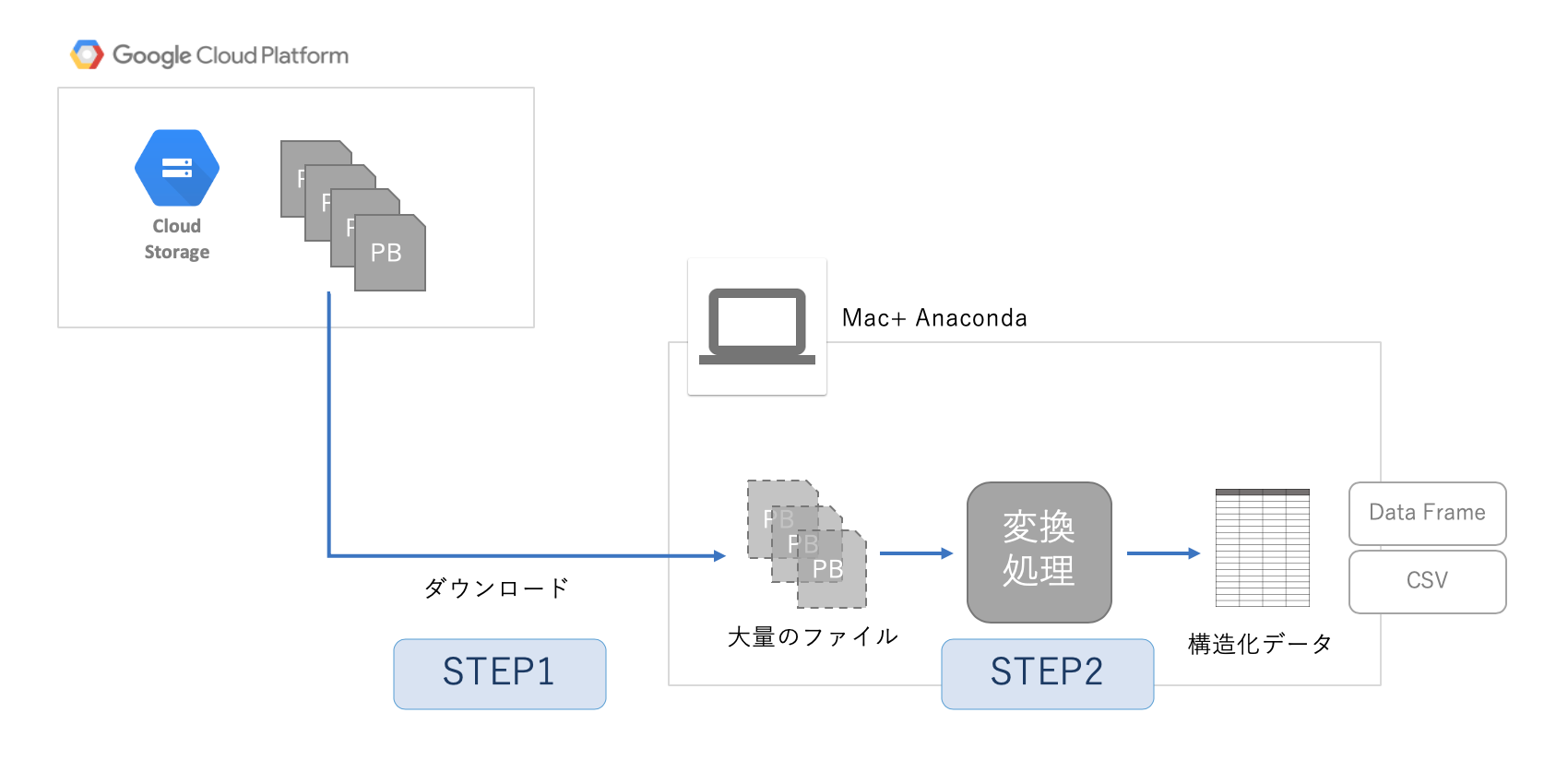
大量のGTFSのファイルに対するETL処理(Python編)
偉大な先人
既に伊藤先生が、GTFSファイルをマージしてPostgreSQL+PostGISに投入する方法、ツールを活用してPostgreSQLにデータを投入する方法を掲載しております。
今回のモチベーション
日本のGTFSデータが、GCSに保存されているとの情報を入手したので、そのデータで遊ぶための準備です。
今回やること
Google Clound Platform のファイルサービスである、Google Cloud Strage 保存されている protocol buffers 形式の大量のファイルを、一気にダウンロードして一纏めのデータに変換します.
STEP1 : gsutilを用いてGCSからデータをダウンロードします
STEP2 : pythonを用いてファイルから構造化データに変換します。 変換対象は、Data Frame形式とCSV種類実装
環境について
今回のコードはJupyter labなどに貼り付けて利用することを想定しています。
単体でツールとしても使えませんし、ライブラリとしても利用できません。
実装した環境はMacです。STEP1 gsutilを用いたデータコピー
GCPにはpython用のツールもありますが、アカウント設定等が煩雑な為、コマンドラインツールであるgsutilを用いて、フォルダの中にあるファイルをまとめてローカルにダウンロードします。
dowload.shgsutil cp -r gs://BACKET_NAME/realtime/unobus.co.jp/vehicle_position/2020/20200801/ ~/dev/unobus/解説
gsutil cpはUNIXのcpコマンドと似ています。-r再帰的に処理するオプションgs://BACKET_NAME/realtime/un....コピー元です。今回はダミーの文字列~/dev/unobus/コピー先です一般的に、
gsutil -m cpとするとマルチスレッドで動作し高速化できます。しかし、今回のバケットのアクセス権限の問題なのか上手く動作しませんでした。STEP2 ファイルの読み込みと構造化
データフレームに変換
pq2df.pyfrom google.transit import gtfs_realtime_pb2 import pandas as pd import numpy as np import os import datetime import time path = '/Users/USER_NAME/dev/unobus/20200801' files = os.listdir(path) feed = gtfs_realtime_pb2.FeedMessage() start = time.time() i = 0; temp_dict = {} for file in files: with open(path+'/'+file, 'rb') as f: data = f.read() feed.ParseFromString(data) for entity in feed.entity: temp_dict[i] = [ entity.id, #車両ID entity.vehicle.vehicle.id, #車両ナンバー entity.vehicle.trip.trip_id, #路線番号? entity.vehicle.timestamp, #車両時刻 entity.vehicle.position.longitude, #車両緯度 entity.vehicle.position.latitude, #車両経度 entity.vehicle.occupancy_status #混雑度 ] i +=1 df = pd.DataFrame.from_dict(temp_dict, orient='index',columns=['id' , 'vehicle_id', 'trip_id','vehicle_timestamp','longitude','latitude','occupancy_status']) elapsed_time = time.time() - start print ("elapsed_time:{0}".format(elapsed_time) + "[sec]")CSVへ出力
pq2csv.pyfrom google.transit import gtfs_realtime_pb2 import pandas as pd import numpy as np import os import datetime import time path = '/Users/USER_NAME/dev/unobus/20200801' csvfilename = 'unobus_20200801.csv' files = os.listdir(path) feed = gtfs_realtime_pb2.FeedMessage() with open(csvfilename, 'a') as csv : start = time.time() for file in files: with open(path+'/'+file, 'rb') as f: data = f.read() feed.ParseFromString(data) for entity in feed.entity: print( entity.id, #車両ID entity.vehicle.vehicle.id, #車両ナンバー entity.vehicle.trip.trip_id, #路線番号? entity.vehicle.timestamp, #車両時刻 entity.vehicle.position.longitude, #車両緯度 entity.vehicle.position.latitude, #車両経度 entity.vehicle.occupancy_status, #混雑度 sep=',',file=csv) elapsed_time = time.time() - start print ("elapsed_time:{0}".format(elapsed_time) + "[sec]")失敗作 データフレームに変換(超低速)
pq2df.pyと比較すると、220倍も遅いコードです。
df.appendがその原因です。pq2df_VeryLowSpeed.pyfrom google.transit import gtfs_realtime_pb2 import pandas as pd import numpy as np import os import datetime import time path = '/Users/USER_NAME/dev/unobus/20200801' files = os.listdir(path) feed = gtfs_realtime_pb2.FeedMessage() df = pd.DataFrame(columns=['id' , 'vehicle_id', 'trip_id','vehicle_timestamp','longitude','latitude','occupancy_status']) start = time.time() for file in files: with open(path+'/'+file, 'rb') as f: data = f.read() feed.ParseFromString(data) for entity in feed.entity: tmp_se = pd.Series( [ entity.id, #車両ID entity.vehicle.vehicle.id, #車両ナンバー entity.vehicle.trip.trip_id, #路線番号? entity.vehicle.timestamp, #車両時刻 entity.vehicle.position.longitude, #車両緯度 entity.vehicle.position.latitude, #車両経度 entity.vehicle.occupancy_status #混雑度 ], index=df.columns ) df = df.append( tmp_se, ignore_index=True ) #ここがだめ!! elapsed_time = time.time() - start print ("elapsed_time:{0}".format(elapsed_time) + "[sec]")終わりに
クラウド上に保存されているGTFSの過去データを、ダウンロードから構造化データへの変換までを実施しました。
DataFrameでデータを追記する処理は書き方によっては非常に処理が遅くなり、苦労しました。微力ではありますが、バスデータ活用に貢献できれば幸いです。
- 投稿日:2020-08-08T21:37:12+09:00
大量のGTFS Realtimeのファイルに対するETL処理(Python編)
偉大な先人
既に伊藤先生が、GTFSファイルをマージしてPostgreSQL+PostGISに投入する方法にて、ツールを活用してPostgreSQLにデータを投入する方法を掲載しております。
今回のモチベーション
日本のGTFSデータが、GCSに保存されているとの情報を入手したので、そのデータで遊ぶための準備です。
今回やること
Google Clound Platform のファイルサービスである、Google Cloud Strage 保存されている protocol buffers 形式の大量のファイルを、一気にダウンロードして一纏めのデータに変換します.
STEP1 : gsutilを用いてGCSからデータをダウンロードします
STEP2 : pythonを用いてファイルから構造化データに変換します。 変換対象は、Data Frame形式とCSVの2種類実装
環境について
今回のコードはJupyter labなどに貼り付けて利用することを想定しています。
単体でツールとしても使えませんし、ライブラリとしても利用できません。
実装した環境はMacです。STEP1 gsutilを用いたデータコピー
GCPにはpython用のツールもありますが、アカウント設定等が煩雑な為、コマンドラインツールであるgsutilを用いて、フォルダの中にあるファイルをまとめてローカルにダウンロードします。
dowload.shgsutil cp -r gs://BACKET_NAME/realtime/unobus.co.jp/vehicle_position/2020/20200801/ ~/dev/unobus/解説
gsutil cpはUNIXのcpコマンドと似ています。-r再帰的に処理するオプションgs://BACKET_NAME/realtime/un....コピー元です。今回はダミーの文字列~/dev/unobus/コピー先です一般的に、
gsutil -m cpとするとマルチスレッドで動作し高速化できます。しかし、今回のバケットのアクセス権限の問題なのか上手く動作しませんでした。STEP2 ファイルの読み込みと構造化
データフレームに変換
pq2df.pyfrom google.transit import gtfs_realtime_pb2 import pandas as pd import numpy as np import os import datetime import time path = '/Users/USER_NAME/dev/unobus/20200801' files = os.listdir(path) feed = gtfs_realtime_pb2.FeedMessage() start = time.time() i = 0; temp_dict = {} for file in files: with open(path+'/'+file, 'rb') as f: data = f.read() feed.ParseFromString(data) for entity in feed.entity: temp_dict[i] = [ entity.id, #車両ID entity.vehicle.vehicle.id, #車両ナンバー entity.vehicle.trip.trip_id, #路線番号? entity.vehicle.timestamp, #車両時刻 entity.vehicle.position.longitude, #車両緯度 entity.vehicle.position.latitude, #車両経度 entity.vehicle.occupancy_status #混雑度 ] i +=1 df = pd.DataFrame.from_dict(temp_dict, orient='index',columns=['id' , 'vehicle_id', 'trip_id','vehicle_timestamp','longitude','latitude','occupancy_status']) elapsed_time = time.time() - start print ("elapsed_time:{0}".format(elapsed_time) + "[sec]")CSVへ出力
pq2csv.pyfrom google.transit import gtfs_realtime_pb2 import pandas as pd import numpy as np import os import datetime import time path = '/Users/USER_NAME/dev/unobus/20200801' csvfilename = 'unobus_20200801.csv' files = os.listdir(path) feed = gtfs_realtime_pb2.FeedMessage() with open(csvfilename, 'a') as csv : start = time.time() for file in files: with open(path+'/'+file, 'rb') as f: data = f.read() feed.ParseFromString(data) for entity in feed.entity: print( entity.id, #車両ID entity.vehicle.vehicle.id, #車両ナンバー entity.vehicle.trip.trip_id, #路線番号? entity.vehicle.timestamp, #車両時刻 entity.vehicle.position.longitude, #車両緯度 entity.vehicle.position.latitude, #車両経度 entity.vehicle.occupancy_status, #混雑度 sep=',',file=csv) elapsed_time = time.time() - start print ("elapsed_time:{0}".format(elapsed_time) + "[sec]")失敗作 データフレームに変換(超低速)
pq2df.pyと比較すると、220倍も遅いコードです。
df.appendがその原因です。pq2df_VeryLowSpeed.pyfrom google.transit import gtfs_realtime_pb2 import pandas as pd import numpy as np import os import datetime import time path = '/Users/USER_NAME/dev/unobus/20200801' files = os.listdir(path) feed = gtfs_realtime_pb2.FeedMessage() df = pd.DataFrame(columns=['id' , 'vehicle_id', 'trip_id','vehicle_timestamp','longitude','latitude','occupancy_status']) start = time.time() for file in files: with open(path+'/'+file, 'rb') as f: data = f.read() feed.ParseFromString(data) for entity in feed.entity: tmp_se = pd.Series( [ entity.id, #車両ID entity.vehicle.vehicle.id, #車両ナンバー entity.vehicle.trip.trip_id, #路線番号? entity.vehicle.timestamp, #車両時刻 entity.vehicle.position.longitude, #車両緯度 entity.vehicle.position.latitude, #車両経度 entity.vehicle.occupancy_status #混雑度 ], index=df.columns ) df = df.append( tmp_se, ignore_index=True ) #ここがだめ!! elapsed_time = time.time() - start print ("elapsed_time:{0}".format(elapsed_time) + "[sec]")終わりに
クラウド上に保存されているGTFSの過去データを、ダウンロードから構造化データへの変換までを実施しました。
DataFrameでデータを追記する処理は書き方によっては非常に処理が遅くなり、苦労しました。微力ではありますが、バスデータ活用に貢献できれば幸いです。
- 投稿日:2020-08-08T20:37:17+09:00
はじめてのPython3 ~番外編:数値ランダム狂想曲~
はじめに
注意点などはこちらを参照してください。
この記事を描こうと思ったきっかけ
Pythonの計算に関して、適当な数値を入れてみてみたい。
そこでランダムな数値を精製してくれるrandomモジュールを使用してみることにしました。
そんなrandomモジュールについての備忘録のようなものです。
(更新予定の途中記事です。)ようこそ、乱数の世界へ
randomモジュールをインポート
randomモジュールはPythonの標準ライブラリに含まれているため、ダウンロードは入りません。
ただし、インポートは必要です。import random # 上部の段にこれを記述randomモジュールの関数
random.random()
0.0以上1.0未満の浮動小数点数float型の乱数を生成します。
import random print(random.random()) # 実行結果↓ # 0.830963714448 # 0.253643551299 # 0.906046050863 などrandom.uniform(a,b)
任意の範囲(a から b の間)の浮動小数点数float型の乱数を生成する。
import random print(random.uniform(10,20)) # 実行結果↓ # 10.4740789672 # 14.8562079851 # 10.2516560825 など・2つの数値は大きさ順不同。
import random print(random.uniform(1,-2)) # 実行結果↓ # -14.6138465193 # -1.29546679922 # 5.63021229752 など・もしも同じ数だったら、その値のみ返す。
import random print(random.uniform(10,10)) # 実行結果↓ # 10.0引数は浮動小数点数float型でも可能。
※ bの値が範囲に含まれるかはドキュメント引用部分より、a + (b-a) * random.random()の丸めに依存。端点の値 b が範囲に含まれるかどうかは、等式 a + (b-a) * random() における浮動小数点の丸めに依存します。 ー Pythonドキュメント:random --- 擬似乱数を生成する より引用
import random print(random.uniform(1.234,4.321)) # 実行結果↓ # 1.93441022683 # 2.75716839399 # 3.91940634098 など参考
https://docs.python.org/ja/3/library/random.html#functions-for-integers
- 投稿日:2020-08-08T20:28:35+09:00
【データサイエンティスト入門】Pythonの基礎♬
今日から、「東京大学のデータサイエンティスト育成講座」を読んで、ちょっと疑問を持ったところや有用だと感じた部分をまとめて行こうと思う。
したがって、あらすじはまんまになると思うが内容は本書とは関係ないと思って読んでいただきたい。
また、環境は問題が発生するまで、Python3.6で実施する。
Python 3.6.10 |Anaconda, Inc.| (default, Jan 7 2020, 15:18:16) [MSC v.1916 64 bit (AMD64)] on win32Chapter1-1 データサイエンティストの仕事
図1-1-1は、...会計士というのが入っているけど、何だろう??
ドメイン知識とかでいいような気がした。Chapter1-2 Pythonの基礎
Jupyter Notebookの使い方から、入るけど割愛します。
1-2-2 Pythonの基礎
1-2-2-1 変数
以下のコードは、
msg = 'test' print(msg) print(msg[0]) print(msg[1]) print(msg[5])結果
test t e Traceback (most recent call last): File "msg-ex.py", line 5, in <module> print(msg[5]) IndexError: string index out of rangeerrorが出たらググれとあるので、ググってみます。
そのものずばりの回答もありますが、今回はよく使うStackOverflowのサイトのものを貼っておきます。
この程度のエラーでも、躓くことはありそうです。
IndexError: string index out of range in Python [closed]
回答は以下のとおりAs other people have indicated, s[s.Length] isn't actually a valid index; indices are in the closed interval [0, length - 1] (i.e. the last valid index is length - 1 and the first index is 0). Note that this isn't true for every language (there are languages where the first index is 1), but it's certainly true for Python.Google翻訳してみると以下のとおりです。
他の人が示したように、s [s.Length]は実際には有効なインデックスではありません。インデックスは閉じた間隔[0、length-1]にあります(つまり、最後の有効なインデックスはlength-1で、最初のインデックスは0です)。これはすべての言語に当てはまるわけではないことに注意してください(最初のインデックスが1である言語があります)が、Pythonには確かに当てはまります。1-2-2-2 演算
data=1 print(data) data = data+10 print(data)結果
1 11ここで大切なお話は、「変数名は大事」というメッセージが大切
以下で確認しておくといいと思います。
Python命名規則一覧
コーディング規約は、一度読んでおくといいと思います。
pythonの標準的なコーディング規約(参考URLの[B-4];p413)
PEP: 8 Python の標準ライブラリに含まれているPythonコードのコーディング規約
PEP 命名規約1-2-2-3 予約語
予約語と組み込み関数名は、変数名として使えない。
Python3.7予約語またはkeyword一覧と組み込み関数取得・確認方法1-2-3 リストと辞書型
data_list = [1,2,3,4,5,6,7,8,9] print(data_list) print(type(data_list)) print(data_list[1]) print(len(data_list))結果
[1, 2, 3, 4, 5, 6, 7, 8, 9] <class 'list'> 2 9listは、以下のような変更ができる。
data_list = [1,2,3,4,5,6,7,8,9] print(data_list*2) data_list.append(4) print(data_list) data_list.remove(5) print(data_list) print(data_list.pop(6)) print(data_list) del data_list[3:6] print(data_list)結果
[1, 2, 3, 4, 5, 6, 7, 8, 9, 1, 2, 3, 4, 5, 6, 7, 8, 9] [1, 2, 3, 4, 5, 6, 7, 8, 9, 4] [1, 2, 3, 4, 6, 7, 8, 9, 4] 8 [1, 2, 3, 4, 6, 7, 9, 4] [1, 2, 3, 9, 4]【参考】
Pythonでリスト(配列)の要素を削除するclear, pop, remove, deltupleは、listに似ているが変更できない。
data_tuple = (1,2,3,4,5,6,7,8,9) print(data_tuple) print(type(data_tuple)) print(data_tuple[1]) print(len(data_tuple))結果
(1, 2, 3, 4, 5, 6, 7, 8, 9) <class 'tuple'> 2 9setは集合であり、要素の重複を許さない
data_set = {1,2,3,4,5,6,7,8,9} print(data_set) print(type(data_set)) #print(data_set[1]) print(len(data_set))結果
コメントアウトの行
TypeError: 'set' object does not support indexing
setはインデックスをサポートしていない{1, 2, 3, 4, 5, 6, 7, 8, 9} <class 'set'> 9class 'list' 'tuple''set'の使い方
listは、自由度高く、要素を追加削除できる。
tupleは、自由度無く、変更できない
setは、重複しない要素(同じ値ではない要素、ユニークな要素)のコレクションで、和集合、積集合、差集合などの集合演算を行うことができる。
要素の重複を許さないので、以下のような変更でユニークな要素に出来る。
組み合わせると、以下のようなことが出来る。data_list = [1,2,3,4,5,6,7,2,3,4,8,9] print(data_list) data_tuple = tuple(data_list) print(data_tuple) data_set = set(data_list) print(data_set) data_list_new = list(data_set) print(data_list_new) data_tuple_new = tuple(data_set) print(data_tuple_new) data_set.discard(6) print(data_set) data_set.add(11) print(data_set)結果
[1, 2, 3, 4, 5, 6, 7, 2, 3, 4, 8, 9] (1, 2, 3, 4, 5, 6, 7, 2, 3, 4, 8, 9) {1, 2, 3, 4, 5, 6, 7, 8, 9} [1, 2, 3, 4, 5, 6, 7, 8, 9] (1, 2, 3, 4, 5, 6, 7, 8, 9) {1, 2, 3, 4, 5, 7, 8, 9} {1, 2, 3, 4, 5, 7, 8, 9, 11}【参考】
Pythonのsetを知る
Python, set型で集合演算(和集合、積集合や部分集合の判定など)辞書型
dict_data = {'apple':100,'banana':100,'orange':300,'mango':400,'melon':500} print(dict_data['apple']) print(dict_data['apple']+dict_data['orange'])結果
100 400辞書型追加
dict_data['りんご'] = 100 print(dict_data)結果
{'apple': 100, 'banana': 100, 'orange': 300, 'mango': 400, 'melon': 500, 'りんご': 100}辞書型削除
removed_value = dict_data.pop('banana') print(removed_value) print(dict_data)結果
100 {'apple': 100, 'orange': 300, 'mango': 400, 'melon': 500, 'りんご': 100}もう一つの削除
del dict_data['mango'] print(dict_data)結果
{'apple': 100, 'orange': 300, 'melon': 500, 'りんご': 100}辞書同士を連結(結合)、複数の要素を追加・更新: update()
連結(結合)
d1 = {'apple': 100, 'banana': 100} d2 = {'orange': 300, 'mango': 400} d3 = {'melon': 500, 'りんご': 100} d1.update(d2) print(d1) d1.update(**d2, **d3) print(d1)結果
{'apple': 100, 'banana': 100, 'orange': 300, 'mango': 400} {'apple': 100, 'banana': 100, 'orange': 300, 'mango': 400, 'melon': 500, 'りんご': 100}複数の要素を追加・更新
d={'apple': 100, 'banana': 100} print(d) d.update([('orange', 300), ('mango', 400), ('melon', 500), ('りんご', 100)]) print(d)結果
{'apple': 100, 'banana': 100} {'apple': 100, 'banana': 100, 'orange': 300, 'mango': 400, 'melon': 500, 'りんご': 100}【参考】
Pythonで辞書に要素を追加、辞書同士を連結(結合)まとめ
・変数
・演算
・予約語と組み込み関数
・リストと集合、そして辞書型
を見た基本に立ち返って、並べてみると分かり易い。
- 投稿日:2020-08-08T20:07:17+09:00
Optunaを使ったxgboostの設定方法
Optunaを使ったxgboostの設定方法
xgboostの回帰について設定してみる。
xgboostについては、他のHPを参考にしましょう。
「ザックリとした『Xgboostとは』& 主要なパラメータについてのメモ」
https://qiita.com/2357gi/items/913af8b813b069617aad後、公式HPのパラメーターのところを参考にしました。
https://xgboost.readthedocs.io/en/latest/parameter.htmlいろいろ入れたけど、決定木系は過学習になりやすいので、それを制御するパラメーターをしっかり設定した方が良いと思ってます。
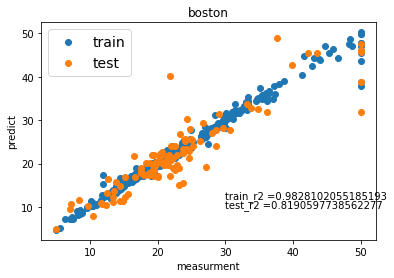
xgboostでは、lambdaとalphaですが、pythonで設定するときは、reg_lambdaとreg_alphaのように、reg_をつけて指定します。# optunaの目的関数を設定する #gtreeのパラメーター設定です。 def objective(trial): eta = trial.suggest_loguniform('eta', 1e-8, 1.0) gamma = trial.suggest_loguniform('gamma', 1e-8, 1.0) max_depth = trial.suggest_int('max_depth', 1, 20) min_child_weight = trial.suggest_loguniform('min_child_weight', 1e-8, 1.0) max_delta_step = trial.suggest_loguniform('max_delta_step', 1e-8, 1.0) subsample = trial.suggest_uniform('subsample', 0.0, 1.0) reg_lambda = trial.suggest_uniform('reg_lambda', 0.0, 1000.0) reg_alpha = trial.suggest_uniform('reg_alpha', 0.0, 1000.0) regr =xgb.XGBRegressor(eta = eta, gamma = gamma, max_depth = max_depth, min_child_weight = min_child_weight, max_delta_step = max_delta_step, subsample = subsample,reg_lambda = reg_lambda,reg_alpha = reg_alpha) score = cross_val_score(regr, X_train, y_train, cv=5, scoring="r2") r2_mean = score.mean() print(r2_mean) return r2_mean# optunaで最適値を見つける study = optuna.create_study(direction='maximize') study.optimize(objective, n_trials=500) # チューニングしたハイパーパラメーターをフィット optimised_rf = xgb.XGBRegressor(eta = study.best_params['eta'],gamma = study.best_params['gamma'], max_depth = study.best_params['max_depth'],min_child_weight = study.best_params['min_child_weight'], max_delta_step = study.best_params['max_delta_step'],subsample = study.best_params['subsample'], reg_lambda = study.best_params['reg_lambda'],reg_alpha = study.best_params['reg_alpha']) optimised_rf.fit(X_train ,y_train)ボストンの結果です。
全部です。
# -*- coding: utf-8 -*- from sklearn import datasets import xgboost as xgb from sklearn.model_selection import train_test_split from sklearn.metrics import r2_score import pandas as pd import optuna import numpy as np import matplotlib.pyplot as plt from sklearn.model_selection import cross_val_score #ボストンのデータセットを読み込む boston = datasets.load_boston() #print(boston['feature_names']) #特徴量と目的変数をわける X = boston['data'] y = boston['target'] X_train, X_test, y_train, y_test = train_test_split(X, y, train_size=0.8) # optunaの目的関数を設定する #gtreeのパラメーター設定です。 def objective(trial): eta = trial.suggest_loguniform('eta', 1e-8, 1.0) gamma = trial.suggest_loguniform('gamma', 1e-8, 1.0) max_depth = trial.suggest_int('max_depth', 1, 20) min_child_weight = trial.suggest_loguniform('min_child_weight', 1e-8, 1.0) max_delta_step = trial.suggest_loguniform('max_delta_step', 1e-8, 1.0) subsample = trial.suggest_uniform('subsample', 0.0, 1.0) reg_lambda = trial.suggest_uniform('reg_lambda', 0.0, 1000.0) reg_alpha = trial.suggest_uniform('reg_alpha', 0.0, 1000.0) regr =xgb.XGBRegressor(eta = eta, gamma = gamma, max_depth = max_depth, min_child_weight = min_child_weight, max_delta_step = max_delta_step, subsample = subsample,reg_lambda = reg_lambda,reg_alpha = reg_alpha) score = cross_val_score(regr, X_train, y_train, cv=5, scoring="r2") r2_mean = score.mean() print(r2_mean) return r2_mean # optunaで最適値を見つける study = optuna.create_study(direction='maximize') study.optimize(objective, n_trials=500) # チューニングしたハイパーパラメーターをフィット optimised_rf = xgb.XGBRegressor(eta = study.best_params['eta'],gamma = study.best_params['gamma'], max_depth = study.best_params['max_depth'],min_child_weight = study.best_params['min_child_weight'], max_delta_step = study.best_params['max_delta_step'],subsample = study.best_params['subsample'], reg_lambda = study.best_params['reg_lambda'],reg_alpha = study.best_params['reg_alpha']) optimised_rf.fit(X_train ,y_train) #結果の表示 print("訓練データにフィット") print("訓練データの精度 =", optimised_rf.score(X_train, y_train)) pre_train = optimised_rf.predict(X_train) print("テストデータにフィット") print("テストデータの精度 =", optimised_rf.score(X_test, y_test)) pre_test = optimised_rf.predict(X_test) #グラフの表示 plt.scatter(y_train, pre_train, marker='o', cmap = "Blue", label="train") plt.scatter(y_test ,pre_test, marker='o', cmap= "Red", label="test") plt.title('boston') plt.xlabel('measurment') plt.ylabel('predict') #ここでテキストは微調整する x = 30 y1 = 12 y2 = 10 s1 = "train_r2 =" + str(optimised_rf.score(X_train, y_train)) s2 = "test_r2 =" + str(optimised_rf.score(X_test, y_test)) plt.text(x, y1, s1) plt.text(x, y2, s2) plt.legend(loc="upper left", fontsize=14) plt.show()
- 投稿日:2020-08-08T19:41:03+09:00
【Python】写真の位置情報のタグ(JPGファイルのGPS Exif)の読み書き
はじめに
これも自分用のメモです。python を使うと楽にできます。注意点として、
- 緯度経度は度分秒なので60進数
- 値は実数ではなく、分数で。(有理数、というのかな)
いくつか関係するライブラリがあります。どれか一つに統一したほうがよいのですが、とりあえず動いたものメモです。(おい)
仕様書?
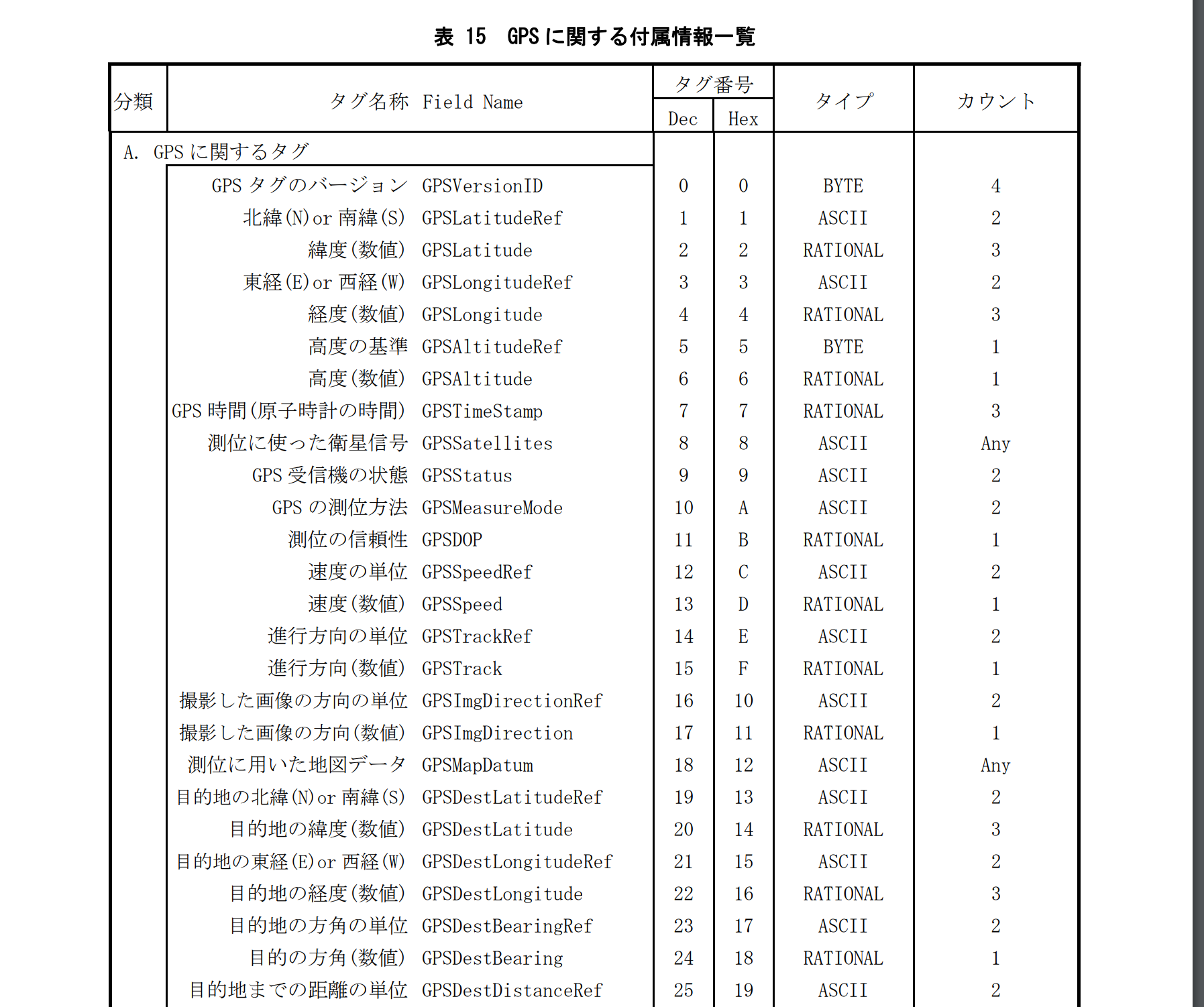
下記の文献を見つけました。
これによるとGPS Info IFDは以下のように定義されているようです。
たくさんありますが、私が扱ったファイルでは0 - 6 までのタグのみでした。GPSの緯度、経度、高さが読み書きできたので、とりあえずは十分でした。ここでGPSLatitudeRefは一文字の'N'か'S'で北緯、南緯を表します。GPSLatitudeは分数で表します。
python で読み書きする
Pillow で緯度経度を読む
Pillow を使用しました。準備で苦労したかは覚えていません。
pip3 install pillow下記で緯度経度を読めました。
read_gpsexif.pyfrom PIL import Image infile = "test.JPG" img = Image.open( infile ) exif = img._getexif() for k, v in exif.items(): if k==34853: print(v)これで
{0: b'\x02\x00\x00\x00', 1: 'N', 2: (35.0, 20.0, 53.51123), 3: 'E', 4: (137.0, 9.0, 17.83123), 5: b'\x00', 6: 256.123}のような結果を得ます。先の仕様書のGPS INfo IFD の定義と見比べると意味が分かると思います。度分秒なので、10進法にするには変換が必要です。例えば
deg,minu,sec = 35.0, 20.0, 53.51366 deg + minu/60.0 + sec/3600.0で
35.34819823888889を得ます。piexif でGPS Exif の緯度経度を書き換える
piexifを利用しました。
度分秒と分数への変換を関数にし、Gps exif を読んで書き換える作戦です。def _to_deg(value, loc): if value < 0: loc_value = loc[0] elif value > 0: loc_value = loc[1] else: loc_value = "" abs_value = abs(value) deg = int(abs_value) t1 = (abs_value-deg)*60 minu = int(t1) sec = round((t1 - minu)* 60, 5) return (deg, min, sec, loc_value) import Fraction def _change_to_rational(number): f = Fraction(str(number)) return (f.numerator, f.denominator)を用いて、以下を実行します。
write_gpsexif.pyimport piexif lat, lng, altitude = 39.123, 139.123, 50.123 jpg_filepath = "test.JPG" lat_deg = to_deg(lat, ["S", "N"]) lng_deg = to_deg(lng, ["W", "E"]) exif_lat = (change_to_rational(lat_deg[0]), change_to_rational(lat_deg[1]), change_to_rational(lat_deg[2])) exif_lng = (change_to_rational(lng_deg[0]), change_to_rational(lng_deg[1]), change_to_rational(lng_deg[2])) gps_ifd = { piexif.GPSIFD.GPSVersionID: (2, 0, 0, 0), piexif.GPSIFD.GPSAltitudeRef: 0, piexif.GPSIFD.GPSAltitude: change_to_rational(round(altitude, 3)), piexif.GPSIFD.GPSLatitudeRef: lat_deg[3], piexif.GPSIFD.GPSLatitude: exif_lat, piexif.GPSIFD.GPSLongitudeRef: lng_deg[3], piexif.GPSIFD.GPSLongitude: exif_lng, } gps_exif = {"GPS": gps_ifd} exif_data = piexif.load(jpg_filepath) exif_data.update(gps_exif) exif_bytes = piexif.dump(exif_data) piexif.insert(exif_bytes, file_name)これは確かStackOverflow を参考にさせていただいたと思うのですが、linkを無くしてしまいました。見つけたら追加します。
まとめ・雑感
必要にせまらて写真のGPSExif の読み書きをした時のメモです。
- Pillow, piexif を利用したが、何となくどちらかに統一して正しく使うのがよさそう。
えいやの作業メモなので、拡充させていきたい、、、という投稿でした。最近雑だな。。^^; (2020/08/08)
- 投稿日:2020-08-08T18:58:46+09:00
【Python】pandas でdtypeを変更
- 投稿日:2020-08-08T18:38:08+09:00
PythonでYahooにメール送信できなかった時の対応。
環境
- Windows 10
- Python 3.8
プログラム
↓を参考にさせていただきました。
https://cercopes-z.com/Python/tips-mail-py.html#yahooしかし、なぜか↓のエラーが出た。
smtplib.SMTPServerDisconnected: Connection unexpectedly closedYahooメール側で外部からのメールを受信できる設定にする必要があった。
https://knowledge.support.yahoo-net.jp/PccMail/s/article/H000007321
- 投稿日:2020-08-08T18:25:19+09:00
【Python】多次元リストのコピー
Pythonの多次元リストのコピーについて、その挙動が自分にとっては理解しづらいので実験結果とともに整理。
結論から言うと、copy()、リスト[:]、copyライブラリのcopy.copy()は浅いコピー(shallow copy)であり、新たな多次元リストを作成し、その後元のリスト中に見つかったリストに対する参照を挿入する。
copyライブラリのcopy.deepcopy()は深いコピー(deep copy)であり、新たな多次元リストを作成し、その後元のリスト中に見つかったリストのコピーを挿入する。
copy()「1層目」というのがいいのか、「1次元目」というのがいいのかよくわからないが…。コピーによって別の多次元リストができるので、IDは当然別のものになる。しかしそのリストの中身は参照なので、コピーの1層目を書き換えてもオリジナルのリストにその影響はないが、2層目以降の書き換えはIDが違うのにもかかわらずオリジナルにも波及する。
copy()lst = [[0]*3 for _ in range(3)] lst1 = lst.copy() lst1[1] = ['r'] * 3 print('lst1 = ', lst1) print('id(lst1) = %d' % id(lst1)) print('lst = ', lst) print('id(lst) = %d' % id(lst)) lst1[0][1] = 'r' print('lst1 = ', lst1) print('id(lst1) = %d' % id(lst1)) print('lst = ', lst) print('id(lst) = %d' % id(lst))出力lst1 = [[0, 0, 0], ['r', 'r', 'r'], [0, 0, 0]] id(lst1) = 139730947400512 lst = [[0, 0, 0], [0, 0, 0], [0, 0, 0]] id(lst) = 139730946890944 lst1 = [[0, 'r', 0], ['r', 'r', 'r'], [0, 0, 0]] id(lst1) = 139730947400512 lst = [[0, 'r', 0], [0, 0, 0], [0, 0, 0]] id(lst) = 139730946890944
リスト[:]
copy()と同様。リスト[lst = [[0]*3 for _ in range(3)] lst1 = lst[:] lst1[1] = ['r'] * 3 print('lst1 = ', lst1) print('id(lst1) = %d' % id(lst1)) print('lst = ', lst) print('id(lst) = %d' % id(lst)) lst1[0][1] = 'r' print('lst1 = ', lst1) print('id(lst1) = %d' % id(lst1)) print('lst = ', lst) print('id(lst) = %d' % id(lst))出力lst1 = [[0, 0, 0], ['r', 'r', 'r'], [0, 0, 0]] id(lst1) = 140313735131456 lst = [[0, 0, 0], [0, 0, 0], [0, 0, 0]] id(lst) = 140313734622336 lst1 = [[0, 'r', 0], ['r', 'r', 'r'], [0, 0, 0]] id(lst1) = 140313735131456 lst = [[0, 'r', 0], [0, 0, 0], [0, 0, 0]] id(lst) = 140313734622336copyライブラリ
copy.copy()
copy()と同様。copy.copy()import copy lst = [[0]*3 for _ in range(3)] lst1 = copy.copy(lst) lst1[1] = ['r'] * 3 print('lst1 = ', lst1) print('id(lst1) = %d' % id(lst1)) print('lst = ', lst) print('id(lst) = %d' % id(lst)) lst1[0][1] = 'r' print('lst1 = ', lst1) print('id(lst1) = %d' % id(lst1)) print('lst = ', lst) print('id(lst) = %d' % id(lst))出力lst1 = [[0, 0, 0], ['r', 'r', 'r'], [0, 0, 0]] id(lst1) = 139783910001664 lst = [[0, 0, 0], [0, 0, 0], [0, 0, 0]] id(lst) = 139783910057280 lst1 = [[0, 'r', 0], ['r', 'r', 'r'], [0, 0, 0]] id(lst1) = 139783910001664 lst = [[0, 'r', 0], [0, 0, 0], [0, 0, 0]] id(lst) = 139783910057280copyライブラリ
copy.deepcopy()コピーによって別の多次元リストができるのでIDは別のものになり、そのリストの中身もコピーされる。いわば、クローンが作られる。全く同じ内容だが別のものなので、それを書き換えてもオリジナルには影響がない。
copy.deepcopy()import copy lst = [[0]*3 for _ in range(3)] lst1 = copy.deepcopy(lst) lst1[1] = ['r'] * 3 print('lst1 = ', lst1) print('id(lst1) = %d' % id(lst1)) print('lst = ', lst) print('id(lst) = %d' % id(lst)) lst1[0][1] = 'r' print('lst1 = ', lst1) print('id(lst1) = %d' % id(lst1)) print('lst = ', lst) print('id(lst) = %d' % id(lst))出力lst1 = [[0, 0, 0], ['r', 'r', 'r'], [0, 0, 0]] id(lst1) = 139629482679488 lst = [[0, 0, 0], [0, 0, 0], [0, 0, 0]] id(lst) = 139629482735040 lst1 = [[0, 'r', 0], ['r', 'r', 'r'], [0, 0, 0]] id(lst1) = 139629482679488 lst = [[0, 0, 0], [0, 0, 0], [0, 0, 0]] id(lst) = 139629482735040参考
Python - リストを複製する
copy --- 浅いコピーおよび深いコピー操作
【Python】 自分で作った関数の引数にリストを代入すると、そのオリジナルのリストが改変されてしまうのを防ぐ方法
- 投稿日:2020-08-08T18:24:28+09:00
【python】WGS84から平面直角座標系への変換
はじめに
測量では成果を緯度経度でなく、平面直角座標系という2次元の平面の座標値で表現します。その相互変換を行う方法のメモです。
日本では現在、19の直角座標系が用いられています。(「分かりやすい平面直角座標系」from 国土地理院)
よくある(?)誤解: 慣性航法で使う座標系とは違う!
座標系の変換というと簡単に聞こえますが、これは
- WGS84の楕円体の極座標(緯度,経度,楕円体高)を地球中心の座標系(ECEF)の直交座標系の座標値XYZに変換
- ECEFの直交座標系の値を、該当する楕円体における接平面に対応するように回転させる
という話とは『異なり』ます。ガウス・クリューゲル投影法という計算方法を使うようなのですが、私は数式が複雑で着いていけませんでした。
当初、自分は上記のような単純な回転計算と誤解していて、周りに「そんなの簡単な計算だよ」などと話していて、思い返すと恥ずかしいです。
国土地理院のサポート
変換するために、国土地理院から提供されているものを利用できます。(さすが公共機関!)
まず、ブラウザに直接座標値を入力して変換することができます。
https://vldb.gsi.go.jp/sokuchi/surveycalc/surveycalc/bl2xyf.html
また、WEB APIからも利用できます。こちらに、提供されている機能と説明があります。アクセスが集中しないように同一アドレスから10秒間で10回の制限がある等の注意も書かれています。
https://vldb.gsi.go.jp/sokuchi/surveycalc/main.html
平面直角座標系の番号を指定して取り出す例です。
use_gsi_webapi.pyimport requests from numpy import round lat, lon = 35.71, 139.74 url = "http://vldb.gsi.go.jp/sokuchi/surveycalc/surveycalc/bl2xy.pl?" params={'latitude':round(lat, 8), 'longitude': round(lon, 8), "refFrame":2, "zone": 9, 'outputType':'json'} res = requests.get(url, params=params) if res.status_code == requests.codes.ok: print(res.json())ここでは次の辞書が返されます。
{'OutputData': { 'publicX': '-32170.0990', 'publicY': '-8445.1361', 'gridConv': '0.054477778', 'scaleFactor': '0.99990088'}}EPSG
日本の平面直角座標系の番号は日本が定めるものですが、国際的にはEPSGとして定めらています。国際的には平面直角座標系の番号ではなくEPSGで参照します。(例えば写真測量のソフトであるmetashape などを使う場合とか。)
EPSG 測地系 EPSG:4326 WGS 84 EPSG:6668 JGD2011 EPSG:6669 JGD2011 / Japan Plane Rectangular CS I EPSG:6670 JGD2011 / Japan Plane Rectangular CS II EPSG:6671 JGD2011 / Japan Plane Rectangular CS III EPSG:6672 JGD2011 / Japan Plane Rectangular CS IV EPSG:6673 JGD2011 / Japan Plane Rectangular CS V EPSG:6674 JGD2011 / Japan Plane Rectangular CS VI EPSG:6675 JGD2011 / Japan Plane Rectangular CS VII EPSG:6676 JGD2011 / Japan Plane Rectangular CS VIII EPSG:6677 JGD2011 / Japan Plane Rectangular CS IX EPSG:6678 JGD2011 / Japan Plane Rectangular CS X EPSG:6679 JGD2011 / Japan Plane Rectangular CS XI EPSG:6680 JGD2011 / Japan Plane Rectangular CS XII EPSG:6681 JGD2011 / Japan Plane Rectangular CS XIII EPSG:6682 JGD2011 / Japan Plane Rectangular CS XIV EPSG:6683 JGD2011 / Japan Plane Rectangular CS XV EPSG:6684 JGD2011 / Japan Plane Rectangular CS XVI EPSG:6685 JGD2011 / Japan Plane Rectangular CS XVII EPSG:6686 JGD2011 / Japan Plane Rectangular CS XVIII EPSG:6687 JGD2011 / Japan Plane Rectangular CS XIX 情報源ですが、本家本元や管理団体などよく理解していません。以下がわりとauthority のような気がします。
- epsg.io で説明を読むことができます。例えばWGS84とか。
- EPSG Geodetic Parameter Registryで検索できます
- spatial reference というページにもリストや検索機能があります。
pyproj を使う
さきほどはWEB API をりようしましたが、これはpython ライブラリを用いてもできます。
インストールで苦労した記憶はありません。test_webapi.pyfrom pyproj import Transformer lat, lon = 35.71, 139.74 wgs84_epsg, rect_epsg = 4326, 6677 tr = Transformer.from_proj(wgs84_epsg, rect_epsg) x, y = tr.transform(lat, lon) print(x, y)実行すると以下を得ます。
-32170.09900022997 -8445.136058616452有効数字は分かりませんが、地理院の結果とは一致しています。(小数点以下4桁、0.1ミリメートル!)
CRSの利用
巷では、CRSを使う方法が紹介されていましたが、利用してみると、、、
test_use_pyproj.pyimport pyproj wgs84=pyproj.CRS("EPSG:4326") jgd2011_9 = pyproj.CRS("EPSG:6677") lat, lon = 35.71, 139.74 print( pyproj.transform(wgs84, jgd2011_9, lat, lon) )実行すると警告が出ますが、同じ結果を得ます。
/usr/bin/ipython3:1: DeprecationWarning: This function is deprecated. See: https://pyproj4.github.io/pyproj/stable/gotchas.html#upgrading-to-pyproj-2-from-pyproj-1 (-32170.09900022997, -8445.136058616452)これは多分、古い方法なのだと思います。しっかりチュートリアルを読まなければ!
https://pyproj4.github.io/pyproj/stable/examples.htmlまとめ・雑感
- WGS84と平面直角座標系の変換は地理院提供のAPIでできるが、pyproj でも同じことはできそう
- pyproj は多機能で4Dtansform など気になる機能もあり、余力があれば調べてみたい
等々(2020/08/08)
- 投稿日:2020-08-08T17:49:42+09:00
Djangoでパスワード変更すると、勝手にログアウトされる
Django==2.2.12でのお話です。
ログインした状態で、自作の画面からユーザパスワードを変更したあと・別のページに遷移しようとすると、まずログイン画面に飛んでしまう
・普段なら問題のないサーバーへのリクエストがエラーを返すといった状況を不思議に思ったアナタ!
実はカスタムのパスワード変更画面を利用している場合、パスワード変更後に強制的にログアウト状態になってしまうんですね。
それまで使用していたセッションが使われなくなるのが原因です。Djangoが用意している
PasswordChangeViewなんかを使用している場合は、パスワード変更後に自動で新しいセッションを作成してくれるので、ログイン状態が続いてくれます。カスタム画面でもそうしたい!という場合は
update_session_auth_hash()関数を使うといいよ。といったことが、ドキュメント
https://docs.djangoproject.com/en/3.0/topics/auth/default/#session-invalidation-on-password-change
に書かれていました...ついでに、このドキュメントに書かれている使用例も貼っておきます。
from django.contrib.auth import update_session_auth_hash def password_change(request): if request.method == 'POST': form = PasswordChangeForm(user=request.user, data=request.POST) if form.is_valid(): form.save() update_session_auth_hash(request, form.user) else: ...
- 投稿日:2020-08-08T17:09:55+09:00
Dockerで作る、環境整備 (PythonでのGrADSの後処理をしたい
何をしたいのか?
- PythonでGrADS用のファイルを読み込んで後処理をしたい
- しかし共有計算資源の環境はクリーンでなければならない
- だがしかし、変換ソフト、ライブラリなどなどインストールしたいものがいっぱいある
ということで、コンテナ仮想化を使おうということです。
Docker
Dockerを使うと何がいいかというと、計算資源を汚すことなく、必要な後処理を行う専用の仮想計算機を作り上げることができます。使うときにコンテナを立ち上げ、処理を行った後は破棄してまた必要になったら立ち上げることができます。
Dockerの場合は立ち上げのコストが非常に低いので、気軽に立ち上げて一つの処理をした後また破棄することが気軽にできます。環境整備手順
Dockerfileを書くと自動化できていいのですが、ここでは単にシェルを立ち上げて整備して、それを単純にコミットしてイメージを作ることにします。
長期的に使いたいなら、バージョンアップも可能になるようにDockerfileを作るべきですが、最初の基本の機能の確認はシェルで行ったほうが理解が深まります。今回はUbuntuをもとに、cdoとpip3, netcdf4をインストールします。
1. もとになるイメージをpull(拾ってくる)して走らせる
今回はUbuntuを使います。
$ docker pull ubuntu/ubuntu$ docker imagesで、イメージが作れたことが確認できます。
イメージを走らせるためには、
$ docker run -it ubuntu/ubuntuとします。
2. 必要なソフトウェアのインストール
pip3とcdoをインストールします。
pip3でnetCDF4もインストールします。# apt update # apt install python3 python3-pip cdo # pip3 install netCDF43. 動作確認
きちんとcdoで変換できてnetCDF4もインポートできるか確認します。
# python3 >>> import netCDF4 as nc >>> ^D # cdo ....4. コミットしてimageを作成する
exitコマンドかCtrl+dで抜けて、イメージを作りましょう。
イメージを作るために必要なコンテナIDは、終了したコンテナを確認するために-aが必要です。# exit (host) $ docker ps -aこのような画面になると思います。
CONTAINER IDはコミットの時に指定するコンテナIDになります。
IMAGEのところにimageの名前があって、STATUSのところでexited ** agoとなっているので、大体どれかはそこを見ればわかると思います。
$ docker commit [container id] [author]/[image名]というようにコミットしてあげましょう。
$ docker imagesで、イメージが作れたことが確認できます。
5. 実際に計算させてみる
実際に計算させるとき、2通りあって、runしてからインタラクティブにシェルから実行することもできます。ただ、今回はrunの際にコマンドを指定して実行させてみます。
プログラム、データ群のディレクトリをDockerへとバインドすることで、実際にコンテナから容易にアクセスることができます。また、グラフの確認にXフォワーディングをします。
$ sudo docker run -e DISPLAY=$DISPLAY --net host -v /tmp/.X11-unix:/tmp/.X11-unix -v $HOME/.Xauthority:/root/.Xauthority -v /home/xxx/:/home/ --shm-size 16g xxx/ubuntu-cdo2 /bin/bash -c "cd /home/speedy-epyc/speedy/python-script; python3 rmse.py"
--shm-sizeは、足りなくなるプログラムがあれば必要に応じてつけてください。
- 投稿日:2020-08-08T16:58:16+09:00
初めてのFlask
Flaskモジュールのインストール
(base) root@e8cf64ce12e9:/home/continuumio#pip install flaskフォルダ構造の作成
Flaskで使用するHtmlファイルは、プロジェクトフォルダに「templates」という名称のフォルダを作成して格納する必要がある。
注:テンプレートという名前だけど、要はFlaskで利用するHtmlファイルはすべてここに格納すれば良い。. ├── __pycache__ ├── hello.py └── templates ├── hello.html └── layout.html余談:上記のようなディレクトリ構造を取得するためには、「tree」コマンドを別途インストールする必要がある。
Docker環境がUbuntuだったので「apt install tree」でインストールできた。Macローカルの場合のインストールは別途作業が必要みたい。各ファイルの役割は下記の通り
- hello.py: アプリケーションのルーティングファイル。
- template/hello.html: 画面の一部を構成するhtmlファイル。とどのつまり画面を構成するパーツ。
- templates/layout.html: すべてのHtmlファイルのベースとしてのhtmlファイル。このファイルの部分部分にhello.htmlのようなパーツとしてのhtmlフィアルが組み込まれてくイメージ。各ファイルの内容
layout.html
layout.html<!DOCTYPE html> <html> <head> <titile>{{ title }}</titile> </head> <body> {% block content %} <!-- main--> {% endblock %} </body> </html>
- {{ val }}: val部分には同名の変数の値が反映される。
- {% block content%}~{% endblock %}:このタグ範囲に拡張パーツとしてのhtmlファイルに記載されている{% block content%}~{% endblock %}のスクリプトが差し込まれるイメージ
hello.html
hello.html<!-- layout.htmlをテンプレートに拡張する--> {% extends "layout.html" %} <!-- block content ~ endblock-の範囲がテンプレートの同宣言範囲に差し込まれる --> {% block content %} <h3>Hello</h3> こんにちは。{{ name }}さん。 {% endblock %}
- extends で指定したhtmlにこのhtml内のblock contentの内容を反映する。継承のイメージ。
hello.py
hello.pyfrom flask import Flask, render_template app = Flask(__name__) @app.route('/') def hello(): name = 'TEST' # return name return render_template('hello.html', tite="flask test", name=name) if __name__ == "__main__": app.run(host='0.0.0.0', port=5000, debug=True)
- 「render_template」をimportすることでJinja2テンプレートの機能が利用できるようになる。
- render_template([呼び出すhtml],[[テンプレートに渡す変数名=値],...]
- 「if name == "main":」 コマンドラインからpythonファイルを実行する際のおまじないという認識でOK。
- app_run([host=0,0,0,0],[port=XXXX],[debug=XXXX])でアプリケーションを起動する。 hostとかポートはここで指定しなくても動かす方法がありそうだが、おいおい調査する。
実行
(base) root@e8cf64ce12e9:/home/continuumio# python hello.py * Serving Flask app "hello" (lazy loading) * Environment: production WARNING: This is a development server. Do not use it in a production deployment. Use a production WSGI server instead. * Debug mode: on * Running on http://0.0.0.0:5000/ (Press CTRL+C to quit) * Restarting with inotify reloader * Debugger is active! * Debugger PIN: 197-268-519画面が表示できた。
最後に
とりあえず最低限の使い方だけわかったけど、実務でFlask使うことってないだろうし、事業会社はDjangoなんでしょうね。
あくまでも、フレームワークの足がかりとしてなら勉強する価値あるかな。
- 投稿日:2020-08-08T16:51:04+09:00
AWS Dynamo DBへのcsvインポートを手軽に実装(Windows・無料)
はじめに
Tech Dive様の記事 を大変参考にさせて頂きました。この場を借りて御礼申し上げます。
この記事は上記内容をWindows環境で実施した際の備忘録になります。
筆者の環境
・Windows 10
・Git Bash手順
Python3 のインストール
Pandasのインストール
pip install pandasaws cliのインストール
インストールしてもGit Bash でawsコマンドが使えなかったので、GitBashで
cmd \\C aws --version
を叩いてから再起動したら使えるようになりましたアクセスキーの確認
AWS ユーザーメニューの「マイセキュリティ資格情報」から、インポート用のアクセスキーを新規作成アクセスキーとシークレットアクセスキーの登録
aws configureを叩き、先ほど作ったアクセスキーを登録aws configure AWS Access Key ID [None]: [アクセスキー] AWS Secret Access Key [None]: [シークレットアクセスキー] Default region name [None]: us-east-2 ※テーブルがあるリージョン Default output format [None]: jsonCSVのヘッダーを変更
先頭行をデータベースのkeyと合わせて、型を()でくくって指定
例) キーが「UserName」(String)なら UserName (S)import_to_dynamodb をclone
git clone https://github.com/hidesan-xyz/import_to_dynamodb.gitcloneしたディレクトリのルートにインポートするcsvファイルを置く
import用shellコマンドの作成
python create_insert_command.py importdata testtable
※第一引数にcsv名(拡張子は記載しない) 第二引数にテーブル名作成されたシェルコマンドの実行
sh ./testtable_import_20200808164839.sh上記手順で完了です。
- 投稿日:2020-08-08T16:48:14+09:00
【Python】写真のxmpタグ
はじめに
写真のJPGファイルに、撮影日時や場所などを記録することができます。ドローン等で撮影された写真にタグ情報が書かれていることがあります。それらは一般的なフォーマットに従って記録されているようです。それらを読み書きしたいのが目的です。
GPS Exif については多くの記述があります。なので、ここでは情報が少ないと思われる xmp についてメモしておきます。なお、読むことはできるのですが、書くところはうまくできていません。(涙)
準備: xmp-toolkit のインストール
XMPとは
どうやら、Adobe が定義し、各ファイルにXML形式のテキストで情報をファイルのヘッダに加えるために用いていたようです。詳しくは分かりません。^^;)
なので、例えば、less コマンドで見ると、テキストのXMLの部分はそのまま読めます。
おじさんはLinux環境(WSL)ですが、
- less でファイルを開き
- 「binaryだけど開くか」と聞かれるのでYes とし
- xml という文字列を検索する(スラッシュ xml リターン)と、<x:xmpmeta xmlns:x="adobe:ns:meta/"> <rdf:RDF xmlns:rdf="http://www.w3.org/1999/02/22-rdf-syntax-ns#"> <rdf:Description rdf:about="DJI Meta Data" xmlns:tiff="http://ns.adobe.com/tiff/1.0/" xmlns:exif="http://ns.adobe.com/exif/1.0/" xmlns:xmp="http://ns.adobe.com/xap/1.0/" xmlns:xmpMM="http://ns.adobe.com/xap/1.0/mm/" xmlns:dc="http://purl.org/dc/elements/1.1/" xmlns:crs="http://ns.adobe.com/camera-raw-settings/1.0/" xmlns:drone-dji="http://www.dji.com/drone-dji/1.0/" xmp:ModifyDate="2018-12-14" xmp:CreateDate="2018-12-14" tiff:Make="DJI" tiff:Model="FC6310R" dc:format="image/jpg" drone-dji:AbsoluteAltitude="+255.92" ...のように見えます。これらの情報を読み書きしたい、というのが目的です。
python では
便利なツールはもちろん存在します。
python3 -m pip install python-xmp-toolkitAdobe からXMP-Toolkit-SDKがソースごと公開もされています。組み込みやスマホアプリで利用できるのかな。
https://github.com/adobe/XMP-Toolkit-SDKxmp のタグの値を読む
使い方はpython-xmp-toolkit のDocに書かれていて、それをフォローするだけで問題なかったです。メモしておきます。
ファイルを読む
read_xmp01.pyfrom libxmp import XMPFiles xmpfile = XMPFiles( file_path="./100_0020_0001.JPG", open_forupdate=True ) xmp = xmpfile.get_xmp()エラーが出なければ読めています。^^;) いろいろxmp の中身を調べることができます。
値の取得
先のサンプルでは
tiff:Make="DJI"
という行がありました。これを "tiff:Make" というkey に対して value である "DJI"を直接取得したい。そのためには、2ステップ必要です。
namespace の取得
まず、tiff のnamespace (URI)を取得します。
In[]: xmp.get_namespace_for_prefix("tiff") Out[]: 'http://ns.adobe.com/tiff/1.0/'あらかじめadobe が定義されているnamespace もありこれはライブラリからも参照できます。
In []: libxmp.consts.XMP_NS_EXIF Out[]: 'http://ns.adobe.com/exif/1.0/'変数名は直接ソースの "Common Namespaces" をみて知ることもできます。
property を取得する
tiff:Make というタグの値を読むには、さきほどのtiffに対応するnamespace を指定してあげます。
In []: xmp.get_property(xmp.get_namespace_for_prefix('tiff'),'Make') Out[]: 'DJI'namespace内にそのキーワードがあるかを事前に確認することで、エラーを未然に防げます。
In []: xmp.does_property_exist(xmp.get_namespace_for_prefix('tiff'),'TEST') Out[]: False書く
正確には「書き換える」ができる「はず」なのですが、実は。。。
まず、xmp の値をsetします。
In []: xmp.set_property(xmp.get_namespace_for_prefix('tiff'),'Make', 'some/makers') In []: xmp.get_property(xmp.get_namespace_for_prefix('tiff'),'Make') Out[]: 'some/makers'ファイルに書くには、xmpfileにput して file を close する必要があるようです。
In []: if xmpfile.can_put_xmp(xmp): ...: xmpfile.put_xmp(xmp) ...: xmpfile.close_file()で良いはずなのですが。。。結果は正しく書き換えられていませんでした。手順が違うのかバグなのか分かりませんが、とりあえずここまでメモしておきます。実行時にエラーは発生しません。
メモ
とりあえず読むことだけできる、という状態でした。以下自分の課題:
- 各 namespace にあるタグを事前にすべて取得できるか?
- 書き込みの動作確認
以上。(2020/08/08)
- 投稿日:2020-08-08T16:08:47+09:00
ABC173Cによる学び(bit全探索、listのコピー、多次元リストの一元化)
ABC173Cは非常に学びが多く、今後の参考になりそうなので個人的な備忘録代わりに。
ABC173Cimport copy # 多次元リストの値渡し/深いコピー(Deep Copy): copy.deepcopy()のため h, w, k = map(int, input().split()) grd = [list(i for i in input()) for _ in range(h)] ans = 0 for i in range(2**h): # 行の選び方が2**h通りで、そのうちi番目 grd1 = copy.deepcopy(grd) # grdの値渡し/深いコピー(Deep Copy) for i1 in range(h): # 2**h通りは2進数にしたときh桁で、そのうちi1桁目 if i >> i1 & 1: # 「2**h通りのうちi番目」のiを2進数にしたとき、どの桁が1になっているか grd1[i1] = ['r'] * w for j in range(2**w): # 列の選び方が2**w通りで、そのうちj番目 grd2 = copy.deepcopy(grd1) # grd1の値渡し/深いコピー(Deep Copy) for j1 in range(w): # 2**w通りは2進数にしたときw桁で、そのうちj1桁目 if j >> j1 & 1: # 「2**w通りのうちj番目」のjを2進数にしたとき、どの桁が1になっているか for n in range(h): grd2[n][j1] = 'r' if sum(grd2,[]).count('#') == k: # sum()で2次元リストを一次元に平坦化 ans += 1 print(ans)bit全探索
bit全探索に関してはいろいろな記事があるが、概して初心者かつPythonしか使えない人には難しい。以下はそんな人にもわかりやすく、まずbit全探索って何をするのかを掴むのにはちょうどよい。
Python de アルゴリズム(bit全探索)listのコピー
リスト.copy()やリスト[:]を使って、オブジェクトの破壊・非破壊など意識して書いたつもりではあったが、思ったとおりに動いてくれなくて(上のコードではgrdが意図せずに書き換わって)困った。
コピーには参照渡し/浅いコピー(Shallow Copy)と値渡し/深いコピー(Deep Copy)があり、更に値渡し/深いコピー(Deep Copy)でもcopy.copy()は一次元リストと多次元リストで挙動が異なる。
Python - リストを複製する
【Python】多次元リストのコピー多次元リストの一元化
この問題のように多次元のリストを一元的に集計したいようなときに便利。いくつかやり方はあるようだが、
sum()の意外な使い方も。
Pythonでflatten(多次元リストを一次元に平坦化)
- 投稿日:2020-08-08T16:08:47+09:00
ABC173Cによる学び(bit全探索、listのコピー、多次元リストの一次元化)
ABC173Cは非常に学びが多く、今後の参考になりそうなので個人的な備忘録代わりに。
ABC173Cimport copy # 多次元リストの値渡し/深いコピー(Deep Copy): copy.deepcopy()のため h, w, k = map(int, input().split()) grd = [list(i for i in input()) for _ in range(h)] ans = 0 for i in range(2**h): # 行の選び方が2**h通りで、そのうちi番目 grd1 = copy.deepcopy(grd) # grdの値渡し/深いコピー(Deep Copy) for i1 in range(h): # 2**h通りは2進数にしたときh桁で、そのうちi1桁目 if i >> i1 & 1: # 「2**h通りのうちi番目」のiを2進数にしたとき、どの桁が1になっているか grd1[i1] = ['r'] * w for j in range(2**w): # 列の選び方が2**w通りで、そのうちj番目 grd2 = copy.deepcopy(grd1) # grd1の値渡し/深いコピー(Deep Copy) for j1 in range(w): # 2**w通りは2進数にしたときw桁で、そのうちj1桁目 if j >> j1 & 1: # 「2**w通りのうちj番目」のjを2進数にしたとき、どの桁が1になっているか for n in range(h): grd2[n][j1] = 'r' if sum(grd2,[]).count('#') == k: # sum()で2次元リストを一次元に平坦化 ans += 1 print(ans)bit全探索
bit全探索に関してはいろいろな記事があるが、概して初心者かつPythonしか使えない人には難しい。以下はそんな人にもわかりやすく、まずbit全探索って何をするのかを掴むのにはちょうどよい。
Python de アルゴリズム(bit全探索)listのコピー
リスト.copy()やリスト[:]を使って、オブジェクトの破壊・非破壊など意識して書いたつもりではあったが、思ったとおりに動いてくれなくて(上のコードではgrdが意図せずに書き換わって)困った。
コピーには参照渡し/浅いコピー(Shallow Copy)と値渡し/深いコピー(Deep Copy)があり、copyライブラリのcopy.copy()とcopy.deepcopy()では特に多次元リストで挙動が異なる。
Python - リストを複製する
【Python】多次元リストのコピー多次元リストの一次元化
この問題のように多次元のリストを一元的に集計したいようなときに便利。いくつかやり方はあるようだが、
sum()の意外な使い方も。
Pythonでflatten(多次元リストを一次元に平坦化)
- 投稿日:2020-08-08T16:08:47+09:00
ABC173Cによる学び(bit全探索、多次元リストのコピー、多次元リストの一次元化)
ABC173Cは非常に学びが多く、今後の参考になりそうなので個人的な備忘録代わりに。
ABC173Cimport copy # 多次元リストの値渡し/深いコピー(Deep Copy): copy.deepcopy()のため h, w, k = map(int, input().split()) grd = [list(i for i in input()) for _ in range(h)] ans = 0 for i in range(2**h): # 行の選び方が2**h通りで、そのうちi番目 grd1 = copy.deepcopy(grd) # grdの値渡し/深いコピー(Deep Copy) for i1 in range(h): # 2**h通りは2進数にしたときh桁で、そのうちi1桁目 if i >> i1 & 1: # 「2**h通りのうちi番目」のiを2進数にしたとき、どの桁が1になっているか grd1[i1] = ['r'] * w for j in range(2**w): # 列の選び方が2**w通りで、そのうちj番目 grd2 = copy.deepcopy(grd1) # grd1の値渡し/深いコピー(Deep Copy) for j1 in range(w): # 2**w通りは2進数にしたときw桁で、そのうちj1桁目 if j >> j1 & 1: # 「2**w通りのうちj番目」のjを2進数にしたとき、どの桁が1になっているか for n in range(h): grd2[n][j1] = 'r' if sum(grd2,[]).count('#') == k: # sum()で2次元リストを一次元に平坦化 ans += 1 print(ans)bit全探索
bit全探索に関してはいろいろな記事があるが、概して初心者かつPythonしか使えない人には難しい。以下はそんな人にもわかりやすく、まずbit全探索って何をするのかを掴むのにはちょうどよい。
Python de アルゴリズム(bit全探索)多次元リストのコピー
リスト.copy()やリスト[:]を使って、オブジェクトの破壊・非破壊など意識して書いたつもりではあったが、思ったとおりに動いてくれなくて(上のコードではgrdが意図せずに書き換わって)困った。
コピーには参照渡し/浅いコピー(Shallow Copy)と値渡し/深いコピー(Deep Copy)があり、copyライブラリのcopy.copy()とcopy.deepcopy()では特に多次元リストで挙動が異なる。
Python - リストを複製する
【Python】多次元リストのコピー多次元リストの一次元化
この問題のように多次元のリストを一元的に集計したいようなときに便利。いくつかやり方はあるようだが、
sum()の意外な使い方も。
Pythonでflatten(多次元リストを一次元に平坦化)
- 投稿日:2020-08-08T16:07:46+09:00
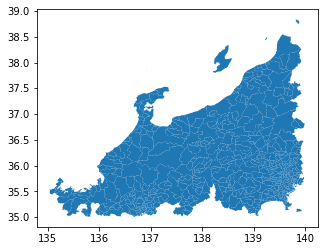
Python環境でGeoSpark(Apache Sedona)をインストールしてみる
0. 経緯
PythonでGeoSpatialなデータを取り扱う際、小〜中規模なものであればGeoPandasをよく使うのですが、大規模なデータを取り扱おうとすると限界があります。
そうすると、軽く調べた感じではPostgreSQLの拡張機能であるPostGISがよく使われている印象で、他にNoSQLだと例えばMongoDBなどでもGeometry型を使えそう(参考)でしたが、いかんせんデータベースを立ち上げてスキーマを用意してテーブルorコレクションを作って、、、というのは結構面倒で、この辺り興味はあるのですがまだ手を出せていません。個人的には大規模なデータ処理にはよくpysparkを使うのでこれを使って何か出来ないか、と探しているとGeoSparkなるものを見つけました。
まだまだ発展途上のライブラリのようですが、見て頂ければ分かる通りで2020-07-19(※記事執筆日時:2020-08-08)にはApache Sedonaの名前でApache Incubatorに登録されています。
(あまり詳しくないのですが、今後うまく軌道に乗れれば正式なApacheプロジェクトになる、ということのようです。また、管理がApache側へ移るので上記のリポジトリ・ドキュメントもそのうち移動されると思います。)大規模なデータを処理出来る可能性を持ちつつ、pandasやgeopandasに比較的近い感覚(スキーマ設計などせず適当な感じでも使える)でデータ処理可能なのはかなり面白そう、ということで少し遊んでみた次第です。
1. 環境構築
※以下、Apache Spark、pyspark自体の初歩的な説明は省略します。
手元の環境:
Linux(Ubuntu20.04)
(省略しますが、Windowsでも大体同じ雰囲気で作れます)Python環境は
pyenv+miniconda3(つまりconda)で用意しましたが、別に何でも良いと思います。1-1. 仮想環境作成
例えば以下のようなYAMLファイルを用意:
create_env.ymlname: geospark_demo channels: - conda-forge - defaults dependencies: - python==3.7.* - pip - jupyterlab # for test - pyspark==2.4.* - openjdk==8.* - pyarrow - pandas - geopandas - folium # for test - matplotlib # for test - descartes # for test - pip: - geospark
folium、matplotlib、descartes、jupyterlabはgeospark的に必須ではないですが、テスト用に可視化する目的で入れていますpysparkとjava8は自前で用意しているなら不要です
- なお、
geospark(1.3.1)が対応するApache Sparkのバージョンは執筆時点(2020年8月)で2.2 - 2.4系までなので、pysparkは2.4系を指定しますこれで
conda env create -f create_env.yml # 作った仮想環境に入る conda activate geospark_demoなどとすると、
geospark_demoという名前のconda仮想環境が作れます。
(パッケージや仮想環境名などの諸々の調整は例えばここなどを参照)
(別にconda使わなくても同等なことは出来ると思います)1-2. 環境変数設定
上記の例(conda仮想環境利用)であれば
PATHの設定や、JAVA_HOMEくらいまでは勝手にやってくれますが、いくつか追加で環境変数を設定する必要があります。まずgeosparkでは内部で
SPARK_HOMEを参照するときがあるので、Apache Sparkのインストール場所を環境変数で設定します。
なお、今回の例のようにcondaなどでApache Sparkを入れている場合、どこにSparkの本体がいるのか分かりづらいときがあるので、例えばここを参考にして# Apache Sparkのインストール場所を確認 echo 'sc.getConf.get("spark.home")' | spark-shell # SPARK_HOME設定 export SPARK_HOME=<上で出てきたパス>みたいにして設定します。筆者は
SPARK_HOME=/home/<ユーザー名>/.pyenv/versions/miniconda3-latest/envs/geospark_demo/lib/python3.7/site-packages/pysparkみたいな感じになりました。また、インストールされている
pyarrowのバージョンが0.15以降の場合、ここに従ってexport ARROW_PRE_0_15_IPC_FORMAT=1を設定しておく必要があります(pyspark2.x系で必要な設定)。
あるいは、pyarrow==0.14.*を指定してインストールしておくようにします。この辺いちいち手でやるのは面倒なので、個人的にはファイルに書いておいて
sourceするようにするか、あるいはDockerでENVなどを使って設定するなどしています。2. 動作確認
公式のGitHub上にpython用のJupyter Notebookと必要なテストデータ(
python/data/に格納)が置いてあるので、それらを使って問題なく動作することを確認します。
例えば、# 作業ディレクトリに移動 cd /path/to/workdir # githubからnotebookをダウンロード wget https://raw.githubusercontent.com/DataSystemsLab/GeoSpark/master/python /GeoSparkCore.ipynb wget https://raw.githubusercontent.com/DataSystemsLab/GeoSpark/master/python/GeoSparkSQL.ipynb # svnを使ってgithubから特定のディレクトリだけダウンロードする svn export https://github.com/DataSystemsLab/GeoSpark/trunk/python/data/のような感じで取得出来ます。
なお、svnを使ったGitHubからのディレクトリのダウンロードはここやここを参考にしました。あとはjupyterlabかjupyter notebookを立ち上げて↑のnotebookを実行します。
これで動作確認をしつつ、どういう雰囲気で使えるものかの参考になると思います。2-1. 少し遊んでみる
↑の動作確認で使ったnotebookや公式ドキュメントのTutorialの方がよっぽどためになるのですが、せっかくなので自分でも少し遊んでみます。
テストデータ
esriジャパンの全国市区町村界データを使います。
リンク先の「ファイルのダウンロード」を押して、「同意する」にチェックをつけるとjapan_ver821.zipの形でshpファイルを入手出来るので、作業ディレクトリ上でunzipして置いておきます。実行
以下のようなことを試してみます:
- shpファイルをGeoPandasで読み込んで、parquetファイル(hiveテーブル)の形で保存
- ↑で作ったparquetファイルを読み込んで、Geometryタイプを含むDataFrameを生成・操作
以下、jupyterlab上で動作確認を行っています。
下準備
# 必要なライブラリのインポート import os import folium import geopandas as gpd from pyspark.sql import SparkSession from geospark.register import GeoSparkRegistrator from geospark.utils import GeoSparkKryoRegistrator, KryoSerializer from geospark.register import upload_jars # sparkセッションの生成 upload_jars() spark = SparkSession.builder.\ master("local[*]").\ appName("TestApp").\ config("spark.serializer", KryoSerializer.getName).\ config("spark.kryo.registrator", GeoSparkKryoRegistrator.getName) .\ getOrCreate() GeoSparkRegistrator.registerAll(spark)テストデータ読み込み
sdf_japan = spark.createDataFrame( # ダウンロード済みのesriジャパンの全国市区町村界データをgeopandasで読み込み gpd.read_file("japan_ver821/japan_ver821.shp") ) # 確認 sdf_japan.show(5) # +-----+------+----------+----+------+----------+--------------------+--------+--------+--------------------+ # |JCODE| KEN| SICHO| GUN|SEIREI|SIKUCHOSON| CITY_ENG| P_NUM| H_NUM| geometry| # +-----+------+----------+----+------+----------+--------------------+--------+--------+--------------------+ # |01101|北海道|石狩振興局|null|札幌市| 中央区|Sapporo-shi, Chuo-ku|235449.0|141734.0|POLYGON ((141.342...| # |01102|北海道|石狩振興局|null|札幌市| 北区|Sapporo-shi, Kita-ku|286112.0|151891.0|POLYGON ((141.408...| # |01103|北海道|石狩振興局|null|札幌市| 東区|Sapporo-shi, Higa...|261777.0|142078.0|POLYGON ((141.446...| # |01104|北海道|石狩振興局|null|札幌市| 白石区|Sapporo-shi, Shir...|212671.0|122062.0|POLYGON ((141.465...| # |01105|北海道|石狩振興局|null|札幌市| 豊平区|Sapporo-shi, Toyo...|222504.0|126579.0|POLYGON ((141.384...| # +-----+------+----------+----+------+----------+--------------------+--------+--------+--------------------+ # only showing top 5 rowsDataFrame保存
# ファイルとして保存(デフォルトではsnappy.parquet形式) sdf_japan.write.save("esri_japan") # hiveテーブル形式で保存(実体ファイルはデフォルトではsnappy.parquet) spark.sql("CREATE DATABASE IF NOT EXISTS geo_test") # 必須でないが、データベース作成 sdf_japan.write.saveAsTable("geo_test.esri_japan") # データベースgeo_test上のテーブルesri_japanとして保存↑saveおよびsaveAsTableのオプションで
formatやcompressionを変えることができ、zlib.orcやjson.gzipなどでも保存出来るようです。(それが嬉しいのかはさておき)読み込み
# ファイル読み込み # 実体ファイルが保存されているディレクトリを指定する。parquet以外の形式で保存した場合はloadのオプションでformatを指定する。 sdf_from_file = spark.read.load("esri_japan") sdf_from_file.show(5) # +-----+------+-----+------+------+----------+--------------+-------+-------+--------------------+ # |JCODE| KEN|SICHO| GUN|SEIREI|SIKUCHOSON| CITY_ENG| P_NUM| H_NUM| geometry| # +-----+------+-----+------+------+----------+--------------+-------+-------+--------------------+ # |32207|島根県| null| null| null| 江津市| Gotsu-shi|23664.0|11513.0|MULTIPOLYGON (((1...| # |32209|島根県| null| null| null| 雲南市| Unnan-shi|38479.0|13786.0|MULTIPOLYGON (((1...| # |32343|島根県| null|仁多郡| null| 奥出雲町| Okuizumo-cho|12694.0| 4782.0|POLYGON ((133.078...| # |32386|島根県| null|飯石郡| null| 飯南町| Iinan-cho| 4898.0| 2072.0|POLYGON ((132.678...| # |32441|島根県| null|邑智郡| null| 川本町|Kawamoto-machi| 3317.0| 1672.0|POLYGON ((132.487...| # +-----+------+-----+------+------+----------+--------------+-------+-------+--------------------+ # only showing top 5 rows # table読み込み sdf_from_table = spark.table("geo_test.esri_japan") # 読み込むテーブル名を指定 sdf_from_table.show(5) # +-----+------+-----+------+------+----------+--------------+-------+-------+--------------------+ # |JCODE| KEN|SICHO| GUN|SEIREI|SIKUCHOSON| CITY_ENG| P_NUM| H_NUM| geometry| # +-----+------+-----+------+------+----------+--------------+-------+-------+--------------------+ # |32207|島根県| null| null| null| 江津市| Gotsu-shi|23664.0|11513.0|MULTIPOLYGON (((1...| # |32209|島根県| null| null| null| 雲南市| Unnan-shi|38479.0|13786.0|MULTIPOLYGON (((1...| # |32343|島根県| null|仁多郡| null| 奥出雲町| Okuizumo-cho|12694.0| 4782.0|POLYGON ((133.078...| # |32386|島根県| null|飯石郡| null| 飯南町| Iinan-cho| 4898.0| 2072.0|POLYGON ((132.678...| # |32441|島根県| null|邑智郡| null| 川本町|Kawamoto-machi| 3317.0| 1672.0|POLYGON ((132.487...| # +-----+------+-----+------+------+----------+--------------+-------+-------+--------------------+ # only showing top 5 rows単純なファイルとしてもテーブル形式でも保存と読み込みが出来ることが確認出来ました。
なお、体感ではgeopandasからpysparkのDataFrameへ変換は遅いように感じたので、geopandas<->pysparkの変換は最小限の回数に抑えた方が良いのだと思いました。※dtypes
sdf = sdf_from_file # 以下、ファイルから読み込んだ方をsdfとする display(sdf.dtypes) # [('JCODE', 'string'), # ('KEN', 'string'), # ('SICHO', 'string'), # ('GUN', 'string'), # ('SEIREI', 'string'), # ('SIKUCHOSON', 'string'), # ('CITY_ENG', 'string'), # ('P_NUM', 'double'), # ('H_NUM', 'double'), # ('geometry', 'udt')]
geometryはgeosparkライブラリ内で定義されたudt型として扱われているようです。
(なので、geosparkの設定をせずにファイルやテーブルを読み込もうとするとエラーが起きます)簡単なクエリの実行と確認など
公式ドキュメントから特に以下を参照:
- https://datasystemslab.github.io/GeoSpark/api/sql/GeoSparkSQL-Constructor/
- https://datasystemslab.github.io/GeoSpark/api/sql/GeoSparkSQL-Function/
- https://datasystemslab.github.io/GeoSpark/api/sql/GeoSparkSQL-Predicate/
# spark sqlを使えるようにDataFrameをTEMP VIEWとして登録しておく sdf.createOrReplaceTempView('esri_japan') # 大元のデータ数確認 sdf.count() # 1907 # 経度:135-140、緯度:35-40の範囲でfilter sdf_filtered = spark.sql(""" SELECT * FROM esri_japan WHERE ST_Contains(ST_PolygonFromEnvelope(135., 35., 140., 40.), esri_japan.geometry) """) sdf_filtered.show(5) # +-----+------+-----+--------+------+----------+----------------+-------+------+--------------------+ # |JCODE| KEN|SICHO| GUN|SEIREI|SIKUCHOSON| CITY_ENG| P_NUM| H_NUM| geometry| # +-----+------+-----+--------+------+----------+----------------+-------+------+--------------------+ # |06401|山形県| null|西置賜郡| null| 小国町| Oguni-machi| 7612.0|3076.0|POLYGON ((139.911...| # |06426|山形県| null|東田川郡| null| 三川町| Mikawa-machi| 7400.0|2387.0|POLYGON ((139.842...| # |07364|福島県| null|南会津郡| null| 檜枝岐村| Hinoemata-mura| 557.0| 202.0|POLYGON ((139.259...| # |07367|福島県| null|南会津郡| null| 只見町| Tadami-machi| 4366.0|1906.0|POLYGON ((139.366...| # |07368|福島県| null|南会津郡| null| 南会津町|Minamiaizu-machi|15679.0|6707.0|POLYGON ((139.530...| # +-----+------+-----+--------+------+----------+----------------+-------+------+--------------------+ # only showing top 5 rows sdf_filtered.count() # 573 <- original: 1907DataFrameの個数も減り(1907 -> 573)、ちゃんとフィルター出来ていそうだが念のため可視化して確認してみる
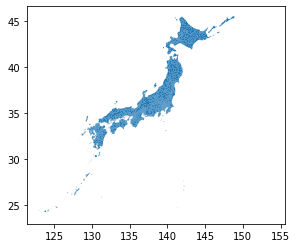
# matplotlib gdf_filtered = gpd.GeoDataFrame( # geopandasに変換しておく sdf_filtered.toPandas(), geometry='geometry' ) gdf_filtered.plot()ちなみにですが、オリジナルのjapan_ver821.shp全体をplotすると
gpd.read_file('japan_ver821/japan_ver821.shp') \ .plot()という感じなので、ちゃんとフィルタリング出来ていそうです。
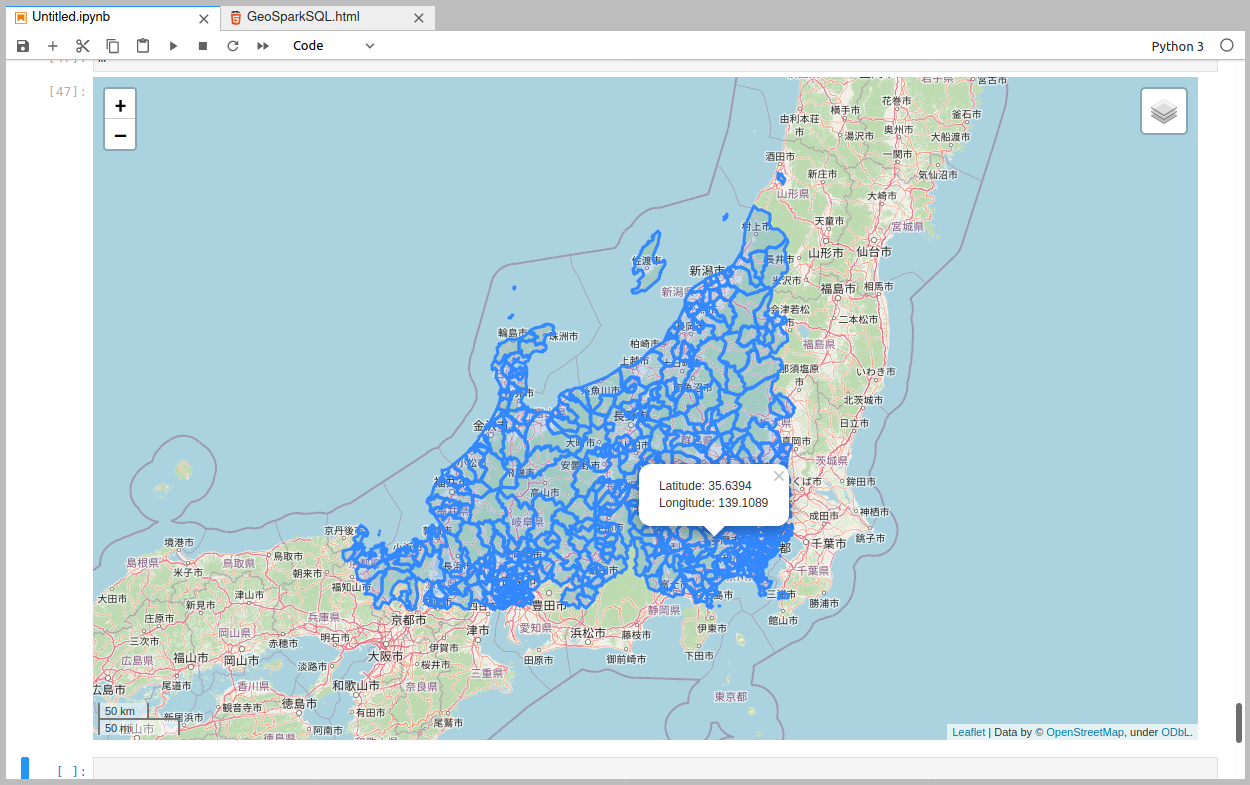
また、インタラクティブに可視化するには例えば
foliumを使って、m = folium.Map( location=(37.5, 137.5), # 緯度、経度の順なので注意 zoom_start=7, control_scale=True, ) m.add_child(folium.LatLngPopup()) # クリックするとポップアップで緯度経度を確認出来る # フィルタリングしたDataFrameをGeoJSONに変換してfoliumに渡す m.add_child( folium.GeoJson(gdf_filtered.to_json()) ) folium.LayerControl().add_to(m) # LayerControlを追加 m.save('df_filterd.html') # 保存 m # jupyter上で表示めでたくfolium上でも可視化出来ました。
3. 雑感
- 今回は小さいデータにごく簡単なクエリかけただけなのであまり嬉しさはなかったものの、PostGISのような雰囲気のことがSpark上で出来そうなのは面白かったです
- 後半で可視化するところは結局geopandas+matplotlibとfoliumでgeosparkあまり関係なかったのですが、可視化用の機能(GeoSpark Viz)もあるようなので、時間があるときに試せたら、と思います
- 序盤で書いたとおりApache Incubatorに入った関係で、今後の発展は期待出来そうですが仕様変更やパッケージ名の変更などがなされていって、今回書いたことがすぐに使えなくなるかもしれない。。。
- 投稿日:2020-08-08T15:49:26+09:00
kerasで指定するbatch_sizeについて
①具体例
batch_sizeはデフォルトでは32。
X_train.shape=(60000,784)の時、
model.fit(X_train,y_train,epochs=4)
とすると、(batch_sizeは指定してないので、32)
Epoch 1/4
1875/1875 [==============================] - 2s 840us/step - loss: 0.8699 - accuracy: 0.82040s
Epoch 2/4
1875/1875 [==============================] - 2s 811us/step - loss: 0.6358 - accuracy: 0.85760s
Epoch 3/4
1875/1875 [==============================] - 2s 829us/step - loss: 0.5283 - accuracy: 0.87400s - loss:
Epoch 4/4
1875/1875 [==============================] - 2s 949us/step - loss: 0.4664 - accuracy: 0.88420s - loss: 0.4674 - accuracy:というような結果が出現
この1875というのは、60000/32の値で、
1875個のデータで学習されたという事
②具体例
上記と同じ条件で、
model.fit(X_train,y_train,epochs=4,batch_size=100)
Epoch 1/4
600/600 [==============================] - 1s 2ms/step - loss: 0.4371 - accuracy: 0.8896
Epoch 2/4
600/600 [==============================] - 1s 2ms/step - loss: 0.4257 - accuracy: 0.8918
Epoch 3/4
600/600 [==============================] - 1s 1ms/step - loss: 0.4156 - accuracy: 0.8934: 0s - loss: 0.4190 - accu
Epoch 4/4
600/600 [==============================] - 1s 1ms/step - loss: 0.4065 - accuracy: 0.8949というような結果が出現。
この600というのは、60000/100の値で、
600個のデータで学習されたという事
- 投稿日:2020-08-08T14:32:41+09:00
Google翻訳とDeepL翻訳をGUIで実行
はじめに
最近は、英語の文献を読むために機械翻訳が手放せません。
こうなると、特に英語のPDFファイルをボタン一つで自動的に翻訳するツールが欲しくなります。
しかし、PDFは本文以外にも脚注やページ番号が混在していて、必要なテキストデータのみを抽出するところが難しいですね。
私は、テキストデータは手動でコピペし、その後の処理をGUIで対話的に実行するツールText_translatorを作成しました。使用するモジュール
- GUIはtkinterで作成
- Google翻訳のAPIはサードパーティ製のgoogletransxを利用
- DeepL翻訳は素直に正規APIを発行して利用
- 月630円+2,500円=3,130円から 下記コードのAPIキーは削除してあります。
- Wordの対訳表作成はpython-docxを利用
コード
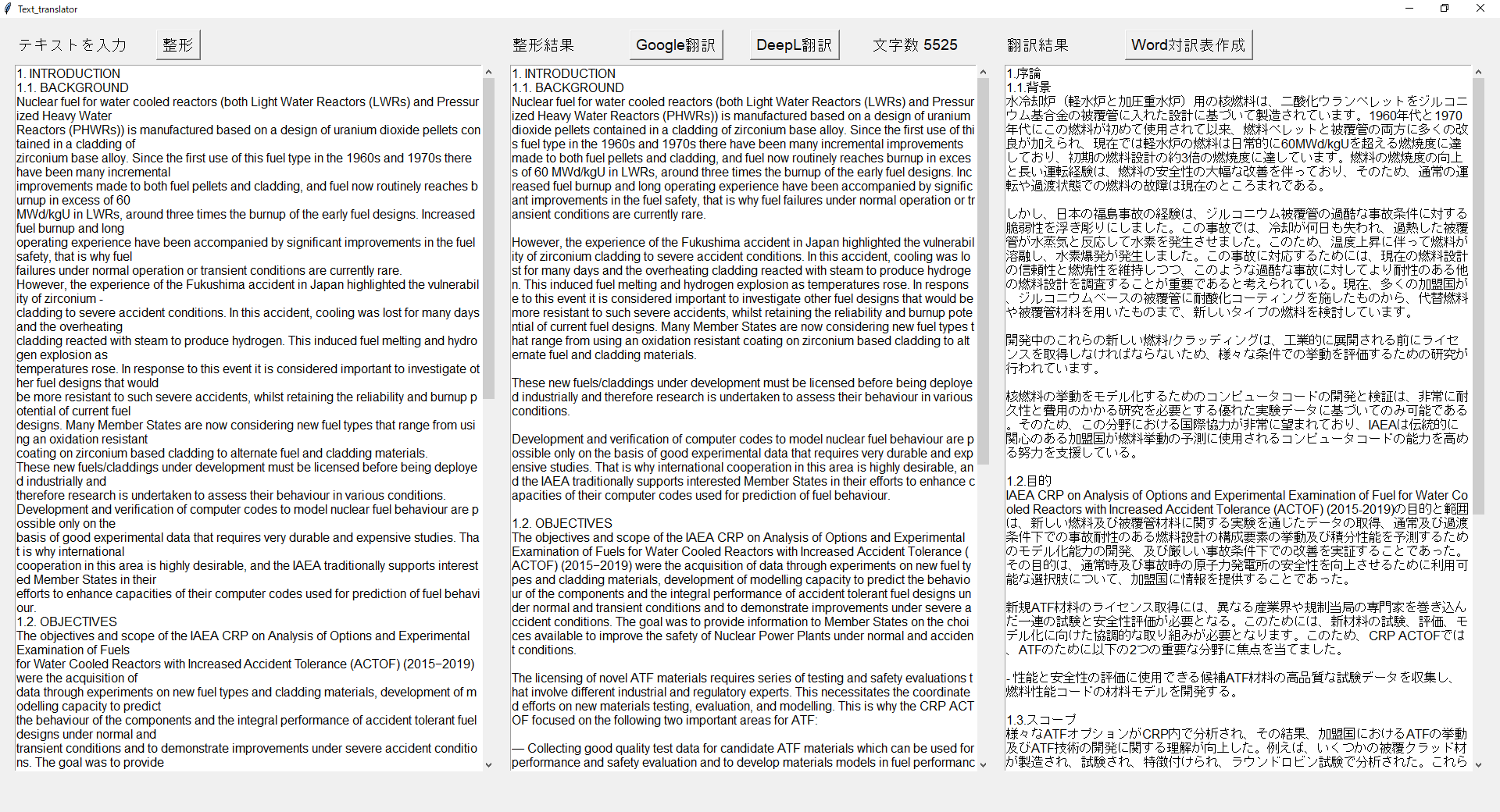
text_translator.py# -*- coding: utf-8 -*- import tkinter as tk import tkinter.scrolledtext as S from tkinter import messagebox as tkMessageBox from tkinter import filedialog as tkFileDialog from googletransx import Translator from docx import Document import os import datetime import requests # ボタン1がクリックされた時の処理 def ButtonClick1(): lines = input_box.get('1.0', 'end -1c') # 入力欄に入力された文字列を取得 # 改行前の「.」、「."」、「.”」、「:」をダミー文字に置き換え words = [".\n",".\"\n",".”\n",":\n"] for i, word in enumerate(words): lines = lines.replace(word,"XXX" + str(i)) lines = lines.replace("-\n", "") # 改行前の「-」を削除 lines = lines.replace("\n", " ") # 改行記号を削除 # ダミー文字を元に戻してさらに空行を追加 for i, word in enumerate(words): lines = lines.replace("XXX" + str(i), word + "\n") lines = lines.encode('utf-8', "ignore").decode('utf-8') # Pythonで取り扱えない文字を削除する。 processed_box.delete('1.0', 'end') # 整形結果欄をクリア processed_box.insert('1.0', lines) # 整形結果欄に整形結果を出力 # ボタン2がクリックされた時の処理 def ButtonClick2(): lines = get_text() while True: if len(lines) >= 5000: # 翻訳文字数が5000字以上の場合 lines1 = lines[:5000].rsplit('\n\n', 1)[0] # 5000字以内の段落 lines2 = lines[:5000].rsplit('\n\n', 1)[1] + lines[5000:] # 残りの段落 translator = Translator() lines1 = translator.translate(lines1, dest='ja').text # Google翻訳 translate_box.insert('end', lines1 + '\n\n') # 翻訳結果欄に表示 lines = lines2 # 残りの段落を設定 else: # 翻訳文字数が5000字未満の場合 translator = Translator() lines = translator.translate(lines, dest='ja').text # Google翻訳 translate_box.insert('end', lines) # 翻訳結果欄に表示 break # ボタン3がクリックされた時の処理 def ButtonClick3(): lines = get_text() # ここはご自分で発行されたKEYを入れてください DEEPL_API_KEY = 'XXXXX' # URLクエリに仕込むパラメータの辞書を作っておく params = { "auth_key": DEEPL_API_KEY, "text": lines, "target_lang": 'JA' # 出力テキストの言語を英語に設定 } # パラメータと一緒にPOSTする request = requests.post("https://api.deepl.com/v2/translate", data=params) result = request.json() lines = result["translations"][0]["text"] translate_box.insert('end', lines) # 翻訳結果欄に表示 # ボタン4がクリックされた時の処理 def ButtonClick4(): edit_text = processed_box.get('1.0', 'end -1c') # 整形結果欄に入力された文字列を取得 translate_text = translate_box.get('1.0', 'end -1c') # 翻訳結果欄に入力された文字列を取得 fTyp=[('wordファイル',"*.docx")] # Word対訳表テンプレートを選択 iDir='.' filename=tkFileDialog.askopenfilename(filetypes=fTyp,initialdir=iDir) document = Document(filename) for paragraph in document.paragraphs: paragraph.text = paragraph.text.replace("原文をここに記載する。",edit_text) # Word対訳表に原文を記載 paragraph.text = paragraph.text.replace("訳文をここに記載する。",translate_text) # Word対訳表に訳文を記載 paragraphs = (paragraph for table in document.tables for row in table.rows for cell in row.cells for paragraph in cell.paragraphs) for paragraph in paragraphs: paragraph.text = paragraph.text.replace("原文をここに記載する。",edit_text) # Word対訳表に原文を記載 paragraph.text = paragraph.text.replace("訳文をここに記載する。",translate_text) # Word対訳表に訳文を記載 dt_now = datetime.datetime.now() # Word対比表の保存 dt_str = str(dt_now.hour).zfill(2)+str(dt_now.minute).zfill(2)+str(dt_now.second).zfill(2) savefilename = filename.replace(u".docx","_replace" + dt_str + ".docx") document.save(savefilename) tkMessageBox.showinfo("作成完了",os.path.basename(savefilename) + " で保存しました。") # 保存結果を表示 def get_text(): lines = processed_box.get('1.0', 'end -1c') # 整形結果欄に入力された文字列を取得 # 文字数を取得して画面に表示 label = tk.Label(root, text = "文字数 " + str(len(lines)) + " ", font = ("Helvetica",14)) label.place(relx = 0.58, y = 20) translate_box.delete('1.0', 'end') # 翻訳結果欄をクリア return lines # メインのプログラム root = tk.Tk() root.geometry("1600x800") root.title("Text_translator") # ラベルの設定 label1 = tk.Label(root, text = "テキストを入力", font = ("Helvetica",14)) label1.place(x = 20, y = 20) label2 = tk.Label(root, text = "整形結果", font = ("Helvetica",14)) label2.place(relx = 0.34, y = 20) label3 = tk.Label(root, text = "翻訳結果", font = ("Helvetica",14)) label3.place(relx = 0.67, y = 20) # ボタンの設定 button1 = tk.Button(root, text = "整形", font = ("Helvetica",14), command = ButtonClick1) button1.place(x = 200, y = 15) button2 = tk.Button(root, text = "Google翻訳", font = ("Helvetica",14), command = ButtonClick2) button2.place(relx = 0.42, y = 15) button3 = tk.Button(root, text = "DeepL翻訳", font = ("Helvetica",14), command = ButtonClick3) button3.place(relx = 0.50, y = 15) button4 = tk.Button(root, text = "Word対訳表作成", font = ("Helvetica",14), command = ButtonClick4) button4.place(relx = 0.75, y = 15) # 入力ボックスの設定 input_box = S.ScrolledText(root, font = ("Helvetica",12)) input_box.place(relheight = 0.89, relwidth = 0.32, relx = 0.01, y = 60) # 整形結果ボックスの設定 processed_box = S.ScrolledText(root, font = ("Helvetica",12)) processed_box.place(relheight = 0.89, relwidth = 0.32, relx = 0.34, y = 60) # 翻訳ボックスの設定 translate_box = S.ScrolledText(root, font = ("Helvetica",12)) translate_box.place(relheight = 0.89, relwidth = 0.32, relx = 0.67, y = 60) root.mainloop()画面イメージと使用法
- 左ウィンドウにテキストをコピペ
-整形ボタンを押すと、テキストの余計な改行が削除され、中ウィンドウに表示される。
- 整形がうまくいっていないところは適宜手動で修正
-Google翻訳またはDeepL翻訳を押すと、右ウィンドウに翻訳結果が表示される。
-Word対訳表作成を押して、別途作成したWordのテンプレートを選択することにより、翻訳前後のWord対訳表が作成される。
- Wordテンプレートには、「原文をここに記載する。」と「訳文をここに記載する。」を記載しておく。それぞれ、原文と訳文に置き換わる。おわり
Google翻訳の自動化ツールはいろいろな人が作成していますが、GUIを使ったツールはあまり見当たりません。
GUIはわかりやすさがウリです。
気に入ったらLGTMお願いします。
- 投稿日:2020-08-08T14:32:23+09:00
pythonによる決定木による二値分類(【高等学校学習指導要領 情報Ⅱ】教員研修用教材)
はじめに
決定木とは決定理論の分野において、決定を行うための木構造のグラフです。
決定木を使う場面として、回帰(回帰木:regression tree)と分類(分類木:classification tree)が存在しますが、分類に関して決定木の活用方法を確認していきたいと思います。
具体的には、文部科学省のページで公開されている情報Ⅱの教員研修教材内の「分類による予測」で取り上げられているものをpythonで実装しつつ、仕組みを確認していけたらと思います。教材
高等学校情報科「情報Ⅱ」教員研修用教材(本編):文部科学省
第3章 情報とデータサイエンス 後半 (PDF:7.6MB)環境
- ipython
- Colaboratory - Google Colab
教材内で取り上げる箇所
学習15 分類による予測:「2. 決定木による二値分類」
のRで書かれたソースコードをpythonで実装しつつ、仕組みを見ていきたいと思います。
今回取り扱うデータ
教材と同じように、kaggleからtitanicデータをダウンロードします。
今回使うのはtitanicの「train.csv」です。https://www.kaggle.com/c/titanic/data
これは、タイタニック号の事故に関して一部の乗客の「生存・死亡」「客室の等級」「性別」「年齢」等が記載されたデータです。
まずは、教材に載っているRでの実装をこちらでpythonに置き換えた実装例を挙げていきながら、決定木について理解を深めたいと思います。pythonでの実装例と結果
データの読み込みと前処理(python)
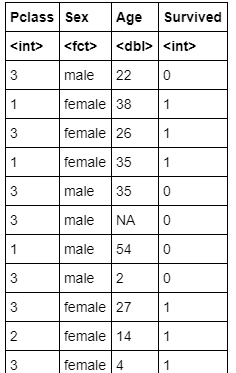
train.csvのうち、今回必要な、Pclass(客室の等級)、Sex(性別)、Age(年齢)、Survived(生存1,死亡0)の情報のみが必要なので、必要な部分のみ抽出します。
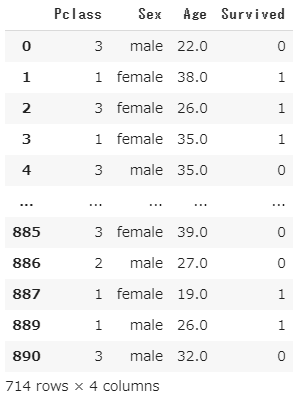
欠損値は'NaN'として取り扱っており、欠損値は取り除く方針で進めます。元データの読み込み・データ抽出・欠損値処理(ソースコード)
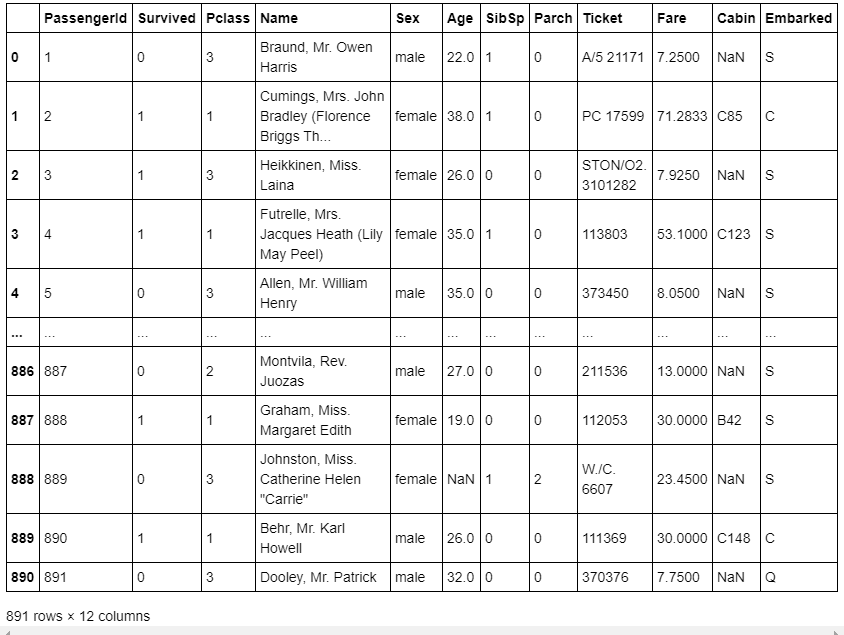
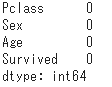
import numpy as np import pandas as pd from IPython.display import display from numpy import nan as NaN titanic_train = pd.read_csv('/content/train.csv') # 元データ表示 display(titanic_train) # Pclass(客室の等級)、Sex(性別)、Age(年齢)、Survived(生存1,死亡0) titanic_data = titanic_train[['Pclass', 'Sex', 'Age', 'Survived']] display(titanic_data) # 欠損値'NaN'は、取り除く titanic_data = titanic_data.dropna() display(titanic_data) # 欠損値取り除かれているかデータ確認 titanic_data.isnull().sum()元データの読み込み・データ抽出・欠損値処理(出力結果)
元データの読み込み
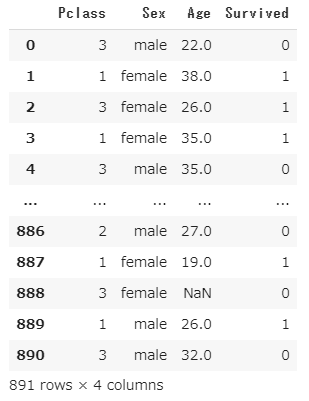
データ抽出
欠損値処理のデータ結果
欠損値取り除かれているかデータ確認
決定木の可視化の実行(ソースコード)
pythonによる決定木の可視化は、見た目がわかりやすいという理由で、dtreevizを使用しようと思います。
dtreevizインストール
!pip install dtreeviz pydotplus決定木の可視化の実行
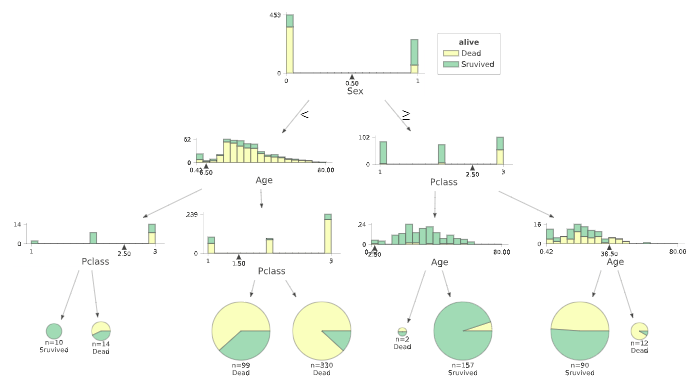
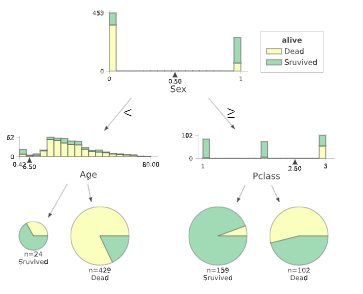
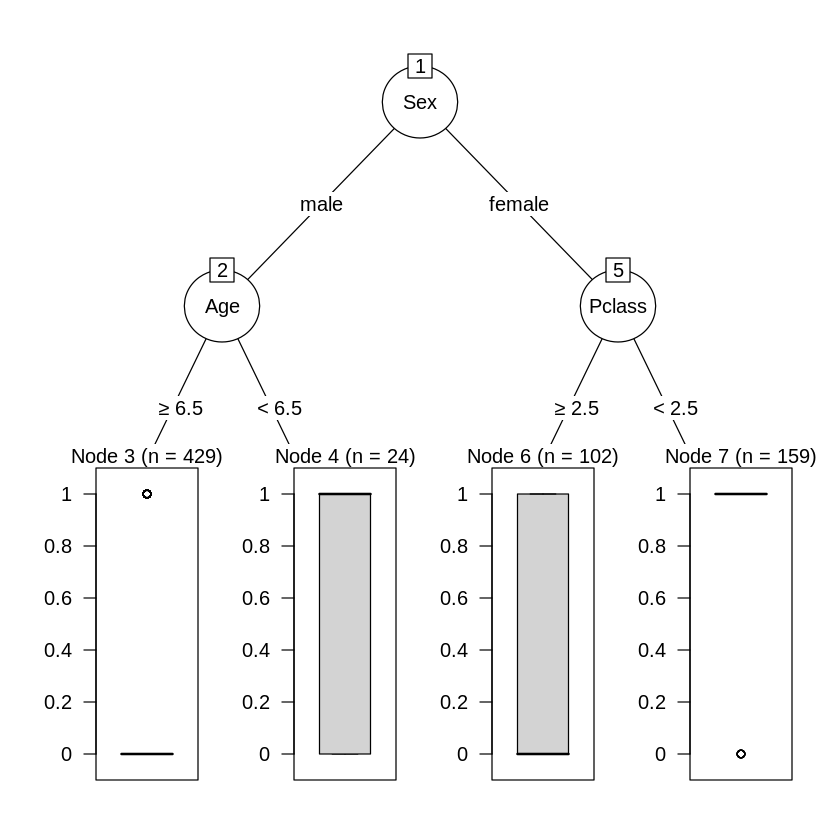
import sklearn.tree as tree from dtreeviz.trees import dtreeviz ##maleを0に、femaleを1に変換 titanic_data["Sex"] = titanic_data["Sex"].map({"male":0,"female":1}) # 'Survived'列を覗いたデータを特徴行列 # 'Survived'列を目的変数 X_train = titanic_data.drop('Survived', axis=1) Y_train = titanic_data['Survived'] # 決定木作成(木の最大の深さは3に指定した) clf = tree.DecisionTreeClassifier(random_state=0, max_depth = 3) model = clf.fit(X_train, Y_train) viz = dtreeviz( model, X_train, Y_train, target_name = 'alive', feature_names = X_train.columns, class_names = ['Dead','Sruvived'] ) # 決定木表示 display(viz)決定木の可視化の実行(出力)
決定木の分析では、どれだけ深い木まで分析をすすめるかという点を考慮する必要があります。
決定木は適度な深さで分析を止めるなどしないと、分析に使った訓練データに過剰に適合してしまう過学習(overfitting)が発生し、汎化性能が低下する恐れがあります。
今回表示の関係で、最大の深さ=3に指定しているので、そこまで深すぎる設定にはなっていませんが、教材では適度な複雑度(complexioty parameter)を指定して、木の剪定(pruning)を行っているので同様に進めたいと思います。剪定
決定木の各ノードの条件分岐がうまい具合に作られているかを見る際に、不純度(impurity)というパラメータが使用される事が多く、この不純度というパラメータは、小さいほどよりシンプルな基準で分類ができているというイメージです。
もう一つ関係する重要な要素として複雑度というパラメータがあり、これは木全体がどれだけ複雑かどうかということを示しているイメージです。
今回のソースコードでは、決定木の生成の際の不純度はジニ不純度というものが使われております。(DecisionTreeClassifier()の引数criterion{“gini”, “entropy”}, default=”gini”)
そして、決定木の生成方法としては、最小コスト複雑度剪定(Minimal cost-complexity pruning)というアルゴリズムを使っております。
これは、最小コスト複雑度剪定と言う通り、木の生成コストと呼ばれる(木の終端のノード数×木の複雑度+木の不純度)を最小とするような決定木を生成するアルゴリズムです。
複雑度が大きいときは、終端のノード数が木の生成コストに強く影響を与え、最小コスト複雑度剪定で決定木を生成する場合、より"小さい木(深さやノード数が小さい)"が生成できます。
逆に複雑度が小さいときは、終端ノード数による木の生成コストの影響が小さくなり、同様に決定木を生成する場合、大きくて複雑な木(深さやノード数が小さい)が生成できます。数式を使わずざっくりとしたイメージの話をしましたが、公式ドキュメントやそれ以外にも詳しく説明しているサイトはたくさんあるので、じっくり調べてみると良いかもしれません。
[参考] https://scikit-learn.org/stable/modules/tree.html#minimal-cost-complexity-pruning剪定(ソースコード)
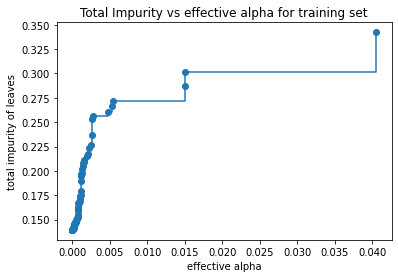
複雑度に関するパラメータと不純度に関するパラメータの関係
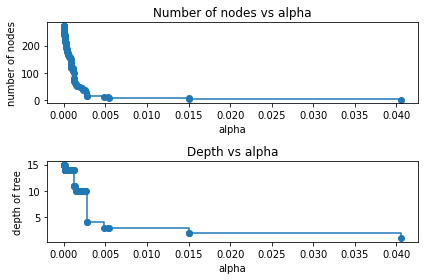
import matplotlib.pyplot as plt # 決定木作成(木の最大の深さ指定なし) clf = tree.DecisionTreeClassifier(random_state=0) model = clf.fit(X_train, Y_train) path = clf.cost_complexity_pruning_path(X_train, Y_train) # ccp_alphas:複雑度に関するパラメータ # impurities:不純度に関するパラメータ ccp_alphas, impurities = path.ccp_alphas, path.impurities fig, ax = plt.subplots() ax.plot(ccp_alphas[:-1], impurities[:-1], marker='o', drawstyle="steps-post") ax.set_xlabel("effective alpha") ax.set_ylabel("total impurity of leaves") ax.set_title("Total Impurity vs effective alpha for training set")複雑度に関するパラメータと生成されるノード数や木の深さの関係
clfs = [] for ccp_alpha in ccp_alphas: clf = tree.DecisionTreeClassifier(random_state=0, ccp_alpha=ccp_alpha) clf.fit(X_train, Y_train) clfs.append(clf) print("Number of nodes in the last tree is: {} with ccp_alpha: {}".format( clfs[-1].tree_.node_count, ccp_alphas[-1])) clfs = clfs[:-1] ccp_alphas = ccp_alphas[:-1] node_counts = [clf.tree_.node_count for clf in clfs] depth = [clf.tree_.max_depth for clf in clfs] fig, ax = plt.subplots(2, 1) ax[0].plot(ccp_alphas, node_counts, marker='o', drawstyle="steps-post") ax[0].set_xlabel("alpha") ax[0].set_ylabel("number of nodes") ax[0].set_title("Number of nodes vs alpha") ax[1].plot(ccp_alphas, depth, marker='o', drawstyle="steps-post") ax[1].set_xlabel("alpha") ax[1].set_ylabel("depth of tree") ax[1].set_title("Depth vs alpha") fig.tight_layout()剪定(出力結果)
複雑度に関するパラメータと不純度に関するパラメータの関係
複雑度に関するパラメータと生成されるノード数や木の深さの関係
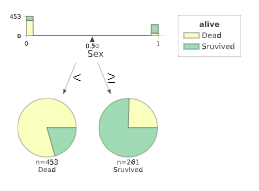
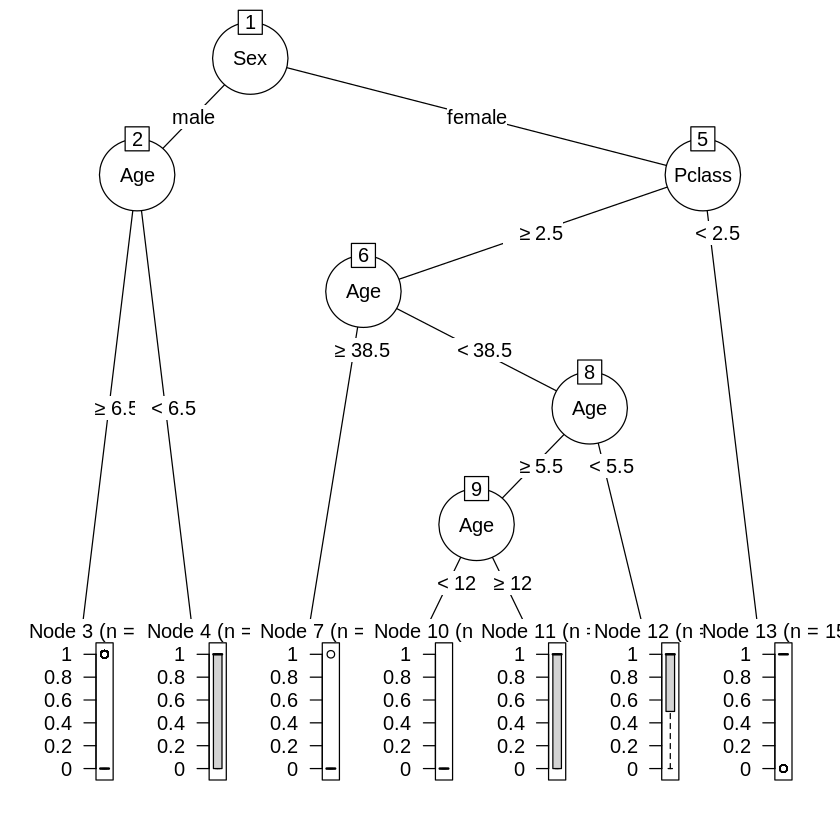
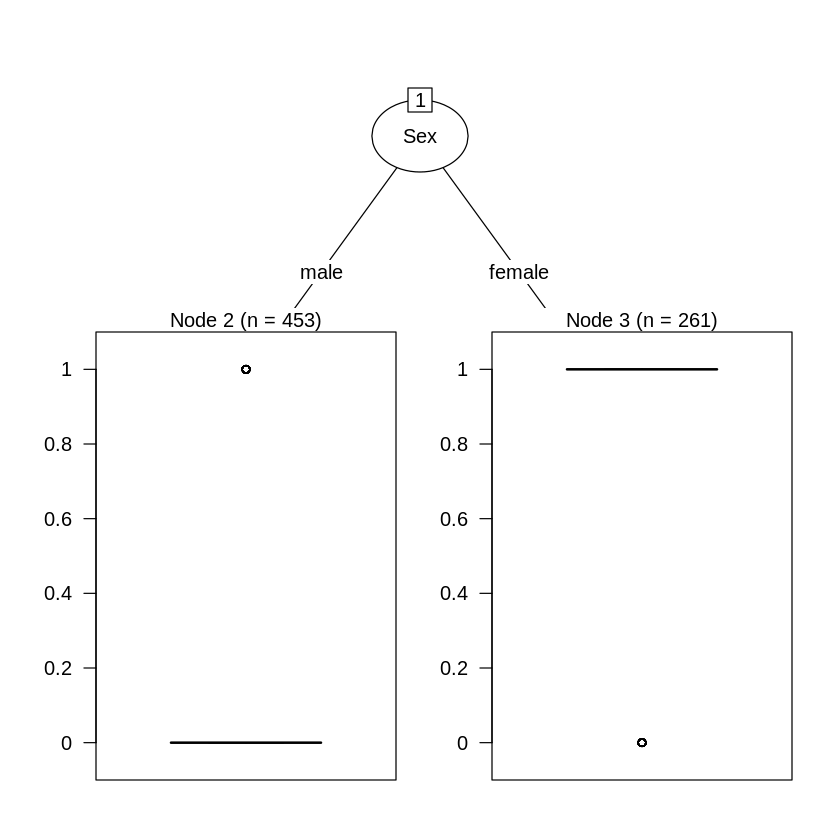
教材では、深さ1~2程度の木になるように剪定を行っているので、複雑度に関するパラメータccp_alphaを0.041あたりだと深さが1、ノード数1くらいで、ccp_alphaが0.0151あたりだと深さ2、ノード数3くらいになりそうであることがわかります。
剪定後の決定木(ソースコード)
ccp_alpha=0.041
clf = tree.DecisionTreeClassifier(ccp_alpha = 0.041) model = clf.fit(X_train, Y_train) viz = dtreeviz( model, X_train, Y_train, target_name = 'alive', feature_names = X_train.columns, class_names = ['Dead','Sruvived'] ) display(viz)ccp_alpha=0.0151
clf = tree.DecisionTreeClassifier(ccp_alpha = 0.0151) model = clf.fit(X_train, Y_train) viz = dtreeviz( model, X_train, Y_train, target_name = 'alive', feature_names = X_train.columns, class_names = ['Dead','Sruvived'] ) display(viz)剪定後の決定木(出力結果)
ccp_alpha=0.041
ccp_alpha=0.0151
これらを見ると、生死を分ける最大要因は、性別であり女性のほうが救助されやすかったことがわかります。
男性であっても、年齢が若い(=子ども)であるほど生存率は高いことがわかります。
女性であれば、客室の等級が高いほど生存率が高いように読み取れます。コメント
教材では以下のような記述になっております。
この事故の生死をこの事故の生死を決める最大の要素は,性別であった。乗務員が積極的に女性や子供を救助したことも読み取れる。また,船室の優劣は生死を決める要因にはなっていないようである。
私のほうで実装し出力させた結果では、船室の優劣も生死を決める要因になっているようにみえました。
pythonであっても、Rであっても決定木の構成は相違ないものでしたので、教材の結果だけを見るだけでなく、実際に実行してみて自分なりに分析してみることが大切だと思いました。[参考]Rでの実装例と結果(教材より)
データの読み込みと前処理(R)
元データの読み込み(ソースコード)
titanic.train<-read.csv("/content/train.csv") # データの場所を指定 str(titanic.train)元データの読み込み(出力結果)
'data.frame': 891 obs. of 12 variables: $ PassengerId: int 1 2 3 4 5 6 7 8 9 10 ... $ Survived : int 0 1 1 1 0 0 0 0 1 1 ... $ Pclass : int 3 1 3 1 3 3 1 3 3 2 ... $ Name : Factor w/ 891 levels "Abbing, Mr. Anthony",..: 109 191 358 277 16 559 520 629 417 581 ... $ Sex : Factor w/ 2 levels "female","male": 2 1 1 1 2 2 2 2 1 1 ... $ Age : num 22 38 26 35 35 NA 54 2 27 14 ... $ SibSp : int 1 1 0 1 0 0 0 3 0 1 ... $ Parch : int 0 0 0 0 0 0 0 1 2 0 ... $ Ticket : Factor w/ 681 levels "110152","110413",..: 524 597 670 50 473 276 86 396 345 133 ... $ Fare : num 7.25 71.28 7.92 53.1 8.05 ... $ Cabin : Factor w/ 148 levels "","A10","A14",..: 1 83 1 57 1 1 131 1 1 1 ... $ Embarked : Factor w/ 4 levels "","C","Q","S": 4 2 4 4 4 3 4 4 4 2 ...データ抽出(ソースコード)
titanic.data<-titanic.train[,c("Pclass","Sex","Age","Survived")] titanic.dataデータ抽出(出力結果)
欠損値(NA)(ソースコード)
titanic.data<-na.omit(titanic.data)決定木の可視化の実行(ソースコード)
install.packages("partykit") library(rpart) library(partykit) titanic.ct<-rpart(Survived~.,data=titanic.data, method="class") plot(as.party(titanic.ct),tp_arg=T)決定木の可視化の実行(出力結果)
分類木のCP(ソースコード)
printcp(titanic.ct)分類木のCP(出力結果)
Classification tree: rpart(formula = Survived ~ ., data = titanic.data, method = "class") Variables actually used in tree construction: [1] Age Pclass Sex Root node error: 290/714 = 0.40616 n= 714 CP nsplit rel error xerror xstd 1 0.458621 0 1.00000 1.00000 0.045252 2 0.027586 1 0.54138 0.54138 0.038162 3 0.012069 3 0.48621 0.53793 0.038074 4 0.010345 5 0.46207 0.53448 0.037986 5 0.010000 6 0.45172 0.53793 0.038074CPを0.028にした場合の分類木(ソースコード)
titanic.ct2<-rpart(Survived~.,data=titanic.data, method="class", cp=0.028) plot(as.party(titanic.ct2))決定木の可視化の実行(出力結果)
CPを0.027にした場合の分類木(ソースコード)
titanic.ct3<-rpart(Survived~.,data=titanic.data, method="class", cp=0.027) plot(as.party(titanic.ct3))決定木の可視化の実行(出力結果)
ソースコード
python版
https://gist.github.com/ereyester/dfb4fd6fb3e58c5d0539866f7e2622b4R版
https://gist.github.com/ereyester/182d5d49ea04be579da2ffc82412a82a
- 投稿日:2020-08-08T14:32:23+09:00
pythonによる決定木による二値分類(【高等学校情報科 情報Ⅱ】教員研修用教材)
はじめに
決定木とは決定理論の分野において、決定を行うための木構造のグラフです。
決定木を使う場面として、回帰(回帰木:regression tree)と分類(分類木:classification tree)が存在しますが、分類に関して決定木の活用方法を確認していきたいと思います。
具体的には、文部科学省のページで公開されている情報Ⅱの教員研修教材内の「分類による予測」で取り上げられているものをpythonで実装しつつ、仕組みを確認していけたらと思います。教材
高等学校情報科「情報Ⅱ」教員研修用教材(本編):文部科学省
第3章 情報とデータサイエンス 後半 (PDF:7.6MB)環境
- ipython
- Colaboratory - Google Colab
教材内で取り上げる箇所
学習15 分類による予測:「2. 決定木による二値分類」
のRで書かれたソースコードをpythonで実装しつつ、仕組みを見ていきたいと思います。
今回取り扱うデータ
教材と同じように、kaggleからtitanicデータをダウンロードします。
今回使うのはtitanicの「train.csv」です。https://www.kaggle.com/c/titanic/data
これは、タイタニック号の事故に関して一部の乗客の「生存・死亡」「客室の等級」「性別」「年齢」等が記載されたデータです。
まずは、教材に載っているRでの実装をこちらでpythonに置き換えた実装例を挙げていきながら、決定木について理解を深めたいと思います。pythonでの実装例と結果
データの読み込みと前処理(python)
train.csvのうち、今回必要な、Pclass(客室の等級)、Sex(性別)、Age(年齢)、Survived(生存1,死亡0)の情報のみが必要なので、必要な部分のみ抽出します。
欠損値は'NaN'として取り扱っており、欠損値は取り除く方針で進めます。元データの読み込み・データ抽出・欠損値処理(ソースコード)
import numpy as np import pandas as pd from IPython.display import display from numpy import nan as NaN titanic_train = pd.read_csv('/content/train.csv') # 元データ表示 display(titanic_train) # Pclass(客室の等級)、Sex(性別)、Age(年齢)、Survived(生存1,死亡0) titanic_data = titanic_train[['Pclass', 'Sex', 'Age', 'Survived']] display(titanic_data) # 欠損値'NaN'は、取り除く titanic_data = titanic_data.dropna() display(titanic_data) # 欠損値取り除かれているかデータ確認 titanic_data.isnull().sum()元データの読み込み・データ抽出・欠損値処理(出力結果)
元データの読み込み
データ抽出
欠損値処理のデータ結果
欠損値取り除かれているかデータ確認
決定木の可視化の実行(ソースコード)
pythonによる決定木の可視化は、見た目がわかりやすいという理由で、dtreevizを使用しようと思います。
dtreevizインストール
!pip install dtreeviz pydotplus決定木の可視化の実行
import sklearn.tree as tree from dtreeviz.trees import dtreeviz ##maleを0に、femaleを1に変換 titanic_data["Sex"] = titanic_data["Sex"].map({"male":0,"female":1}) # 'Survived'列を覗いたデータを特徴行列 # 'Survived'列を目的変数 X_train = titanic_data.drop('Survived', axis=1) Y_train = titanic_data['Survived'] # 決定木作成(木の最大の深さは3に指定した) clf = tree.DecisionTreeClassifier(random_state=0, max_depth = 3) model = clf.fit(X_train, Y_train) viz = dtreeviz( model, X_train, Y_train, target_name = 'alive', feature_names = X_train.columns, class_names = ['Dead','Sruvived'] ) # 決定木表示 display(viz)決定木の可視化の実行(出力)
決定木の分析では、どれだけ深い木まで分析をすすめるかという点を考慮する必要があります。
決定木は適度な深さで分析を止めるなどしないと、分析に使った訓練データに過剰に適合してしまう過学習(overfitting)が発生し、汎化性能が低下する恐れがあります。
今回表示の関係で、最大の深さ=3に指定しているので、そこまで深すぎる設定にはなっていませんが、教材では適度な複雑度(complexioty parameter)を指定して、木の剪定(pruning)を行っているので同様に進めたいと思います。剪定
決定木の各ノードの条件分岐がうまい具合に作られているかを見る際に、不純度(impurity)というパラメータが使用される事が多く、この不純度というパラメータは、小さいほどよりシンプルな基準で分類ができているというイメージです。
もう一つ関係する重要な要素として複雑度というパラメータがあり、これは木全体がどれだけ複雑かどうかということを示しているイメージです。
今回のソースコードでは、決定木の生成の際の不純度はジニ不純度というものが使われております。(DecisionTreeClassifier()の引数criterion{“gini”, “entropy”}, default=”gini”)
そして、決定木の生成方法としては、最小コスト複雑度剪定(Minimal cost-complexity pruning)というアルゴリズムを使っております。
これは、最小コスト複雑度剪定と言う通り、木の生成コストと呼ばれる(木の終端のノード数×木の複雑度+木の不純度)を最小とするような決定木を生成するアルゴリズムです。
複雑度が大きいときは、終端のノード数が木の生成コストに強く影響を与え、最小コスト複雑度剪定で決定木を生成する場合、より"小さい木(深さやノード数が小さい)"が生成できます。
逆に複雑度が小さいときは、終端ノード数による木の生成コストの影響が小さくなり、同様に決定木を生成する場合、大きくて複雑な木(深さやノード数が小さい)が生成できます。数式を使わずざっくりとしたイメージの話をしましたが、公式ドキュメントやそれ以外にも詳しく説明しているサイトはたくさんあるので、じっくり調べてみると良いかもしれません。
[参考] https://scikit-learn.org/stable/modules/tree.html#minimal-cost-complexity-pruning剪定(ソースコード)
複雑度に関するパラメータと不純度に関するパラメータの関係
import matplotlib.pyplot as plt # 決定木作成(木の最大の深さ指定なし) clf = tree.DecisionTreeClassifier(random_state=0) model = clf.fit(X_train, Y_train) path = clf.cost_complexity_pruning_path(X_train, Y_train) # ccp_alphas:複雑度に関するパラメータ # impurities:不純度に関するパラメータ ccp_alphas, impurities = path.ccp_alphas, path.impurities fig, ax = plt.subplots() ax.plot(ccp_alphas[:-1], impurities[:-1], marker='o', drawstyle="steps-post") ax.set_xlabel("effective alpha") ax.set_ylabel("total impurity of leaves") ax.set_title("Total Impurity vs effective alpha for training set")複雑度に関するパラメータと生成されるノード数や木の深さの関係
clfs = [] for ccp_alpha in ccp_alphas: clf = tree.DecisionTreeClassifier(random_state=0, ccp_alpha=ccp_alpha) clf.fit(X_train, Y_train) clfs.append(clf) print("Number of nodes in the last tree is: {} with ccp_alpha: {}".format( clfs[-1].tree_.node_count, ccp_alphas[-1])) clfs = clfs[:-1] ccp_alphas = ccp_alphas[:-1] node_counts = [clf.tree_.node_count for clf in clfs] depth = [clf.tree_.max_depth for clf in clfs] fig, ax = plt.subplots(2, 1) ax[0].plot(ccp_alphas, node_counts, marker='o', drawstyle="steps-post") ax[0].set_xlabel("alpha") ax[0].set_ylabel("number of nodes") ax[0].set_title("Number of nodes vs alpha") ax[1].plot(ccp_alphas, depth, marker='o', drawstyle="steps-post") ax[1].set_xlabel("alpha") ax[1].set_ylabel("depth of tree") ax[1].set_title("Depth vs alpha") fig.tight_layout()剪定(出力結果)
複雑度に関するパラメータと不純度に関するパラメータの関係
複雑度に関するパラメータと生成されるノード数や木の深さの関係
教材では、深さ1~2程度の木になるように剪定を行っているので、複雑度に関するパラメータccp_alphaを0.041あたりだと深さが1、ノード数1くらいで、ccp_alphaが0.0151あたりだと深さ2、ノード数3くらいになりそうであることがわかります。
剪定後の決定木(ソースコード)
ccp_alpha=0.041
clf = tree.DecisionTreeClassifier(ccp_alpha = 0.041) model = clf.fit(X_train, Y_train) viz = dtreeviz( model, X_train, Y_train, target_name = 'alive', feature_names = X_train.columns, class_names = ['Dead','Sruvived'] ) display(viz)ccp_alpha=0.0151
clf = tree.DecisionTreeClassifier(ccp_alpha = 0.0151) model = clf.fit(X_train, Y_train) viz = dtreeviz( model, X_train, Y_train, target_name = 'alive', feature_names = X_train.columns, class_names = ['Dead','Sruvived'] ) display(viz)剪定後の決定木(出力結果)
ccp_alpha=0.041
ccp_alpha=0.0151
これらを見ると、生死を分ける最大要因は、性別であり女性のほうが救助されやすかったことがわかります。
男性であっても、年齢が若い(=子ども)であるほど生存率は高いことがわかります。
女性であれば、客室の等級が高いほど生存率が高いように読み取れます。コメント
教材では以下のような記述になっております。
この事故の生死をこの事故の生死を決める最大の要素は,性別であった。乗務員が積極的に女性や子供を救助したことも読み取れる。また,船室の優劣は生死を決める要因にはなっていないようである。
私のほうで実装し出力させた結果では、船室の優劣も生死を決める要因になっているようにみえました。
pythonであっても、Rであっても決定木の構成は相違ないものでしたので、教材の結果だけを見るだけでなく、実際に実行してみて自分なりに分析してみることが大切だと思いました。[参考]Rでの実装例と結果(教材より)
データの読み込みと前処理(R)
元データの読み込み(ソースコード)
titanic.train<-read.csv("/content/train.csv") # データの場所を指定 str(titanic.train)元データの読み込み(出力結果)
'data.frame': 891 obs. of 12 variables: $ PassengerId: int 1 2 3 4 5 6 7 8 9 10 ... $ Survived : int 0 1 1 1 0 0 0 0 1 1 ... $ Pclass : int 3 1 3 1 3 3 1 3 3 2 ... $ Name : Factor w/ 891 levels "Abbing, Mr. Anthony",..: 109 191 358 277 16 559 520 629 417 581 ... $ Sex : Factor w/ 2 levels "female","male": 2 1 1 1 2 2 2 2 1 1 ... $ Age : num 22 38 26 35 35 NA 54 2 27 14 ... $ SibSp : int 1 1 0 1 0 0 0 3 0 1 ... $ Parch : int 0 0 0 0 0 0 0 1 2 0 ... $ Ticket : Factor w/ 681 levels "110152","110413",..: 524 597 670 50 473 276 86 396 345 133 ... $ Fare : num 7.25 71.28 7.92 53.1 8.05 ... $ Cabin : Factor w/ 148 levels "","A10","A14",..: 1 83 1 57 1 1 131 1 1 1 ... $ Embarked : Factor w/ 4 levels "","C","Q","S": 4 2 4 4 4 3 4 4 4 2 ...データ抽出(ソースコード)
titanic.data<-titanic.train[,c("Pclass","Sex","Age","Survived")] titanic.dataデータ抽出(出力結果)
欠損値(NA)(ソースコード)
titanic.data<-na.omit(titanic.data)決定木の可視化の実行(ソースコード)
install.packages("partykit") library(rpart) library(partykit) titanic.ct<-rpart(Survived~.,data=titanic.data, method="class") plot(as.party(titanic.ct),tp_arg=T)決定木の可視化の実行(出力結果)
分類木のCP(ソースコード)
printcp(titanic.ct)分類木のCP(出力結果)
Classification tree: rpart(formula = Survived ~ ., data = titanic.data, method = "class") Variables actually used in tree construction: [1] Age Pclass Sex Root node error: 290/714 = 0.40616 n= 714 CP nsplit rel error xerror xstd 1 0.458621 0 1.00000 1.00000 0.045252 2 0.027586 1 0.54138 0.54138 0.038162 3 0.012069 3 0.48621 0.53793 0.038074 4 0.010345 5 0.46207 0.53448 0.037986 5 0.010000 6 0.45172 0.53793 0.038074CPを0.028にした場合の分類木(ソースコード)
titanic.ct2<-rpart(Survived~.,data=titanic.data, method="class", cp=0.028) plot(as.party(titanic.ct2))決定木の可視化の実行(出力結果)
CPを0.027にした場合の分類木(ソースコード)
titanic.ct3<-rpart(Survived~.,data=titanic.data, method="class", cp=0.027) plot(as.party(titanic.ct3))決定木の可視化の実行(出力結果)
ソースコード
python版
https://gist.github.com/ereyester/dfb4fd6fb3e58c5d0539866f7e2622b4R版
https://gist.github.com/ereyester/182d5d49ea04be579da2ffc82412a82a
- 投稿日:2020-08-08T14:31:42+09:00
日本語(カタカナ)を音節単位で分かち書き【Python】
はじめに
日本語(カタカナ文字列)を音節単位で分かち書き(音節分かち書き)するpythonの関数を作りました。
元文 モーラ分かち書き 音節分かち書き ガッキューシンブン ガ/ッ/キュ/ー/シ/ン/ブ/ン ガッ/キュー/シン/ブン アウトバーン ア/ウ/ト/バ/ー/ン ア/ウ/ト/バーン 日本語の音韻の代表的な分割単位としてモーラと音節があります。モーラはいわゆる俳句の「5・7・5」を数えるときの区切り方で、長音(ー)、促音(ッ)、撥音(ン)も1拍と数えます。それに対し、音節では長音、促音、撥音は単体で数えられず、直前の単一で音節となれるカナと合わせてひとつの拍と見なされます。「バーン」のように長音、促音、撥音が連続した場合は3以上のモーラ数で1音節となります。詳細はモーラ - Wikipediaを御覧ください。
本稿では音節単位での分かち書きについて説明します。
モーラ単位での分割については下記で説明しています。
日本語(カタカナ)をモーラ単位で分かち書き【Python】環境
- macOS Catalina 10.15.4
- python3.8.0
方針
考えやすくするために、入力は記号を含まない全角カタカナの文字列とします。また、長音で表せるところは長音に変換されているものとします。これは例えば「ガッキュウ」は「ガッキュー」のように表現されているという意味です。
なお、漢字仮名交じり文を発音のカタカナ文字列に変換する方法は別記事にまとめましたので、もしよければ御覧ください。ただし、MeCabを使っていますので、辞書にない言葉は変換できません。このとき、音節の構成条件を下記のように定義します。
【下記1-4と「ー/ッ/ン」の連続(0文字含む)文字列】
1. ウ段+「ァ/ィ/ェ/ォ」
2. イ段(「イ」を除く)+「ャ/ュ/ェ/ョ」
3. 「テ/デ」+「ィ/ュ」
4. 上記以外の大文字カナ1文字これは
正規表現 意味 ① [ウクスツヌフムユルグズヅブプヴ][ァィェォ] ウ段+「ァ/ィ/ェ/ォ」 ② [イキシシニヒミリギジヂビピ][ャュェョ] イ段(「イ」を除く)+「ャ/ュ/ェ/ョ」 ③ [テデ][ィュ] 「テ/デ」+「ィ/ュ」 ④ [アイウエオカ-ヂツ-モヤユヨ-ヲヴ] ①②③以外の大文字カナ1文字 ⑤ [ーッン]* 「ー/ッ/ン」の連続文字列(0文字含む) としたとき、
'(①|②|③|④)⑤'のように書けます。コード
import re #「((ウ段+「ァ/ィ/ェ/ォ」)|(イ段(「イ」を除く)+「ャ/ュ/ェ/ョ」)|( 「テ/デ」+「ィ/ュ」)|(大文字カナ))(「ー/ッ/ン」の連続文字列(0文字含む))」の正規表現 c1 = '[ウクスツヌフムユルグズヅブプヴ][ァィェォ]' #ウ段+「ァ/ィ/ェ/ォ」 c2 = '[イキシシニヒミリギジヂビピ][ャュェョ]' #イ段(「イ」を除く)+「ャ/ュ/ェ/ョ」 c3 = '[テデ][ィュ]' #「テ/デ」+「ィ/ュ」 c4 = '[アイウエオカ-ヂツ-モヤユヨ-ヲヴ]' #大文字カナ c5 = '[ーッン]*' #「ー/ッ/ン」の連続文字列(0文字含む) cond = '(?:'+c1+'|'+c2+'|'+c3+'|'+c4+')'+c5 #(?:)はサブパターンの参照を避けるカッコ cond = '('+cond+')' re_syllable = re.compile(cond) def syllableWakachi(kana_text): return re_syllable.findall(kana_text) text = 'シンシュンシャンソンショー' print(text) print(syllableWakachi(text)) print('') text = 'トーキョートッキョキョカキョク' print(text) print(syllableWakachi(text)) print('') text = 'アウトバーン' print(text) print(syllableWakachi(text)) print('') text = 'ガッキュウホウカイ' print(text) print(syllableWakachi(text))出力は下記です。
シンシュンシャンソンショー ['シン', 'シュン', 'シャン', 'ソン', 'ショー'] トーキョートッキョキョカキョク ['トー', 'キョー', 'トッ', 'キョ', 'キョ', 'カ', 'キョ', 'ク'] アウトバーン ['ア', 'ウ', 'ト', 'バーン'] ガッキュウホウカイ ['ガッ', 'キュ', 'ウ', 'ホ', 'ウ', 'カ', 'イ']
- 投稿日:2020-08-08T14:11:34+09:00
matplotlibとcartopyでEarth風の流れのアニメーションを描く
次のようなEarth風(https://earth.nullschool.net/)
の流れのアニメーションをつくる。
元のプログラムは
https://github.com/rougier/windmap
から得て、それをCartopy(https://scitools.org.uk/cartopy/docs/latest/)
で地図上に落とせるようにした(ついでながら元のプログラムではアニメーションの動きが期待とは逆向きだったので符号などを逆転させた)。
作ったPythonコードのJupyter Notebookはgithubに置いた。
https://github.com/tmiyama/windmapアニメーションの肝はカラーで、それがなければ一本の流跡線になる。カラーで白から黒に色を変える事で、点線のようにし、それを少しずつずらすことで流れていくようなアニメーションにしている。