- 投稿日:2020-05-20T23:51:25+09:00
ゼロから始めるLeetCode Day31「581. Shortest Unsorted Continuous Subarray」
概要
海外ではエンジニアの面接においてコーディングテストというものが行われるらしく、多くの場合、特定の関数やクラスをお題に沿って実装するという物がメインである。
その対策としてLeetCodeなるサイトで対策を行うようだ。
早い話が本場でも行われているようなコーディングテストに耐えうるようなアルゴリズム力を鍛えるサイト。
せっかくだし人並みのアルゴリズム力くらいは持っておいた方がいいだろうということで不定期に問題を解いてその時に考えたやり方をメモ的に書いていこうかと思います。
前回
ゼロから始めるLeetCode Day30「234. Palindrome Linked List」基本的にeasyのacceptanceが高い順から解いていこうかと思います。
Twitterやってます。
問題
581. Shortest Unsorted Continuous Subarray
難易度はeasy。
Top 100 Liked Questionsからの抜粋です。問題としては、整数の配列が与えられます。
この配列を昇順で並べ替えると、配列全体を昇順で並べ替えられる連続したサブ配列を見つけてください。
それらの中で最短のサブの配列を見つけ、その長さを返してください、という問題です。例をみながら考えてみましょう。
Example 1:
Input: [2, 6, 4, 8, 10, 9, 15]
Output: 5
Explanation: You need to sort [6, 4, 8, 10, 9] in ascending order to make the whole array sorted in ascending order.この中で並べ替えれば配列全体が昇順に整頓されるサブ配列の中最短のものは
6から9までですので、その要素数を取得して5を返しています。解法
ソートした変数と最初から要素を取得する変数
startと最後から要素を取得する変数lastを用意し、仮にソート済みの配列と元の配列の値が一致しないならばその要素のインデックスをstartに代入します。それをlastの方でも行い、その差分+1が求められている要素数の長さになります。最後に、
last-startが成り立つならばlast - start + 1を、それ以外は0を返すという関数を実装しました。class Solution: def findUnsortedSubarray(self, nums: List[int]) -> int: start = last = 0 num = sorted(nums) for i in range(len(nums)): if nums[i] != num[i]: start = i break for i in range(len(nums)-1,-1,-1): if nums[i] != num[i]: last = i break return last - start + 1 if last - start else 0 # Runtime: 196 ms, faster than 98.85% of Python3 online submissions for Shortest Unsorted Continuous Subarray. # Memory Usage: 15.2 MB, less than 5.00% of Python3 online submissions for Shortest Unsorted Continuous Subarray.かなり単純な考え方でしたが思ったより良さげですね。
Top Liked Questionsのeasyもかなり少なくなってきたので今後はMediumばかりになる気がします。Hardに関しては、ちゃんと解けるか分かりませんが機会があれば解きます。
- 投稿日:2020-05-20T23:08:29+09:00
競プロ用二分探索まとめ
いつ使うのか
N個の要素に対して繰り返し大小判定をし、
- 条件を満たす最大/最小の値を求めるとき。
- 条件を満たす要素を数え上げるとき。
- ソートしたリストのK番目を求めるとき。(リストのK番目の数字以下の数字の個数<=Kを満たす最大の値)
何がいいのか
通常、一個一個見ていくとO(N)のオーダーがかかるところをO(logN)に短縮することができる。
どうするのか
- 予めN個の要素をソートしておく。
- ソートした全要素の両端より
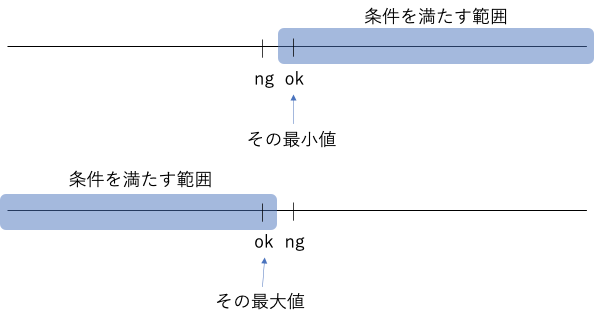
外側の値を2つ選ぶ。(=初期値を両端としてしまうとその点は評価されないため全てTrue/Falseだった場合に不正になる)- 次の操作を繰り返し、最終的には条件を満たすokと満たさないngの境界を求める。
- 2値の平均値(中点)が条件を満たすか判定し、満たすならokをその中点の値とする。満たさないならngを中点とする。
- okとngの絶対値の差が1未満になったら繰り返しを終了する。(=境界が求まった)
イメージ
考えること
ソートしたリストを条件によって篩にかけた時にどちら側がTrue/Falseになるか。
テンプレート
条件判定def is_ok(mid: int): # 検索対象の外にあるものは先に弾く(リストのインデックスなんかだとout of rangeするので) if mid < 0: return True | False # ※1 elif mid >= N: return True | False return True | False #判定条件の結果※1 : 最終的なリストの状態の左側を指定。
*True ----||---- False→True
*False ----||---- True→False探索実行部分def binSearch(ok: int, ng: int): #print(ok, ng) # はじめの2値の状態 while abs(ok - ng) > 1: # 終了条件(差が1となり境界を見つけた時) mid = (ok + ng) // 2 if is_ok(mid): ok = mid # midが条件を満たすならmidまではokなのでokの方を真ん中まで持っていく else: ng = mid # midが条件を満たさないならmidまではngなのでngの方を真ん中まで持っていく #print(ok, ng) # 半分に切り分ける毎の2値の状態 return ok # 取り出すのは基本的にokの方。(問題によりけり)実行# 探索範囲が0 ~ Nまでの場合。 INF = N + 1 binSearch(-1, INF)
- 実行する時は、両端より±1した場所から始める(なお、ここはどれだけ外の値を入れても問題ない)
- 呼び出す時はokとngに対応した引数の順で入れること。(あたりまえ)
使用例
1 条件を満たす最大の値を求めるシンプルなパターン
INF = 10 ** 9 + 1 def main(): A, B, X = map(int, input().split()) # True - ng - ok - False def is_ok(mid: int): # 判定する条件 if mid < 0: return True elif mid >= INF: return False return A * mid + B * len(str(mid)) <= X def binSearch(ok: int, ng: int): #print(ok, ng) while abs(ok - ng) > 1: mid = (ok + ng) // 2 if is_ok(mid): ok = mid else: ng = mid #print(ok, ng) return ok print(binSearch(0, INF)) return2 条件を満たすものを数え上げるパターン(閾値以上/以下の両方をそれぞれ二分探索)
※ ポイント:3つの変動値がある時には中央を固定することでパターン数を抑えられる。
def solve(N: int, A: "List[int]", B: "List[int]", C: "List[int]"): # 最大値 + 1を設定 INF = N + 1 A.sort() B.sort() C.sort() # True - ok - ng - False def is_ok(mid: int, b: int): # 判定する条件 if mid < 0: return True elif mid >= N: return False return A[mid] < b # False - ng - ok - True def is_ok2(mid: int, b: int): # 判定する条件 if mid < 0: return False elif mid >= N: return True return C[mid] > b def binSearch(ok: int, ng: int, b: int): #print(ok, ng) while abs(ok - ng) > 1: mid = (ok + ng) // 2 if is_ok(mid, b): ok = mid else: ng = mid #print(ok, ng) return ok def binSearch2(ok: int, ng: int, b: int): #print(ok, ng) while abs(ok - ng) > 1: mid = (ok + ng) // 2 if is_ok2(mid, b): ok = mid else: ng = mid #print(ok, ng) return ok sum = 0 for b in B: a = binSearch(-1, INF, b) - 0 + 1 c = N - 1 - binSearch2(INF, -1, b) + 1 sum += a * c #print(b, "->", a, c) print(sum) return3 ソートしたリストのK番目を求めるとき。(K番目の数字X以下の個数がK個以上を満たす最小のX)
より厳密には「一列に並べた時にK番目を求める問題。ただし膨大すぎて全ての要素を列挙できないとき。」
この問題はN^2個の数をソートしてK番目を求めればいいが最大 9 * 10^8 個はTLEする。
そこで問題の捉え方を変える。
K番目の数字Xを求める。= その数字X以下の個数がK個ある。
積X以下の個数がK個以上かどうかを条件として二分探索。1,1,2,2,2,2,2,4,4 の5番目(K = 5)を求める時。
X = 1では2個 ( < 5 ) →ダメ
X = 2では7個 ( >= 5 ) →ok5番目は2とわかる。
INF = 10 ** 18 + 1 def main(): N, K = map(int, input().split()) A = sorted([int(i) for i in input().split()]) B = sorted([int(i) for i in input().split()]) # False - ng - ok - True def is_ok(mid: int): # 判定する条件 if mid < 0: return False elif mid >= INF: return True count = 0 for a in A: maxb = mid // a # 各aに対して積を取った時に値mid以下となる最大のbの値 count += bisect_right(B, maxb) # ソート済みのBからそのインデックスを求めれば、そこより手前のbは全てaとの積がn以下のbとなり個数がでる。 return count >= K def binSearch(ok: int, ng: int): #print(ok, ng) while abs(ok - ng) > 1: mid = (ok + ng) // 2 if is_ok(mid): ok = mid else: ng = mid #print(ok, ng) return ok print(binSearch(INF, 0)) return参考
- 投稿日:2020-05-20T23:08:02+09:00
AtomでAnaconda Promptを使いながらコマンドマクロを使いたいんだ!
0.概要
ここでは,エディタのAtomにplatformio-ide-terminalパッケージをインストールしAtom上でターミナル操作を行う際に,『デフォルト端末をAnaconda Promptにする』のと『コマンドマクロファイルを起動時に自動で書き込む』のを,同時に実施するための解決方法をまとめています.
1.背景
Atomは様々なパッケージをインストールすることで,自分好みに環境をカスタマイズできる非常に自由度の高いエディタです.自分もまだ使い始めて間もないですが,すでにその使いやすさの虜になっています.
このパッケージの中で,特に気に入っているものが,platformio-ide-terminalです.こいつのおかげで,いままで煩雑だった,ターミナルとエディタ間の切り替えが無くなり,すべてエディタ上の操作で完結させることができます.
これから構築しようとしている開発環境ではワケあってAnacondaの利用が必須であったため,下記参照ページの設定方法に従い,Atom上でのデフォルト端末をAnaconda Promptに設定しました.
参照URL:https://qiita.com/daizutabi/items/6e4e7042b764b61adf27
2.環境
OS:Windows10 Enterprise 1809 64bit
Atom:1.47.0
platformio-ide-terminal 2.10.0
Anaconda Navigater 1.9.123.困ったこと
さて,ここで困ったことが発生しました.というのもここ最近,Linux OSの環境で端末操作を行うことも増えてきたこともあり,コマンドプロンプト上では,Linuxライクな端末コマンドが使えるように設定したコマンドマクロファイルを活用していました.(コマンドマクロについては手前味噌ですが以下のページをご参照ください.)
参照;https://qiita.com/titanium0715/items/8bbad95f939017302d38
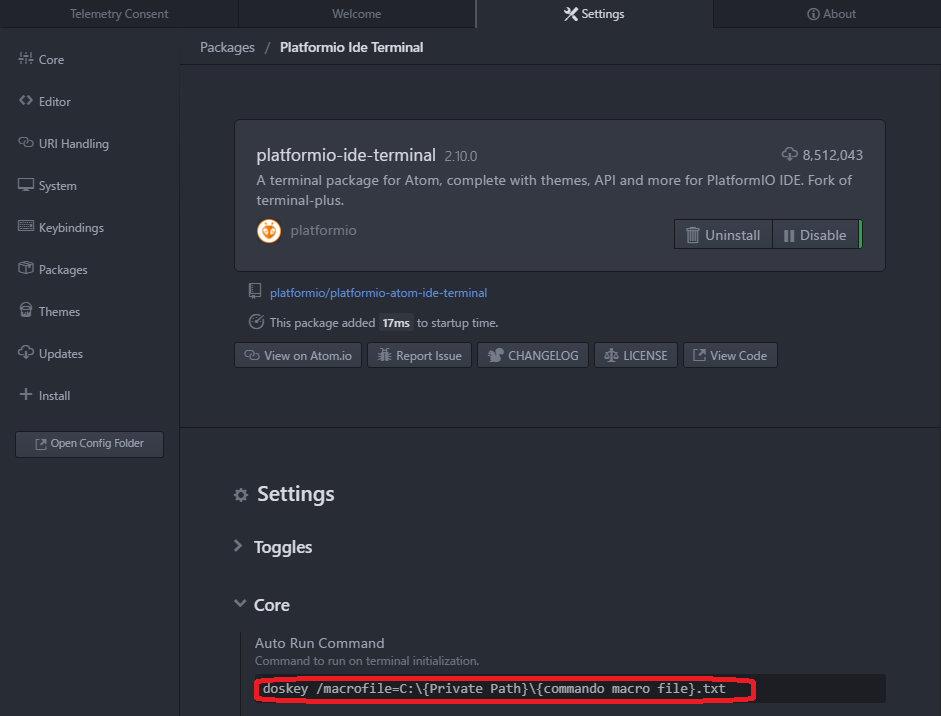
上記参照ページでは,コマンドプロンプトのショートカットのプロパティ上で,kオプションを追加することで対応していました.ところが,Atomではショートカットを介さずに直接cmd.exeを読み込みに行ってしまうため,上記解決方法を使用することはできません.なのでこれを解決するためには,platformio-ide-terminalのsettingより,Auto Run Commandに①
doskey /macrofile=C:\{Private Path}\{コマンドマクロファイル}.txtを設定してやる必要があります.しかし,1.背景に挙げた参照URLにもありますように,Atom上でAnaconda Promptを動かそうとするには,Auto Run Commandには②
{Private Path}\{miniconda or Anaconda}\Scripts\activate.bat rootを記載する必要があります.Atom上で,Anaconda Promptを使いながら,マクロファイルの設定も行いたい・・・.じゃあ①と②を二つとも書けばいいじゃん!って思ったのですが,単純に横に並べただけでは,Atom上で端末が立ち上がらなくなったり,どちらか一方の指示だけが通ってしまっていたりと,うまくいきませんでした.
4.解決方法
いろいろと試した結果,以下の書き方でAuto Run Commandを記載してやれば,どちらの設定も反映させることができました!
doskey /macrofile=C:{Private Path 1}{コマンドマクロファイル}.txt & C:{Private Path 1}\Anaconda3\Scripts\activate.bat root
ポイントは,『二つのコマンドを"&"でつなぐこと』,あとは『①⇒②の順番で書く』というところです.順番についてはかなり悩まされまして,②⇒①の順番で書いていたときは何をやっても適切な設定が行えてませんでした.以後,要注意です.
バッチファイルの文法とかが分かっていれば,こんな苦労はしなかったのかもしれないなぁ.
- 投稿日:2020-05-20T23:08:02+09:00
AtomでAnaconda Promptを使いながらコマンドマクロファイル自動書き込みをやりたいんだ!
0.概要
ここでは,platformio-ide-terminalパッケージを用いてAtom上でターミナル操作を行う際に,『デフォルト端末をAnaconda Promptにする』のと『コマンドマクロファイルを端末起動時に自動で書き込む』のを,同時に実施するための解決方法をまとめています.
1.背景
Atomは様々なパッケージをインストールすることで,自分好みに環境をカスタマイズできる非常に自由度の高いエディタです.自分もまだ使い始めて間もないですが,すでにその使いやすさの虜になっています.
このパッケージの中で,特に気に入っているものが,platformio-ide-terminalです.こいつのおかげで,いままで煩雑だった,ターミナルとエディタ間の切り替えが無くなり,すべてエディタ上の操作で完結させることができます.
これから構築しようとしている開発環境ではワケあってAnacondaの利用が必須であったため,下記参照ページの設定方法に従い,Atom上でのデフォルト端末をAnaconda Promptに設定しました.
参照URL:https://qiita.com/daizutabi/items/6e4e7042b764b61adf27
2.環境
OS:Windows10 Enterprise 1809 64bit
Atom:1.47.0
platformio-ide-terminal 2.10.0
Anaconda Navigater 1.9.123.困ったこと
さて,ここで困ったことが発生しました.というのもここ最近,Linux OSの環境で端末操作を行うことも増えてきたこともあり,コマンドプロンプト上では,Linuxライクな端末コマンドが使えるように設定したコマンドマクロファイルを活用していました.(コマンドマクロについては手前味噌ですが以下のページをご参照ください.)
参照;https://qiita.com/titanium0715/items/8bbad95f939017302d38
上記参照ページでは,コマンドプロンプトのショートカットのプロパティ上で,kオプションを追加することで対応していました.ところが,Atomではショートカットを介さずに直接cmd.exeを読み込みに行ってしまうため,上記解決方法を使用することはできません.なのでこれを解決するためには,platformio-ide-terminalのsettingより,Auto Run Commandに①
doskey /macrofile=C:\{Private Path}\{コマンドマクロファイル}.txtを設定してやる必要があります.しかし,1.背景に挙げた参照URLにもありますように,Atom上でAnaconda Promptを動かそうとするには,Auto Run Commandには②
{Private Path}\{miniconda or Anaconda}\Scripts\activate.bat rootを記載する必要があります.Atom上で,Anaconda Promptを使いながら,マクロファイルの設定も行いたい・・・.じゃあ①と②を二つとも書けばいいじゃん!って思ったのですが,単純に横に並べただけでは,Atom上で端末が立ち上がらなくなったり,どちらか一方の指示だけが通ってしまっていたりと,うまくいきませんでした.
4.解決方法
いろいろと試した結果,以下の書き方でAuto Run Commandを記載してやれば,どちらの設定も反映させることができました!
doskey /macrofile=C:{Private Path 1}{コマンドマクロファイル}.txt & C:{Private Path 1}\Anaconda3\Scripts\activate.bat root
ポイントは,『二つのコマンドを"&"でつなぐこと』,あとは『①⇒②の順番で書く』というところです.順番についてはかなり悩まされまして,②⇒①の順番で書いていたときは何をやっても適切な設定が行えてませんでした.以後,要注意です.
バッチファイルの文法とかが分かっていれば,こんな苦労はしなかったのかもしれないなぁ.
- 投稿日:2020-05-20T23:05:19+09:00
【記録用】Pandas備忘録
今回はPandasを使った際に、すぐに分からなかった事象のまとめです。
結構Pandasってどうやって操作すればいいのかな?って迷うことも多いので少しでも参考になれば幸いです。使用するデータセット作成
- まずは適当にデータセットを作成する。本記事はこの簡単なデータを例に書いていく
index = ['製品A', '製品B', '製品C'] columns = ['春', '夏', '秋', '冬'] data = np.array([ [0,10,20,30], [0,0,100,20], [50,100,20,40], ]) df = pd.DataFrame(data, index=index, columns=columns) df.head()
春 夏 秋 冬 製品A 0 10 20 30 製品B 0 0 100 20 製品C 50 100 20 40 ケース①各行の最大値,最小値に対応しているカラム名、そしてその値を抜き出して追加
- つまり製品Aなら冬が一番大きく製品Cなら夏が一番大きい数字なので、新しいカラム「売れる季節」にその季節を挿入する
for index, row in df.iterrows(): # df.ix[index, '売れる季節'] = row.argmax() df.ix[index, '売れる季節'] = row.idxmax() #どっちでもできる # df.ix[index, '売れない季節'] = row.argmin() df.ix[index, '売れない季節'] = row.idxmin() #どっちでもできる df.ix[index, 'MAX'] = row.max() df.ix[index, 'MIN'] = row.min() df.head()
春 夏 秋 冬 売れる季節 売れない季節 MAX MIN 製品A 0 10 20 30 冬 春 30 0 製品B 0 0 100 20 秋 春 100 0 製品C 50 100 20 40 夏 秋 100 20 ケース②各行で最初に正の整数になるカラム名を抜き出して追加
- つまり各製品が初めて売れ始めた季節を出したい。Aなら夏、Bなら秋という感じ
for index, row in df.iterrows(): #index[0]で最初に条件が成立したものを取り出す df.ix[index, '売れ始め季節'] = row[row > 0].index[0] df.head()
春 夏 秋 冬 売れ始め季節 製品A 0 10 20 30 夏 製品B 0 0 100 20 秋 製品C 50 100 20 40 春 ケース③0が1つも入ってない行を抜き出したい
- つまり年中売れている製品Cの行を抜き出したい
#not_zero_df = df.query('春 > 0 and 夏 > 0 and 秋 > 0 and 冬 > 0') #not_zero_df = df.query('春 != 0 and 夏 != 0 and 秋 != 0 and 冬 != 0') # ↑でもいいが変数として外だしも可能。下で「@」をつければいい hoge1,hoge2,hoge3,hoge4 = 0,0,0,0 not_zero_df = df.query('春 > @hoge1 and 夏 > @hoge2 and 秋 > @hoge3 and 冬 > @hoge4') not_zero_df.head()
春 夏 秋 冬 製品C 50 100 20 40
- ちなみに夏のみに着目して0以外を抽出したい場合は同じくこう書けばいい(夏が0でない行を取り出す)
hoge = 0 not_zero_df = df.query('夏 != @hoge') not_zero_df.head()
春 夏 秋 冬 製品A 0 10 20 30 製品C 50 100 20 30
今後も随時追記していく予定です
- 投稿日:2020-05-20T22:53:49+09:00
jupyter Nteractをherokuサーバーで立ち上げてみた
きっかけ
flask, herokuを利用し、ちまちまとテキストマイニング・可視化webアプリを作っていた。
ある日、Nteractを知る。Nteract
https://github.com/nteract/nteract
https://blog.nteract.io/designing-the-nteract-data-explorer-f4476d53f897
自動インタラクティブ可視化機能がついたjupyter notebookだとおもいねぇ。「おう、pandasのDataFrameを表示するのと同じ手間で、これだけ可視化できるのか。
これは使ってみるしかあるまい。
・・・重いデータを使うと落ちるな・・・
いくらか弱点はありそうだが、webアプリとできれば、ハードを選ばずどこからでもやりたいときに速やかに可視化できるようになる。制限の多いGoogle Colaboratoryよりある意味便利かな。」というわけで、heroku上でNteractを稼働させてみた。
*なぜかNteractの情報はあまりないのよね・・・jupyterの機能拡張でインタラクティブな可視化が簡単にできるようになっていたりするのかな?
構成
heroku: アプリケーションサーバーとして
Nteract: heroku上で起動させる
github private: ファイルサーバーとしてコード
- 次のファイルを作成しデプロイ。heroku openまたは指定されたアドレスを開けば動く。
- 注意:パスワードなどセキュリティ関連の処理は除いています。足すことを強くお勧めします。
- 注意:githubのpriveteをファイルサーバー代わりとしていますが、その説明は省きます。
フォルダ構成
xxx-xxx (任意)
┣ Procfile
┣ requirements.txt
┣ start_jupyter
┗ (任意のipynbファイルなど)requirements.txt 要事増減させてくださいgunicorn==19.9.0 click==7.1.1 Flask==1.1.2 itsdangerous==1.1.0 Jinja2==2.11.2 MarkupSafe==1.1.1 numpy==1.18.3 pandas==1.0.3 plotly==4.6.0 python-dateutil==2.8.1 pytz==2019.3 retrying==1.3.3 six==1.14.0 Werkzeug==1.0.1 xlrd==1.2.0 nteract_on_jupyter matplotlib PyGithubProcfileweb: chmod +x start_jupyter ; ./start_jupyterstart_jupyter#!/usr/bin/env bash jupyter nteract --no-browser --ip=* --port=$PORT$ git init $ heroku create xxx-xxx $ git add . $ git commit -m "first" $ git push heroku master稼働例
参照
要事、mecabなど入れておいてもよいでしょう。
HerokuにMeCabを入れる際ハマっていた記録
https://qiita.com/kzuzuo/items/1b3e9c9af57bd4464690
- 投稿日:2020-05-20T22:53:49+09:00
jupyter nteractをherokuサーバーで立ち上げてみた
きっかけ
flask, herokuを利用し、ちまちまとテキストマイニング・可視化webアプリを作っていた。
ある日、nteractを知る。nteract
https://github.com/nteract/nteract
https://blog.nteract.io/designing-the-nteract-data-explorer-f4476d53f897
自動インタラクティブ可視化機能がついたjupyter notebookだとおもいねぇ。「おう、pandasのDataFrameを表示するのと同じ手間で、これだけ可視化できるのか。
これは使ってみるしかあるまい。
・・・重いデータを使うと落ちるな・・・
いくらか弱点はありそうだが、webアプリとできれば、ハードを選ばずどこからでもやりたいときに速やかに可視化できるようになる。制限の多いGoogle Colaboratoryよりある意味便利かな。」というわけで、heroku上でnteractを稼働させてみた。
参照
*なぜかnteractの情報はあまりないのよね・・・
Pythonを使った機械学習の勉強にはJupyter NotebookをHerokuの無料枠で立ててスマホからでも実行できるようにして共有しよう
https://qiita.com/G-awa/items/8530a10cb847e4080df3Deploy a Jupyter Notebook Online with Voila and Heroku
https://pythonforundergradengineers.com/deploy-jupyter-notebook-voila-heroku.html構成
heroku: アプリケーションサーバーとして
nteract: heroku上で起動させる
github private: ファイルサーバーとしてコード
- 次のファイルを作成しデプロイ。heroku openまたは指定されたアドレスを開けば動く。
- 注意:パスワードなどセキュリティ関連の処理は除いています。足すことを強くお勧めします。
- 注意:githubのpriveteをファイルサーバー代わりとしていますが、その説明は省きます。
フォルダ構成
xxxxxx (任意)
┣ Procfile
┣ requirements.txt
┣ start_jupyter
┗ (任意のipynbファイルなど)requirements.txt 要事増減させてくださいgunicorn==19.9.0 click==7.1.1 Flask==1.1.2 itsdangerous==1.1.0 Jinja2==2.11.2 MarkupSafe==1.1.1 numpy==1.18.3 pandas==1.0.3 plotly==4.6.0 python-dateutil==2.8.1 pytz==2019.3 retrying==1.3.3 six==1.14.0 Werkzeug==1.0.1 xlrd==1.2.0 nteract_on_jupyter matplotlib PyGithubProcfileweb: chmod +x start_jupyter ; start_jupyterstart_jupyter#!/usr/bin/env bash jupyter nteract --no-browser --ip=* --port=$PORT$ cd xxxxxx $ git init $ heroku create xxxxxx $ git add . $ git commit -m "first" $ git push heroku master稼働例
参考
要事、mecabなど入れておいてもよいでしょう。
HerokuにMeCabを入れる際ハマっていた記録
https://qiita.com/kzuzuo/items/1b3e9c9af57bd4464690次、nteractベースに変更しようか・・・
特許など比較的長文の文章間 類似可視化手法: tfidf/cluster vis: tfidf-word2vec-clustering visualization
https://qiita.com/kzuzuo/items/8a80d8974bf3a7db7e54
- 投稿日:2020-05-20T22:12:31+09:00
alpineでC言語依存モジュールを pip install した時の時間を計測してみた
Let's 計測
alpineでC言語依存モジュールを pip install すると激重になる話 のおまけ
素直にpip installした場合に、どれくらい時間がかかるか試してみた。
手元にあった機械学習用のrequirements.txtで実行してみると以下のようなログがでてきたのでBuilding wheels for collected packages: backcall, h5py, kiwisolver, matplotlib, numpy, pandas, pathspec, Pillow, PyYAML, pyzmq, regex, retrying, scikit-learn, scipy, tornado, typed-ast開発時にも使用頻度高そうなモジュールだけピックアップして計測することにした。
- 実行してみるリスト
- numpy
- pandas
- Pillow
- scipy
- scikit-learn
- h5py
- matplotlib
- regex
実験環境
手元で実行できる環境が二種類あったので両方とも試してみる。
- case1: MacBookAir 2015 13inch intel-corei5 8GB DDR3 ・ docker desktop
- case2: AMD Ryzen9 3900X, M.2 ASM2NE6500GTTD, 8x2GB DDR4 ・ ubuntu18.04(WSL2 integration)
計測方法
timeコマンドを頭につける事で実行時間を計測する。
どちらのケースも同一のdockerfileで実行しており、RUN命令文中にtime pip install hogeを埋め込む形を取っている。FROM python:3.8-alpine3.11 RUN apk update \ && apk add --virtual .build --no-cache openblas-dev lapack-dev freetype-dev \ gfortran libxml2 g++ gcc zip unzip cmake make \ libpng-dev openssl-dev musl libffi-dev python3-dev libxslt-dev \ libxml2-dev jpeg-dev \ && apk add --no-cache -X http://dl-cdn.alpinelinux.org/alpine/edge/testing hdf5-dev RUN pip install --upgrade --no-cache-dir pip setuptools wheel && \ time pip install --no-cache-dir Cython && \ ...こんな感じ。
docker build中に出力されるログをまとめたのが次の表となる。結果
※ Cythonはalpineでもコンパイル不要のモジュールであり、
Cython-0.29.16-py2.py3-none-any.whlのような.whl形式で降ってきます。非依存モジュールとの比較としてここに載せています。1
module / time case1 case2 real user sys real user sys Cython 3.07s 2.33s 0.42s 1.52s 0.99s 0.07s numpy 5m 28.27s 7m 6.90s 22.11s 2m 11.15s 3m 23.69s 6.05s pandas 31m 46.58s 30m 36.25s 0m 53.83s 14m 8.24s 13m 53.10s 15.88s Pillow 50.81s 44.09s 5.99s 24.79s 19.88s 1.69s scipy 30m 45.99s 36m 29.33s 1m 45.89s 12m 52.81s 17m 54.87s 42.58s scikit-learn 14m 38.63s 14m 3.37s 28.98s 6m 33.10s 6m 24.84s 10.11s h5py 3m 45.58s 3m 34.79s 9.30s 1m 45.87s 1m 42.42s 4.18s matplotlib 2m 51.50s 2m 35.53s 13.59s 1m 21.77s 1m 13.70s 6.75s regex 30.52s 28.84s 1.24s 13.75s 13.06s 0.34s おっっっっそい
ものっっっっっそい、おっっっっっっそいcase1だとnumpyだけでも5分かかるし、pandas・scipyに至っては30分を超えている。scikit-learnに至ってはnumpy・scipyに依存しているので実際のところ一時間は覚悟しないといけない。
挙げた全部合わせると一時間半超だ。
これがどういう事になるか。一日8時間作業すると仮定すると一日たったの5回程度しかデプロイする事ができないことになる
気軽に開発環境に最新ブランチを充てたり、軽微なバグ修正の確認すら困難になる。作業時間も含めると更に減る。
一応、メモリが倍になると処理速度も倍になるので時間の短縮はできなくもない。
awsを使ったりしているのならメモリ量が多いインスタンスタイプを選択することで時短は可能だ。
しかし、性能的にcase1の二倍以上のcase2だと半分以下の時間で済むとはいえ、全部で40分ほどかかっているのは事実。
一日あたり最高で12回ほどのデプロイしかできない。
ちなみに、表の上から順番に
pip installしていったので一部モジュールの実行時間には依存モジュールのインストール実行時間も含まれる。
kiwisolverもtar.gzで降ってきているようなのでmatplotlib単体はもう少し早いはず。Successfully built pandas Installing collected packages: six, python-dateutil, pytz, pandas Successfully installed pandas-0.25.3 python-dateutil-2.8.1 pytz-2019.3 six-1.14.0 ------ Successfully built scikit-learn Installing collected packages: joblib, scikit-learn Successfully installed joblib-0.14.1 scikit-learn-0.22.2.post1 ----- Successfully built matplotlib kiwisolver Installing collected packages: cycler, kiwisolver, pyparsing, matplotlib Successfully installed cycler-0.10.0 kiwisolver-1.2.0 matplotlib-3.2.1まとめ
軽いものでも1個あたり1~2分かかるレベルなので、なるべくC言語依存モジュールはビルドしないで済む運用を考える必要がある。
condaを使う、は今無しで余談pip
pip install時はモジュールのwheel化が完了した直後に、再度whlを展開し直して必要な生成物をまるっとsite-package直下に移動する。pythonで書かれたコードからビルドされた共有ライブラリ郡*.soも含めて全てだ。
alpineのようにpython3.8のバイナリが/usr/local/bin直下にいるとすると/usr/local/lib/python3.8/site-packages/に移動する。再展開は手間のように見えるがこれはPEPに則った安全なモジュールの導入法である。PEP491にもそう書かれている2
試しに、ビルド済みのwhlファイルを
unzipして.soファイルを検索してみる。./numpy/linalg/lapack_lite.cpython-38-x86_64-linux-gnu.so ./numpy/linalg/_umath_linalg.cpython-38-x86_64-linux-gnu.so ./numpy/core/_operand_flag_tests.cpython-38-x86_64-linux-gnu.so ...それぞれのwhlファイルを
pip installで導入後にライブラリパスに対して.soファイルを検索してみると、モジュール毎にビルド済みファイルが存在しているのを確認できる。/usr/local/lib/python3.8/site-packages/numpy/linalg/lapack_lite.cpython-38-x86_64-linux-gnu.so /usr/local/lib/python3.8/site-packages/numpy/linalg/_umath_linalg.cpython-38-x86_64-linux-gnu.so /usr/local/lib/python3.8/site-packages/numpy/core/_operand_flag_tests.cpython-38-x86_64-linux-gnu.so ... /usr/local/lib/python3.8/site-packages/pandas/io/sas/_sas.cpython-38-x86_64-linux-gnu.so /usr/local/lib/python3.8/site-packages/pandas/_libs/hashtable.cpython-38-x86_64-linux-gnu.so ...これでようやくpythonでimportできる訳だが、
裏を返せばpythonがモジュールを検索するパスに生成物さえ居れば使用できる。
なので元記事の回避法では、python関連以外は後に入れること前提で/usr/local/直下をdockerのマルチステージビルドを使ってえいやでコピペしている。参考文献
- Wheel files : What is the meaning of “none-any” in protobuf-3.4.0-py2.py3-none-any.whl
- PEP 425 -- Compatibility Tags for Built Distributions
- PEP 491 -- The Wheel Binary Package Format 1.9
- wheelのありがたさとAnacondaへの要望
- wheel・PEP関連ですごく参考にしました。
- ちなみに現在だと
conda skeletonでPyPiのwheelを導入できる
- ただし、conda独自にビルドし直して導入している模様。個人的には ソウジャナイ感
- anacondaにsentencepieceをwheelファイル経由でインストールする
- 投稿日:2020-05-20T21:50:50+09:00
pip install のエラーの解決策 【Python】【Mac】
pythonエンジニアで初心者の自分が困ってて解決したことをシェアします。
pip install のエラー
pip is configured with locations that require TLS/SSL, however the ssl module in Python is not available. Retrying (Retry(total=0, connect=None, read=None, redirect=None, status=None)) after connection broken by 'SSLError("Can't connect to HTTPS URL because the SSL module is not available.")': /simple/pip/などのエラーが出た時の対処法
根本的な原因が全く分からないので、とりあえずエラー文章やsslとは、など初めて見る単語を調べてました。解決策自体はいろいろあるみたいです。
-環境-
macOS 10.14.6
python 3.6.6
venv(仮想環境)原因
おそらくpip/pythonが古いopenssl(1.0.2s)を参照しているのではありませんか? 情報を見る限り、質問者の方のopensslは最新版になっているので参照エラーが起きているように思います
下記でsslが使えるかチェックします。
pyimport sslTraceback (most recent call last): File "<stdin>", line 1, in <module> File "/Users/[username]/.pyenv/versions/3.7.0/lib/python3.7/ssl.py", line 98, in <module> import _ssl # if we can't import it, let the error propagate ImportError: dlopen(/Users/[username]/.pyenv/versions/3.7.0/lib/python3.7/lib-dynload/_ssl.cpython-37m-darwin.so, 2): Library not loaded: /usr/local/opt/openssl/lib/libssl.1.0.0.dylib Referenced from: /Users/LO/.pyenv/versions/3.7.0/lib/python3.7/lib-dynload/_ssl.cpython-37m-darwin.so Reason: image not foundエラーが出たので
結論
sh$ brew switch openssl 1.0.2spip install でSSL関連のエラーが出たらこれを読め!!!
これだけです。
sslが切り替わってなかったんですね。
解決してみると原因はシンプルで分かりやすかったです。
自分なりに解釈してるところが、あるかもしれませんが助けになれば幸いです。
- 投稿日:2020-05-20T21:39:20+09:00
ダイクストラ法(Dijkstra's Algorithm)をpythonで実装する
やったこと
ダイクストラ法についての勉強を兼ねて、ダイクストラ法をpythonで実装してみた。
例題としては、「Dijkstra AtCoder」でググって最初に出てきた「AtCoder Beginner Contest 035 の D -トレジャーハント」を用いた。ダイクストラ法とDFSやBFSの違い
ダイクストラ法とDFS、BFSの違いがよくわからなくなったので、調べた結果を簡単にまとめる。
(DFS、BFSについては下記で実装)
深さ優先探索(DFS)と幅優先探索(BFS)をpythonで実装する
- DFS(深さ優先探索)・BFS(幅優先探索)
- 重みのないグラフについての探索を行うためのアルゴリズム
- ダイクストラ法
- 重み付きグラフについての探索を行うためのアルゴリズム(キュー(正確には重み付きキュー)を用いるため、BFSの拡張版のイメージ)
Dijkstra's Algorithm
メモ
start→目的地に着くまでの最短経路を
g_list,go
目的地→startに着くまでの最短経路をb_list,backで管理# ABC035 D トレジャーハント import heapq def dijkstra(s, n, c_list): _list = [float("Inf")]*n _list[s] = 0 hq = [[0,s]] heapq.heapify(hq) while len(hq) > 0: _ci, _i = heapq.heappop(hq) if _list[_i] < _ci: continue for _cj,_j in c_list[_i]: if _list[_j] > (_list[_i] + _cj): _list[_j] = _list[_i] + _cj heapq.heappush(hq, [_list[_j],_j]) return _list n, m, t = map(int, input().split()) a_list = [int(x) for x in input().split()] g_list = [[] for i in range(n)] b_list = [[] for i in range(n)] for i in range(m): _a, _b, _c = map(int, input().split()) g_list[_a-1].append([_c,_b-1]) b_list[_b-1].append([_c,_a-1]) go = dijkstra(0, n, g_list) back = dijkstra(0, n, b_list) ans = 0 for i in range(n): _tmp = t - (go[i] + back[i]) if a_list[i]*_tmp > ans: ans = a_list[i]*_tmp print(ans)
- 投稿日:2020-05-20T21:23:03+09:00
[numpy][scipy]対角行列クロネッカー積パフォーマンス天下一武闘会
クロネッカー積
Wikipediaを見よ。
ざっくりいうと、2つの行列やベクトルの各要素の組み合わせを作っていく計算で、AxB行列とCxD行列のクロネッカー積はACxBD行列になって、繰り返しでクロネッカー積を取っていけばゴリゴリと行列が巨大になっていきます。何に使うの? って感じですが、量子コンピュータをシミュレーションする上ではかなり使います。
複数の系をつなげて、ひとつの系として見るには、2つの量子状態のクロネッカー積をとることになるからです。戦いに挑むプレイヤー
彼らで、パフォーマンス天下一武闘会をやっていきます。
プレイヤー 説明 普通の2次元配列 (np.array) ぼくら大好きnumpyの2次元配列 対角要素だけの1次元配列(np.array) 対角行列同士のクロネッカー積は対角行列なので、対角要素だけ1次元配列で取り出せばよくないですか? 1次元配列(np.array) + scipy.linalg.kronnumpyに np.kron関数が用意されているんですが、scipyにも似た関数があるので、そっちも使ってみましたscipy.sparseのdia行列本命登場。scipyの疎行列のひとつ。対角圧縮格納行列という形式 scipy.sparseのcoo行列scipyの疎行列のひとつ。座標とデータを格納する方式 scipy.sparseのlil行列scipyの疎行列のひとつ。リストのリストとしてデータを格納 scipy.sparseのdok行列scipyの疎行列のひとつ。dictでデータを格納 疎行列が一杯出てきますね。
「0がいっぱいある行列って、持つの無駄じゃん」ってことで、それをなんとかしたやつが疎行列なんですが。こいつら、種類が多すぎて大変なんですよ。結果
つまり、2x2行列とのクロネッカー積をとっていって、行列の次元を倍々にしてみましょう。
from time import perf_counter from contextlib import contextmanager import numpy as np import scipy.linalg from scipy.sparse import eye, kron, dia_matrix, coo_matrix, lil_matrix, dok_matrix @contextmanager def timer(ls): t0 = perf_counter() yield ls.append(perf_counter() - t0) fmt = ['dia', 'coo', 'lil', 'dok'] mat1 = [np.eye(2), np.ones(2), np.ones(2).reshape((-1, 1))] for f in fmt: mat1.append(eye(2, format=f)) mat2 = [x.copy() for x in mat1] print('n\tdim\tnp.kron (mat)\tnp.kron (diag)\tscipy.linalg.kron\tdia\tcoo\tlil\tdok') for i in range(15): time = [] with timer(time): mat1[0] = np.kron(mat1[0], mat2[0]) with timer(time): mat1[1] = np.kron(mat1[1], mat2[1]) with timer(time): mat1[2] = scipy.linalg.kron(mat1[2], mat2[2]) for j, f in enumerate(fmt, start=3): with timer(time): mat1[j] = kron(mat1[j], mat2[j], format=f) print(i, len(mat1[1]), *time, sep='\t')こんなコードを動かしてみました。
結果、こうなりました
dim np.kron (mat) np.kron (diag) scipy.linalg.kron dia coo lil dok 4 7.621300028404221e-05 4.2740000935737044e-05 1.618100213818252e-05 0.00034239199885632843 0.00010936500621028244 0.00040860599983716384 0.0002625720007927157 8 3.6978999560233206e-05 2.091900387313217e-05 1.4868004654999822e-05 0.00031008200312498957 0.00010704099986469373 0.00037807899934705347 0.0002334279997739941 16 3.546700463630259e-05 1.7302001651842147e-05 1.6571000742260367e-05 0.000312356001813896 0.00011324300430715084 0.0003890799998771399 0.000256961997365579 32 5.516300007002428e-05 2.5807996280491352e-05 2.214199776062742e-05 0.00030120500014163554 0.00010780199954751879 0.0004031159987789579 0.0005835240008309484 64 0.00010105999535880983 2.7360998501535505e-05 2.941599814221263e-05 0.00030793799669481814 0.0001086229967768304 0.00042860399844357744 0.0002643059997353703 128 0.00020814999879803509 4.005499795312062e-05 5.749799311161041e-05 0.00031550200219498947 0.00010875400039367378 0.0005037450027884915 0.00027479499840410426 256 0.0007497959959437139 6.817800021963194e-05 7.954899774631485e-05 0.0003335859946673736 0.00011099899711553007 0.0014838429997325875 0.0003385250020073727 512 0.00291782299609622 0.00013850899995304644 0.00015007200272521004 0.0004066420005983673 0.00011842299863928929 0.000978336000116542 0.00039470100455218926 1024 0.009874209994450212 0.0002527940014260821 0.0002898239981732331 0.0010409530004835688 0.0001297330018132925 0.0016027960009523667 0.0006418839984689839 2048 0.0948937740031397 0.0004355569981271401 0.000531908000994008 0.0004898590050288476 0.00015769599849591032 0.0030208060052245855 0.0009902669989969581 4096 0.4371686599988607 0.0008414180047111586 0.0010229589970549569 0.0006520530005218461 0.00018501699378248304 0.005185828005778603 0.0017490499958512373 8192 1.5510790590051329 0.0017292539996560663 0.0019197299989173189 0.00098355500085745 0.00034314399817958474 0.014461956001468934 0.002987473999382928 16384 5.583846742003516 0.0033475189993623644 0.004184869001619518 0.0011626810010056943 0.0003905220000888221 0.01908096900297096 0.006053636003343854メモリがだいぶつらくなってきました。
要素数が万のオーダーになってくると、2次元配列で持つのがつらくなります。
脱落させて次を見てみましょう。dim np.kron (diag) scipy.linalg.kron dia coo lil dok ## 略 16384 0.003250737994676456 0.0040016369966906495 0.0011192689999006689 0.0003866760016535409 0.01951057599944761 0.005936435001785867 32768 0.006436383002437651 0.008126152002660092 0.00257632300053956 0.0006475750051322393 0.04585514999780571 0.012816850001399871 65536 0.013466698997945059 0.016394651000155136 0.0036876980011584237 0.0012796200026059523 0.09913738199975342 0.027271613995253574 131072 0.027018057997338474 0.03451431899884483 0.007149820994527545 0.002364355997997336 0.20790910699724918 0.07132508500217227 262144 0.054609892998996656 0.07083234099991387 0.013791570003377274 0.004519036003330257 0.4256131450019893 0.15278254599979846 524288 0.11130649899860146 0.14217567000014242 0.029465308994986117 0.008979752994491719 0.8125153309956659 0.3088614529988263 1048576 0.22045852500014007 0.2833445610012859 0.060802528001659084 0.018181282001023646 1.6133216590023949 0.6439245029978338 2097152 0.4423036929947557 0.5692931669982499 0.13147616000060225 0.037687891999667045 3.204140270005155 1.342386914002418 4194304 0.8839434900000924 1.1379078309983015 0.265317548000894 0.07555588000104763 6.303365648003819 2.8236029719992075 8388608 1.7452782509935787 2.242528515998856 0.5512261999974726 0.14783157800411573 12.572135554000852 5.75408922600036 16777216 3.614834731000883 4.505664156000421 1.1048625590046868 0.3020367110002553 25.316714471002342 11.952817262994358lilとdokが厳しくなってきました。
脱落させて、他やっていきましょう。dim np.kron (diag) scipy.linalg.kron dia coo ## 略 16777216 3.6877648160007084 4.646497107998584 1.0930974110015086 0.29926496299594874 33554432 7.348309517001326 9.31613551099872 2.176151612002286 0.591943232997437 67108864 14.420524740002293 18.4003505590008 4.722769480998977 1.2042434280010639多分、numpy.kronがいちばんシンプルなことやるだけで済むはずなんですが、遅いです。
あと、scipy.linalg.kronも、numpy.kronとの違いがわからないので使ってみましたが、あんまりパフォーマンス上のメリットはなさそうです。もう結果は見えてきてますが、決勝戦やりましょう。
diaは対角圧縮という名前がついていて、対角行列に強いんじゃないかと期待したんですが、残念ながら勝てるか怪しいですね。dim dia coo ## 略 67108864 4.889362873000209 1.2848818929996924 134217728 11.382003072998486 2.5668652079984895時間はもう少し頑張れそうですが、メモリが厳しくなってきました。
cooがだいぶ早いです。優勝: coo行列
coo行列がだいぶ速い、という結果になりました。
正直、理由はよくわからないですが、ライブラリのうちCで書かれている比率が高いとか、そういうようなことだと思っています。
- 投稿日:2020-05-20T21:19:55+09:00
データの可視化と相関関係の把握を同時に行う
はじめに
データ分析の際にはグラフを用いてデータを可視化すると思います。そのとき、2変数の相関を表す統計量も同時に表示できたら便利ですよね。そこで、変数の内容(カテゴリか数値か)に応じて適切なグラフに適切な統計量を表示できるようにしました。
これまでのおさらい
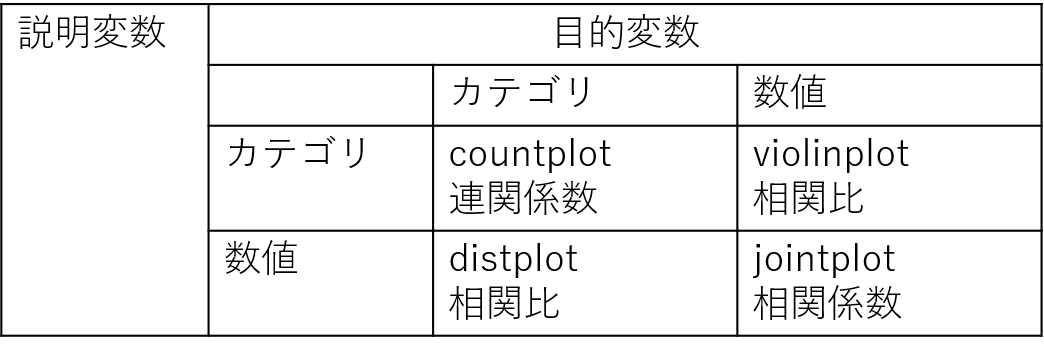
これまで記事にした、変数の内容別の適切なグラフ化メソッドと、相関を表す統計量についてまとめます。詳細は下記のリンクをご覧ください。
説明変数、目的変数別データの可視化方法
カテゴリ変数についても相関を求める方法適切なグラフに適切な統計量を記載
以前作成した、「変数の内容(カテゴリか数値か)に応じて適切なグラフを描く」メソッドに手を入れて、適切なグラフに適切な統計量を記載します(参照:pandas DataFrameを適切なグラフを自動で選んで可視化するメソッドを作った)。
import matplotlib.pyplot as plt import seaborn as sns import scipy.stats as st def visualize_data(data, target_col, categorical_keys=None): keys=data.keys() if categorical_keys is None: categorical_keys=keys[[is_categorical(data, key) for key in keys]] for key in keys: if key==target_col: continue length=10 subplot_size=(length, length/2) if (key in categorical_keys) and (target_col in categorical_keys): r=cramerV(key, target_col, data) fig, axes=plt.subplots(1, 2, figsize=subplot_size) sns.countplot(x=key, data=data, ax=axes[0]) sns.countplot(x=key, data=data, hue=target_col, ax=axes[1]) plt.title(r) plt.tight_layout() plt.show() elif (key in categorical_keys) and not (target_col in categorical_keys): r=correlation_ratio(cat_key=key, num_key=target_col, data=data) fig, axes=plt.subplots(1, 2, figsize=subplot_size) sns.countplot(x=key, data=data, ax=axes[0]) sns.violinplot(x=key, y=target_col, data=data, ax=axes[1]) plt.title(r) plt.tight_layout() plt.show() elif not (key in categorical_keys) and (target_col in categorical_keys): r=correlation_ratio(cat_key=target_col, num_key=key, data=data) fig, axes=plt.subplots(1, 2, figsize=subplot_size) sns.distplot(data[key], ax=axes[0], kde=False) g=sns.FacetGrid(data, hue=target_col) g.map(sns.distplot, key, ax=axes[1], kde=False) axes[1].set_title(r) axes[1].legend() plt.tight_layout() plt.close() plt.show() else: r=data.corr().loc[key, target_col] sg=sns.jointplot(x=key, y=target_col, data=data, height=length*2/3) plt.title(r) plt.show()なお、途中以下のメソッドを使っています。
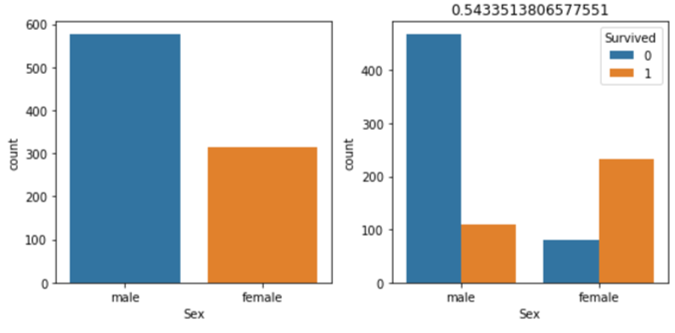
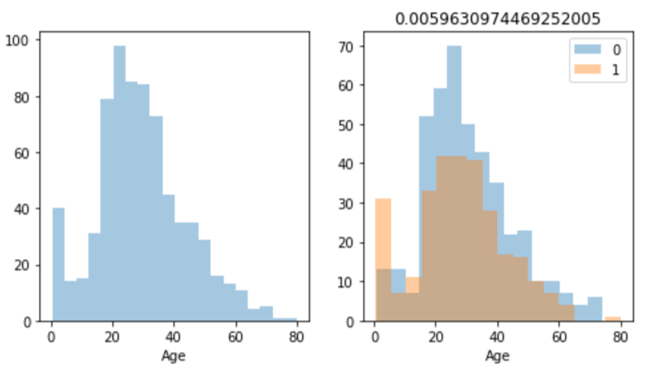
def is_categorical(data, key): #カテゴリ変数かどうかを判定 col_type=data[key].dtype if col_type=='int': nunique=data[key].nunique() return nunique<6 elif col_type=="float": return False else: return True def correlation_ratio(cat_key, num_key, data): #相関比を求める categorical=data[cat_key] numerical=data[num_key] mean=numerical.dropna().mean() all_var=((numerical-mean)**2).sum() unique_cat=pd.Series(categorical.unique()) unique_cat=list(unique_cat.dropna()) categorical_num=[numerical[categorical==cat] for cat in unique_cat] categorical_var=[len(x.dropna())*(x.dropna().mean()-mean)**2 for x in categorical_num] r=sum(categorical_var)/all_var return r def cramerV(x, y, data): #連関係数を求める table=pd.crosstab(data[x], data[y]) x2, p, dof, e=st.chi2_contingency(table, False) n=table.sum().sum() r=np.sqrt(x2/(n*(np.min(table.shape)-1))) return rtitanicデータに適用してみます(結果は一部のみ表示)。
train_data=pd.read_csv("train.csv") train_data=train_data.drop(["PassengerId", "Name", "Ticket", "Cabin"], axis=1) categories=["Survived", "Pclass", "Sex", "Embarked"] visualize_data(train_data, "Survived", categories)最後に
これまで作ってきたメソッドをまとめてみました。これでデータの可視化と相関関係の把握が1度にできるようになりました。ソースコードはgithubに置いてあるので自由に使ってください!
- 投稿日:2020-05-20T21:18:01+09:00
[Python]辞書
辞書の作り方
{キー1:値1 , キー2:値2}
リストとカッコ {} が違うことに注意fruits = {"apple":"りんご" , "banana":"バナナ" , "strawberry":"イチゴ"}出力
辞書[キー]
出力する時のカッコ [] に注意fruits = {"apple":"りんご" , "banana":"バナナ" , "strawberry":"イチゴ"} print("appleは" + fruits["apple"] + "です")出力結果
appleはりんごです
要素の更新
辞書[キー] = 値fruits = {"apple":"りんご" , "banana":"バナナ" , "strawberry":"イチゴ"} fruits["banana"] = "ばなな"要素の追加
辞書[新しいキー] = 値fruits = {"apple":"りんご" , "banana":"バナナ" , "strawberry":"イチゴ"} fruits["kiwi"] = "キウイ"
- 投稿日:2020-05-20T21:03:55+09:00
BlenderをPythonから扱うときにパラメータがわからない問題
BlenderのPython API
BlenderにはPythonのAPIがあり、ドキュメントが公開されている。
なんだ、ドキュメントあるんだったら余裕じゃん、と思ってGUIからパラメータ名を見てドキュメントを検索するも、引っかからないことが多い気がする。
なぜなのか。
単純な話、GUIに表示されているプロパティ名とpython APIでのプロパティ名が異なるからである。具体例
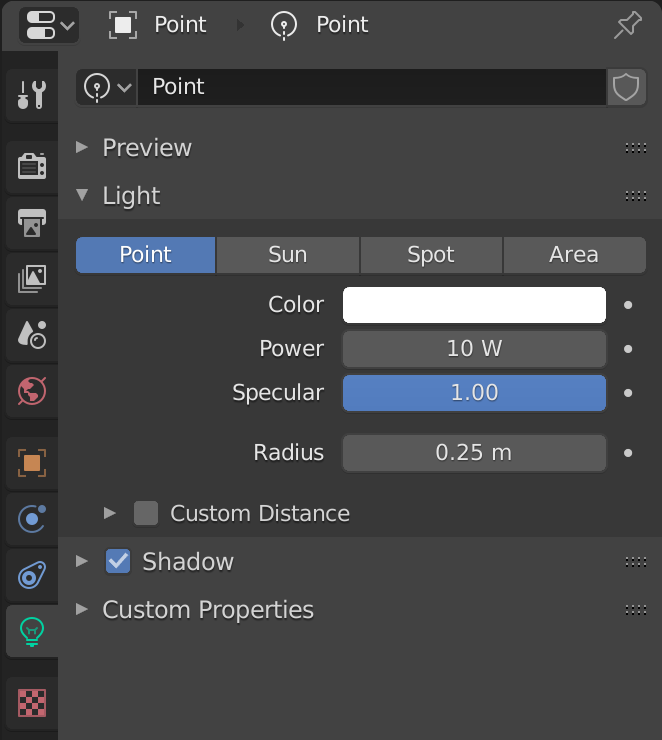
例えば、光源で表示されているPower、これはAPIだとenergyである。いやいやいや。
Radiusはshadow_soft_size。もう何が何だか。
上図の光源を作成するのをコードに落とすと以下のようになるlight_add.pybpy.ops.object.light_add(type='POINT') light_obj = bpy.context.active_object light = light_obj.data light.color = (1.0, 1.0, 1.0) light.energy = 10 light.specular_factor = 1.0 light.shadow_soft_size = 0.25解決方法
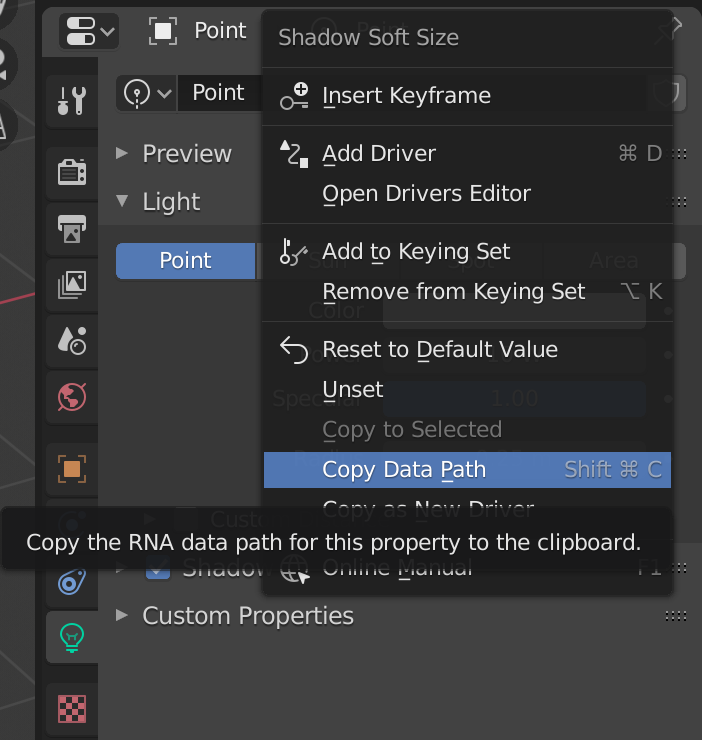
どうやって調べればいいかというと、
1. プロパティの上で右クリックしてCopy Data Pathを選択する。あるいは右にあるコマンドを叩く(macOSならShift+Command+C)
2. これをコード上にペーストする。
それだけ。ここにたどりつくのに莫大な時間を費やしたのでここに書いておく。
- 投稿日:2020-05-20T20:59:56+09:00
[OpenCV] 写真の中からマルフク看板を探す実験
はじめに
OpenCVを使った画像処理の実験。
物体検出の練習として、一部界隈で熱狂的なファンを持つ(?)マルフク看板を写真の画像から探してみます。
といっても物体検出というほど立派なことをしていないので、いくらでも改善の余地がありそうですが、まずは取っ掛かりとして。マルフク看板?
こんなの。
民家や小屋に貼られているのを見たことのある方もいらっしゃると思います。
最近見なくなったとお思いの方も多いかもしれません。ここではあまり書いていませんでしたが、ファンサイトやTwitterのBotも作ってたりする筋金入りのマルフク看板ファンなのであります。(宣伝)
検証環境
- Windows 10 Home (1903)
- Python 3.6.8
- opencv-python==4.2.0.34 (pipからインストール)
- imutils==0.5.3 (同上)
問題設定
今回は入力された画像に対して、マルフク看板の写っている領域を検出して四角で囲んでみます。看板自体は長方形ですが、撮影の角度によって多少歪んだ形になっているでしょう。
最初の画像はこんな感じになると良いですね。自分で作らなくてもいい?
既存の技術やモデルで間に合うならそれでよいので、最初にMask R-CNNの学習済みモデルを試してみたのですが、どうも微妙な結果でした。
matterport/Mask_RCNN: Mask R-CNN for object detection and instance segmentation on Keras and TensorFlow
stop sign、赤と白ですし気持ちは分かりますが…。他の画像だと何も検出されない場合もありました。
「マルフク看板」なんてクラス、あるわけないもんなあ。【手動ポスト】最近Botの中の人は深層学習で遊んでいるのですが、マルフク看板のある風景を物体検出させたらどうなるのかやってみました。マルフク看板には残念ながら何の結果もつかず。マルフク看板自動検出への道は遠いか。使ったプログラムはこれです。 https://t.co/aFUoKs05UJ pic.twitter.com/6BiyNyDs44
— 電話の金融マルフクbot (@029bot) March 26, 2020というわけで、やはり何か考える必要があるっぽいです…。
OpenCVで物体検出(もどき)
四角や円を検出するチュートリアルがあるので、同様の方法でマルフク看板の領域を検出してみたいと思います。
なお「もどき」と書いているのは、いろいろな物体クラスを用意しているわけでもなければ、輪郭抽出以上のことをしているわけでもないからです。。。以下のチュートリアルの内容をベースに作ってみます。
OpenCV shape detection - PyImageSearchチュートリアルの内容の他、必要に応じてこちらのページを参考にしました。
機械学習のためのOpenCV入門 - Qiitaまずはモジュールのインポートと、引数解析・画像読み込み部分です。
import argparse import imutils import cv2 # 引数解析 ap = argparse.ArgumentParser() ap.add_argument("-i", "--image", required=True, help="path to the input image") args = vars(ap.parse_args()) # 画像読み込み image = cv2.imread(args["image"])2値化
まずは画像を2値化して、背景部分が黒、物体部分(マルフク看板の領域)が白になるようにします。
以下のコードを実行すると、2値化された画像がウィンドウに表示され、何かキーを入力するとウィンドウが閉じます。gray = cv2.cvtColor(image, cv2.COLOR_BGR2GRAY) blurred = cv2.bilateralFilter(gray, 25, 15, 15) thresh = cv2.threshold(blurred, 160, 255, cv2.THRESH_BINARY)[1] cv2.imshow("Thresh", thresh) cv2.waitKey(0)カラー画像をグレースケール化し、画素値がある閾値以上かどうかで白・黒に塗り分けます。
この方法で問題なのは、「赤で描かれている部分が黒(背景)になってしまう」という点です。このままだと赤い部分は看板の領域の外になってしまいます。
cv2.threshold()のパラメータも関係ないわけではありませんが、赤色がそれなりに濃く描かれているので、赤い部分が白くなるようにパラメータを調整すると、大部分が白になってしまいます。そこで、前処理として 赤色を消す 方法を試してみたいと思います。
光の赤色に見える部分は、絵の具(インク)の三原色でいえば「マゼンタ」と「イエロー」を混ぜたものになります。1
つまり、マゼンタとイエローを消すことによって、赤い部分を白くすることができそうです。
光の三原色 (RGB) で言えば、マゼンタを消すには緑 (G) を最大にすればよく、イエローを消すには青 (B) を最大にすればよいので、マゼンタとイエローを消すと結局RGBの赤 (R) の成分だけが効いてきます。よって、Rの値だけを残すことによって目的を達成できます。ということで、グレースケール化する際に
cv2.cvtColor()を使わず、RGBの「赤」の成分を取り出す方法を試します。gray = image[:, :, 2] blurred = cv2.bilateralFilter(gray, 25, 15, 15) thresh = cv2.threshold(blurred, 160, 255, cv2.THRESH_BINARY)[1] cv2.imshow("Thresh", thresh) cv2.waitKey(0)結果はこんな感じ。良いですね。
ちなみに、赤の成分を取り出した時点の画像 (gray) はこうなっています。文字が読める程度に色は残っていますが、背景と比べると薄くなっていることが分かります。
こんな感じで色あせて赤色が抜けた看板は普通に見かける気がする…。
輪郭抽出
今度は、先ほど作成した白黒の2値画像から、物体の輪郭を抽出します。
cv2.findContours()を使えば輪郭抽出は簡単にできるので、その結果を画面に描画して確認してみましょう。output = image.copy() # 輪郭抽出 cnts = cv2.findContours(thresh.copy(), cv2.RETR_LIST, cv2.CHAIN_APPROX_SIMPLE) cnts = imutils.grab_contours(cnts) # 画像に輪郭を描画 for c in cnts: cv2.drawContours(output, [c], -1, (0, 255, 0), 2) cv2.imshow("Image", output) cv2.waitKey(0)なんか随分ぐちゃぐちゃですね…。
先ほどの2値画像に境界線を素直に引いた結果ではあるのですが、これでは使い勝手が悪いですし、ノイズの影響もあって変なところに線が出ていたりするので、もう少し改良しましょう。輪郭の多角形近似
輪郭抽出の結果は点の集合になっていますが、最終的には輪郭は四角形(綺麗な長方形とは限りませんが)を想定しているので、多角形で近似した方がノイズの影響を小さくできてよいでしょう。
output = image.copy() # 輪郭抽出 cnts = cv2.findContours(thresh.copy(), cv2.RETR_LIST, cv2.CHAIN_APPROX_SIMPLE) cnts = imutils.grab_contours(cnts) # 画像に輪郭を描画 for c in cnts: # 輪郭を多角形で近似 epsilon = 0.1*cv2.arcLength(c,True) c = cv2.approxPolyDP(c,epsilon,True) cv2.drawContours(output, [c], -1, (0, 255, 0), 2) cv2.imshow("Image", output) cv2.waitKey(0)看板の縁の部分の線がギザギザしていましたが、そのギザギザ感が取れて直線的になりましたね。
一方、背景の関係ないところに線が出る状況はそのままです。小さい領域を削除
アドホックな方法ではありますが、ノイズから誤検出される領域は小さいことが多いので、面積が一定値未満の領域は表示しないようにします。
output = image.copy() # 輪郭抽出 cnts = cv2.findContours(thresh.copy(), cv2.RETR_LIST, cv2.CHAIN_APPROX_SIMPLE) cnts = imutils.grab_contours(cnts) # 画像に輪郭を描画 for c in cnts: # 輪郭を多角形で近似 epsilon = 0.1 * cv2.arcLength(c, True) c = cv2.approxPolyDP(c, epsilon, True) # 小さい領域を表示しない area = cv2.contourArea(c) if area < 400: continue cv2.drawContours(output, [c], -1, (0, 255, 0), 2) cv2.imshow("Image", output) cv2.waitKey(0)形状の情報でフィルタリング
先ほどの例ではうまくいくのですが、他の画像を試すと変な輪郭が残ることがあります。
以下のように、画像の縁と屋根で囲まれた三角形の部分を検出してしまっています。
そこで、形状の情報を利用してフィルタリングしてみます。
マルフク看板の実物は「幅900mm × 高さ600mm」の長方形です。このとき、面積 $S$ と周囲の長さ $L$ の関係として\frac{\sqrt S}{L} = \frac{\sqrt{900 \times 600}}{2 \times (900 + 600)} = \frac{\sqrt 6}{10} \approx 0.245が成り立ちます。この値は幅と高さを同じ倍率で大きくしても変わりませんので、形状を表す特徴として使えそうです。2
output = image.copy() # 輪郭抽出 cnts = cv2.findContours(thresh.copy(), cv2.RETR_LIST, cv2.CHAIN_APPROX_SIMPLE) cnts = imutils.grab_contours(cnts) # 画像に輪郭を描画 for c in cnts: # 輪郭を多角形で近似 epsilon = 0.1 * cv2.arcLength(c, True) c = cv2.approxPolyDP(c, epsilon, True) # 小さい領域を表示しない area = cv2.contourArea(c) if area < 400: continue # 周囲の長さを計算 perimeter = cv2.arcLength(c, True) # 輪郭が3:2の長方形に近い場合のみ表示 if perimeter == 0: # ゼロ割り防止 continue if 0.23 < area ** 0.5 / perimeter < 0.26: # approx sqrt(6)/10 cv2.drawContours(output, [c], -1, (0, 255, 0), 2) cv2.imshow("Image", output) cv2.waitKey(0)これで右上の三角形の輪郭は消え、良い感じに看板の部分だけ残ります。
まだ形状しか見ていませんが、実際には枠の中の領域を取り出して物体検出(マルフク看板かどうか?)を実行することになるのでしょう。2値化の閾値パラメータを与えないようにする
最初の方に出てきた以下のコードです。Rの値が160より大きいか小さいかで白と黒を塗り分けています。
thresh = cv2.threshold(blurred, 160, 255, cv2.THRESH_BINARY)[1]少し考えれば分かるように、画像の明るさなどの条件によって最適な閾値は変化します。
OpenCVには、閾値を自動で決める方法として「大津の方法(大津の2値化)」3という有名な方法が実装されているので、試してみます。# 大津の方法 thresh = cv2.threshold(blurred, 0, 255, cv2.THRESH_BINARY+cv2.THRESH_OTSU)[1]結果はこちら。
自動でパラメータを決めることなしに輪郭を抽出できました。
電気メーターの部分に枠が出てしまっていますが、後で分類すればよいことにして、とりあえず目を瞑ります。。。課題
結構うまく動いているように見えますが、実はこんな画像をうまく処理できません。
小屋に貼られている白い板が実はマルフク看板です。皆様ご存知の通り(?)、放置されたマルフク看板はこんな感じで読めなくなってしまいます。しかし真っ白ならばむしろ検出には都合がよいはず。
ではなぜ白い領域に枠が出てこないかというと、2値化の結果
こうなってしまったからです。要するに背景のトタン板の色が明るいことが原因ですね。この画像の場合、パラメータを適切に決めることができればうまくいきます。
#thresh = cv2.threshold(blurred, 0, 255, cv2.THRESH_BINARY+cv2.THRESH_OTSU)[1] thresh = cv2.threshold(blurred, 190, 255, cv2.THRESH_BINARY)[1]推測ですが、「大津の方法」では輝度のヒストグラムに「背景」と「物体」という2つのピークがあることを仮定しているため、明るいトタン板の色が「物体」側に取り込まれてしまったと考えられます。
他の画像を使った場合でも、うまくいったりいかなかったり。改善は今後の課題ですね。コードまとめ
marufuku.pyimport argparse import imutils import cv2 # 引数解析 ap = argparse.ArgumentParser() ap.add_argument("-i", "--image", required=True, help="path to the input image") args = vars(ap.parse_args()) # 画像読み込み image = cv2.imread(args["image"]) gray = image[:, :, 2] blurred = cv2.bilateralFilter(gray, 25, 15, 15) # 大津の方法 thresh = cv2.threshold(blurred, 0, 255, cv2.THRESH_BINARY+cv2.THRESH_OTSU)[1] cv2.imshow("Thresh", thresh) cv2.waitKey(0) output = image.copy() # 輪郭抽出 cnts = cv2.findContours(thresh.copy(), cv2.RETR_LIST, cv2.CHAIN_APPROX_SIMPLE) cnts = imutils.grab_contours(cnts) # 画像に輪郭を描画 for c in cnts: # 輪郭を多角形で近似 epsilon = 0.1 * cv2.arcLength(c, True) c = cv2.approxPolyDP(c, epsilon, True) # 小さい領域を表示しない area = cv2.contourArea(c) if area < 400: continue # 周囲の長さを計算 perimeter = cv2.arcLength(c, True) # 輪郭が3:2の長方形に近い場合のみ表示 if perimeter == 0: # ゼロ割り防止 continue if 0.23 < area ** 0.5 / perimeter < 0.26: # approx sqrt(6)/10 cv2.drawContours(output, [c], -1, (0, 255, 0), 2) cv2.imshow("Image", output) cv2.waitKey(0)以下のようにコマンドを実行すると動きます。
python marufuku.py -i image.pngまとめ
物体検出の初歩の初歩ではありますが、マルフク看板を題材に輪郭抽出だけ試してみました。
実際に使えるものにするには、背景色への頑健性も課題ですが、抽出した領域に対するクラス分類器の学習(データを集めて)も必要になってくると思います。
パラメータを全自動で決めることにこだわらず、目視確認しながらパラメータ調整しつつ領域を切り出して、Mask R-CNNなどの学習データを作る方針が良いのかな、などと考えています。
実は「円形度」を $4\pi$ で割って正の平方根をとったものと等しくなります。→円形度とは何? Weblio辞書 ↩
大津展之: 判別および最小2乗規準に基づく自動しきい値選定法, 電子通信学会論文誌 D, Vol. 63, No. 4, pp.349-356, 1980 ↩
- 投稿日:2020-05-20T20:56:17+09:00
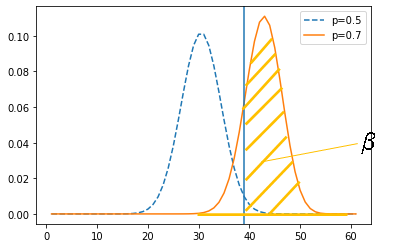
(Python)期待値・モンテカルロサンプリングを丁寧に理解しようとした
はじめに
ベイズ統計学を学ぶ上で期待値をおさらいしました。
下記本を参考にしました。
期待値とは
期待値(expectation)とは、ある関数$f(x)$の確率分布$p(x)$のもとでの$f(x)$の平均値のことを指します。表記としては、$E[f]$と書きます。離散分布では以下のように表します。
E[f] = \sum_x p(x)f(x)一方、連続変数の場合は積分として表すことができます。
E[f] = \int p(x)f(x)dxエントロピー
確率分布$p(x)$に対する次のような期待値をエントロピー(entropy)と呼びます。
\begin{align} H[p(x)]& = - \sum_x p(x) ln(p(x))\\ \end{align}有限和での近似(モンテカルロサンプリング)
分布$p(x)$から独立に抽出されたサンプルの集合を$\bf {z}^{(n)} (n = 1,...,N)$としたとき、期待値は下記のように近似できます。
E[f] = \frac{1}{L} \sum_{n=1}^{N}f(\bf{z}^{(N)})となります。
ここで例題で考えます。
例題
$p(x=1)=0.3,p(x=1)=0.7$となるような離散分布を考えます。エントロピーの定義からエントロピーは
\begin{align} H[p(x)]& = - \sum_x p(x) ln(p(x))\\ &=-(p(x=1)lnp(x=1) + p(x=0)lnp(x=0) )\\ &= -(\frac{3}{10}ln\frac{3}{10}+\frac{7}{10}ln\frac{7}{10})\\ &=0.610 \end{align}となります。
さて、これを有限和で近似する場合の計算をしてみます。random.uniformメソッドにより0~1の無作為な値を出力させて、それが$p(x=1)=0.3$よりも大きくなるかどうかで$x=1$か$x=2$かどうかを判別させています。
そして、$x=1,2$となる回数をcntで数えさせることをしています。
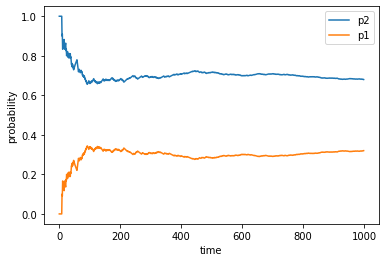
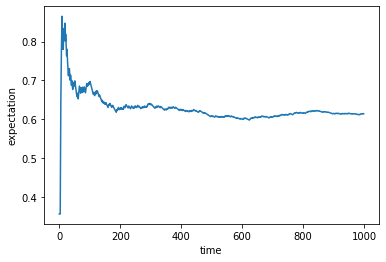
下記のようなプログラムになります。試しに1000回計算しています。cnt = [] proba_1 =[] proba_2 =[] time = 1000 a = random.uniform(0,1) exp =[] for i in range(time): a = random.uniform(0,1) if a > p1: cnt = np.append(cnt,1) else: cnt = np.append(cnt, 0) proba_1 = np.append(proba_1, (i+1-sum(cnt))/(i+1)) proba_2 = np.append(proba_2, sum(cnt)/(i+1)) exp = np.append(exp, -(((i+1-sum(cnt))*math.log(p1))+((sum(cnt))*math.log(p2)))/(i+1)) plt.xlabel('time') plt.ylabel('probability') plt.plot(time_plot, proba_2, label="p2") plt.plot(time_plot, proba_1, label="p1") plt.legend()$p(x=1)=0.3,p(x=2)=0.7$に大よそ100回以降収束していくことが分かりました。
こちらも100回以降で元々求めていた期待値(=エントロピー)の0.61あたりに収束していることが分かりました。
確かにこの期待値近似の手法が問題ないことが確認することができました。
終わりに
今回は非常に簡単な例題だったため、算出及び確かめることが容易でした。しかし、実際の問題では期待値が解析的に求めることが難しい場合がほとんどです。
従い、このモンテカルロサンプリングで近似することを覚えておくと有益なのだと思います。
プログラ全文はこちらです。
https://github.com/Fumio-eisan/VI20200520
- 投稿日:2020-05-20T20:49:51+09:00
[Python]リスト
リスト
リスト作り方
[要素1 , 要素2 , 要素3]
, で区切る。リスト例
文字列のリスト
["sushi" , "ramen"]数値のリスト
[100 , 200]変数にリストを代入
fruits = ["apple" , "banana" , "strawberry"]リストの出力、要素の取得
リストの出力fruits = ["apple" , "banana" , "strawberry"] print(fruits)出力結果
["apple" , "banana" , "strawberry"]
リストの要素の取得
変数[インデックス番号]
変数はリストを代入したものfruits = ["apple" , "banana" , "strawberry"] print("好きな果物は" + fruits[1] + "です")出力結果
好きな食べ物はbananaです
**要素の上書き**
リスト[インデックス番号] = 値fruits = ["apple" , "banana" , "strawberry"] fruits[1] = "kiwi"**要素の追加**
リスト.append(値)fruits = ["apple" , "banana" , "strawberry"] fruits.append("kiwi")
- 投稿日:2020-05-20T20:32:41+09:00
DjangoとNewsAPIを使って携帯大手3社のニュースをそれぞれ取得する
はじめに
プログラミング初心者なので暖かい目でこの記事を見ていただけると幸いです。
環境
Windows10
Python 3.7.6
newsapi-python 0.2.6
Django 3.0.6
DateTime 4.3また作成時のディレクトリの名前は以下の通りになります。
sample_news ├─news ├─sample_news ├─static └─manage.pyNewsAPIの導入
NewsAPIのサイトに行きメールアドレスを入力してAPIキーを取得し、そのAPIキーはメモしておいてください。
その後pipでNewsAPIのライブラリをインストールしてください。pip install newsapi-pythonsample_newsにあるsetting.pyに以下のコードを追加してNewsAPIを使えるようにしてください。
api_keyは自分のメモしたキーを入力して下さい。setting.pyfrom newsapi import NewsApiClient newsapi=NewsApiClient(api_key='xxxxxxxxxxxxxxxxxxxxxx') INSTALLED_APPS = [ 'django.contrib.admin', 'django.contrib.auth', 'django.contrib.contenttypes', 'django.contrib.sessions', 'django.contrib.messages', 'django.contrib.staticfiles', 'news.apps.NewsConfig', 'newsapi', ]これでNewsAPIが使えるようになりました。
ニュースアプリの作成
ルーティングの設定
ルーティングを設定するためにsample_newsのurls.pyに以下のコードを追加してください。
sample_news/urls.pyfrom django.contrib import admin from django.urls import path,include urlpatterns = [ path('admin/', admin.site.urls), path('',include('news.urls')), ]次にnewsにurls.pyを作成し以下のコードを入力しました。
news/urls.pyfrom django.urls import path from.import views app_name = 'news' urlpatterns = [ path('', views.IndexView.as_view(), name="index"), path('docomo/',views.DocomoView.as_view(),name="docomo"), path('au/',views.AuView.as_view(),name="au"), path('softbank/',views.SoftbankView.as_view(),name="softbank") ]NewsAPIでニュースを取得する
NewsAPIではトップニュースを取得する方法と過去の一定の期間のニュースを取得する方法の2つがあります。
トップニュースを取得した場合はニュースがなかった場合何も表示されなかったので一定期間のニュースを取得する方法を採用しました。NewsAPIは課金しなかった場合30日前までのニュースを取得できます。
今回はキーワード、期間、表示する順番、ニュースの数を指定しました。
ほかにもタイトルのキーワード、ソース、ドメイン、言語(日本語は不可)などがあります。news/views.pyfrom django.views import generic from newsapi import NewsApiClient from datetime import datetime, date, timedelta class IndexView(generic.TemplateView): template_name = "index.html" class DocomoView(generic.TemplateView): template_name = "docomo.html" def get_context_data(self, **kwargs): context=super().get_context_data(**kwargs) today = date.today() monthago = today-timedelta(days=30) newsapi = NewsApiClient(api_key='e56b4ed3346745e3800bc521d085e04b') context['all_article']=newsapi.get_everything(q='ドコモ', from_param=monthago, to=today, sort_by='publishedAt', page_size=40) return context class AuView(generic.TemplateView): template_name = "au.html" def get_context_data(self, **kwargs): context=super().get_context_data(**kwargs) today = date.today() monthago = today-timedelta(days=30) newsapi = NewsApiClient(api_key='e56b4ed3346745e3800bc521d085e04b') context['all_article']=newsapi.get_everything(q='KDDI', from_param=monthago, to=today, sort_by='publishedAt', page_size=40) return context class SoftbankView(generic.TemplateView): template_name = "softbank.html" def get_context_data(self, **kwargs): context=super().get_context_data(**kwargs) today = date.today() monthago = today-timedelta(days=30) newsapi = NewsApiClient(api_key='e56b4ed3346745e3800bc521d085e04b') context['all_article']=newsapi.get_everything(q='ソフトバンク', from_param=monthago, to=today, sort_by='publishedAt', page_size=40) return contextこれでニュースを取得するプログラムは完成したのであとはコーティングをするだけです。
取得したニュースを表示する
今回はトップページ(index.html)、ドコモのニュースを表示するページ(docomo.html)、auのニュースを表示するページ(au.html)、ソフトバンクのニュース(softbank.html)を表示するページの4つのページと、各ページに共通する部分(base.html)の5つのhtmlファイルを作成しました。
templates ├─au.html ├─base.html ├─docomo.html ├─index.html └─softbank.htmlドコモのニュースを表示するページは以下の通りです。
今回はタイトルとurlとニュースの画像を取得して表示するようにしました。
docomo.html{% extends 'base.html' %} {% block title %}Sample_News | SOFTBANK{% endblock %} {% block contents %} <div class="grid"> {% for context in all_article.articles %} <div class="news"> <div class="news-wrapper"> <img class="news-img" src="{{ context.urlToImage }}" alt=""> <a class="news-title" href="{{ context.url }}">{{ context.title }}</a> </div> </div> {% endfor %} </div> {% endblock %}このような感じでauとソフトバンクのhtmlファイルも記述します。
最後にCSSでのコーディングをして完成です。
まとめ
今回はdjangoを勉強した後に初めて作ったwebアプリなので参考書などをたくさん活用して作りました。勉強した後に復讐のために自分でwebアプリを作るとより理解が深められると自分で感じることができました。
検索の単語をドコモはドコモ、auはau、ソフトバンクはソフトバンクでやったのですが、ドコモはドコモのニュースのみが出力されたのですが、auは海外のニュースが、ソフトバンクは野球のニュースが出力されたのでそこを改善していきたいと思います。
また記事を書くのも初めてなので至らぬことがあれば教えていただけるとありがたいです。
参考
- 投稿日:2020-05-20T20:32:36+09:00
Pythonで作成したデータの可視化画像をTypetalkに送る方法
目的
データをmatplotlibを使って可視化した後、その結果を定期的にTypetalkに送りたいと思ったので作りました。
ローカルに作成した画像を保存せずにそのまま送信する方法になります。必要なライブラリ
作り方
import requests import json import matplotlib.pyplot as plt import io image_buffer = io.BytesIO() plt.plot([1,2,3]) # 折れ線グラフを書く plt.savefig(image_buffer, format='jpeg') # ここで書き出し token = '<Typetalkのbotのトークンを取得して代入してください>' topic_id = '<Typetalkの投稿するトピックIDを代入してください>' # メッセージ送信用のURL message_url = f"https://typetalk.com/api/v1/topics/{topic_id}?typetalkToken={token}" # ファイルアップロード用のURL upload_file_url = f'https://typetalk.com/api/v1/topics/{topic_id}/attachments?typetalkToken={token}' files = {'file': ('scatter.jpeg', image_buffer.getvalue(), 'image/jpeg')} upload_file_result = requests.post(upload_file_url, files=files) # 実行の確認 print(upload_file_result.status_code) print(upload_file_result.content) fileKey = json.loads(upload_file_result.content)['fileKey'] message_result = requests.post(url_message, {'message': '今日の結果!', 'fileKeys[0]': fileKey}) # 実行の確認 print(message_result.status_code) print(message_result.content)実行結果
これで定期的なデータのモニタリングができる!
- 投稿日:2020-05-20T20:24:46+09:00
GCPのCloud Vision APIの使い方
ドキュメントが少し分かりにくかったのでまとめてみる。
APIのパラメータ
- Type
- maxResults
- model ### GCPのCloud Vision APIのTYPEには二種類ある。
- テキスト検出「TEXT_DETECTION」(大きな画像内のテキストのスパース領域向けに最適化されている)
- ドキュメントテキスト検出「DOCUMENT_TEXT_DETECTION」(高密度テキストに適している)
OCRの出力の構造はどちらも、
TextAnnotation -> Page -> Block -> Paragraph -> Word -> Symbolになっている
必要なものをインポート
import base64 import json from requests import Request, Session from io import BytesIO from PIL import Image import numpy as npAPIキーの獲得
Google Cloud Vision APIのOCRを使ってPythonから文字認識する方法
APIの使い方
def recognize_image1(input_image):#最後にstr_encode_fileに変える #pathからbase64にする場合 def pil_image_to_base64(img_path): pil_image = Image.open(img_path) buffered = BytesIO() pil_image.save(buffered, format="PNG") str_encode_file = base64.b64encode(buffered.getvalue()).decode("utf-8") return str_encode_file #arrayからbase64にする場合 def array_to_base64(img_array): pil_image = Image.fromarray(np.uint8(img_array)) buffered = BytesIO() pil_image.save(buffered, format="PNG") str_encode_file = base64.b64encode(buffered.getvalue()).decode("utf-8") return str_encode_file def get_fullTextAnnotation(json_data): text_dict = json.loads(json_data) try: text = text_dict["responses"][0]["fullTextAnnotation"]["text"] return text except: print(None) return None str_encode_file = pil_image_to_base64(input_image) # input_imageを画像のPATHにしたい時はこっちを選択 #str_encode_file = array_to_base64(input_image)# input_imageをarrayにしたい時はこっちを選択 str_url = "https://vision.googleapis.com/v1/images:annotate?key=" str_api_key = ""#APIキーをここに入れる str_headers = {'Content-Type': 'application/json'} str_json_data = { 'requests': [ { 'image': { 'content': str_encode_file }, 'features': [ { 'type': "DOCUMENT_TEXT_DETECTION",#ここでtypeを選択 'maxResults': 1 } ] } ] } obj_session = Session() obj_request = Request("POST", str_url + str_api_key, data=json.dumps(str_json_data), headers=str_headers ) obj_prepped = obj_session.prepare_request(obj_request) obj_response = obj_session.send(obj_prepped, verify=True, timeout=60 ) if obj_response.status_code == 200: text = get_fullTextAnnotation(obj_response.text) return text else: return "error"参考
- 投稿日:2020-05-20T19:41:08+09:00
Pythonのリストのコピーを行う際に注意すること
はじめに
Pythonでリストのコピーを行う際に注意することをメモします。詳しい内容は「値渡し・参照渡し」などで検索してみてください。
注意すること
言葉で説明するよりも先に、以下のコードをご覧ください。
X = 2 Y = X Y = 5 print('Y = ', Y) print('X = ', X)これを実行すると、もちろん
Y = 5 X = 2という結果が得られます。
次に、以下のコードを実行してみてくだいさい。i = [1, 2, 3, 4, 5] j = i j[0] = 100 print('j = ', j) print('i = ', i)ここでは、iというリストを決め、jにiを代入し、jのインデックス番号0の値を100にし、iとjをそれぞれ出力するものです。その結果は、
j = [100, 2, 3, 4, 5] i = [100, 2, 3, 4, 5]となり、 iの方も変更がされてしまいました。
この原因は、次のコードで確かめることができます。print(id(X)) print(id(Y)) print(id(i)) print(id(j))4436251808 4436251904 4304286176 4304286176となり、値を代入したXとYはそれぞれ違うidを持つのに対し、リストを代入したiとjは同じidを持つことがわかりました。そのため、リストではjで行った変更はiにも反映されることになります。
そのため、リストのコピーを行う際は、j = i.copy()最後に
リストをコピーすることは少ないと聞いたので、あまり使用頻度は高くないと思いますが、バグに繋がらないよう、注意してください。
- 投稿日:2020-05-20T18:33:27+09:00
バッチコミット作成例(配列を刻みながら処理する方法をいくつか)
大量のデータをDBへ格納する際など、バッチオブジェクトに一定量の書き込み処理を蓄積し、定期的にコミットするという処理を行うことがあります。
上記を行うための、配列を一定間隔で刻みながら処理する方法のPythonのコードによるメモです。
もっと良い方法をご存知でしたらご指摘いただけると幸いです。更新:指摘を頂き、方法4を追加(@shiracamusさんありがとうございます)
方法1(専用のカウンタを使う)
data = ['a', 'b', 'c', 'd', 'e', 'f', 'g', 'h'] q = [] batch_size = 3 batch_count = 0 for d in data: print("{}".format(d)) q.append(d) batch_count += 1 if batch_count == batch_size: print("commit {}".format(q)) q = [] batch_count = 0 print("commit {}".format(q)) > python sample1.py a b c commit ['a', 'b', 'c'] d e f commit ['d', 'e', 'f'] g h commit ['g', 'h']方法2(インデックスと剰余を使う)
data = ['a', 'b', 'c', 'd', 'e', 'f', 'g', 'h'] q = [] batch_size = 3 for i, d in enumerate(data): print(d) q.append(d) if (i + 1) % batch_size == 0: print("commit {}".format(q)) q = [] print("commit {}".format(q)) > python sample2.py a b c commit ['a', 'b', 'c'] d e f commit ['d', 'e', 'f'] g h commit ['g', 'h']方法3(最後のコミットを工夫する)
最後のcommitの記述をなくせてコードの見通しが良い方法です。
予めデータサイズを取得する必要があるのでイテラブルなデータには使えないのと、判定処理の効率が悪いのが弱点です。
data = ['a', 'b', 'c', 'd', 'e', 'f', 'g', 'h'] q = [] last = len(data) batch_size = 3 for i, d in enumerate(data): print(d) q.append(d) if ((i + 1) % batch_size == 0) | ((i + 1) == last): print("commit {}".format(q)) q = [] > python sample3.py a b c commit ['a', 'b', 'c'] d e f commit ['d', 'e', 'f'] g h commit ['g', 'h']方法3+アルファ
Pythonのenumerateは第二引数で開始番号を指定できるので、方法3はもう少しシンプルにできます。
data = ['a', 'b', 'c', 'd', 'e', 'f', 'g', 'h'] q = [] last = len(data) batch_size = 3 for n, d in enumerate(data, 1): print(d) q.append(d) if (n % batch_size == 0) | (n == last): print("commit {}".format(q)) q = [] > python sample3.py a b c commit ['a', 'b', 'c'] d e f commit ['d', 'e', 'f'] g h commit ['g', 'h']方法4(スライスを利用する)
オシャレな方法(@shiracamusさん、ありがとうございます)
バッチオブジェクトのqに配列で渡せない場合はfor inなどでバラす。data = ['a', 'b', 'c', 'd', 'e', 'f', 'g', 'h'] batch_size = 3 for i in range(0, len(data), batch_size): q = data[i:i+batch_size] print("commit", q) > python sample3.py commit ['a', 'b', 'c'] commit ['d', 'e', 'f'] commit ['g', 'h']参考
方法4で、GCPのfirestoreにバッチインサートの上限の500件づつデータを追加するサンプルです。
db = firestore.Client() records_collection = db.collection("<COLLECTION NAME>") batch = db.batch() batch_size = 500 for i in range(0, len(data), batch_size): for row in data[i:i + batch_size]: batch.set(records_collection.document(), row) print('committing...') batch.commit() batch = db.batch()
- 投稿日:2020-05-20T17:39:22+09:00
5分でできるgRPC チュートリアル
概要
gRPC について調べたことをまとめました。
また、公式のチュートリアルをpythonで動かしてみました。適宜触り次第更新していきます。
gRPCとは
gRPCとは、オープンソースの高機能な Remote Procedure Call フレームワークです。gRPCにより、クライアント/サーバーのアプリケーション間の通信が透明になり、かつ簡単に構築できます。
とあります。気になるので公式のウェブサイトをみてみましょう。
gRPCを使うことで何がうれしいのか
gRPCを使うと、リモート上のサーバの関数をローカルのもののように呼ぶことができ、これにより分散されたサービスやアプリケーションを作るのが楽になります。
gPRCをサポートした言語で書けるので、gPRCサーバを Javaで書き、clientをpythonで書く、みたいなことも容易にできます。ここでの違う言語間のコミュニケーションの複雑さが、 gPRCによってハンドリングされます。Protocol Buffer について
gPRCではProtocol Bufferというものがデフォルトでは使われ(jsonなどのフォーマットを使うことも可能です)ます。Protocol Bufferは .protoファイルで定義されますが、その形式はXMLファイルなどに比べてとても軽い(3~10倍軽い)、また、高速(20~100倍速い)です。この.protoファイルにサービスで使うメソッドや、データのやりとりに使う構造体を定義すると、そのシリアライゼーションやディシリアライゼーションは、protoコンパイラを通すことで簡単に行うことができます。それにより、各言語で転送される構造体を扱うアクセッサ(クラス)が定義され、構造体のプロパティなどを設定できるセッターやゲッターなども勝手に作られます。
したがって、.protoファイルを定義することで、各言語間であってもサービスを簡単に構築できる、また、シリアライゼーションなどの差異も吸収してくれるということです。
すごい!!というわけで、PythonとC++でチュートリアルやってみます。
gRPCチュートリアル
Python
必要なものをpip install します。
pip install grpcio grpcio-toolsこのとき、最新のバージョンが入ってないかも知れないので、
grpcioのバージョンの確認をきちんとしときましょう!必要なレポジトリをクローンします。
git clone -b v1.28.1 https://github.com/grpc/grpc && cd grpc/examples/python/helloworld2つターミナル開いて、片方で
python greeter_server.pyもう片方で
python greeter_client.pyをやってみます。
クライアント側で、Greeter client received: Hello, you!と出れば成功です!!
さらに、gRPCのサービスにもう1つメソッドを追加してみます。
ここでは、もともとあった
SayHelloに加え、SayHelloAgainを追加します。examples/protos/helloworld.proto// The greeting service definition. service Greeter { // Sends a greeting rpc SayHello (HelloRequest) returns (HelloReply) {} // Sends another greeting rpc SayHelloAgain (HelloRequest) returns (HelloReply) {} } // The request message containing the user's name. message HelloRequest { string name = 1; } // The response message containing the greetings message HelloReply { string message = 1; }サービスを更新します。
examples/python/helloworldで、python -m grpc_tools.protoc -I../../protos --python_out=. --grpc_python_out=. ../../protos/helloworld.protoを実行し、
greeter_server.py及びgreeter_client.pyを更新します。greeter_server.pyclass Greeter(helloworld_pb2_grpc.GreeterServicer): def SayHello(self, request, context): return helloworld_pb2.HelloReply(message='Hello, %s!' % request.name) def SayHelloAgain(self, request, context): return helloworld_pb2.HelloReply(message='Hello again, %s!' % request.name)greeter_client.pydef run(): channel = grpc.insecure_channel('localhost:50051') stub = helloworld_pb2_grpc.GreeterStub(channel) response = stub.SayHello(helloworld_pb2.HelloRequest(name='you')) print("Greeter client received: " + response.message) response = stub.SayHelloAgain(helloworld_pb2.HelloRequest(name='you')) print("Greeter client received: " + response.message)以上ができたら、以前の
2つターミナル開いて、片方で
python greeter_server.pyもう片方で
python greeter_client.pyを実行します。
Greeter client received: Hello, you! Greeter client received: Hello again, you!こう出るはずです!
今回は、gRPCについて調べ、
公式のチュートリアルを走らせ、.protoファイルなどについて少し理解を深められたかと思います。
引き続きいろんなことを試し次第更新、もしくは別記事に書きたいと思います!!今回はこの辺で。
おわり。
- 投稿日:2020-05-20T17:30:58+09:00
Pythonで線形代数をやる(1)
線形代数
理系大学で絶対に習う線形代数をわかりやすく、かつ論理的にまとめる。ちなみにそれをPythonで実装。たまに、Juliaで実装するかも。。。
・Pythonで動かして学ぶ!あたらしい数学の教科書 -機械学習・深層学習に必要な基礎知識-
・世界基準MIT教科書 ストラング線形代数イントロダクション
を基に線形代数を理解し、pythonで実装。環境
・JupyterNotebook
・言語:Python3, Julia線形結合(ベクトル入門)
線形結合はスカラー積とベクトル和によって構成される。
2つのスカラーをa,bとして、ベクトル和をv,wとする。スカラー積は、
av, bw のように掛けられたもの。
ベクトル和は、
v + w のように足されたもの。
これら二つの組み合わせを線形結合という。よって、
av + bw*
のような形のことである。ちなみにこれは項数が2つなので、2次元面を張る。(v=wの場合、違うが。)pythonで実装
import numpy as np # ベクトル v = np.array([1, 2, 3]) w = np.array([4, 5, 6]) # スカラー a = 2 b = 5 #線形結合の形 linearcomb = a * v + b * w print(linearcomb)juliaで実装
# ベクトル v = [1, 2, 3] w = [4, 5, 6] # スカラー a = 2 b = 5 linearcomb = a * v + b * w print(linearcomb)どちらも以下のようにターミナルに出てくる
[22 29 36]ここまで。
- 投稿日:2020-05-20T17:09:09+09:00
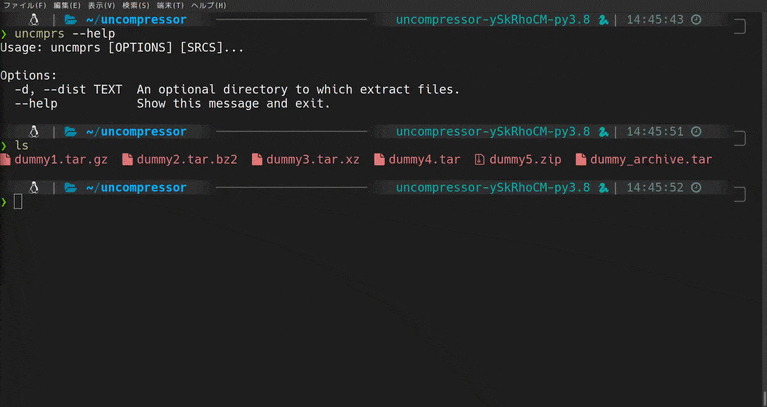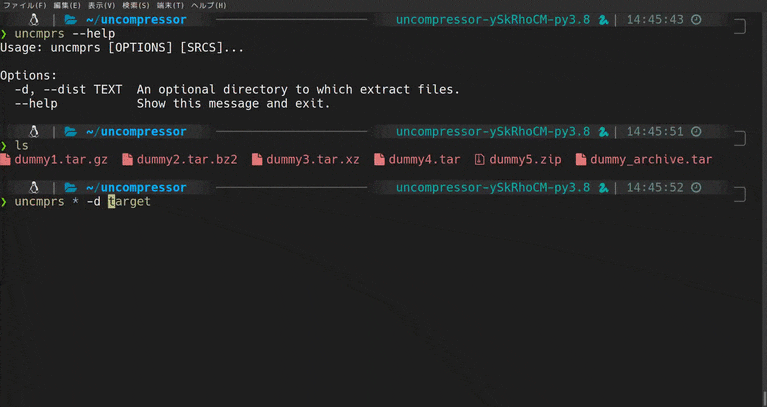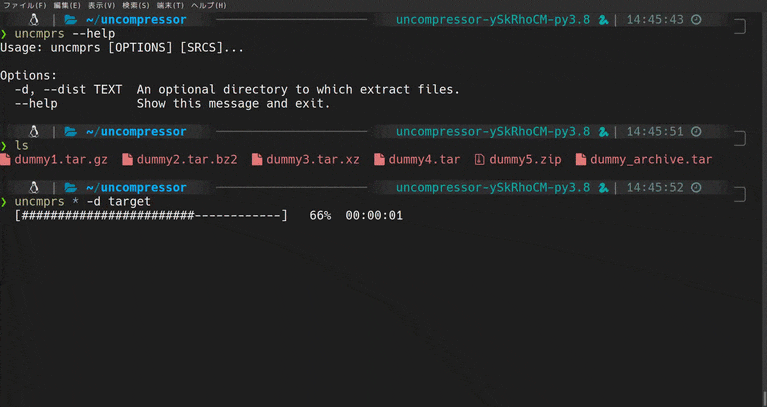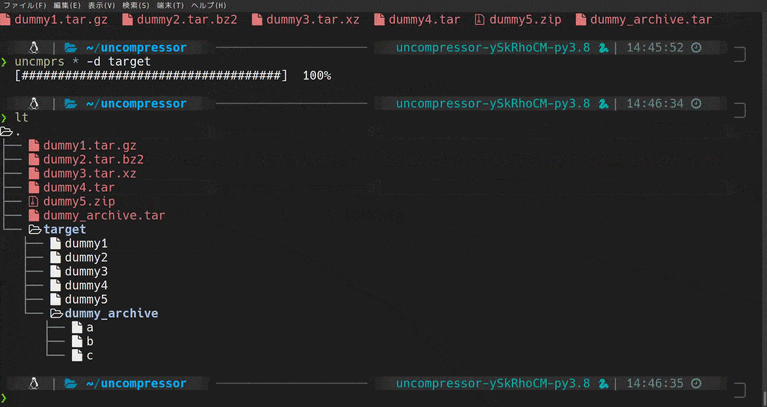
CLIで解凍がちょっと楽になるツールをつくった(Python3)
uncompressorってなに
Python製のCLIファイル解凍ツールです。
動機
僕はGUIよりCUIを好んで使います。
慣れればGUIより作業が捗るという理由からなのですが、いまいちGUIよりスマートじゃないなって思うところもあります。
そしてこれは、CUIでファイルの解凍を行ったことがある人なら誰しもが一度は感じたことがあるんじゃないかと思うのですが、
圧縮/解凍系コマンドって覚えられなくない?
.zip形式のファイルの解凍はunzipですが、.tarだとtar -xvfになるのどうして??ってはじめのうちは思いましたよね。tarはtarでも
gzip,bzip2等、圧縮方法が変わればオプションも変わるので辛い。よく使うコマンドなら多少複雑でもオプションは覚えるけど、解凍ってそんなに頻繁にするもんじゃないし、、、こんな悩みを解決するために作ったのがこのuncompressorです。
デモ
使い方
使い方は至ってシンプルです。
uncmprs {解凍したいファイル} [-d|--dist 解凍先のディレクトリ]インストール
pip install uncompressorでインストールできます。さいごに
バグ等の報告いただけると助かります。
githubはこちら
- 投稿日:2020-05-20T17:00:56+09:00
ubuntu20.04でseleniumをfirefox headlessで動かしてみる
参考にしたサイト
https://qiita.com/derodero24/items/9b38bc4ca99bfd8d5ea3
もろもろのダウンロードとインストール
#sudo apt-install python3-pip firefox-geckodriver -y #sudo pip3 install seleniumFirefox起動まで。
from selenium import webdriver from selenium.webdriver.firefox.options import Options options = Options() options.binary_location = '/usr/bin/firefox' options.add_argument('-headless') driver = webdriver.Firefox(options=options) #あとは参考サイトと同じソースで動きます。メモ
geckodriverとは、Webdriverからfirefoxを使うためのwapperみたいなものらしい。
- 投稿日:2020-05-20T16:24:04+09:00
[Python]変数(定義、出力、値変更)
変数定義
変数名 = 値
"" は '' でも可能。ただ、"" の方が半角全角の判断がしやすい。
2語以上は _ で区切ると良い。name = "chikazu" user_name = "chikazu"変数の出力
変数の場合は "" 又は `` はつけない。
print(name)変数値の変更
①price=100 を 200に上書き。
そのまま定義するだけ。price = 100 print(price) price = 200 print(price)②price=100 に数値を演算したものを変数に代入する。
例は + しているが他の四則演算でも可能。
今回は 100+200 で 300が出力される。price = 100 print(price) price += 200 print(price)
- 投稿日:2020-05-20T15:40:49+09:00
scipyを使って特徴量の相関を考慮したマハラノビス距離を計算する
はじめに
本連載について
こんにちは,(株)日立製作所 Lumada Data Science Lab. の露木です。
化学プラントや工場設備,あるいはもっと身近なモーターや冷蔵庫などの故障予兆検知を行う際に,振動や音,温度,圧力,電圧,消費電力のような値を取得できる複数のセンサーで測定した多次元の時系列数値データを分析することがあります。このような故障予兆検知は,機械学習の分野では異常検知問題として解くことができます。
そこで本連載では時系列数値データの異常検知を最終目標とし,数回の記事に分けてアルゴリズムの基礎的な説明と実装を示していきます。初回の本稿では,後に説明する異常検知アルゴリズムの出発点となるマハラノビス距離について取り上げます。
- 第1回. scipyを使って特徴量の相関を考慮したマハラノビス距離を計算する (本稿)
- 第2回. ホテリングの$T^2$法による多変量正規分布を仮定した異常検知
- 第3回. GMMによる多峰性分布にもとづいたデータの異常検知
- 第4回. 移動窓を使った多次元時系列データの異常検知
想定シナリオ
まず最初に何かの機器が故障する具体的なシナリオを想定して,時系列数値データの異常検知問題に必要な要素技術を考えます。
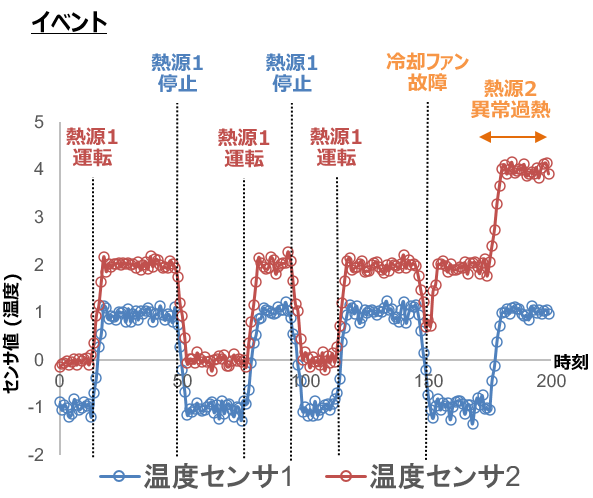
本連載では,図1のように2つの熱源と冷却ファン,2つの温度センサがあるような仮想的な機器を想定します。この機器では,熱源1に接続された運転スイッチが時刻とともに運転,停止,運転,停止と繰り返しており,それに対応して熱源1の温度も上下していきます。一方,熱源2は常に運転状態にありますが,冷却ファンによって一定以下の温度に抑えられています。
ここで,熱源2の冷却ファンが故障したとします。この故障を2つの温度センサの値から見つけ出すことが最終目標です。
2つの温度センサの値を,横軸に時刻をとった時系列数値データとして可視化すると図2のようなグラフになります。正常時は熱源1の運転状態に応じて,2つの温度センサの値が連動して上下します。そして時刻150で冷却ファンが故障すると,熱源2の加熱に対応して温度センサ2の値のみが上昇する異常値が記録されます。
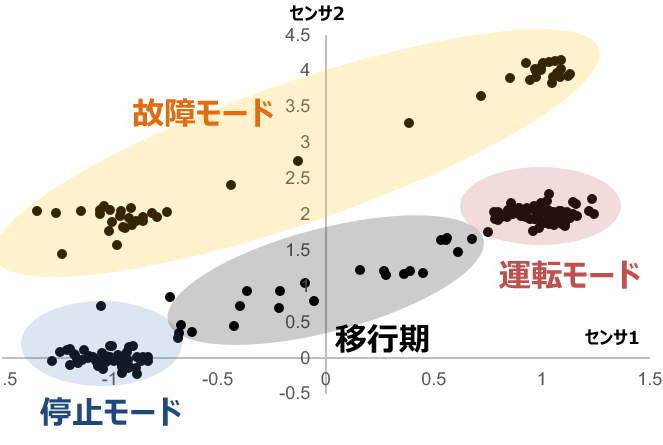
時系列データを各時刻でのセンサ値の組を持つベクトルとして捉え,散布図の形式で可視化すると,異常値の存在がより明確になります。図2と同じデータについて,今度は縦軸・横軸に2つの温度センサの値をとって可視化すると図3のような散布図を得られます。このように可視化すると,データ分析によって熱源1の「運転モード」と「停止モード」,そしてその間の「移行期」および熱源2が過熱した「故障モード」の4つに分類できるように見えてきます。
利用可能なデータは温度センサの値のみであるため,機械学習の言葉で言えば教師なしの分類問題を解くことになります。より具体的に言えば,クラスタリングによって与えられた特徴量空間における「正常なデータ」の分布を学習し,そこから遠く離れたデータを「異常なデータ」とみなせば良いと言えます。これを実現するためには要素技術として以下の4つが必要です。
- 特徴量空間における距離の定義
- 正常データの分布を仮定し,異常なデータと正常なデータを分ける閾値を設定する方法
- 複数のモードに対応できるクラスタリング
- 時系列データに対応するための移動窓の活用
上記の1~4を実装する方法について,先に示した本連載の第1回~第4回で順に説明していきます。
今回扱う内容
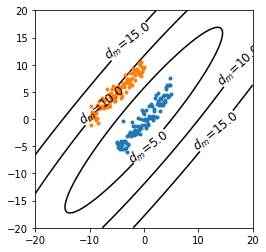
初回である本稿では特徴量空間における距離の定義を扱います。距離の定義は,正常なデータと異常なデータを正しく分離するために重要な役割を果たします。
図3のように横に細長い「移行期」や「故障モード」を分離したクラスタリングは,等方的なユークリッド距離では難しく,マハラノビス距離が有効です。マハラノビス距離は母集団の分布を考慮し,特徴量の間に強い相関がある場合でも正常データと異常データを分離しやすい特徴を持った距離の定義方法です。実際に,マハラノビス距離は異常検知のアルゴリズムとして有名なホテリングの $T^2$ 法やGMM (混合正規分布モデル) の内部で使われています。その理論的な詳細は成書 1 や多くのWeb記事 2 3 4 に譲るとして,本稿ではscipyの距離関数 5 を利用した実装方法を示します。
マハラノビス距離の特徴
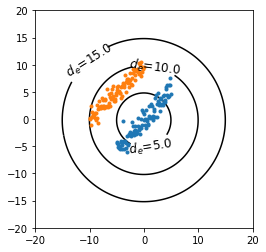
ユークリッド距離の欠点
距離の定義としてユークリッド距離はもっとも著名であり,比較対象として適任といえます。${\bf x}$と${\bf y}$の2つのベクトル間のユークリッド距離$d_e$は次式で計算できます。
d_{e} = \sqrt{ \sum_i ({x}_i - {y}_i)^2}しかし,先に述べたようにユークリッド距離は背景にある母集団の分布を考慮した距離ではありません。特に,特徴量に強い相関がある場合,ユークリッド距離にもとづいたクラスタリングではうまく分離できないことがあります。例えば図4のように,原点 (青色のデータ群の標本平均) を中心としたユークリッド距離による分離を考えます。図4では,ユークリッド距離 $d_e=5$ の等高線では青色のデータ群を包み込むには小さすぎる一方で, $d_e=10$ ではオレンジ色のデータ群も含んでしまいます。
マハラノビス距離の利点
このような問題に対処するため,異常検知の分野ではマハラノビス距離がよく使われます。 1 マハラノビス距離$d_m$は分散共分散行列$\Sigma$を用いて次式で定義されます。
d_{m} = \sqrt{ ({\bf x} - {\bf y})^{\rm T} \Sigma^{-1} ({\bf x} - {\bf y}) }定義式に母集団の分散が含まれることから感覚的にも理解できますが,以下の図5のように原点を中心としたマハラノビス距離の等高線図は分散の大きい方向に引き伸ばされた形式になります。これにより,ユークリッド距離では難しかった青色のデータ群とオレンジ色のデータ群の分離が可能になります。
scipy (Python) による実装
マハラノビス距離の計算方法
最初に結論を述べると,scipyに組み込みの関数 scipy.spatial.distance.mahalanobis() を使えば,以下のように簡単にマハラノビス距離を計算できます。
ただし,numpyのcov関数 はデフォルトで不偏分散を計算する (つまり,$1/(N-1)$で行列要素が規格化されている) ことに注意してください。cov関数に
bias=Trueの引数を与えれば標本分散を計算します (つまり,$1/N$で規格化する)。 6import numpy as np from scipy.spatial import distance X = np.array([[0, 0], [1, 1], [2, 2]]) # 分散共分散行列を計算 cov = np.cov(X.T) # 分散共分散行列の逆行列を計算 cov_i = np.linalg.pinv(cov) # 2つの標本 [1, 1] と [0, 0] のマハラノビス距離を計算する d = distance.mahalanobis([1, 1], [0,0], cov_i) print("マハラノビス距離の計算結果: %1.2f" % d)マハラノビス距離の計算結果: 1.00
なお,scipyにはユークリッド距離を計算する関数 scipy.spatial.distance.euclidean() も用意されており,こちらも簡単に計算できます。 2つの標本 [0, 0] と [1, 1] のマハラノビス距離は
1.00でしたが, ユークリッド距離は1.41となり,母集団の分散に応じてマハラノビス距離で数えたほうがユークリッド距離よりも近いということになりました。# 2つの標本 [1, 1] と [0, 0] のユークリッド距離を計算する d = distance.euclidean([1, 1], [0, 0]) print("ユークリッド距離の計算結果: %1.2f" % d)ユークリッド距離の計算結果: 1.41
トイデータを使った2種類の距離の比較
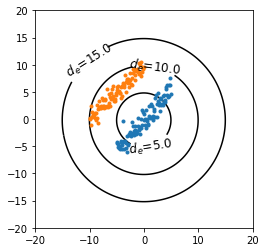
トイデータの性質
マハラノビス距離の計算方法を示したので,次に本稿の冒頭で示した等高線図を描いてユークリッド距離とマハラノビス距離の比較を行います。まずは人工的な右肩上がりの成分に正規分布のノイズを足したトイデータを作成します。
X1 = np.array([(x, x + np.random.normal()) for x in np.arange(-5, 5, 0.1)]) X2 = np.array([(x, 10 + x + np.random.normal()) for x in np.arange(-10, 0, 0.1)])このトイデータは以下の図6のように右肩上がりに偏った分布のデータセットです。
import matplotlib.pyplot as plt # グラフ設定 myfig = plt.figure() myax = myfig.add_subplot(1, 1, 1) myax.set_aspect('equal') plt.ylim(-20, 20) plt.xlim(-20, 20) # 散布図を描画 plt.plot(X1[:,0], X1[:,1], '.') plt.plot(X2[:,0], X2[:,1], '.') plt.show()ユークリッド距離では適切に分離できない
まず,青色のデータ群の中心点 (標本平均) からのユークリッド距離を等高線図として可視化します。図7を見ると,やはりユークリッド距離の等高線は2つのデータ群にまたがっており,回のトイデータのような強い相関を持った特徴量空間に広がった2つのデータ群をうまく分離できないといえます。
# 青色のデータ群の中心 (標本平均) を計算 mean = X1[:,0].mean(), X1[:,1].mean() # z軸値 (標本平均からのユークリッド距離) を計算 z = np.array( [ [(i, j, distance.euclidean([i, j], mean)) for i in np.linspace(-20, 20, 100)] for j in np.linspace(-20, 20, 100)] ) # グラフ設定 myfig = plt.figure() myax = myfig.add_subplot(1, 1, 1) myax.set_aspect('equal') plt.ylim(-20, 20) plt.xlim(-20, 20) # 等高線図を描画 cont = plt.contour(z.transpose()[0], z.transpose()[1], z.transpose()[2], levels=[5,10,15], colors=['k']) cont.clabel(fmt=' $d_e$=%1.1f ', fontsize=12) # 散布図を描画 plt.plot(X1[:,0], X1[:,1], '.') plt.plot(X2[:,0], X2[:,1], '.') plt.show()マハラノビス距離なら適切に分離できる
次に同じ可視化をマハラノビス距離について行います。可視化で得た図8を見れば,マハラノビス距離であれば母集団の分布を考慮するので$d_m=5$の等高線で2つのデータ群を適切に分離できていることがわかります。
# 分散共分散行列を計算 cov = np.cov(X1.T) # z軸値 (標本平均からのマハラノビス距離) を計算 z = np.array( [ [(i, j, distance.mahalanobis([i,j], mean, np.linalg.pinv(cov))) for i in np.linspace(-20, 20, 100)] for j in np.linspace(-20, 20, 100)] ) # グラフ描画 myfig = plt.figure() myax = myfig.add_subplot(1, 1, 1) myax.set_aspect('equal') plt.ylim(-20, 20) plt.xlim(-20, 20) # 等高線図を描画 cont = plt.contour(z.transpose()[0], z.transpose()[1], z.transpose()[2], levels=[5,10,15], colors=['k']) cont.clabel(fmt='$d_m$=%1.1f', fontsize=12) # 散布図の描画 plt.plot(X1[:,0], X1[:,1], '.') plt.plot(X2[:,0], X2[:,1], '.') plt.show()おわりに
本稿で計算したマハラノビス距離を使えばデータを分離できますが,これだけではデータの中心や異常データと正常データを分ける閾値を決定できません。そこで,次回の記事では,ホテリングの$T^2$法 1 を実装する方法を説明します。ホテリングの$T^2$法は,正常データについて正規分布を仮定した上で,データが出現する確率にもとづいて異常度の閾値を決定して異常検知を行う方法です。なお,ホテリングの$T^2$法では異常度としてマハラノビス距離の2乗値を使います。
また,混合正規分布モデル (GMM) によるクラスタリングでも一般にマハラノビス距離が使われます。一方,クラスタリングの手法として著名なK-Means法は,ユークリッド距離を内部的に使う事が多いアルゴリズムです。一般に,K-meansよりもGMMのほうが性能が良くなることが多い 7 のですが,その一因としてマハラノビス距離の採用が挙げられます。
参考文献
井手, 入門 機械学習による異常検知 Rによる実践ガイド, コロナ社, 2015. ↩
Comparing different clustering algorithms on toy datasets scikit-learn 0.22.2 documentation ↩
- 投稿日:2020-05-20T14:22:42+09:00
Python SDKを使ってImage Searchの類似検索を試す【検索編】
今回の目的(Python SDKを使って検索してみる)
前回、Image Searchの設定と画像取り込みまで終わりました。今回は、前回作ったImage Searchにプログラムでアクセスしてみようと思います。
実装環境の構築
今回はPoetryを使ってでプロジェクト作成します。Python、Poetyの開発環境が整っていない方はインストールを行っておいてください。(サンプルはPoetyで作成していますが、pipenvでも構いません)
参考:Windows+Scoopの場合
PS C:\Users\user> pip install poetry Collecting poetry Downloading https://files.pythonhosted.org/packages/19/52/ffdd04d17616cbfa47e0c145d0df2d1cf4280015c39db0a055fbfb06a8e2/poetry-1.0.5-py2.py3-none-any.whl (220kB) |████████████████████████████████| 225kB 3.3MB/s Collecting clikit<0.5.0,>=0.4.2 (from poetry) Downloading https://files.pythonhosted.org/packages/7a/45/91c67a4f623bd09e98fe72c01e0139a9ae70bffdd658dce12e4a8be3c1e3/clikit-0.4.3-py2.py3-none-any.whl (88kB) |████████████████████████████████| 92kB 6.1MB/s Collecting tomlkit<0.6.0,>=0.5.11 (from poetry) Downloading https://files.pythonhosted.org/packages/7d/8c/c3ee9cd41b2df781b2dc39c31209724b4f04a3110b46531de2e661ace186/tomlkit-0.5.11-py2.py3-none-any.whl Collecting requests-toolbelt<0.9.0,>=0.8.0 (from poetry) Downloading https://files.pythonhosted.org/packages/97/8a/d710f792d6f6ecc089c5e55b66e66c3f2f35516a1ede5a8f54c13350ffb0/requests_toolbelt-0.8.0-py2.py3-none-any.whl (54kB) |████████████████████████████████| 61kB ... Collecting shellingham<2.0,>=1.1 (from poetry) Downloading https://files.pythonhosted.org/packages/9f/6b/160e80c5386f7820f0a9919cc9a14e5aef2953dc477f0d5ddf3f4f2b62d0/shellingham-1.3.2-py2.py3-none-any.whl Collecting pyparsing<3.0,>=2.2 (from poetry) Downloading https://files.pythonhosted.org/packages/8a/bb/488841f56197b13700afd5658fc279a2025a39e22449b7cf29864669b15d/pyparsing-2.4.7-py2.py3-none-any.whl (67kB) |████████████████████████████████| 71kB 4.5MB/s Collecting keyring<21.0.0,>=20.0.1; python_version >= "3.5" and python_version < "4.0" (from poetry) Downloading https://files.pythonhosted.org/packages/f1/07/0afb82d449d210a332d126978634470abdd0c754128a9ead8bbe78eb1b43/keyring-20.0.1-py2.py3-none-any.whl Collecting html5lib<2.0,>=1.0 (from poetry) Downloading https://files.pythonhosted.org/packages/a5/62/bbd2be0e7943ec8504b517e62bab011b4946e1258842bc159e5dfde15b96/html5lib-1.0.1-py2.py3-none-any.whl (117kB) |████████████████████████████████| 122kB 6.8MB/s Collecting pkginfo<2.0,>=1.4 (from poetry) Downloading https://files.pythonhosted.org/packages/e6/d5/451b913307b478c49eb29084916639dc53a88489b993530fed0a66bab8b9/pkginfo-1.5.0.1-py2.py3-none-any.whl Collecting cleo<0.8.0,>=0.7.6 (from poetry) Downloading https://files.pythonhosted.org/packages/4b/9d/10a5923c14c4f0faa98216af5262938f468af76101d9cd124ad2054943c7/cleo-0.7.6-py2.py3-none-any.whl Collecting pexpect<5.0.0,>=4.7.0 (from poetry) Downloading https://files.pythonhosted.org/packages/39/7b/88dbb785881c28a102619d46423cb853b46dbccc70d3ac362d99773a78ce/pexpect-4.8.0-py2.py3-none-any.whl (59kB) |████████████████████████████████| 61kB 1.9MB/s Collecting pyrsistent<0.15.0,>=0.14.2 (from poetry) Downloading https://files.pythonhosted.org/packages/8c/46/4e93ab8a379d7efe93f20a0fb8a27bdfe88942cc954ab0210c3164e783e0/pyrsistent-0.14.11.tar.gz (104kB) |████████████████████████████████| 112kB 3.3MB/s Collecting jsonschema<4.0,>=3.1 (from poetry) Downloading https://files.pythonhosted.org/packages/c5/8f/51e89ce52a085483359217bc72cdbf6e75ee595d5b1d4b5ade40c7e018b8/jsonschema-3.2.0-py2.py3-none-any.whl (56kB) |████████████████████████████████| 61kB 3.8MB/s Collecting cachecontrol[filecache]<0.13.0,>=0.12.4 (from poetry) Downloading https://files.pythonhosted.org/packages/18/71/0a9df4206a5dc5ae7609c41efddab2270a2c1ff61d39de7591dc7302ef89/CacheControl-0.12.6-py2.py3-none-any.whl Collecting cachy<0.4.0,>=0.3.0 (from poetry) Downloading https://files.pythonhosted.org/packages/82/e6/badd9af6feee43e76c3445b2621a60d3d99fe0e33fffa8df43590212ea63/cachy-0.3.0-py2.py3-none-any.whl Collecting requests<3.0,>=2.18 (from poetry) Downloading https://files.pythonhosted.org/packages/1a/70/1935c770cb3be6e3a8b78ced23d7e0f3b187f5cbfab4749523ed65d7c9b1/requests-2.23.0-py2.py3-none-any.whl (58kB) |████████████████████████████████| 61kB 3.8MB/s Collecting pastel<0.3.0,>=0.2.0 (from clikit<0.5.0,>=0.4.2->poetry) Downloading https://files.pythonhosted.org/packages/06/cd/999508cef5e92fea672a420e3164bb02c01a763d5d6d5149312194c9178c/pastel-0.2.0-py2.py3-none-any.whl Collecting pylev<2.0,>=1.3 (from clikit<0.5.0,>=0.4.2->poetry) Downloading https://files.pythonhosted.org/packages/40/1c/7dff1d242bf1e19f9c6202f0ba4e6fd18cc7ecb8bc85b17b2d16c806e228/pylev-1.3.0-py2.py3-none-any.whl Collecting pywin32-ctypes!=0.1.0,!=0.1.1; sys_platform == "win32" (from keyring<21.0.0,>=20.0.1; python_version >= "3.5" and python_version < "4.0"->poetry) Downloading https://files.pythonhosted.org/packages/9e/4b/3ab2720f1fa4b4bc924ef1932b842edf10007e4547ea8157b0b9fc78599a/pywin32_ctypes-0.2.0-py2.py3-none-any.whl Collecting six>=1.9 (from html5lib<2.0,>=1.0->poetry) Downloading https://files.pythonhosted.org/packages/65/eb/1f97cb97bfc2390a276969c6fae16075da282f5058082d4cb10c6c5c1dba/six-1.14.0-py2.py3-none-any.whl Collecting webencodings (from html5lib<2.0,>=1.0->poetry) Downloading https://files.pythonhosted.org/packages/f4/24/2a3e3df732393fed8b3ebf2ec078f05546de641fe1b667ee316ec1dcf3b7/webencodings-0.5.1-py2.py3-none-any.whl Collecting ptyprocess>=0.5 (from pexpect<5.0.0,>=4.7.0->poetry) Downloading https://files.pythonhosted.org/packages/d1/29/605c2cc68a9992d18dada28206eeada56ea4bd07a239669da41674648b6f/ptyprocess-0.6.0-py2.py3-none-any.whl Requirement already satisfied: setuptools in c:\users\user\scoop\apps\python\3.8.2\lib\site-packages (from jsonschema<4.0,>=3.1->poetry) (41.2.0) Collecting attrs>=17.4.0 (from jsonschema<4.0,>=3.1->poetry) Downloading https://files.pythonhosted.org/packages/a2/db/4313ab3be961f7a763066401fb77f7748373b6094076ae2bda2806988af6/attrs-19.3.0-py2.py3-none-any.whl Collecting msgpack>=0.5.2 (from cachecontrol[filecache]<0.13.0,>=0.12.4->poetry) Downloading https://files.pythonhosted.org/packages/54/df/51af837ceaeda25563c99e13f772859784dd64fe6add68bfeade022b6aee/msgpack-1.0.0-cp38-cp38-win_amd64.whl (73kB) |████████████████████████████████| 81kB 5.1MB/s Collecting lockfile>=0.9; extra == "filecache" (from cachecontrol[filecache]<0.13.0,>=0.12.4->poetry) Downloading https://files.pythonhosted.org/packages/c8/22/9460e311f340cb62d26a38c419b1381b8593b0bb6b5d1f056938b086d362/lockfile-0.12.2-py2.py3-none-any.whl Collecting idna<3,>=2.5 (from requests<3.0,>=2.18->poetry) Downloading https://files.pythonhosted.org/packages/89/e3/afebe61c546d18fb1709a61bee788254b40e736cff7271c7de5de2dc4128/idna-2.9-py2.py3-none-any.whl (58kB) |████████████████████████████████| 61kB 3.8MB/s Collecting certifi>=2017.4.17 (from requests<3.0,>=2.18->poetry) Downloading https://files.pythonhosted.org/packages/57/2b/26e37a4b034800c960a00c4e1b3d9ca5d7014e983e6e729e33ea2f36426c/certifi-2020.4.5.1-py2.py3-none-any.whl (157kB) |████████████████████████████████| 163kB ... Collecting chardet<4,>=3.0.2 (from requests<3.0,>=2.18->poetry) Downloading https://files.pythonhosted.org/packages/bc/a9/01ffebfb562e4274b6487b4bb1ddec7ca55ec7510b22e4c51f14098443b8/chardet-3.0.4-py2.py3-none-any.whl (133kB) |████████████████████████████████| 143kB 6.4MB/s Collecting urllib3!=1.25.0,!=1.25.1,<1.26,>=1.21.1 (from requests<3.0,>=2.18->poetry) Downloading https://files.pythonhosted.org/packages/e1/e5/df302e8017440f111c11cc41a6b432838672f5a70aa29227bf58149dc72f/urllib3-1.25.9-py2.py3-none-any.whl (126kB) |████████████████████████████████| 133kB 6.4MB/s Installing collected packages: pastel, pylev, clikit, tomlkit, idna, certifi, chardet, urllib3, requests, requests-toolbelt, shellingham, pyparsing, pywin32-ctypes, keyring, six, webencodings, html5lib, pkginfo, cleo, ptyprocess, pexpect, pyrsistent, attrs, jsonschema, msgpack, lockfile, cachecontrol, cachy, poetry WARNING: The script chardetect.exe is installed in 'c:\users\user\scoop\apps\python\3.8.2\Scripts' which is not on PATH. Consider adding this directory to PATH or, if you prefer to suppress this warning, use --no-warn-script-location. WARNING: The script keyring.exe is installed in 'c:\users\user\scoop\apps\python\3.8.2\Scripts' which is not on PATH. Consider adding this directory to PATH or, if you prefer to suppress this warning, use --no-warn-script-location. WARNING: The script pkginfo.exe is installed in 'c:\users\user\scoop\apps\python\3.8.2\Scripts' which is not on PATH. Consider adding this directory to PATH or, if you prefer to suppress this warning, use --no-warn-script-location. Running setup.py install for pyrsistent ... done WARNING: The script jsonschema.exe is installed in 'c:\users\user\scoop\apps\python\3.8.2\Scripts' which is not on PATH. Consider adding this directory to PATH or, if you prefer to suppress this warning, use --no-warn-script-location. WARNING: The script doesitcache.exe is installed in 'c:\users\user\scoop\apps\python\3.8.2\Scripts' which is not on PATH. Consider adding this directory to PATH or, if you prefer to suppress this warning, use --no-warn-script-location. WARNING: The script poetry.exe is installed in 'c:\users\user\scoop\apps\python\3.8.2\Scripts' which is not on PATH. Consider adding this directory to PATH or, if you prefer to suppress this warning, use --no-warn-script-location. Successfully installed attrs-19.3.0 cachecontrol-0.12.6 cachy-0.3.0 certifi-2020.4.5.1 chardet-3.0.4 cleo-0.7.6 clikit-0.4.3 html5lib-1.0.1 idna-2.9 jsonschema-3.2.0 keyring-20.0.1 lockfile-0.12.2 msgpack-1.0.0 pastel-0.2.0 pexpect-4.8.0 pkginfo-1.5.0.1 poetry-1.0.5 ptyprocess-0.6.0 pylev-1.3.0 pyparsing-2.4.7 pyrsistent-0.14.11 pywin32-ctypes-0.2.0 requests-2.23.0 requests-toolbelt-0.8.0 shellingham-1.3.2 six-1.14.0 tomlkit-0.5.11 urllib3-1.25.9 webencodings-0.5.1 WARNING: You are using pip version 19.2.3, however version 20.1 is available. You should consider upgrading via the 'python -m pip install --upgrade pip' command.Python プロジェクトの作成
それでは、Pythonのプロジェクトを作ってみましょう
Python プロジェクトの作成
PS C:\Users\user\Documents\py-sample> poetry new sample Created package sample in sample PS C:\Users\user\Documents\py-sample> ls Directory: C:\Users\user\Documents\py-sample Mode LastWriteTime Length Name ---- ------------- ------ ---- d---- 2020/05/14 16:34 sample PS C:\Users\user\Documents\py-sample> cd .\sample\ PS C:\Users\user\Documents\py-sample\sample> ls Directory: C:\Users\user\Documents\py-sample\sample Mode LastWriteTime Length Name ---- ------------- ------ ---- d---- 2020/05/14 16:34 sample d---- 2020/05/14 16:34 tests -a--- 2020/05/14 16:34 295 pyproject.toml -a--- 2020/05/14 16:34 0 README.rst依存関係の追加
Alibaba CloudのSDKを追加します。
PS C:\Users\user\Documents\py-sample\sample> poetry add aliyun-python-sdk-core Using version ^2.13.16 for aliyun-python-sdk-core Updating dependencies Resolving dependencies... Writing lock file Package operations: 3 installs, 0 updates, 0 removals - Installing jmespath (0.10.0) - Installing pycryptodome (3.9.7) - Installing aliyun-python-sdk-core (2.13.16)PS C:\Users\user\Documents\py-sample\sample> poetry add aliyun-python-sdk-imagesearch Using version ^2.0.0 for aliyun-python-sdk-imagesearch Updating dependencies Resolving dependencies... Writing lock file Package operations: 1 install, 0 updates, 0 removals - Installing aliyun-python-sdk-imagesearch (2.0.0)前回作成したImage Searchにアクセスする
アクセスキーとシークレットキーは前回作成したものを使用します。
また、リージョンとエンドポイントについては、以下の表から設定します。今回は日本(東京)を設定します。
リージョン エンドポイント シンガポール imagesearch.ap-southeast-1.aliyuncs.com 中国 (香港) imagesearch.cn-hongkong.aliyuncs.com 日本 (東京) imagesearch.ap-northeast-1.aliyuncs.com オーストラリア (シドニー) imagesearch.ap-southeast-2.aliyuncs.com imagesearch.py# -*- coding: utf8 -*- from aliyunsdkcore.client import AcsClient import base64 import aliyunsdkimagesearch.request.v20190325.AddImageRequest as AddImageRequest import aliyunsdkimagesearch.request.v20190325.DeleteImageRequest as DeleteImageRequest import aliyunsdkimagesearch.request.v20190325.SearchImageRequest as SearchImageRequest # AccessKey ACCESS_KEY = "XXXXXXXXXXXXXXX" SEACRET_KEY = "YYYYYYYYYYYYYY" # AcsClient インスタンスの作成 client = AcsClient(ACCESS_KEY, SEACRET_KEY, "ap-northeast-1") # 検索 request = SearchImageRequest.SearchImageRequest() request.set_endpoint("imagesearch.ap-northeast-1.aliyuncs.com") request.set_InstanceName("itemsearch") with open('search01.jpg', 'rb') as imgfile: encoded_pic_content = base64.b64encode(imgfile.read()) request.set_PicContent(encoded_pic_content) response = client.do_action_with_exception(request) print(response)result.jsonb'{"Msg":"success","Head":{"DocsFound":11,"DocsReturn":10,"SearchTime":100},"RequestId":"D56A620C-3EF5-461C-85FE-AFC89B6C79BB","Auctions":[{"CategoryId":9,"PicName":"12.jpg","CustomContent":"k1:v12,k2:v211,k3:v311","ProductId":"1011","SortExprValues":"3.07306838035583;217"},{"CategoryId":9,"PicName":"11.jpg","CustomContent":"k1:v11,k2:v210,k3:v310","ProductId":"1010","SortExprValues":"2.97270393371582;222"},{"CategoryId":9,"PicName":"09.jpg","CustomContent":"k1:v09,k2:v208,k3:v308","ProductId":"1008","SortExprValues":"2.87724995613098;238"},{"CategoryId":9,"PicName":"10.jpg","CustomContent":"k1:v10,k2:v209,k3:v309","ProductId":"1009","SortExprValues":"2.79507827758789;235"},{"CategoryId":9,"PicName":"02.jpg","CustomContent":"k1:v02,k2:v201,k3:v301","ProductId":"1001","SortExprValues":"2.67687916755676;251"},{"CategoryId":9,"PicName":"05.jpg","CustomContent":"k1:v05,k2:v204,k3:v304","ProductId":"1004","SortExprValues":"2.67470407485962;249"},{"CategoryId":9,"PicName":"06.jpg","CustomContent":"k1:v06,k2:v205,k3:v305","ProductId":"1005","SortExprValues":"2.66586232185364;254"},{"CategoryId":9,"PicName":"04.jpg","CustomContent":"k1:v04,k2:v203,k3:v303","ProductId":"1003","SortExprValues":"2.63756942749023;255"},{"CategoryId":9,"PicName":"01.jpg","CustomContent":"k1:v01,k2:v200,k3:v300","ProductId":"1000","SortExprValues":"2.57631182670593;270"},{"CategoryId":9,"PicName":"07.jpg","CustomContent":"k1:v07,k2:v206,k3:v306","ProductId":"1006","SortExprValues":"2.52564144134521;253"}],"PicInfo":{"CategoryId":9,"AllCategories":[{"Id":0,"Name":"Tops"},{"Id":1,"Name":"Dress"},{"Id":2,"Name":"Bottoms"},{"Id":3,"Name":"Bag"},{"Id":4,"Name":"Shoes"},{"Id":5,"Name":"Accessories"},{"Id":6,"Name":"Snack"},{"Id":7,"Name":"Makeup"},{"Id":8,"Name":"Bottle"},{"Id":9,"Name":"Furniture"},{"Id":20,"Name":"Toy"},{"Id":21,"Name":"Underwear"},{"Id":22,"Name":"Digital device"},{"Id":88888888,"Name":"Other"}],"Region":"140,474,36,578"},"Code":0,"Success":true}'前回、管理画面から投げた結果と同じ結果が出力されました。Pythonでも簡単に検索する事ができました。
- 投稿日:2020-05-20T14:13:26+09:00
statsmodelsによるディッキーフラー検定
この記事は数日前に投稿したものを取り下げ、再度改良して掲載しています。
分析の対象となる時系列データがランダムウォークにしたがうかどうかを知ることはつぎの2つの点から重要です。
1) 発生したトレンドが見せかけのトレンドか、トレンドはたまたま生じたものなのか、
2) 2つの時系列が相関を示したときにそれらは見せかけの相関か、たまたま発生したものではないのか、
という点です。多くの経済時系列や株価のレベル(原系列)はランダムウォークにしたがいます。ランダムウォークにしたがう時系列は時間の経過にともない平均や分散が以前のものと異なってしまう非定常時系列です。ランダムウォークでは時間の経過とともに平均は確率的に変化し、分散は時間の経過の平方根に比例して増大します。非定常時系列にもいろいろあります。時間の経過とともに確実に平均値が上昇したり下落するトレンド定常性もその1つです。ランダムウォークであるかどうかを判定するにはその時系列が単位根を持つかどうかで判断します。その方法としてディッキーフラー検定があります。しかし、検出力が低いとよく指摘されています。そこで少し調べてみようと思います。
DF検定に移る前に
まず1次の自己回帰モデル(AR(1))を
$y_t=\rho y_{t-1}+u_t$
とします。$\rho$が1であればこのAR(1)は単位根をもつといいます。$y_t$はランダムウォークです。$\Delta y_t=y_t-y_{t-1}$が時間の経過に対して平均値と分散が一定の定常過程であるとき、それを1次の和分といいます。この単位根過程は
$y_1=y_0+e_1$
$y_2=y_1+e_2$
$y_3=y_2+e_3$
...
となり、t=2とt=3を書き換えると
$y_2=(y_0+e_1)+e2=x_0+e1+e2$
$y_3=(y_0+e_1+e_2)+e_3=y_0+e_1+e_2+e_3$
となります。一般化すると
$y_t=y_0+\sum_{i=1}^te_i$
となり初期値と乱数の和になります。$\rho>1$では時系列は発散してしまうので、$\rho<1$とします。そうするとAR(1)モデルも同様に
$y_1=ρ\cdot y_0+e_1$
$y_2=ρ\cdot y_1+e_2$
$y_3=ρ\cdot y_2+e_3$
$y_2$と$y_3$を書き換えると
$y_2=ρ\cdot (ρ\cdot y_0+e_1)+e2=ρ^2\cdot y_0+ρ\cdot e_1+e2$
$y_3=ρ\cdot (ρ^2\cdot y_0 +ρ\cdot e_1+e_2)+e_3$
$=ρ^3y_0+ρ^2\cdot (e_1)+ρ\cdot (e_2)+e_3$
$=ρ^3y_0+\sum_{i=1}^3ρ^{3-i}\cdot (e_i)$
となります。一般化すると
$y_t=ρ^ty_0+\sum_{i=1}^tρ^{t-i}\cdot (e_i)$
となります。ここまでをpythonを使って確かめましょう。
import numpy as np import matplotlib.pyplot as plt def generate(y0,rho,sigma,nsample): e = np.random.normal(size=nsample) y=[] y.append(rho*y0+e[0]) for i,u in enumerate(e[1:]): y.append(rho*y[i]+u) plt.plot(y) generate(1,1,1,100)つぎに$rho=0.9$にしてみます。
generate(1,0.9,1,100)
ある値に収束していくことが分かります。つぎにどちらもsigma=0にしてみましょう。generate(1,1,0,100)generate(1,0.9,0,100)ランダムウォークでは1のままで、AR(1)ではゼロに収束していきます。これでは不便なので、AR(1)モデルをつぎのように書き換えます。
$y_t=c+\rho y_{t-1}+u_t$
とします。つぎにこの系列を足し合わせます。
$\sum_{t=2}^n y_t=\sum_{t=2}^n c+\sum_{t=2}^n\rho y_{t-1}+\sum_{t=2}^nu_t$$y_t$の平均値を$\mu$とすると、nが十分に大きければ、$\sum_{t=2}^nu_t=0$なので
$\mu=c+\rho \mu$となります。したがって
$c=\mu(1-\rho)$
となります。これを使って関数generateを書き換えましょう。def generate(y0,rho,c,sigma,nsample): e = np.random.normal(size=nsample) y=[] y.append(rho*y0+e[0]*sigma+c) for i,u in enumerate(e[1:]): y.append(rho*y[i]+u*sigma+c) plt.plot(y) generate(1,0.9,0.1,0,100)となりました。これでAR(1)は時間が経過しても平均値は一定です。sigmaを小さくして確認してみましょう。
generate(1,0.9,0.1,0.01,100)何となく、中心回帰しているのが分かると思います。AR(1)モデルはある条件を満たせば定常(弱定常)になります。
このcのことをドリフト率と呼んだりします。ドリフト率というと平均が変化する率のことです。しかし、ここでは変化しません。ただし、cが
$c>\mu(1-\rho)$であったり、$c<\mu(1-\rho)$であると変化する率になります。確率論では、確率的ドリフトは、確率過程の平均値の変化のことです。同じような概念でドリフト率がありますが、これは確率的に変化しません。非確率的なパラメータです。
長期にわたる事象に関する長期的な研究では、時系列の特性を、トレンド成分、周期成分、および確率的な成分(確率的ドリフト)に分解して概念化することがよくあります。周期的および確率的ドリフトの成分は、自己相関分析と傾向(トレンド)の階差を取ることで識別されます。自己相関分析は、適合モデルの正しい位相を特定するのに役立ち、階差を繰り返すことは確率的ドリフト成分をホワイトノイズに変換します。
トレンド定常
トレンド定常過程$y_t$は、$y_t = f(t)+ u_t$に従います。ここで、tは時間、fは決定論的関数、$u_t$は長期的な平均値がゼロとなる定常確率変数です。この場合、確率的項は定常的であり、したがって確率的ドリフトはありません。確定的な成分f(t)は長期的に一定に保たれる平均値を持ちませんが、時系列自体がドリフトする可能性があります。この確定的ドリフトは、tで$y_t$を回帰し、定常な残差がえられれば、データから削除して求めることができます。もっとも簡単なトレンド定常過程は$y_t=d \cdot t +u_t$です。$d$は定数です。
階差定常
単位根(定常)過程は、$y_t = y_ {t-1} + c + u_t$に従います。$u_t$は長期的にはゼロとなる平均値をもつ定常確率変数です。ここでcは非確率的ドリフトパラメーターです。ランダムショック$u_t$がなくても、yの平均は期間ごとにcだけ変化します。この非定常性は1次の階差を取ることでデータから削除できます。階差された変数$z_t = y_t-y_{t-1}=c+u_t$はcの長期的に一定となる平均値を持ち、したがってこの平均値にドリフトはありません。つぎにc = 0として考えてみます。この単位根過程は、$y_t = y_ {t-1} + c + u_t$です。$u_t$は定常過程ですが、その確率的なショック$u_t$の存在により、ドリフト、特に確率論的ドリフトが$y_t$に生じます。一度発生したゼロ以外の値uは同じ期間のyに組み込まれます。これは、1期間後にyの1期間遅れの値になり、次の期のy値にも影響します。 yは次々に新しいy値に影響を与えます。したがって、最初のショックがyに影響した後、その値はyの平均に永久に組み込まれるため、確率的なドリフトが生じるのです。ですからこのドリフトも、最初にyを微分してドリフトしないzを取得することで除くことができます。この確率的ドリフトは$c\ne0$でも生じます。ですから$y_t = y_ {t-1} + c + u_t$でも生じます。cは非確率的ドリフトパラメーターと説明しました。なぜ確定的なドリフトではないのでしょうか?実は、$c+u_t$から生じるドリフトは確率過程の一部です。
numpy.random.normal
numpyのリファレンスから
numpy.random.normal(loc=0.0, scale=1.0, size=None)
loc float or array_like of floats: 分布の平均値
scalefloat or array_like of floats:分布の標準偏差
です。ここでlocの意味を考えてみます。この分布から生成される標本の平均値は$N(μ,σ^2/n)$に近似的に従います。その仕組みをみてみましょう。あえてloc=1,scale=1.0とします。n=2としてその標本平均と標準偏差を求めてみました。ただし、計算方法は2つです。1つは
$z_t$の平均と標準偏差を求めます。
もう1つは
それぞれの観測値$z_t$から1を引きその標本の平均と標準偏差を求めます。そしてその標本平均に1を足します。import numpy as np import matplotlib.pyplot as plt s = np.random.normal(1, 1, 2) print(np.mean(s),np.std(s)) o=np.ones(len(s)) Print(np.mean(s-o)+1,np.std(s-o)) # 0.9255104221128653 0.5256849930003175 # 0.9255104221128653 0.5256849930003175 # 標本から1を引いて、その平均値に1を足した。結果は同じになりました。これはnp.random.normal(1,1,n)が1/n+np.random.normal(0,1,n)で乱数を生成していることが分かります。しかし、これはコンピュータの中での話です。実際にはどちらが確率的ドリフトを作り出しているかは判別不能なはずです。そこでn=2とn=100の場合について1000000回データを生成してその結果がどうなるかを見てみましょう。
階差定常過程の標本の算術平均の分布
階差された変数$z_t = y_t-y_{t-1}=c+u_t$はcの長期的に一定となる平均値を持ちます。その標本平均はnが大きければ、$N(μ,σ^2/n)$に近似的に従います。したがって、平均値は一定ではありません。しかし、この場合には平均値にドリフトはありません。長期的に一定となる平均値をもちます。
m1=[] trial=1000000 for i in range(trial): s = np.random.normal(1, 1, 2) m1.append(np.mean(s)) m2=[] for i in range(trial): s = np.random.normal(1, 1, 100) m2.append(np.mean(s)) bins=np.linspace(np.min(s),np.max(s),1000) plt.hist(m1,bins=bins,label='n=2') plt.hist(m2,bins=bins,label='n=100',alpha=0.5) plt.legend() plt.show()標本平均も標準偏差も分布を形成し、一意には定まりません。一意に定まるためにはnはもっともっと大きくする必要があります。したがって、時系列の特性を、トレンド成分、周期成分、および確率的な成分(確率的ドリフト)に分解して概念化しますが、確率的ドリフトとドリフト率の扱いには注意が必要です。
単位根過程では確率的ドリフトが発生します。もし、確定的なドリフト率があったとしてもそれは識別が不可能です。
最終日の単位根をもつyの分布
単位根過程では標本の数が増えるにつれて標準偏差は大きくなっていきます。標本の大きさがnのyの平均値はc=0ではゼロですが、標準偏差$\sqrt{n \cdot \sigma^2}$が大きくなった分だけ、その分布は広がります。
m1=[] trial=1000000 for i in range(trial): s = np.cumsum(np.random.normal(0, 1, 2)) m1.append(s[-1]) m2=[] for i in range(trial): s = np.cumsum(np.random.normal(0, 1, 100)) m2.append(s[-1]) bins=np.linspace(np.min(m2),np.max(m2),1000) plt.hist(m1,bins=bins,label='n=2') plt.hist(m2,bins=bins,label='n=100',alpha=0.5) plt.legend() plt.show()先ほどとは異なる分布になりました。標本の大きさが大きいほうの分布の広がりが大きくなります。ためしに1つのsの系列をプロットして見ましょう。
plt.plot(s)この場合、階段的にそれぞれの標本のレベルが変化していきます。この変化は確率的なショック$u_t$によるものです。それが加算されていったものがプロットされています。確率的ドリフトの系列がプロットされています。これが大きなトレンドを形成することがあります。それはn=100の分布図でいえば両端の領域に相当します。どの範囲かはトレンドの定義によります。そのトレンドは確率的トレンドです。これはトレンド定常性がつくる確定的トレンドと明確に区別されます。
1次の自己回帰過程
1次の自己回帰過程はつぎに様に書き換えることができます。
$x_1=ρ\cdot x_0+y_1+e_1$
$x_2=ρ\cdot x_1+y_2+e_2$
$x_3=ρ\cdot x_2+y_3+e_3$$x_2=ρ\cdot (ρ\cdot x_0+y_1+e_1)+e2=ρ^2\cdot x_0+ρ\cdot (y_1+e1)+y_2+e2$
$x_3=ρ\cdot (ρ\cdot ρ\cdot x_0+ρ\cdot (y_1+e_1)+y_2+e_2)+y_3+e_3$
$=ρ^3x_0+ρ^2\cdot (y_1+e_1)+ρ\cdot (y_2+e_2)+y_3+e_3$
$=ρ^3x_0+\sum_{i=1}^n\rho^{n-i}(y_i+e_i)$
ここで$y_i=c+d\cdot i$$\rho=1$のときランダムウォーク過程となります。
# 初期化 import numpy as np import statsmodels.api as sm import matplotlib.pyplot as plt from scipy.stats import norm from statsmodels.tsa.stattools import adfuller #np.random.seed(0)# AR(1)過程の生成 # x0は価格の初期値 # nsampleは1回の試行の期間の数 # rho=1: random walk ## c=c1-rho はドリフト、c1=1のときはドリフト無しランダムウォーク dは確定的トレンド # rho<1で1次の自己回帰モデル ## x0とcとdの効果は時間の経過とともに消滅 def ar1(rho,x0,c1,d,sigma,nsample): e = np.random.normal(size=nsample)#random variable rhoo=np.array([rho ** num for num in range(nsample)]) #effects of rho rhoo_0=np.array([rho**(num+1) for num in range(nsample)]) #effects of rho t=np.arange(nsample) #deterministic trend one=np.ones(nsample) #effect of x0 x00=x0*one*rhoo_0 #effect of rho to x0 c=c1-rho # constant of autoregressive mdoel c0=np.cumsum(c*one*rhoo) #effect of rho to c d0=(d*t*rhoo) # effect of rho to determinist trend y=np.cumsum(rhoo*e*sigma)+x00+c0+d0 return yAR(1)過程の最終日のyの分布
m1=[] trial=1000000 nsample=250 x0=1 for i in range(trial): s = np.cumsum(np.random.normal(0, 1, nsample)) m1.append(s[-1]+x0) def generate(rho,x0,c1,sigma,nsample,trial): m=[] for i in range(trial): e = np.random.normal(0, 1, nsample) rhoo=np.array([rho ** num for num in range(nsample)]) #effects of rho rhoo_0=np.array([rho**(num+1) for num in range(nsample)]) #effects of rho one=np.ones(nsample) #effect of x0 x00=x0*one*rhoo_0 #effect of rho to x0 c=c1-rho # constant of autoregressive mdoel c0=np.cumsum(c*one*rhoo) #effect of rho to c s=np.cumsum(rhoo*e*sigma)+x00+c0 m.append(s[-1]) return m c1=1 x0=1 rho=0.99 sigma=1 m2=generate(rho,x0,c1,sigma,nsample,trial) rho=0.9 m3=generate(rho,x0,c1,sigma,nsample,trial) rho=0.5 m4=generate(rho,x0,c1,sigma,nsample,trial) bins=np.linspace(np.min(m1),np.max(m1),1000) plt.hist(m1,bins=bins,label='rw') plt.hist(m2,bins=bins,label='ar1 rho=0.99',alpha=0.5) plt.hist(m3,bins=bins,label='ar1 rho=0.9',alpha=0.5) plt.hist(m4,bins=bins,label='ar1 rho=0.5',alpha=0.5) plt.legend() plt.show()
1次の自己回帰モデルでは$\rho$が1より小さくなるにしたがい、初期値の周りをうろうろするのが分かります。$\rho$が1に近づくとランダムウォークとの区別はつきにくくなります。つぎの図はn=7とした場合です。7日程度ですとどの$\rho$でも広がり具合は似たり寄ったりです。検出力(statsmodelsによる統計的仮説検定 入門)
仮説検定では帰無仮説と対立仮説を立てます。帰無仮説は母数で構成します。母数は一般的には未知なので推定値を用いることもあります。標本はこの母集団から抽出されたものとして扱います。つぎの図では、その母数の分布を青い点線で示しています。標本からはその統計量の標本分布が得られます。それを橙の線で示しています。この2つを比べます。2つの分布が近ければ、この2つの分布を違うと判断するのが難しく、離れていれば違うと判断できます。
この判断を有意水準を用いて行うことができます。有意水準$\alpha$は母集団の右端の面積のことです。10%,5%などが用いられます。青い分布の青い垂直の線の右側の面積です。青い垂直の線は帰無仮説が棄却される境界を示しています。この線よりも内側を採択域、外側を棄却域といいます。標本が得られると平均、分散などの統計量を計算することができます。p値は、その統計量を下限として帰無仮説を棄却した場合の棄却される確率を母数の分布から算出したものです。つぎに図でいうと赤い垂直の線から右の部分の面積です。この値が判断基準である有意水準より小さければ、標本(統計量)は母集団(母数)とは異なる分布から得られたと判断します。図では赤い垂直の線は青い垂直の線の左側にありますが、橙色の標本分布の平均値が青い垂直の線の右側にくれば、そのp値は有意水準よりも小さくなることは明確です。
誤った帰無仮説を正しく棄却できる力を検出力といいます。検出力$\beta$は青い垂直の線の橙の分布の右側の面積に相当します。$\beta$の部分が大きければ帰無仮説を棄却した時に対立仮説が正しい確率が大きくなります。
この$\beta$を大きくするためには2つの分布が大きく離れている、または分布の中心の位置が大きく異なることが考えられます。もう1つは標本の大きさが大きいことです。そうすれば分布の形状は狭くなり、標本の大きさが分布を線とみなせるほど大きくなれば、2つの直線を比べることになります。
Dicky-fuller検定
単純なAR(1)過程を
$ y_t= \rho y_{t-1}+u_t$
とし、両辺から$y_{t-1}$を差し引いて
$y_t-y_{t-1}=(\rho-1)y_{t-1}+u_t$
とします。$\delta=\rho-1$として、帰無仮説はARモデルに単位根が存在するとします。
$H_0$:$\delta=0$
$H_1$: $\delta<0$
となります。3つのタイプがあります。
1)ドリフト無しランダムウォーク
$\Delta y_t=\delta y_{t-1}+u_t$
2) ドリフト付きランダムウォーク
$\Delta y_t=c+\delta y_{t-1}+u_t$
3) ドリフト付きランダムウォーク+時間トレンド
$\Delta y_t=c+c\cdot t+\delta y_{t-1}+u_t$
これらは左辺が一次の差分なので、差分がドリフトを持つ、または時間トレンドをもつという意味です。# Dicky-Fuller検定のテスト def adfcheck(a0,mu,sigma,nsample,trial): prw=0 prwc=0 prwct=0 for i in range(trial): y=ar1(a0,1,mu,sigma,nsample) rw=sm.tsa.adfuller(y,maxlag=0,regression='nc')[1] rwc=sm.tsa.adfuller(y,maxlag=0,regression='c')[1] rwct=sm.tsa.adfuller(y,maxlag=0,regression='ct')[1] if rw>0.05: prw+=1 if rwc>0.05: prwc+=1 if rwct>0.05: prwct+=1 print('nsample',nsample,prw/trial,prwc/trial,prwct/trial)ランダウウォークの検定
ドリフト無しランダムウォーク
rho=1 c1=1 c1=(c1-1)/250+1 d=0 d=d/250 sigma=0.4/np.sqrt(250) trial=10000 for i in [7,20,250]: adfcheck(rho,x0,c1,d,sigma,i,trial) # nsample 7 0.9448 0.8878 0.8528 # nsample 20 0.9635 0.9309 0.921 # nsample 250 0.9562 0.9509 0.9485ドリフト付きランダムウォーク
rho=1 c1=1.1 c1=(c1-1)/250+1 d=0 d=d/250 sigma=0.4/np.sqrt(250) trial=10000 for i in [7,20,250]: adfcheck(rho,x0,c1,d,sigma,i,trial) # nsample 7 0.9489 0.8889 0.8524 # nsample 20 0.9649 0.9312 0.921 # nsample 250 0.9727 0.9447 0.9454ランダムウォーク+ドリフト+確定的トレンド
rho=1 c1=1.1 c1=(c1-1)/250+1 d=0.9 d=d/250 sigma=0.4/np.sqrt(250) trial=10000 for i in [7,20,250]: adfcheck(rho,x0,c1,d,sigma,i,trial) # nsample 7 0.9971 0.948 0.8581 # nsample 20 1.0 1.0 0.983 # nsample 250 1.0 1.0 0.9998ランダムウォークが棄却されるべき場合
ドリフト無しAR(1)モデル
rho=0.99 c1=rho #c1=(c1-1)/250+1 d=0 d=d/250 sigma=0.4/np.sqrt(250) trial=10000 for i in [7,20,250]: adfcheck(rho,x0,c1,d,sigma,i,trial) # nsample 7 0.7797 0.9012 0.8573 # nsample 20 0.6188 0.9473 0.9225 # nsample 250 0.0144 0.7876 0.944ドリフト付きAR(1)モデル
rho=0.90 c1=1.1 c1=(c1-1)/250+1 d=0 d=d/250 sigma=0.4/np.sqrt(250) trial=10000 for i in [7,20,250]: adfcheck(rho,x0,c1,d,sigma,i,trial) # nsample 7 0.9715 0.8868 0.8539 # nsample 20 0.9989 0.9123 0.912 # nsample 250 1.0 0.0306 0.1622AR(1)モデル+ドリフト+確定的トレンド
rho=0.90 c1=1.1 c1=(c1-1)/250+1 d=1 d=d/250 sigma=0.4/np.sqrt(250) trial=10000 for i in [7,20,250]: adfcheck(rho,x0,c1,d,sigma,i,trial) # nsample 7 0.999 0.9404 0.8576 # nsample 20 1.0 1.0 0.918 # nsample 250 1.0 1.0 0.0044正しい仕様のモデルを使うこと、標本の大きさは大きいほうが良いこと、そして帰無仮説を棄却するときにはρは1から離れていたほうが良いことが分かります。
ノイズの影響について
コーシー分布を使ってファットテイルのノイズを発生させそれを使って時系列を生成した時にどのような影響が出るかを調べてみます。
まずコーシー分布の分布をみます。from scipy.stats import cauchy trial=1000000 m1=[] for i in range(trial): s = np.random.normal(0, 1, 250) m1.append(np.mean(s)) m2=[] for i in range(trial): s=[] i=1 while i<=250: s0 = (cauchy.rvs(0,1,1)) if abs(s0)<10: s.append(s0) i+=1 m2.append(np.mean(s)) bins=np.linspace(np.min(m2),np.max(m2),1000) plt.hist(m1,bins=bins,label='normal') plt.hist(m2,bins=bins,label='abs(cauchy) <10',alpha=0.5) plt.legend() plt.show()ファットテイルが強いことが分かります。これをノイズに見立てます。
つぎにコーシ分布から乱数の和を形成し、その最後の値の分布をみます。
from scipy.stats import cauchy m1=[] trial=1000000 for i in range(trial): s = np.cumsum(np.random.normal(0, 1, 250)) m1.append(s[-1]) m2=[] for i in range(trial): s=[] i=1 while i<=250: s0 = (cauchy.rvs(0,1,1)) if abs(s0)<10: s.append(s0) i+=1 m2.append(np.cumsum(s)[-1]) bins=np.linspace(np.min(m2),np.max(m2),1000) plt.hist(m1,bins=bins,label='normal') plt.hist(m2,bins=bins,label='cauchy',alpha=0.5) plt.legend() plt.show()つぎにコーシー分布を使ったAR1関数を作ってみます。
def ar1_c(rho,x0,c1,d,sigma,nsample): e = cauchy.rvs(loc=0,scale=1,size=nsample) c=c1-rho # constant of autoregressive mdoel y=[] y.append(e[0]+x0*rho+c+d) for i,ee in enumerate(e[1:]): y.append(rho*y[i]+ee+c+d*(i+1)) return yできた1次の自己回帰時系列を用いてDF検定を行います。
def adfcheck_c(rho,x0,c1,d,sigma,nsample,trial): prw=0 prwc=0 prwct=0 for i in range(trial): y=ar1_c(rho,x0,c1,d,sigma,nsample) rw=sm.tsa.adfuller(y,maxlag=0,regression='nc')[1] rwc=sm.tsa.adfuller(y,maxlag=0,regression='c')[1] rwct=sm.tsa.adfuller(y,maxlag=0,regression='ct')[1] if rw>0.05: prw+=1 if rwc>0.05: prwc+=1 if rwct>0.05: prwct+=1 print('nsample',nsample,prw/trial,prwc/trial,prwct/trial)rho=0.90 c1=1.1 c1=(c1-1)/250+1 d=1 d=d/250 sigma=0.4/np.sqrt(250) trial=10000 for i in [7,20,250]: adfcheck_c(rho,x0,c1,d,sigma,i,trial) # nsample 7 0.973 0.8775 0.8377 # nsample 20 0.9873 0.9588 0.9143 # nsample 250 0.8816 0.8425 0.0897ファットテイルの影響で、この場合はドリフト付き時間トレンドなのですが、標本の大きさが250のときに5%有意水準で帰無仮説を棄却できません。rho=0.85ではつぎの結果です。
nsample 7 0.9717 0.8801 0.8499
nsample 20 0.987 0.9507 0.903
nsample 250 0.8203 0.7057 0.0079標本の大きさが250のときに5%有意水準で帰無仮説を棄却できています。
つぎにコーシー分布のファットテイルの部分を少し削ってみましょう。
def cau(cut,nsample): s=[] i=1 while i<=nsample: s0 = cauchy.rvs(0,1,1) if abs(s0)<cut: s.append(s0[0]) i+=1 return s def ar1_c2(rho,x0,c1,d,sigma,nsample): e = cau(10,nsample) c=c1-rho # constant of autoregressive mdoel y=[] y.append(e[0]*sigma+x0*rho+c+d) for i,ee in enumerate(e[1:]): y.append(rho*y[i]+ee*sigma+c+d*(i+1)) return y def adfcheck_c2(rho,x0,c1,d,sigma,nsample,trial): prw=0 prwc=0 prwct=0 for i in range(trial): y=ar1_c2(rho,x0,c1,d,sigma,nsample) rw=sm.tsa.adfuller(y,maxlag=0,regression='nc')[1] rwc=sm.tsa.adfuller(y,maxlag=0,regression='c')[1] rwct=sm.tsa.adfuller(y,maxlag=0,regression='ct')[1] if rw>0.05: prw+=1 if rwc>0.05: prwc+=1 if rwct>0.05: prwct+=1 print('nsample',nsample,prw/trial,prwc/trial,prwct/trial)rho=0.90 c1=1.1 c1=(c1-1)/250+1 d=1 d=d/250 sigma=0.4/np.sqrt(250) trial=10000 for i in [7,20,250]: adfcheck_c2(rho,x0,c1,d,sigma,i,trial) # nsample 7 0.9969 0.9005 0.8462 # nsample 20 1.0 0.9912 0.9182 # nsample 250 1.0 1.0 0.0931先ほどとは違った結果になりました。帰無仮説を棄却できるのはドリフト付き時間トレンドだけになりましたが、有意水準は10%です。rho=0.85にすると
nsample 7 0.9967 0.9012 0.8466
nsample 20 1.0 0.9833 0.9121
nsample 250 1.0 1.0 0.0028という結果になります。
つぎに拡張ディッキーフラー検定を試してみます。
$\Delta y_t=c+c\cdot t+\delta y_{t-1}+\delta_1\Delta y_{t-1}+,\cdots,+\delta_{p-1}\Delta y_{t-p+1}+u_t$
で遅延の1階差分を増やすことで自己相関などの構造的な効果を取り除いています。def adfcheck_c3(rho,x0,c1,d,sigma,nsample,trial): prw=0 prwc=0 prwct=0 for i in range(trial): y=ar1_c2(rho,x0,c1,d,sigma,nsample) rw=sm.tsa.adfuller(y,regression='nc')[1] rwc=sm.tsa.adfuller(y,regression='c')[1] rwct=sm.tsa.adfuller(y,regression='ct')[1] if rw>0.05: prw+=1 if rwc>0.05: prwc+=1 if rwct>0.05: prwct+=1 print('nsample',nsample,prw/trial,prwc/trial,prwct/trial)rho=0.90 c1=1.1 c1=(c1-1)/250+1 d=1 d=d/250 sigma=0.4/np.sqrt(250) trial=10000 for i in [7,20,250]: adfcheck_c3(rho,x0,c1,d,sigma,i,trial) # nsample 7 0.94 0.8187 0.8469 # nsample 20 1.0 0.873 0.7969 # nsample 250 1.0 1.0 0.1253rho=0.90では正しく判断できません。rho=0.85にすると結果は
nsample 7 0.9321 0.8109 0.8429
nsample 20 1.0 0.8512 0.7942
nsample 250 1.0 1.0 0.0252となりドリフト+時間トレンドを棄却できています。
標準偏差がゼロまたは小さい時のドリフト、時間トレンドの検出
# driftのみ、標準偏差はゼロ rho=0 c1=1.1 c1=(c1-1)/250+1 d=0 d=d/250 sigma=0#.4/np.sqrt(250) trial=1#0000 for i in [7,20,250]: adfcheck(rho,x0,c1,d,sigma,i,trial)nsample 7 0.0 0.0 0.0
nsample 20 0.0 0.0 0.0
nsample 250 0.0 0.0 0.0ドリフトだけがあるときにはすべてで帰無仮説は棄却されます。
# driftと時間トレンド、標準偏差はゼロ rho=0 c1=1.1 c1=(c1-1)/250+1 d=1 d=d/250 sigma=0#.4/np.sqrt(250) trial=1#0000 for i in [7,20,250]: adfcheck(rho,x0,c1,d,sigma,i,trial)nsample 7 1.0 1.0 0.0
nsample 20 1.0 1.0 0.0
nsample 250 1.0 1.0 0.0
AR(1)+ドリフト+時間トレンドが標本の大きさに関係なく帰無仮説を棄却つぎはドリフトつきに若干の標準偏差を加えます。
# drift+ very small sigma rho=0 c1=1.1 c1=(c1-1)/250+1 d=0 d=d/250 sigma=0.004#.4/np.sqrt(250) trial=10000 for i in [7,20,250]: adfcheck(rho,x0,c1,d,sigma,i,trial)nsample 7 0.9986 0.6379 0.7223
nsample 20 1.0 0.0396 0.1234
nsample 250 1.0 0.0 0.0つぎはドリフト+時間トレンドに若干の標準偏差を加えます。
# drift+ very small sigma rho=0 c1=1.1 c1=(c1-1)/250+1 d=1 d=d/250 sigma=0.004#.4/np.sqrt(250) trial=10000 for i in [7,20,250]: adfcheck(rho,x0,c1,d,sigma,i,trial)nsample 7 1.0 0.9947 0.6877
nsample 20 1.0 1.0 0.1095
nsample 250 1.0 1.0 0.0参考
statsmodels.tsa.stattools.adfuller
http://web.econ.ku.dk/metrics/Econometrics2_05_II/Slides/08_unitroottests_2pp.pdf