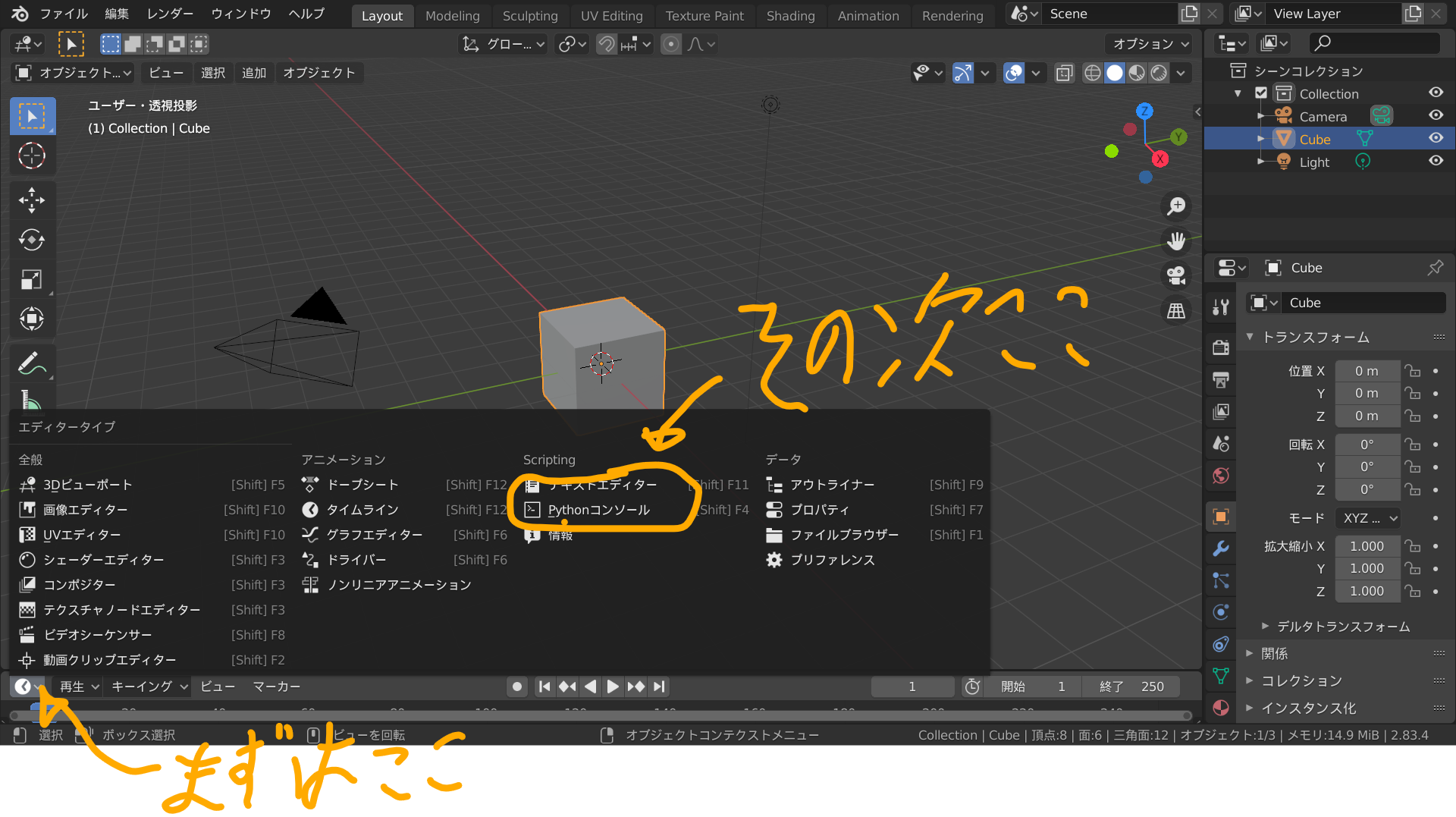
- 投稿日:2020-08-11T23:52:01+09:00
pythonの基礎: 関数
関数、組込関数
関数: プログラムのまとまりを定義したもの
組込関数: pythonそのものに予め設定されてる関数
変数のプログラム版だと考えればいいかと
メソッド
特定の値に大して処理を行う物をメソッドといいます。
メソッド値.メソッド名()インスタンスメソッド
値の要素などを取り出すインスタンス変数というものもあります。
インスタンス変数は関数ではなく変数なので引数を持ちません。インスタンス変数値.インスタンス変数名appendを例に出すと以下のようになります。
append# append()は、リストに新しい要素を1つだけ追加したい場合に使用するメソッドです。 al = ["1","2","3","4","5"] al.append("f") print(al) # ["1","2","3","4","5","f"]が出力される文字列型のメソッド
文字の拡大と、数えを行うメソッドです。
コード 内容 upper 文字の拡大 count 文字数を数える format 文字列に変数を打ち込む city = "Nagoya" print(city.upper()) # 出力: 「NAGOYA」 print(city.count("N")) # 出力: 「1」 print("私は{}生まれ、{}出身".format("愛知", "名古屋")) # 出力: 「私は愛知生まれ、名古屋出身」 # 埋め込む箇所を{}で指定。引数は文字列型でなくてもよし # {}に挿入する値は、挿入順番の指定、同値の繰返しも可能。 print("私は{1}生まれ、{0}育ち、{1}市民".format("愛知", "名古屋")) # 出力: 「私は名古屋生まれ、愛知育ち、名古屋市民」リスト型のメソッド
コード 内容 index インデックス番号を返す count 数える sort 並べ替える reverse 逆に並べ替える sorted 元の値を変えないで並べ変え index() # インデックス番号にあるのか探すためのメソッド count() # いくつかあるか出力するメソッドal = ["0", "1", "2", "3", "3"] print(al.index("0")) # 出力: 「0」 print(al.count("3")) # 出力: 「2」sort() # これはリストの中を小さい順に並べ替えてくれます。 reverse() # リストの要素の順番を反対 に # sort()とreverse()はリストの中身が直接変更される。 # 戻り値なしなので、print(list.sort())としてもNoneが返る soted() # 組込関数のsorted()で元の値はそのまま# sort()の利用例 list = [1, 3, 2, 4] list.sort() print(list) # [1, 2, 3, 4]と表示されます。 # reverse()の利用例 list = ["か", "き", "く", "け", "こ"] list.reverse() print(list) #出力: ["こ", "け", "く", "き", "か"]関数
「 def 関数名(): 」def eat(): print ("食べる") # インデントで処理の範囲を明示 # 関数の呼び出しは「関数名()」となる eat() # 出力: 食べる引数
引数(argument)を設定しておけば、
関数内でその値を使用できるdef 関数名(引数):def ii(n): print(n + "です") ii("yellow") # 出力: yellowです # 引数を変数で指定すると、その都度出力結果を変えれる def ii(n): print(n + "です") name = "yellow" ii(name) # yellowです name = "black" ii(name) # 出力: black # 関数内で定義した変数,引数は、関数内でのみ使用可能複数の引数
引数はカンマで区切り
複数指定するdef ii(sei, name): print("名字は" + sei + "で、名前は" + name + "です。") ii("j", "bb") # 「名字はjで、名前はbbです。」と出力される引数の初期値
初期(デフォルト)値を設定すると
空欄の場合、自動的に初期値が設定
引数 = 初期値 という形で設定されるdef ii(family="j", first="bb"): print("名字は" + family + "で、名前は" + first + "です。") ii("b") # 「名字はbで、名前はbbです。」と出力される # もしfirstの引数のみ渡したい場合はii(first="cc")と指定 # なお、初期値を設定した引数の後ろの引数にも、必ず初期値を設定する必要があります。 # したがって、次のように後ろの引数にだけ初期値を設定することは可能です。 def ii(family, first="aa"): print("名字は" + family + "で、名前は"+ first + "です。") # 前の引数にだけ設定の時、後ろの引数に設定しないとエラーになる。 def ii(family="zz", first): print("名字は" + family + "で、名前は" + first + "です。")return
関数で定義した変数や引数は関数外では使用できない
returnによって、関数の呼び出し元に戻り値を渡すことができるdef ii(family = "jj", first = "bb"): comment = "名字は" + family + "で、名前は" + first + "です。" print(ii("ss")) # Noneが出力されるdef ii(family = "jj", first = "bb"): comment = "名字は" + family + "で、名前は" + first + "です。" return comment #関数に戻り値を渡す print(ii("ss")) # 「名字はssで、名前はbbです。」と出力される
- 投稿日:2020-08-11T22:58:52+09:00
PyGMTの導入
PythonでGMTがつかえるらしいので入れてみる。
https://www.pygmt.org/dev/install.html仮想環境の構築
conda create -n pygmt python=3.8.2 conda activate pygmt conda install pip numpy pandas xarray netcdf4 packaging gmt conda install pygmt -c conda-forge conda install ipython jupyter jupyterlab -c conda-forgeサンプルテスト
チュートリアルにしたがってすすめる。
conda install pytest pytest-mpl ipython
pythonを起動して、import pygmt pygmt.show_versions() pygmt.test()返ってきたメッセージ
============================== 53 failed, 212 passed, 1 skipped, 353 warnings in 83.38s (0:01:23) ============================== Traceback (most recent call last): File "<stdin>", line 1, in <module> File "/home/kanon/local/anaconda3/envs/pygmt/lib/python3.8/site-packages/pygmt/__init__.py", line 187, in test assert status == 0, "Some tests have failed." AssertionError: Some tests have failed.けっこう失敗している気がするけど、これでいいのか?
GMT5も入れているので、チュートリアルにある通りGMT_LIBRARY_PATHも設定してみたが、failedの数は変わらなかった。とりあえず地図!
チュートリアル通りに、絵を描いてみた。
import pygmt fig = pygmt.Figure() fig.basemap(region=[-90,-70,0,20], projection="M8i", frame=True) fig.coast(shorelines=True) fig.show() fig.savefig("test_pygmt.png")うまくかけました!
ただし、JupyterLabがDark modeだとなにも見えない・・・oh
- 投稿日:2020-08-11T22:30:28+09:00
【coding interview】enigma暗号機の実装(Python)
はじめに
酒井潤さんがYoutubeで紹介していたエニグマ暗号機を実装してみました。
今回、このコーディングを行った理由としては、シリコンバレーの人の構文等を参考にしたかったのとオブジェクティブな実装の練習です。酒井さんのYoutubeはこちらになります。
また、オリラジの中田さんがYoutube大学でエニグマについて説明してくれていて面白かったのでこちらも載せておきます。結論
コーディングの仕方等はかなり参考になったし、暇な時間にやるのにそこそこ楽しかった。
今回は実際に作ったけどGithubに載せるほどではないし、載せないのはもったいなかったのでQiitaの記事として投稿しました。
自力で最初に実装したコードは少し汚かったので、酒井さんの動画を見ながら実装したコードを下に載せておくのでよかったら参考にしてみてください。ファイル構成
ファイル構成は以下のようになります。
enigma.py内でモデルをオブジェクティブに定義、main.pyでモデルを構築します。egnima ├── enigma.py └── main.pyEnigma.py
PlugBoard, Rotate, Reflector, Enigmaの4つのクラスを作ります。
それぞれの詳細に関しては酒井さんのYoutubeまたはWikiを参考にされるといいかと思います。enigma.pyimport string ALPHABET = string.ascii_uppercase class PlugBoard(object): def __init__(self, map_alphabet): self.alphabet = ALPHABET self.forward_map = {} self.backward_map = {} self.mapping(map_alphabet) def mapping(self, map_alphabet): self.forward_map = dict(zip(self.alphabet, map_alphabet)) self.backward_map = {v: k for k , v in self.forward_map.items()} def forward(self, index_num): char = self.alphabet[index_num] char = self.forward_map[char] return self.alphabet.index(char) def backward(self, index_num): char = self.alphabet[index_num] char = self.backward_map[char] return self.alphabet.index(char) class Rotor(PlugBoard): def __init__(self, map_alphabet, offset=0): super().__init__(map_alphabet) self.offset = offset self.rotations = 0 def rotate(self, offset=None): if offset is None: offset = self.offset self.alphabet = self.alphabet[offset:] + self.alphabet[:offset] self.rotations += offset return self.rotations def reset(self): self.rotationations = 0 self.alphabet = ALPHABET class Reflector(object): def __init__(self, map_alphabet): self.map = dict(zip(ALPHABET, map_alphabet)) for x, y in self.map.items(): if x != self.map[y]: raise ValueError(x, y) def reflect(self, index_num): reflected_char = self.map[ALPHABET[index_num]] return ALPHABET.index(reflected_char) class Enigam(object): def __init__(self, plug_board, rotors, reflector): self.plug_board = plug_board self.rotors = rotors self.reflector = reflector def encrypt(self, text): return ''.join([self.go_through(c) for c in list(text)]) def decrypt(self, text): for rotor in self.rotors: rotor.reset() return ''.join([self.go_through(c) for c in list(text)]) def go_through(self, char): char = char.upper() if char not in ALPHABET: return char index_num = ALPHABET.index(char) index_num = self.plug_board.forward(index_num) for rotor in self.rotors: index_num = rotor.forward(index_num) index_num = self.reflector.reflect(index_num) for rotor in reversed(self.rotors): index_num = rotor.backward(index_num) index_num = self.plug_board.backward(index_num) char = ALPHABET[index_num] for rotor in reversed(self.rotors): if rotor.rotate() % len(ALPHABET) != 0: break return charmain.py
main.pyimport random from enigma import PlugBoard, ALPHABET, Rotor, Reflector, Enigam if __name__ == '__main__': # outputs : XYGBWNSCMQFJLHEVRIZODAPUTK get_random_alphabet = lambda : ''.join( random.sample(ALPHABET, len(ALPHABET))) p = PlugBoard(get_random_alphabet()) r1 = Rotor(get_random_alphabet(), 3) r2 = Rotor(get_random_alphabet(), 2) r3 = Rotor(get_random_alphabet(), 1) # reflected alphabets list for the Rflection r = list(ALPHABET) indexes = [i for i in range(len(ALPHABET))] for _ in range(int(len(indexes)/2)): x = indexes.pop(random.randint(0, len(indexes)-1)) y = indexes.pop(random.randint(0, len(indexes)-1)) r[x], r[y] = r[y], r[x] reflector = Reflector(''.join(r)) # Define Enigam machine = Enigam( p, [r1, r2, r3], reflector) text = 'PYTHON' encrypted = machine.encrypt(text) decrypted = machine.decrypt(encrypted) print(f' Text : {text}') # Text : PYTHON print(f'Encrypted text : {encrypted}') # Encrypted text : TDZXZS print(f'Decrypted text : {decrypted}') # Decrypted text : PYTH '''
- 投稿日:2020-08-11T21:26:58+09:00
[AWS] SAM + Lambda(Python)で、PythonライブラリをLayerに追加して利用してみる
SAMのインストール
「[AWS] Serverless Application Model (SAM) でAPI Gateway + Lambda + DynamoDBなサンプルを作成してみる」を参照ください。
なお、今回のサンプルに限っていうと、DynamoDBをローカルに持つ必要はないので、Dockerのインストールは不要です。
プロジェクトの作成
では、例によって、HelloWorldベースのプロジェクトを作成していきましょう。
$ sam init --runtime=python3.8 Which template source would you like to use? 1 - AWS Quick Start Templates 2 - Custom Template Location Choice: 1 Project name [sam-app]: Cloning app templates from https://github.com/awslabs/aws-sam-cli-app-templates.git AWS quick start application templates: 1 - Hello World Example 2 - EventBridge Hello World 3 - EventBridge App from scratch (100+ Event Schemas) 4 - Step Functions Sample App (Stock Trader) 5 - Elastic File System Sample App Template selection: 1 ----------------------- Generating application: ----------------------- Name: sam-app Runtime: python3.8 Dependency Manager: pip Application Template: hello-world Output Directory: . Next steps can be found in the README file at ./sam-app/README.mdLayerを定義する
まず、Layer用のディレクトリを作成します。
ディレクトリは、プロジェクト直下で、ディレクトリ名は後ほど定義に記述するので、自由につけて大丈夫です。
とりあえず「hello_world_layer」とでもしておきます。$ mkdir hello_world_layer続いて、template.yamlの
Resources配下に、Layerの定義を追記します。
今回は「HelloWorldLayer」という名称で作成しておきます。template.yml変更前Resources: HelloWorldFunction: Type: AWS::Serverless::Function # More info about Function Resource: https://github.com/awslabs/serverless-application-model/blob/master/versions/2016-10-31.md#awsserverlessfunction Properties: CodeUri: hello_world/ Handler: app.lambda_handler Runtime: python3.8 Events: HelloWorld: Type: Api # More info about API Event Source: https://github.com/awslabs/serverless-application-model/blob/master/versions/2016-10-31.md#api Properties: Path: /hello Method: gettemplate.yml変更後Resources: HelloWorldFunction: Type: AWS::Serverless::Function # More info about Function Resource: https://github.com/awslabs/serverless-application-model/blob/master/versions/2016-10-31.md#awsserverlessfunction Properties: CodeUri: hello_world/ Handler: app.lambda_handler Runtime: python3.8 Events: HelloWorld: Type: Api # More info about API Event Source: https://github.com/awslabs/serverless-application-model/blob/master/versions/2016-10-31.md#api Properties: Path: /hello Method: get Layers: - !Ref HelloWorldLayer HelloWorldLayer: Type: AWS::Serverless::LayerVersion Properties: Description: Layer description ContentUri: hello_world_layer/ CompatibleRuntimes: - python3.8 Metadata: BuildMethod: python3.8NumPyを追加してみる
次に、ライブラリを追加します。
利用したいライブラリを記述してください。
今回のサンプルでは、機械学習でよく使用する数値計算モジュールのNumPyを追加してみたいと思います。
hello_world_layerに、requirements.txtを作成し、以下のように記述して、ライブラリを追加します。$ touch hello_world_layer/requirements.txthello_world_layer/requirements.txtnumpyLayerクラスを作成する
hello_world_layer配下に、numpyの処理をするユーザクラスを作成してみましょう。
名前は適当にuser_numpyとでもしておきましょうか。$ touch hello_world_layer/user_numpy.pyhello_world_layer/user_numpy.pyimport numpy as np class UserNumpy(): def __init__(self): super().__init__() def array(self): return np.array([0,1,2])Layerクラスのメソッドを呼び出す
最後に、Functionの方で、追加したLayerメソッドを呼び出してみましょう。
既存のhello_world/app.pyを以下のように変更してみましょう。helloi_world/app.pyimport json from user_numpy import UserNumpy def lambda_handler(event, context): un = UserNumpy() un_str = [str(n) for n in un.array()] return { "statusCode": 200, "body": json.dumps({ "message": ','.join(un_str) }), }UserNumpyのarrayメソッドは、Numpyの
array([0,1,2])の結果を返します。
これは数値の配列なので、一度文字列の配列に変換したあとで、文字列かしてJsonデータとして返すようにしています。ビルド
では、ビルドしてみましょう。
$ sam build Building function 'HelloWorldFunction' Running PythonPipBuilder:ResolveDependencies Running PythonPipBuilder:CopySource Building layer 'HelloWorldLayer' Running PythonPipBuilder:ResolveDependencies Running PythonPipBuilder:CopySource Build Succeeded Built Artifacts : .aws-sam/build Built Template : .aws-sam/build/template.yaml Commands you can use next ========================= [*] Invoke Function: sam local invoke [*] Deploy: sam deploy --guidedデプロイ
次にデプロイです。
事前に、AWSのクレデンシャルを設定しておいてください。$ sam deploy --guided Configuring SAM deploy ====================== Looking for samconfig.toml : Found Reading default arguments : Success Setting default arguments for 'sam deploy' ========================================= Stack Name [sam-app]: AWS Region [ap-northeast-1]: #Shows you resources changes to be deployed and require a 'Y' to initiate deploy Confirm changes before deploy [Y/n]: y #SAM needs permission to be able to create roles to connect to the resources in your template Allow SAM CLI IAM role creation [Y/n]: y HelloWorldFunction may not have authorization defined, Is this okay? [y/N]: y Save arguments to samconfig.toml [Y/n]: y Looking for resources needed for deployment: Not found. Creating the required resources... Successfully created! Managed S3 bucket: aws-sam-cli-managed-default-samclisourcebucket-fqjmg5s30i1w A different default S3 bucket can be set in samconfig.toml Deploying with following values =============================== Stack name : sam-app Region : ap-northeast-1 Confirm changeset : True Deployment s3 bucket : aws-sam-cli-managed-default-samclisourcebucket-fqjmg5s30i1w Capabilities : ["CAPABILITY_IAM"] Parameter overrides : {} Initiating deployment ===================== Saved arguments to config file Running 'sam deploy' for future deployments will use the parameters saved above. The above parameters can be changed by modifying samconfig.toml Learn more about samconfig.toml syntax at https://docs.aws.amazon.com/serverless-application-model/latest/developerguide/serverless-sam-cli-config.html Uploading to sam-app/ec3e7cb3e8873a2b56d81ae66ba15ec5 262144 / 538577.0 (48.67Uploading to sam-app/ec3e7cb3e8873a2b56d81ae66ba15ec5 524288 / 538577.0 (97.35Uploading to sam-app/ec3e7cb3e8873a2b56d81ae66ba15ec5 538577 / 538577.0 (100.00%) Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 262144 / 14534393.0 (1.8Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 524288 / 14534393.0 (3.6Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 786432 / 14534393.0 (5.4Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 1048576 / 14534393.0 (7.Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 1310720 / 14534393.0 (9.Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 1572864 / 14534393.0 (10Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 1835008 / 14534393.0 (12Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 2097152 / 14534393.0 (14Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 2359296 / 14534393.0 (16Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 2621440 / 14534393.0 (18Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 2883584 / 14534393.0 (19Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 3145728 / 14534393.0 (21Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 3407872 / 14534393.0 (23Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 3670016 / 14534393.0 (25Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 3932160 / 14534393.0 (27Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 4194304 / 14534393.0 (28Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 4456448 / 14534393.0 (30Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 4718592 / 14534393.0 (32Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 4980736 / 14534393.0 (34Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 5242880 / 14534393.0 (36Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 5505024 / 14534393.0 (37Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 5767168 / 14534393.0 (39Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 6029312 / 14534393.0 (41Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 6145785 / 14534393.0 (42Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 6407929 / 14534393.0 (44Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 6670073 / 14534393.0 (45Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 6932217 / 14534393.0 (47Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 7194361 / 14534393.0 (49Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 7456505 / 14534393.0 (51Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 7718649 / 14534393.0 (53Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 7980793 / 14534393.0 (54Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 8242937 / 14534393.0 (56Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 8505081 / 14534393.0 (58Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 8767225 / 14534393.0 (60Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 9029369 / 14534393.0 (62Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 9291513 / 14534393.0 (63Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 9553657 / 14534393.0 (65Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 9815801 / 14534393.0 (67Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 10077945 / 14534393.0 (6Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 10340089 / 14534393.0 (7Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 10602233 / 14534393.0 (7Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 10864377 / 14534393.0 (7Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 11126521 / 14534393.0 (7Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 11388665 / 14534393.0 (7Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 11650809 / 14534393.0 (8Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 11912953 / 14534393.0 (8Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 12175097 / 14534393.0 (8Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 12437241 / 14534393.0 (8Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 12699385 / 14534393.0 (8Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 12961529 / 14534393.0 (8Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 13223673 / 14534393.0 (9Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 13485817 / 14534393.0 (9Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 13747961 / 14534393.0 (9Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 14010105 / 14534393.0 (9Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 14272249 / 14534393.0 (9Uploading to sam-app/04efcd43f42b7344dff7e02a5e9d321c 14534393 / 14534393.0 (100.00%) Deploying with following values =============================== Stack name : sam-app Region : ap-northeast-1 Confirm changeset : True Deployment s3 bucket : aws-sam-cli-managed-default-samclisourcebucket-fqjmg5s30i1w Capabilities : ["CAPABILITY_IAM"] Parameter overrides : {} Initiating deployment ===================== HelloWorldFunction may not have authorization defined. Uploading to sam-app/5c2143d527c3bab330c5c2145019dcb1.template 1455 / 1455.0 (100.00%) Waiting for changeset to be created.. CloudFormation stack changeset ------------------------------------------------------------------------------------------------ Operation LogicalResourceId ResourceType ------------------------------------------------------------------------------------------------ + Add HelloWorldFunctionHelloWorldPe AWS::Lambda::Permission rmissionProd + Add HelloWorldFunctionRole AWS::IAM::Role + Add HelloWorldFunction AWS::Lambda::Function + Add HelloWorldLayer8a10103d68 AWS::Lambda::LayerVersion + Add ServerlessRestApiDeployment47f AWS::ApiGateway::Deployment c2d5f9d + Add ServerlessRestApiProdStage AWS::ApiGateway::Stage + Add ServerlessRestApi AWS::ApiGateway::RestApi ------------------------------------------------------------------------------------------------ Changeset created successfully. arn:aws:cloudformation:ap-northeast-1:767054379442:changeSet/samcli-deploy1597147509/8e325896-aa90-4379-afa2-44147e907291 Previewing CloudFormation changeset before deployment ====================================================== Deploy this changeset? [y/N]: y 2020-08-11 21:05:21 - Waiting for stack create/update to complete CloudFormation events from changeset ------------------------------------------------------------------------------------------------- ResourceStatus ResourceType LogicalResourceId ResourceStatusReason ------------------------------------------------------------------------------------------------- CREATE_IN_PROGRESS AWS::Lambda::LayerVers HelloWorldLayer8a10103 - ion d68 CREATE_IN_PROGRESS AWS::IAM::Role HelloWorldFunctionRole Resource creation Initiated CREATE_IN_PROGRESS AWS::IAM::Role HelloWorldFunctionRole - CREATE_IN_PROGRESS AWS::Lambda::LayerVers HelloWorldLayer8a10103 Resource creation ion d68 Initiated CREATE_COMPLETE AWS::Lambda::LayerVers HelloWorldLayer8a10103 - ion d68 CREATE_COMPLETE AWS::IAM::Role HelloWorldFunctionRole - CREATE_IN_PROGRESS AWS::Lambda::Function HelloWorldFunction - CREATE_IN_PROGRESS AWS::Lambda::Function HelloWorldFunction Resource creation Initiated CREATE_COMPLETE AWS::Lambda::Function HelloWorldFunction - CREATE_IN_PROGRESS AWS::ApiGateway::RestA ServerlessRestApi - pi CREATE_IN_PROGRESS AWS::ApiGateway::RestA ServerlessRestApi Resource creation pi Initiated CREATE_COMPLETE AWS::ApiGateway::RestA ServerlessRestApi - pi CREATE_IN_PROGRESS AWS::ApiGateway::Deplo ServerlessRestApiDeplo - yment yment47fc2d5f9d CREATE_IN_PROGRESS AWS::Lambda::Permissio HelloWorldFunctionHell Resource creation n oWorldPermissionProd Initiated CREATE_IN_PROGRESS AWS::Lambda::Permissio HelloWorldFunctionHell - n oWorldPermissionProd CREATE_COMPLETE AWS::ApiGateway::Deplo ServerlessRestApiDeplo - yment yment47fc2d5f9d CREATE_IN_PROGRESS AWS::ApiGateway::Deplo ServerlessRestApiDeplo Resource creation yment yment47fc2d5f9d Initiated CREATE_IN_PROGRESS AWS::ApiGateway::Stage ServerlessRestApiProdS - tage CREATE_IN_PROGRESS AWS::ApiGateway::Stage ServerlessRestApiProdS Resource creation tage Initiated CREATE_COMPLETE AWS::ApiGateway::Stage ServerlessRestApiProdS - tage CREATE_COMPLETE AWS::Lambda::Permissio HelloWorldFunctionHell - n oWorldPermissionProd CREATE_COMPLETE AWS::CloudFormation::S sam-app - tack ------------------------------------------------------------------------------------------------- CloudFormation outputs from deployed stack ------------------------------------------------------------------------------------------------- Outputs ------------------------------------------------------------------------------------------------- Key HelloWorldFunctionIamRole Description Implicit IAM Role created for Hello World function Value arn:aws:iam::767054379442:role/sam-app-HelloWorldFunctionRole-9AHTS9BZMUQL Key HelloWorldApi Description API Gateway endpoint URL for Prod stage for Hello World function Value https://co2snvo0lk.execute-api.ap-northeast-1.amazonaws.com/Prod/hello/ Key HelloWorldFunction Description Hello World Lambda Function ARN Value arn:aws:lambda:ap-northeast-1:767054379442:function:sam-app- HelloWorldFunction-1GVL3Y25TQYGZ ------------------------------------------------------------------------------------------------- Successfully created/updated stack - sam-app in ap-northeast-1実行
最後に、Deploy時に出力されるAPI Gatewayのエンドポイントにアクセスしてみます。
$ curl https://co2snvo0lk.execute-api.ap-northeast-1.amazonaws.com/Prod/hello/ {"message": "0,1,2"}レスポンスに、
array([0,1,2])の結果が文字列として返されていることが確認できましたね!!まとめ
Layerにライブラリを追加し、それを呼び出す共通モジュールを作ることは、簡単であることがわかったと思います。
サーバーレスの場合、なるべく疎結合に設計するのが望ましいですが、同一ドメイン内での共通ロジックをLayerに配置すること自体は間違いではないと思います。ただし、Lambdaの制約として
- デプロイパッケージサイズ
- 圧縮済みで50MB
- 解凍後さで250MB
という制約がありますので、これを考慮しながら、必要なライブラリのみを取り込むようにしましょう。
- 投稿日:2020-08-11T21:11:23+09:00
4x4x4ルービックキューブを解くプログラムを書こう! 3.実装
この記事はなに?
私は現在4x4x4ルービックキューブ(ルービックリベンジ)を解くロボットを作り、世界記録を目指しています。ロボットを作る上で一番のハードルであったのが4x4x4キューブを現実的な時間に、なるべく少ない手数で解くアルゴリズムを実装することです。
調べてみると4x4x4については文献や先人が少ないことがわかると思います。そんな貴重な資料の一つになれば嬉しいと思い、この記事を書いています。
GitHubはこちら
この記事の内容は完全ではありません。一部効率の悪いところを含んでいるかもしれません。今後改良したら随時追記していきます。↓競技用4x4x4ルービックキューブとプログラム制作のために番号を振られた競技用4x4x4ルービックキューブ
全貌
この記事集は全部で3つの記事から構成されています。
1. 概要
2. アルゴリズム
3. 実装(本記事)この記事で話すこと
この記事では前回に説明したアルゴリズムを実際に実装するときのやり方、およびその際のTIPSをお話しします。実装について手取り足取り説明するのは冗長になってしまうので避けます。こちらの記事に2x2x2ルービックキューブについての解説を詳しく書きました(実際のコードも載っています)のでこちらも参照いただければと思います。
インデックス化
細かくフェーズを分けて探索することで、探索すべき領域が非常に小さくなりました。そのおかげで考えうるそのフェーズの状態に順番に番号をつけられるようになります。これによっていちいちパズルの動きをシミュレートするなどというコストの大きい作業をする必要がなくなり、ただただ配列参照をするだけでパズルを動かすのと等価な作業が行えます。
ただし例えばEPとCPを一緒に考えると$10^6-10^7$個前後の要素数の配列に収まらず、メモリをとても食べてしまいます。そこで例えばEPだけ、CPだけで番号付けをし、2つの数字でパズルの状態を表したりするフェーズもあります。番号の付け方はなんでも良いのですが、わかりやすいようにEPとCPであればパズルの順列番号(階乗進数)、EOとCOであればそれぞれ2進数、3進数、センターであれば重複あり階乗進数で表すと良いと思います。階乗進数についてはこちらがわかりやすいです。
IDA*探索
実際の探索ではIDA*探索(Iterative Deepening A*)と呼ばれる探索を使うことになると思います。これまで幾度となく私の記事の中で登場している文句ですが、こちらの記事から引用すると、
IDA*を一言で表すと、「最大深さを制限した深さ優先探索(DFS)を、深さを増やしながら繰り返す」です。
IDA*のからくりは、一度探索したノードを忘れることにあります。ノードを忘れてしまえば、メモリを解放できますよね。IDA*では深さ$N-1$までで深さ優先探索を行って解が見つからなかったら、最大深さを$N$に増やしてまた一から探索をやり直します。こうすることで、
- 返される結果は必ず最短手数
- メモリ使用量がとても少ない
という恩恵があります。
ただし、深さの浅いノード(状態)については何度も同じ探索を繰り返してしまうため、計算量は若干増大します。しかしパズルの状態は深さに対して指数関数として増大するため、計算量の増大はそこまで大きくありません。
IDA*のミソは、現在の状態からフェーズ完成までの「距離」を推測することにあります。この「距離」は今回の場合は解けるまでの手数です。具体的には、パズルの状態をインデックス$i$で表すとすれば、
$depth \geq f(i) = g(i) + h(i)$
となるように探索を進めます。詳しく説明しましょう。
- $depth$は探索する際の深さ(=手数)の上限です。
- $f(i)$はインデックス$i$の状態を経過する解が何手かかるのかの推定値です。
- $g(i)$は初期状態からインデックス$i$の状態に至るまでにかかった手数です。
- $h(i)$はインデックス$i$の状態からフェーズ完成状態までにかかる手数の推定値です。
この式を満たしながら$depth$を0から1ずつ増やしていけば、手数の短い解が見つかります。
なお、インデックス$i$の状態からフェーズ完成にかかる実際の手数を$h^\star(i)$としたとき、常に$h(i)\leq h^\star(i)$を満たす場合に最適解が見つかります。つまり(そのフェーズ内では)最小手数の解が見つかります。この場合をadmissibleであると言います。同じ状態を重複して探索しないようにする
同じ状態を何回も探索したら非効率ですよね。でも状態をどこかに保存しておくと膨大なメモリを食ってしまいます。そこで、ルービックキューブを解くにあたっては手順を最適化したら同じ手順になる場合を回避することでこれを実装します。
もちろんこれだけでは「違う手順を回したが同じ状態になる」場合を回避できません。しかし、これだけでもかなりの確率で同じ状態を探索せずに済むとのことです(3x3についてですがこちらに書いてあります。)。4x4x4ルービックキューブの場合には、以下の2つの場合に探索をしないようにすると良いです。
- 同じ面を連続して回す場合
- 同じ軸を回す場合、内層->外層(または外層->内層: どっちにするかは最初に決めておく)の順番で回すとき
完成までの手数の推定値を返す関数
IDA*探索では$h(i)$が重要だと話しました。ここでは$h(i)$の算出手法について(私自身研究中ですが)軽くお話しします。
まず、各インデックスについて完成状態から何手で揃うのかを事前計算してテーブルにしておきます。私の場合はこれをcsvにしました。
実際にこの前計算した値を使う場合、往々にしてインデックスは複数あります($n$個あるとします)から、事前計算したテーブルから取り出せる値は$n$個あります。この$n$個の「距離」を使って、最終的にその盤面が完成からどれくらい遠いのかを表す距離を1つ出力しなくてはいけません。やってみたことをひたすら羅列します。$n$個の距離を$L=(l_0, l_1, \dots l_{n-1})$とします。また、$\mathrm{sd}(L)$は$L$の標準偏差を表すとします。
- $h(i)=\max(L)$
- $h(i)=\sum L$
- $h(i)=\max(L)+a\ \mathrm{sd}(L)$
- $h(i)=\sqrt{\sum_j l_j^2}$
- $t=\mathrm{pow}\left(a,-\left(\frac{\max(L)}{b\ \mathrm{sd}(L)}-c\right)^d\right)$として$h(i)=(1-t)\max(L)+t\sqrt{\sum_j l_j^2}$また、$\mathrm{sd}(L)=0$のときは$h(i)=\max(L)$
1つ目はadmissibleなことが保証されますが$h(i)$が$h^\star(i)$を大きく下回ることが多いため、計算量が大きくなりがちです。
2つ目はマンハッタン距離を使っていますが、admissibleを破りすぎてしまいます。簡単な例では同じ回転Rを回せば揃う$n$個の評価項目$(l_0, l_1, \dots l_{n-1})$について、$h^\star(i)=1$であるにも関わらず$h(i)=n$と返してしまいます。admissibleを破ると解の手数が増えるだけでなく、探索する空間が大きくなってしまうので計算量の増大にも繋がりがちです。
3つ目は、$L$のばらつきが大きいと$h^\star(i)$は大きくなる傾向があるという仮説のもと考えた式です。ですがあまりうまくいきませんでした。
4つ目はユークリッド距離です。2つ目よりはマシですがadmissibleを破ってしまう可能性が残っています。
5つ目が現時点で使っている方法です。定数$a, b, c, d$を調整して、$0<t<1$を作ります。この$t$を使って$L$の最大値とユークリッド距離を内分します。$t$は、$\max(L)$に比べて$\mathrm{sd}(L)$が大きいときにユークリッド距離に寄った値を返すという方針で計算しています。この関数$h(i)$を勝手にNyanyan's Functionと言っています。疑似コード
私がCythonで書いたプログラムのPythonチックな擬似コードです。
def phase_search(phase, indexes, depth, h_i): # phase: フェーズ, indexes: パズルの状態インデックス, depth: 残り回せる手数, h_i: indexesの状態でのh(indexes) global path if depth == 0: # 残り手数0の場合には解にたどり着いたかを返す return h_i == 0 twist = successor[phase][0] # twistは回す手 while twist <= successor[phase][-1] : # successorは回転の候補 if skip(twist, path): # 前の手で回したのと同じ面を回すなどはしない twist = skip_axis[phase][twist] # 同じ面を回さなくなるまでtwistを進める continue next_indexes = move(indexes, phase, twist) # 配列参照によってパズルを動かす。indexesの定義はフェーズによって異なるのでフェーズを引数にとる next_h_i = h(indexes, phase) # h(i)を返す。h(i)もフェーズごとに定義が異なるのでフェーズを引数に取る。 if next_h_i > depth or next_h_i > h_i + 1: # 明らかに遠回りをしようとしている場合はスキップ twist = skip_axis[phase][twist] path.append(twist) # ここまで回してきた手順にtwistを追加する if phase_search(phase, next_indexes, depth - 1, next_h_i): # 再帰で次の手を探索する return True # 解が見つかった path.pop() # ここまで回してきた手順から今回した手順を取り除く twist = next_twist(twist, phase) # 次の手 return False # 解が見つからなかった def solver(puzzle): # puzzle: パズルのすべての状態を表したクラスのオブジェクトなど global path solution = [] # 解 for phase in range(6): # フェーズをforで回す indexes = initialize_indexes(puzzle, phase) # パズルの状態をインデックスに変換 h_i = h(indexes, phase) for depth in range(60): # depthを回す。なお60は適当 path = [] # pathを空にする if phase_search(phase, indexes, depth, h_i): # IDA*探索を行う for twist in path: # フェーズが終わった状態までパズルをシミュレート puzzle = puzzle.move(twist) solution.extend(path) # solutionに今のフェーズの解を付け加える break # 解が見つかったのでbreakして次のフェーズへ return solution # 解を返すまとめ
ここまで3回の記事で私が4x4x4ルービックキューブを解くプログラムを書く上で学んだことをまとめて書いてきました。皆様の参考になれば幸いです。
- 投稿日:2020-08-11T20:29:53+09:00
djangoでスキーマに動的テーブル作成、そして動的Model生成
概要
DjangoのORMは素晴らしいが、動的に作成したテーブルや、postgresの特定スキーマ内のテーブル等、少し想定と異なるテーブルに対してModelを作成しようとすると、なかなか情報が少なく難しい。
また、作成済のテーブルを流用し、別のスキーマにテーブルを作成するなど少々込み入ったことをしようとすると、調査に時間がかかり、とたんに開発の手が数日止まってしまう。
この記事は、これら課題に直面し、四苦八苦しながら、以下を実現しようとした記録である。
<実現したいこと>
- 既存のModelと同じ定義で、スキーマのみが異なるModelを、既存Modelを流用して作成したい。
- 作成したModelを使って、スキーマにテーブルを動的に作成したい。
- 作成したModelを使って、DjangoのORM機能を使って、検索や登録を行いたい。まとめると、少々こみった要件に対し、DjangのModel機能は秀逸なので、テーブル作成や検索などの恩恵を最大限に受けたいわけで。
そもそもの動機
なぜPostgresのスキーマを使いたいのか
- PostgreSQL 10.5文書のスキーマ用途の一つである「1つのデータベースを多数のユーザが互いに干渉することなく使用できるようにする」を実現したいためである。今回、いろんなユーザが利用できる解析サービスの実現を目指しているが、ユーザのデータ同士の干渉をなくしたいため、同じテーブル構成のスキーマを各ユーザ毎に用意したいのだ。
なぜ既存テーブルを流用したいのか。
- ユーザ登録がされた時点で、そのユーザ用のスキーマや、そのスキーマの中のテーブルを動的に作りたい。この時、わざわざcreate文を用意することなく、すでに存在するModelから作りたいのである。だってcreate文ってプログラムと二重管理になるじゃん。
環境
今回の環境は以下のとおりである。
- python 3.7.6
- django 3.0.8
- Postgres 12やり方
基本、参考文献の応用で実現できた。以下に詳細を記載する。
流用元となるModelの定義
まず、以降の説明のため、流用元となるModelの例を以下の通りとする。
app/models/model.pyclass Compound(models.Model): name = models.CharField(max_length=1024) smiles = models.CharField(max_length=4000, null=True) standard_inchi = models.CharField(max_length=4000, default=None, null=True) mol = models.TextField(null=True) image = models.BinaryField(null=True)流用元のModelは、Djangoのmigration管理下にあるため、makemigration, migrateによりModelに対するテーブルを作成できるはずだ。そして、特別な設定をしない限り、そのテーブルはpublicスキーマに作成される。
Modelを動的に作成する関数を作成
次に今回の肝となる関数を説明する。
通常Djangoでは、あらかじめModelクラスをソースにベタうちして作っておく必要があるが、今回はDynamic models を参考に、所望のModelクラスを動的に作成する関数を用意した。
具体的は、スキーマ、テーブル名、流用元のModelクラスを指定してあらたなModelを作成できるようにした。ソースは以下のとおりである。
なお、関数のためどこに作成してもよいが、今回流用元と同じモジュールに定義している。app/models/model.pydef create_model_by_prototype(model_name, schema_name, table_name, prototype): import copy class Meta: pass setattr(Meta, "db_table", schema_name + "\".\"" + table_name) fields = {} for field in prototype._meta.fields: fields[field.name] = copy.deepcopy(field) attrs = {'__module__': "app.models.models", 'Meta': Meta} attrs.update(fields) model = type(model_name, (models.Model,), attrs) return model解説
具体的な利用例は後で見ていくこととし、ソースについて簡単に解説する。
- 引数として、schema_nameにはテーブルを作成するスキーマ、table_nameにはテーブル名、prototypeにはひな型とするModelクラスを指定する。
Metaというクラスに、モデルを生成するための属性を設定していくsetattrで、Modelの管理するテーブル名を表す属性"db_table"に対し、引数で指定されたテーブル名を設定しているfor field in prototype._meta.fields:というループで、流用元のModelのfieldを、今回作成するモデルのfieldに設定するための準備を行っている。- 最終的に、
type(model_name, (models.Model,), attrs)で、準備した属性情報を元に、新たなModelクラスを生成し、返却している。ハンズオン
さて、作成した関数を用いた関数の利用イメージを、django shellによるハンズオンで見ていこう。
流用元となるModel、テーブルの作成
これは、Django の makemigrations, migrateを実行するだけなので省略する。
新規スキーマの作成
新たなテーブルを格納するためのスキーマは、今回面倒だったので直接Postgresに接続し作成した。スキーマ名をuser01とした。
create schema user01;Django shellの起動
$ python manage.py shell Python 3.7.6 | packaged by conda-forge | (default, Jun 1 2020, 18:11:50) [MSC v.1916 64 bit (AMD64)] Type 'copyright', 'credits' or 'license' for more information IPython 7.16.1 -- An enhanced Interactive Python. Type '?' for help.モジュールのインポート
続いて、関数を利用するために必要なモジュールのインポートを行う。
from chempre.models.models import Compound from chempre.models import models新規Modelの動的作成
さぁ、いよいよ作成した関数を使って、Modelを動的に作成してみよう。
ここでは、CompoundというModelを流用し、同じ定義を持つmy_compoundというテーブルを、先ほど作成したuser01スキーマに作成する。model= models.create_model_by_prototype("MyCompound", "user01", "my_compound", Compound)テーブルの作成
続いて、Modelに対応するテーブルを作成する。今回のModelはmigrationの管理対象外であるため、通常とは異なる作成方法となる。その手順は以下の通りだ。
from django.db import connection with connection.schema_editor() as schema_editor: schema_editor.create_model(model)これでスキーマuser01にテーブルが作成される。実際にPostgresにpsqlで接続してみると確かに作成されている。
Modelへのデータの登録
続いてデータの登録を行ってみよう。フィールドの指定が面倒だったので、ここではnameフィールドのみ設定したレコードを作成している。
model.objects.create(name='compound name')Modelへの検索
テーブルが作成されたかどうか検索して確認してみよう。
compound=model.objects.get(id=1)[ print(compound.name)登録されていれば、'compund name'という値が表示されるはずだ。
また、実際にPostgresにpsqlで接続してみるも、確かにレコードが作成されている。おわりに
無事、当初掲げていた実現したいことを全て実現することができた。この方法でガンガンModelを量産し、Djangoライフを楽しみたい。
Djangoは、お作法が多く、覚えることも山ほどあるが、この手のフレームワークにありがちな、ガチガチで融通が利かないといったことは全くなく、カスタマイズも柔軟にできそうな印象だ。参考文献
- 投稿日:2020-08-11T20:29:53+09:00
Djangoでスキーマに動的テーブル作成、そして動的Model生成
概要
DjangoのORM機能は素晴らしいが、動的に作成したテーブルや、postgresの特定スキーマ内のテーブル等、少し想定と異なるテーブルに対してModelを作成しようとすると、なかなか情報が少ない。
また、作成済のテーブルを流用し、別のスキーマに同じ定義のテーブルを作成するなど少々込み入ったことをしようとすると、調査に時間がかかり、とたんに開発の手が数日止まってしまう。
この記事は、これら課題に直面し、四苦八苦しながら、以下を実現しようとした記録である。
<実現したいこと>
- 既存のModelと同じ定義で、スキーマのみが異なるModelを、既存Modelを流用して動的に作成したい。
- 作成したModelを使って、スキーマにテーブルを動的に作成したい。
- 作成したModelを使って、DjangoのORM機能を使って、検索や登録を行いたい。要するに、少々こみった要件ではあるが、DjangoのModel機能は秀逸なので、テーブル作成や検索などの恩恵を最大限に受けたいわけで。
そもそもの動機
なぜPostgresのスキーマを使いたいのか
- PostgreSQL 10.5文書に記載されているスキーマ用途の一つである「1つのデータベースを多数のユーザが互いに干渉することなく使用できるようにする」を実現したいためである。今回、いろんなユーザが利用できる解析サービスの実現を目指しているが、ユーザのデータ同士の干渉をなくしたいため、同じテーブル構成のスキーマを各ユーザ毎に用意したいのだ。
なぜ既存テーブルを流用したいのか。
- ユーザ登録がされた時点で、そのユーザ用のスキーマや、そのスキーマの中のテーブルを動的に作りたい。この時、わざわざCreate文を用意することなく、既に定義済のModelから作りたいのである。だってCreate文ってプログラムとの二重管理になるじゃん。
環境
今回の環境は以下のとおりである。
- python 3.7.6
- django 3.0.8
- Postgres 12やり方
基本、参考文献の応用で実現できた。以下に詳細を記載する。
流用元となるModelの定義
まず、以降の説明のため、流用元となるModelの例を以下の通りとする。
app/models/model.pyclass Compound(models.Model): name = models.CharField(max_length=1024) smiles = models.CharField(max_length=4000, null=True) standard_inchi = models.CharField(max_length=4000, default=None, null=True) mol = models.TextField(null=True) image = models.BinaryField(null=True)流用元のModelは、Djangoのmigration管理下にあるため、makemigration, migrateによりModelに対するテーブルを作成できるはずだ。そして、特別な設定をしない限り、そのテーブルはpublicスキーマに作成される。
Modelを動的に作成する関数を作成
次に今回の肝となる関数を説明する。
通常Djangoでは、あらかじめModelクラスをソースにベタうちして作っておく必要があるが、今回はDynamic models を参考に、所望のModelクラスを動的に作成する関数を用意した。
具体的は、スキーマ、テーブル名、流用元のModelクラスを指定してあらたなModelを作成できるようにした。ソースは以下のとおりである。
なお、関数のためどこに作成してもよいが、今回流用元と同じモジュールに定義している。app/models/model.pydef create_model_by_prototype(model_name, schema_name, table_name, prototype): import copy class Meta: pass setattr(Meta, "db_table", schema_name + "\".\"" + table_name) fields = {} for field in prototype._meta.fields: fields[field.name] = copy.deepcopy(field) attrs = {'__module__': "app.models.models", 'Meta': Meta} attrs.update(fields) model = type(model_name, (models.Model,), attrs) return model解説
具体的な利用例は後で見ていくこととし、ソースについて簡単に解説する。
- 引数として、schema_nameにはテーブルを作成するスキーマ、table_nameにはテーブル名、prototypeにはひな型とするModelクラスを指定する。
Metaというクラスに、モデルを生成するための属性を設定していくsetattrで、Modelの管理するテーブル名を表す属性"db_table"に対し、引数で指定されたテーブル名を設定しているfor field in prototype._meta.fields:というループで、流用元のModelのfieldを、今回作成するモデルのfieldに設定するための準備を行っている。- 最終的に、
type(model_name, (models.Model,), attrs)で、準備した属性情報を元に、新たなModelクラスを生成し、返却している。ハンズオン
さて、作成した関数を用いた関数の利用イメージを、django shellによるハンズオンで見ていこう。
流用元となるModel、テーブルの作成
これは、Django の makemigrations, migrateを実行するだけなので省略する。
新規スキーマの作成
新たなテーブルを格納するためのスキーマは、今回面倒だったので直接Postgresに接続し作成した。スキーマ名をuser01とした。
create schema user01;Django shellの起動
$ python manage.py shell Python 3.7.6 | packaged by conda-forge | (default, Jun 1 2020, 18:11:50) [MSC v.1916 64 bit (AMD64)] Type 'copyright', 'credits' or 'license' for more information IPython 7.16.1 -- An enhanced Interactive Python. Type '?' for help.モジュールのインポート
続いて、関数を利用するために必要なモジュールのインポートを行う。
from chempre.models.models import Compound from chempre.models import models新規Modelの動的作成
さぁ、いよいよ作成した関数を使って、Modelを動的に作成してみよう。
ここでは、CompoundというModelを流用し、同じ定義を持つmy_compoundというテーブルを、先ほど作成したuser01スキーマに作成する。model= models.create_model_by_prototype("MyCompound", "user01", "my_compound", Compound)テーブルの作成
続いて、Modelに対応するテーブルを作成する。今回のModelはmigrationの管理対象外であるため、通常とは異なる作成方法となる。その手順は以下の通りだ。
from django.db import connection with connection.schema_editor() as schema_editor: schema_editor.create_model(model)これでスキーマuser01にテーブルが作成される。実際にPostgresにpsqlで接続してみると確かに作成されている。
Modelへのデータの登録
続いてデータの登録を行ってみよう。フィールドの指定が面倒だったので、ここではnameフィールドのみ設定したレコードを作成している。
model.objects.create(name='compound name')Modelへの検索
テーブルが作成されたかどうか検索して確認してみよう。
compound=model.objects.get(id=1)[ print(compound.name)登録されていれば、'compund name'という値が表示されるはずだ。
また、実際にPostgresにpsqlで接続してみるも、確かにレコードが作成されている。おわりに
無事、当初掲げていた実現したいことを全て実現することができた。この方法でガンガンModelを量産し、Djangoライフを楽しみたい。
Djangoは、お作法が多く、覚えることも山ほどあるが、この手のフレームワークにありがちな、ガチガチで融通が利かないといったことは全くなく、カスタマイズも柔軟にできそうな印象だ。参考文献
- 投稿日:2020-08-11T20:29:53+09:00
Djangoでスキーマに動的テーブル作成、動的Model生成
概要
DjangoのORM機能は素晴らしいが、動的に作成したテーブルや、postgresの特定スキーマ内のテーブル等、少し想定と異なるテーブルに対してModelを作成しようとすると、なかなか情報が少ない。
また、作成済のテーブルを流用し、別のスキーマに同じ定義のテーブルを作成するなど少々込み入ったことをしようとすると、調査に時間がかかり、とたんに開発の手が数日止まってしまう。
この記事は、これら課題に直面し、四苦八苦しながら、以下を実現しようとした記録である。
<実現したいこと>
- 既存のModelと同じ定義で、スキーマのみが異なるModelを、既存Modelを流用して動的に作成したい。
- 作成したModelを使って、スキーマにテーブルを動的に作成したい。
- 作成したModelを使って、DjangoのORM機能を使って、検索や登録を行いたい。
要するに、少々こみった要件ではあるが、DjangoのModelクラスを利用したORM機能は秀逸なので、テーブル作成やレコード登録、検索などの恩恵を最大限に受けたいわけで。
そもそもの動機
なぜPostgresのスキーマを使いたいのか
- PostgreSQL 10.5文書に記載されているスキーマ用途の一つである「1つのデータベースを多数のユーザが互いに干渉することなく使用できるようにする」を実現したいためである。今回、いろんなユーザが利用できる解析サービスの実現を目指しているが、ユーザのデータ同士の干渉をなくしたいため、同じテーブル構成のスキーマをユーザ毎に用意したいのだ。
なぜ既存テーブル・既存Modelを流用したいのか。
- ユーザ登録がされた時点で、そのユーザ用のスキーマや、そのスキーマの中のテーブルを動的に作りたい。この時、わざわざCreate文を用意することなく、既に定義済のModelから作りたいのである。だってCreate文とModelのソースでDDLの二重管理になるじゃん。
環境
今回の環境は以下のとおりである。
- python 3.7.6
- django 3.0.8
- Postgres 12やり方
結論からいうと、参考文献の応用で実現できた。以下に詳細を記載する。
流用元となるModelの定義
まず、以降の説明のため、流用元となるModelの例を以下の通りとする。
app/models/model.pyclass Compound(models.Model): name = models.CharField(max_length=1024) smiles = models.CharField(max_length=4000, null=True) standard_inchi = models.CharField(max_length=4000, default=None, null=True) mol = models.TextField(null=True) image = models.BinaryField(null=True)流用元のModelは、Djangoのmigration管理下にあるため、makemigration, migrateによりModelに対するテーブルを作成できるはずだ。そして、特別な設定をしない限り、そのテーブルはpublicスキーマに作成される。
Modelを動的に作成する関数を作成
次に今回の肝となる関数を説明する。
通常Djangoでは、あらかじめModelクラスをソースにベタうちして作っておく必要があるが、今回はDynamic models を参考に、所望のModelクラスを動的に作成する関数を用意した。
具体的は、スキーマ、テーブル名、流用元のModelクラスを指定してあらたなModelを作成できるようにした。ソースは以下のとおりである。
なお、関数のためどこに作成してもよいが、今回流用元と同じモジュールに定義している。app/models/model.pydef create_model_by_prototype(model_name, schema_name, table_name, prototype): import copy class Meta: pass setattr(Meta, "db_table", schema_name + "\".\"" + table_name) fields = {} for field in prototype._meta.fields: fields[field.name] = copy.deepcopy(field) attrs = {'__module__': "app.models.models", 'Meta': Meta} attrs.update(fields) model = type(model_name, (models.Model,), attrs) return model解説
具体的な利用例は後で見ていくこととし、ソースについて簡単に解説する。
- 引数として、schema_nameにはテーブルを作成するスキーマ、table_nameにはテーブル名、prototypeにはひな型とするModelクラスを指定する。
Metaというクラスに、モデルを生成するための属性を設定していくsetattrで、Modelの管理するテーブル名を表す属性"db_table"に対し、引数で指定されたテーブル名を設定しているfor field in prototype._meta.fields:というループで、流用元のModelのfieldを、今回作成するモデルのfieldに設定するための準備を行っている。- 最終的に、
type(model_name, (models.Model,), attrs)で、準備した属性情報を元に、新たなModelクラスを生成し、返却している。ハンズオン
さて、作成した関数を用いた関数の利用イメージを、django shellによるハンズオンで見ていこう。
流用元となるModel、テーブルの作成
これは、Django の makemigrations, migrateを実行するだけなので省略する。
新規スキーマの作成
新たなテーブルを格納するためのスキーマは、今回面倒だったので直接Postgresに接続し作成した。スキーマ名をuser01とした。
create schema user01;Django shellの起動
$ python manage.py shell Python 3.7.6 | packaged by conda-forge | (default, Jun 1 2020, 18:11:50) [MSC v.1916 64 bit (AMD64)] Type 'copyright', 'credits' or 'license' for more information IPython 7.16.1 -- An enhanced Interactive Python. Type '?' for help.モジュールのインポート
続いて、関数を利用するために必要なモジュールのインポートを行う。
from chempre.models.models import Compound from chempre.models import models新規Modelの動的作成
さぁ、いよいよ作成した関数を使って、Modelを動的に作成してみよう。
ここでは、CompoundというModelを流用し、同じ定義を持つmy_compoundというテーブルを、先ほど作成したuser01スキーマに作成する。model= models.create_model_by_prototype("MyCompound", "user01", "my_compound", Compound)テーブルの作成
続いて、Modelに対応するテーブルを作成する。今回のModelはmigrationの管理対象外であるため、通常とは異なる作成方法となる。その手順は以下の通りだ。
from django.db import connection with connection.schema_editor() as schema_editor: schema_editor.create_model(model)これでスキーマuser01にテーブルが作成される。実際にPostgresにpsqlで接続してみると確かに作成されている。
Modelへのデータの登録
続いてデータの登録を行ってみよう。フィールドの指定が面倒だったので、ここではnameフィールドのみ設定したレコードを作成している。
model.objects.create(name='compound name')Modelへの検索
テーブルが作成されたかどうか検索して確認してみよう。
compound=model.objects.get(id=1)[ print(compound.name)登録されていれば、'compund name'という値が表示されるはずだ。
また、実際にPostgresにpsqlで接続してみるも、確かにレコードが作成されている。おわりに
無事、当初掲げていた実現したいことを全て実現することができた。この方法でガンガンModelを量産し、Djangoライフを楽しみたい。
Djangoは、お作法が多く、覚えることも山ほどあるが、この手のフレームワークにありがちな、ガチガチで融通が利かないといったことは全くなく、カスタマイズも柔軟にできそうな印象だ。参考文献
- 投稿日:2020-08-11T20:29:53+09:00
Djangoでスキーマに動的にテーブル作成、動的にModel生成
概要
DjangoのORM機能は素晴らしいが、動的に作成したテーブルや、postgresの特定スキーマ内のテーブル等、少し想定と異なるテーブルに対してModelを作成しようとすると、なかなか情報が少ない。
また、作成済のテーブルを流用し、別のスキーマに同じ定義のテーブルを作成するなど少々込み入ったことをしようとすると、調査に時間がかかり、とたんに開発の手が数日止まってしまう。
この記事は、これら課題に直面し、四苦八苦しながら、以下を実現しようとした記録である。
<実現したいこと>
- 既存のModelと同じ定義で、スキーマのみが異なるModelを、既存Modelを流用して動的に作成したい。
- 作成したModelを使って、作成したModelとは異なるスキーマにテーブルを動的に作成したい。
- 作成したModelを使って、DjangoのORM機能を使って、検索や登録を行いたい。
要するに、少々こみった要件ではあるが、DjangoのModelクラスを利用したORM機能は秀逸なので、テーブル作成やレコード登録、検索などの恩恵を最大限に受けたいわけで。
そもそもの動機
なぜPostgresの標準スキーマ以外のスキーマを使いたいのか
- PostgreSQL 10.5文書に記載されているスキーマ用途の一つである「1つのデータベースを多数のユーザが互いに干渉することなく使用できるようにする」を実現したいためである。今回、いろんなユーザが利用できる解析サービスの実現を目指しているが、ユーザのデータ同士の干渉をなくしたいため、同じテーブル構成のスキーマをユーザ毎に用意したいのだ。
なぜ既存テーブル・既存Modelを流用したいのか。
- ユーザ登録がされた時点で、そのユーザ用のスキーマや、そのスキーマの中のテーブルを動的に作りたい。この時、わざわざCreate文を用意することなく、既に定義済のModelから作りたいのである。だってCreate文とModelのソースでDDLの二重管理になるじゃん。
環境
今回の環境は以下のとおりである。
- python 3.7.6
- django 3.0.8
- Postgres 12やり方
結論からいうと、参考文献の応用で実現できた。以下に詳細を記載する。
流用元となるModelの定義
まず、以降の説明のため、流用元となるModelの例を以下の通りとする。
app/models/model.pyclass Compound(models.Model): name = models.CharField(max_length=1024) smiles = models.CharField(max_length=4000, null=True) standard_inchi = models.CharField(max_length=4000, default=None, null=True) mol = models.TextField(null=True) image = models.BinaryField(null=True)流用元のModelは、Djangoのmigration管理下にあるため、makemigration, migrateによりModelに対するテーブルを作成できるはずだ。そして、特別な設定をしない限り、そのテーブルはpublicスキーマに作成される。
Modelを動的に作成する関数を作成
次に今回の肝となる関数を説明する。
通常Djangoでは、あらかじめModelクラスをソースにベタうちして作っておく必要があるが、今回はDynamic models を参考に、所望のModelクラスを動的に作成する関数を用意した。
具体的は、スキーマ、テーブル名、流用元のModelクラスを指定してあらたなModelを作成できるようにした。ソースは以下のとおりである。
なお、関数のためどこに作成してもよいが、今回流用元と同じモジュールに定義している。app/models/model.pydef create_model_by_prototype(model_name, schema_name, table_name, prototype): import copy class Meta: pass setattr(Meta, "db_table", schema_name + "\".\"" + table_name) fields = {} for field in prototype._meta.fields: fields[field.name] = copy.deepcopy(field) attrs = {'__module__': "app.models.models", 'Meta': Meta} attrs.update(fields) model = type(model_name, (models.Model,), attrs) return model解説
具体的な利用例は後で見ていくこととし、ソースについて簡単に解説する。
- 引数として、schema_nameにはテーブルを作成するスキーマ、table_nameにはテーブル名、prototypeにはひな型とするModelクラスを指定する。
Metaというクラスに、モデルを生成するための属性を設定していくsetattrで、Modelの管理するテーブル名を表す属性"db_table"に対し、引数で指定されたテーブル名を設定しているfor field in prototype._meta.fields:というループで、流用元のModelのfieldを、今回作成するモデルのfieldに設定するための準備を行っている。- 最終的に、
type(model_name, (models.Model,), attrs)で、準備した属性情報を元に、新たなModelクラスを生成し、返却している。ハンズオン
さて、作成した関数を用いた関数の利用イメージを、django shellによるハンズオンで見ていこう。
流用元となるModel、テーブルの作成
これは、Django の makemigrations, migrateを実行するだけなので省略する。
新規スキーマの作成
新たなテーブルを格納するためのスキーマは、今回面倒だったので直接Postgresに接続し作成した。スキーマ名をuser01とした。
create schema user01;Django shellの起動
$ python manage.py shell Python 3.7.6 | packaged by conda-forge | (default, Jun 1 2020, 18:11:50) [MSC v.1916 64 bit (AMD64)] Type 'copyright', 'credits' or 'license' for more information IPython 7.16.1 -- An enhanced Interactive Python. Type '?' for help.モジュールのインポート
続いて、関数を利用するために必要なモジュールのインポートを行う。
from chempre.models.models import Compound from chempre.models import models新規Modelの動的作成
さぁ、いよいよ作成した関数を使って、Modelを動的に作成してみよう。
ここでは、CompoundというModelを流用し、同じ定義を持つmy_compoundというテーブルを、先ほど作成したuser01スキーマに作成する。model= models.create_model_by_prototype("MyCompound", "user01", "my_compound", Compound)テーブルの作成
続いて、Modelに対応するテーブルを作成する。今回のModelはmigrationの管理対象外であるため、通常とは異なる作成方法となる。その手順は以下の通りだ。
from django.db import connection with connection.schema_editor() as schema_editor: schema_editor.create_model(model)これでスキーマuser01にテーブルが作成される。実際にPostgresにpsqlで接続してみると確かに作成されている。
Modelへのデータの登録
続いてデータの登録を行ってみよう。フィールドの指定が面倒だったので、ここではnameフィールドのみ設定したレコードを作成している。
model.objects.create(name='compound name')Modelへの検索
テーブルが作成されたかどうか検索して確認してみよう。
compound=model.objects.get(id=1)[ print(compound.name)登録されていれば、'compund name'という値が表示されるはずだ。
また、実際にPostgresにpsqlで接続してみるも、確かにレコードが作成されている。おわりに
無事、当初掲げていた実現したいことを全て実現することができた。この方法でガンガンModelを量産し、Djangoライフを楽しみたい。
Djangoは、お作法が多く、覚えることも山ほどあるが、この手のフレームワークにありがちな、ガチガチで融通が利かないといったことは全くなく、カスタマイズも柔軟にできそうな印象だ。参考文献
- 投稿日:2020-08-11T19:24:56+09:00
Scrapyの始め方
公式ドキュメント:
https://doc-ja-scrapy.readthedocs.io/ja/latest/index.html
クローリングを実行するまで
# プロジェクト作成 $ scrapy startproject <project name> # 設定 $ cat setting.py DOWNLOAD_DELAY = 1 FEED_EXPORT_ENCODING = "utf-8" # spider作成 $ scrapy genspider <mydomain> <mydomain.com> # parseの処理を書いてクローリング実行 $ scrappy crawl <spider name>parseの例
def parse(self, response): for sel in response.css('#gmap_list > li > a'): next_page = response.urljoin(sel.css('a::attr("href")').get()) yield scrapy.Request(next_page, callback=self.parse_detail) def parse_detail(self, response): ''' 詳細ページのparse処理 '''ORM
使用するならOratorがシンプルで使いやすそう。
https://orator-orm.com/docs/0.9/basic_usage.html
- 投稿日:2020-08-11T19:13:03+09:00
ベイズ推論での解析(1)・・AとBのどちらが良いか
ベイズ推論解析例題;:AとBのどちらが良いか
Pythonで体験するベイズ推論より
ベイズ推論でサイトAとサイトBのどちらが良いか?という問題です。
問題:サイトAの真のコンバージョンは?
サイトAを見たユーザーが最終的に資料請求や購入など利益につながるアクション(これをコンバージョンという)につながる確率を$p_A$とする。
$N$人がサイトAを見てそのうち$n$人がコンバージョンにつながったとする。
そうすると、p_A=n/Nと思うかもしれないが、それは違う。つまり、$p_A$と$n/N$が等しいかどうかわからないからである。観測された頻度と真の頻度には差があり、真の頻度は事象が発生する確率と解釈できる。つまり、サイトAを見たから、アクションにつながったかどうかはわからないからである。
例えば、サイコロを例にとると、サイコロを振って1が出る真の頻度は$1/6$であるが、実際にサイコロを6回降っても一回も1が出ないかもしれない。それが観測された頻度である。
残念ながら現実は複雑すぎてノイズも避けられないので、真の頻度はわからないので、観測された頻度から真の頻度を推定するしかない。ベイズ統計から、事前分布と観測データ($n/N$)から真の頻度($p_A$)を推定するのである。まず未知数$p_A$について事前分布を考えないといけない。データを持っていない時に、$p_A$をどう思っているか?まだ確信が持てないので、$p_A$は[0,1]の一様分布に従うと仮定しよう。
変数pに@pm.Uniformで、0から1の一様分布を与える.import pymc3 as pm # The parameters are the bounds of the Uniform. with pm.Model() as model: p = pm.Uniform('p', lower=0, upper=1)例えば、$p_A=0.05$であり、$N=1500$ユーザーがサイトAを見た時に、ユーザーがコンバージョン(サイトを見て購入や資料請求)したのが何人か、シミュレーションしてみよう。実際には$p_A$はわからないが、とりあえず知っていると仮定してシミュレーションして、観測データを作る。
N人がいるので、N回の試行のシミュレーションするために、ベルヌーイ分布を使う。ベルヌイー分布は二値変数(0か1かのどちらかを取る変数)についての確率分布であるため、コンバージョンしたか、しないかの計算に良い確率分布である。
X\sim \text{Ber}(p)この時、$X$は確率$p$で$1$確率$1-p$で$0$である。
これをシミュレーションする
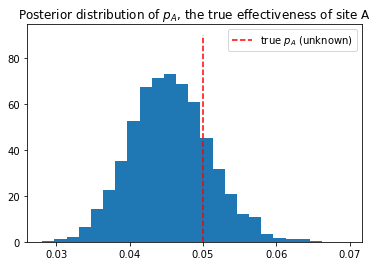
pythonスクリプトは、import scipy.stats as stats import numpy as np #set constants p_true = 0.05 # remember, this is unknown. N = 1500 # sample N Bernoulli random variables from Ber(0.05). # each random variable has a 0.05 chance of being a 1. # this is the data-generation step occurrences = stats.bernoulli.rvs(p_true, size=N) print(occurrences) # Remember: Python treats True == 1, and False == 0 print(np.sum(occurrences)) #[0 0 0 ... 0 0 0] #67 # Occurrences.mean is equal to n/N. print("What is the observed frequency in Group A? %.4f" % np.mean(occurrences)) print("Does this equal the true frequency? %s" % (np.mean(occurrences) == p_true)) #What is the observed frequency in Group A? 0.0447 #Does this equal the true frequency? Falseこれで観測データ(occurrences)ができたので、観測データを
PyMCのobs変数に設定して推論アルゴリズムを実行し、未知である$p_A$の事後分布のグラフを作る。#include the observations, which are Bernoulli with model: obs = pm.Bernoulli("obs", p, observed=occurrences) # To be explained in chapter 3 step = pm.Metropolis() trace = pm.sample(18000, step=step,cores =1) burned_trace = trace[1000:] import matplotlib.pyplot as plt plt.title("Posterior distribution of $p_A$, the true effectiveness of site A") plt.vlines(p_true, 0, 90, linestyle="--",color="r", label="true $p_A$ (unknown)") plt.hist(burned_trace["p"], bins=25, histtype="bar",density=True) plt.legend() plt.show()今回は、真の値の事前確率0.5に対し、事後確率のグラフはこのようなグラフになっています。これは繰り返せば、毎回微妙に違うグラフになります。
問題:サイトAとサイトBを比較すると、どちらが良いサイト?
サイトBも同様に計算すれば、$p_B$の事後確率を求めることができる。
またそこで、$delta=p_A-p_B$を計算することにした。$p_A$と同様に$p_B$も真の値はわからない。仮に$p_B=0.4$とした。そうなると$delta=0.1$となる。先ほどの$p_A$と同様に$p_B$と$delta$を計算する。再度、すべてを計算してみた。
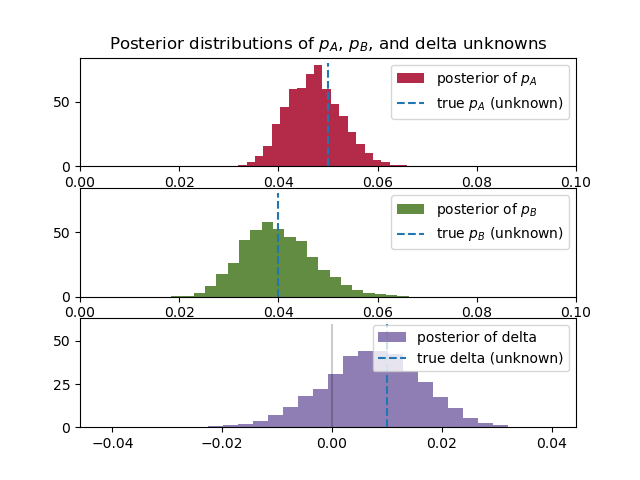
# -*- coding: utf-8 -*- import pymc3 as pm import scipy.stats as stats import numpy as np import matplotlib.pyplot as plt #these two quantities are unknown to us. true_p_A = 0.05 true_p_B = 0.04 #notice the unequal sample sizes -- no problem in Bayesian analysis. N_A = 1500 N_B = 750 #generate some observations observations_A = stats.bernoulli.rvs(true_p_A, size=N_A) observations_B = stats.bernoulli.rvs(true_p_B, size=N_B) print("Obs from Site A: ", observations_A[:30], "...") print("Obs from Site B: ", observations_B[:30], "...") #Obs from Site A: [0 0 0 0 0 0 0 0 0 1 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0] ... #Obs from Site B: [0 0 0 0 0 0 0 0 0 0 0 0 1 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0] ... print(np.mean(observations_A)) #0.04666666666666667 print(np.mean(observations_B)) #0.03866666666666667 # Set up the pymc3 model. Again assume Uniform priors for p_A and p_B. with pm.Model() as model: p_A = pm.Uniform("p_A", 0, 1) p_B = pm.Uniform("p_B", 0, 1) # Define the deterministic delta function. This is our unknown of interest. delta = pm.Deterministic("delta", p_A - p_B) # Set of observations, in this case we have two observation datasets. obs_A = pm.Bernoulli("obs_A", p_A, observed=observations_A) obs_B = pm.Bernoulli("obs_B", p_B, observed=observations_B) # To be explained in chapter 3. step = pm.Metropolis() trace = pm.sample(20000, step=step,cores=1) burned_trace=trace[1000:] p_A_samples = burned_trace["p_A"] p_B_samples = burned_trace["p_B"] delta_samples = burned_trace["delta"] #histogram of posteriors ax = plt.subplot(311) plt.xlim(0, .1) plt.hist(p_A_samples, histtype='stepfilled', bins=25, alpha=0.85, label="posterior of $p_A$", color="#A60628", density=True) plt.vlines(true_p_A, 0, 80, linestyle="--", label="true $p_A$ (unknown)") plt.legend(loc="upper right") plt.title("Posterior distributions of $p_A$, $p_B$, and delta unknowns") ax = plt.subplot(312) plt.xlim(0, .1) plt.hist(p_B_samples, histtype='stepfilled', bins=25, alpha=0.85, label="posterior of $p_B$", color="#467821", density=True) plt.vlines(true_p_B, 0, 80, linestyle="--", label="true $p_B$ (unknown)") plt.legend(loc="upper right") ax = plt.subplot(313) plt.hist(delta_samples, histtype='stepfilled', bins=30, alpha=0.85, label="posterior of delta", color="#7A68A6", density=True) plt.vlines(true_p_A - true_p_B, 0, 60, linestyle="--", label="true delta (unknown)") plt.vlines(0, 0, 60, color="black", alpha=0.2) plt.legend(loc="upper right"); plt.show() # Count the number of samples less than 0, i.e. the area under the curve # before 0, represent the probability that site A is worse than site B. print("Probability site A is WORSE than site B: %.3f" % \ np.mean(delta_samples < 0)) print("Probability site A is BETTER than site B: %.3f" % \ np.mean(delta_samples > 0)) #Probability site A is WORSE than site B: 0.201 #Probability site A is BETTER than site B: 0.799若干$p_B$の事後分布のすそ野が広いことがわかる。これはサイトBの方がデータが少ないためである。これは、サイトBのデータが少ないため、$p_B$の値に自信が持てないことを示している。
また、3つ目のグラフ$delta=p_A-p_B$は、0より大きい値を示している。これはつまりサイトAの方がコンバージョンとしてユーザーの反応が良いことを示している。
$p_A$、$p_B$やデータ数の値を変えて計算をすると、より理解が深まります。
- 投稿日:2020-08-11T19:13:03+09:00
ベイズ推論での解析・・AとBのどちらが良いか
ベイズ推論解析例題;:AとBのどちらが良いか
Pythonで体験するベイズ推論より
ベイズ推論でサイトAとサイトBのどちらが良いか?という問題です。
問題:サイトAの真のコンバージョンは?
サイトAを見たユーザーが最終的に資料請求や購入など利益につながるアクション(これをコンバージョンという)につながる確率を$p_A$とする。
$N$人がサイトAを見てそのうち$n$人がコンバージョンにつながったとする。
そうすると、p_A=n/Nと思うかもしれないが、それは違う。つまり、$p_A$と$n/N$が等しいかどうかわからないからである。観測された頻度と真の頻度には差があり、真の頻度は事象が発生する確率と解釈できる。つまり、サイトAを見たから、アクションにつながったかどうかはわからないからである。
例えば、サイコロを例にとると、サイコロを振って1が出る真の頻度は$1/6$であるが、実際にサイコロを6回降っても一回も1が出ないかもしれない。それが観測された頻度である。
残念ながら現実は複雑すぎてノイズも避けられないので、真の頻度はわからないので、観測された頻度から真の頻度を推定するしかない。ベイズ統計から、事前分布と観測データ($n/N$)から真の頻度($p_A$)を推定するのである。まず未知数$p_A$について事前分布を考えないといけない。データを持っていない時に、$p_A$をどう思っているか?まだ確信が持てないので、$p_A$は[0,1]の一様分布に従うと仮定しよう。
変数pに@pm.Uniformで、0から1の一様分布を与える.import pymc3 as pm # The parameters are the bounds of the Uniform. with pm.Model() as model: p = pm.Uniform('p', lower=0, upper=1)例えば、$p_A=0.05$であり、$N=1500$ユーザーがサイトAを見た時に、ユーザーがコンバージョン(サイトを見て購入や資料請求)したのが何人か、シミュレーションしてみよう。実際には$p_A$はわからないが、とりあえず知っていると仮定してシミュレーションして、観測データを作る。
N人がいるので、N回の試行のシミュレーションするために、ベルヌーイ分布を使う。ベルヌイー分布は二値変数(0か1かのどちらかを取る変数)についての確率分布であるため、コンバージョンしたか、しないかの計算に良い確率分布である。
X\sim \text{Ber}(p)この時、$X$は確率$p$で$1$確率$1-p$で$0$である。
これをシミュレーションする
pythonスクリプトは、import scipy.stats as stats import numpy as np #set constants p_true = 0.05 # remember, this is unknown. N = 1500 # sample N Bernoulli random variables from Ber(0.05). # each random variable has a 0.05 chance of being a 1. # this is the data-generation step occurrences = stats.bernoulli.rvs(p_true, size=N) print(occurrences) # Remember: Python treats True == 1, and False == 0 print(np.sum(occurrences)) #[0 0 0 ... 0 0 0] #67 # Occurrences.mean is equal to n/N. print("What is the observed frequency in Group A? %.4f" % np.mean(occurrences)) print("Does this equal the true frequency? %s" % (np.mean(occurrences) == p_true)) #What is the observed frequency in Group A? 0.0447 #Does this equal the true frequency? Falseこれで観測データ(occurrences)ができたので、観測データを
PyMCのobs変数に設定して推論アルゴリズムを実行し、未知である$p_A$の事後分布のグラフを作る。#include the observations, which are Bernoulli with model: obs = pm.Bernoulli("obs", p, observed=occurrences) # To be explained in chapter 3 step = pm.Metropolis() trace = pm.sample(18000, step=step,cores =1) burned_trace = trace[1000:] import matplotlib.pyplot as plt plt.title("Posterior distribution of $p_A$, the true effectiveness of site A") plt.vlines(p_true, 0, 90, linestyle="--",color="r", label="true $p_A$ (unknown)") plt.hist(burned_trace["p"], bins=25, histtype="bar",density=True) plt.legend() plt.show()今回は、真の値の事前確率0.5に対し、事後確率のグラフはこのようなグラフになっています。これは繰り返せば、毎回微妙に違うグラフになります。
問題:サイトAとサイトBを比較すると、どちらが良いサイト?
サイトBも同様に計算すれば、$p_B$の事後確率を求めることができる。
またそこで、$delta=p_A-p_B$を計算することにした。$p_A$と同様に$p_B$も真の値はわからない。仮に$p_B=0.4$とした。そうなると$delta=0.1$となる。先ほどの$p_A$と同様に$p_B$と$delta$を計算する。再度、すべてを計算してみた。
# -*- coding: utf-8 -*- import pymc3 as pm import scipy.stats as stats import numpy as np import matplotlib.pyplot as plt #these two quantities are unknown to us. true_p_A = 0.05 true_p_B = 0.04 #notice the unequal sample sizes -- no problem in Bayesian analysis. N_A = 1500 N_B = 750 #generate some observations observations_A = stats.bernoulli.rvs(true_p_A, size=N_A) observations_B = stats.bernoulli.rvs(true_p_B, size=N_B) print("Obs from Site A: ", observations_A[:30], "...") print("Obs from Site B: ", observations_B[:30], "...") #Obs from Site A: [0 0 0 0 0 0 0 0 0 1 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0] ... #Obs from Site B: [0 0 0 0 0 0 0 0 0 0 0 0 1 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0] ... print(np.mean(observations_A)) #0.04666666666666667 print(np.mean(observations_B)) #0.03866666666666667 # Set up the pymc3 model. Again assume Uniform priors for p_A and p_B. with pm.Model() as model: p_A = pm.Uniform("p_A", 0, 1) p_B = pm.Uniform("p_B", 0, 1) # Define the deterministic delta function. This is our unknown of interest. delta = pm.Deterministic("delta", p_A - p_B) # Set of observations, in this case we have two observation datasets. obs_A = pm.Bernoulli("obs_A", p_A, observed=observations_A) obs_B = pm.Bernoulli("obs_B", p_B, observed=observations_B) # To be explained in chapter 3. step = pm.Metropolis() trace = pm.sample(20000, step=step,cores=1) burned_trace=trace[1000:] p_A_samples = burned_trace["p_A"] p_B_samples = burned_trace["p_B"] delta_samples = burned_trace["delta"] #histogram of posteriors ax = plt.subplot(311) plt.xlim(0, .1) plt.hist(p_A_samples, histtype='stepfilled', bins=25, alpha=0.85, label="posterior of $p_A$", color="#A60628", density=True) plt.vlines(true_p_A, 0, 80, linestyle="--", label="true $p_A$ (unknown)") plt.legend(loc="upper right") plt.title("Posterior distributions of $p_A$, $p_B$, and delta unknowns") ax = plt.subplot(312) plt.xlim(0, .1) plt.hist(p_B_samples, histtype='stepfilled', bins=25, alpha=0.85, label="posterior of $p_B$", color="#467821", density=True) plt.vlines(true_p_B, 0, 80, linestyle="--", label="true $p_B$ (unknown)") plt.legend(loc="upper right") ax = plt.subplot(313) plt.hist(delta_samples, histtype='stepfilled', bins=30, alpha=0.85, label="posterior of delta", color="#7A68A6", density=True) plt.vlines(true_p_A - true_p_B, 0, 60, linestyle="--", label="true delta (unknown)") plt.vlines(0, 0, 60, color="black", alpha=0.2) plt.legend(loc="upper right"); plt.show() # Count the number of samples less than 0, i.e. the area under the curve # before 0, represent the probability that site A is worse than site B. print("Probability site A is WORSE than site B: %.3f" % \ np.mean(delta_samples < 0)) print("Probability site A is BETTER than site B: %.3f" % \ np.mean(delta_samples > 0)) #Probability site A is WORSE than site B: 0.201 #Probability site A is BETTER than site B: 0.799若干$p_B$の事後分布のすそ野が広いことがわかる。これはサイトBの方がデータが少ないためである。これは、サイトBのデータが少ないため、$p_B$の値に自信が持てないことを示している。
また、3つ目のグラフ$delta=p_A-p_B$は、0より大きい値を示している。これはつまりサイトAの方がコンバージョンとしてユーザーの反応が良いことを示している。
$p_A$、$p_B$やデータ数の値を変えて計算をすると、より理解が深まります。
- 投稿日:2020-08-11T19:02:52+09:00
iPadで機械学習ができるiOS/iPadOSアプリ「Juno」
Junoについて
https://apps.apple.com/jp/app/juno/id1462586500
appstoreで1840円でDLできます。
このアプリ自体は昔からあるんですが、最近のアプデでついにscikit-learnが使えるようになったということなので購入して実際に試してみます。
このアプリ、基本的にはiOS/iPadOSでJupyterNotebookを編集できるというアプリになっていて、
同様にJupyterが使えるcarnetsと言うアプリがあり、無料ということもあってそちらを使っていましたが、scikit-learnは使用できませんでした。
動作チェック
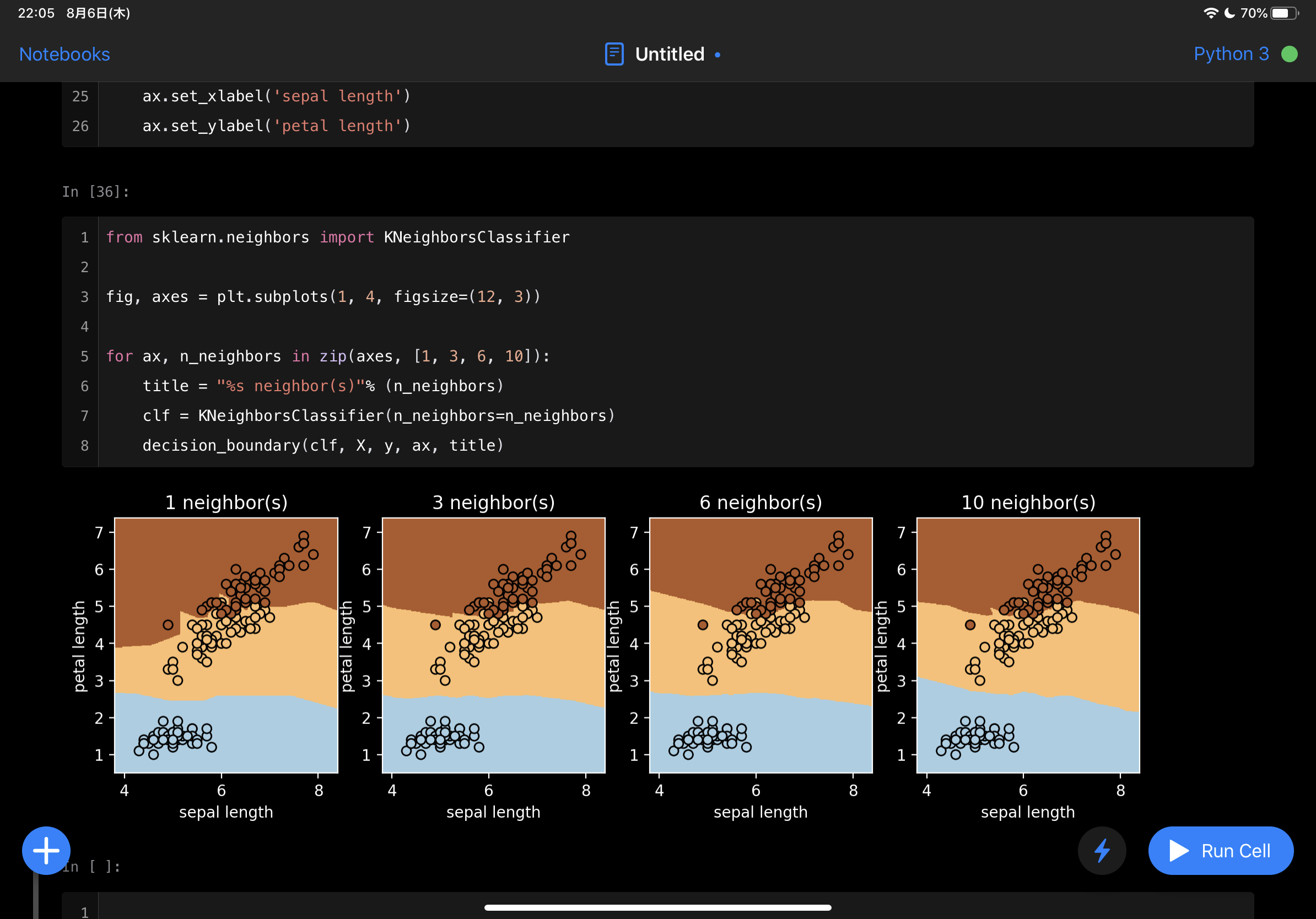
動作チェックはこちらの記事のコードをそのままやっていきます。
結論から言うと、全て動作します。
メイン画面はこんな感じ。PCのJupyterの画面よりもiPad用アプリとして洗練されたイメージ。
ちなみに前述のcarnetsはPCのJupyterと同じような画面構成になっています。
テーマは白背景と黒背景選べます。
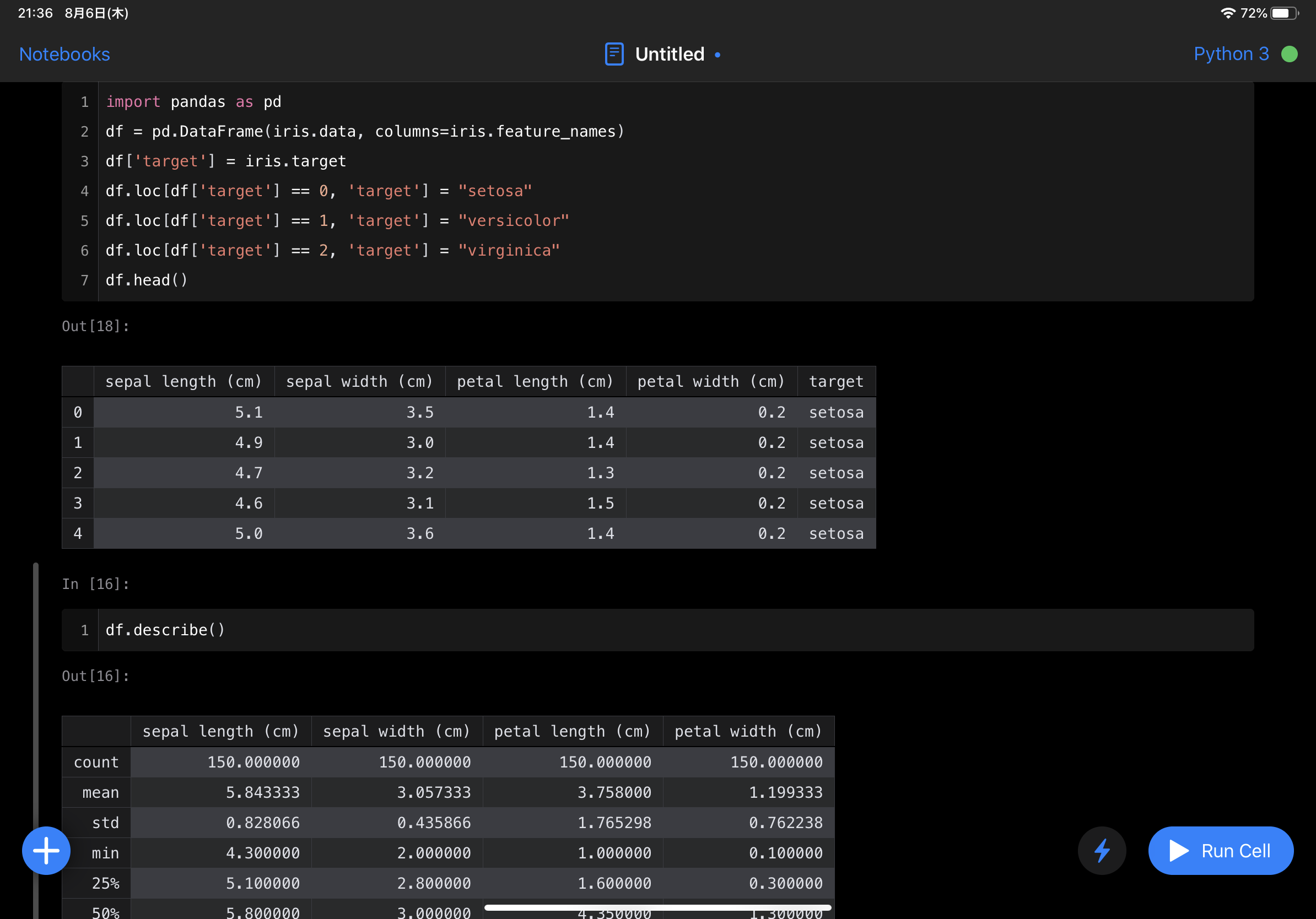
当然sklearnがインポートできていますね。
補完も効きます。
pandasの表も綺麗に表示されます。
iPad専用のapple公式キーボードだとescキーがないですが、escキーを'@'などに割り当てたりすることもでき、ホットキーも充実しています。
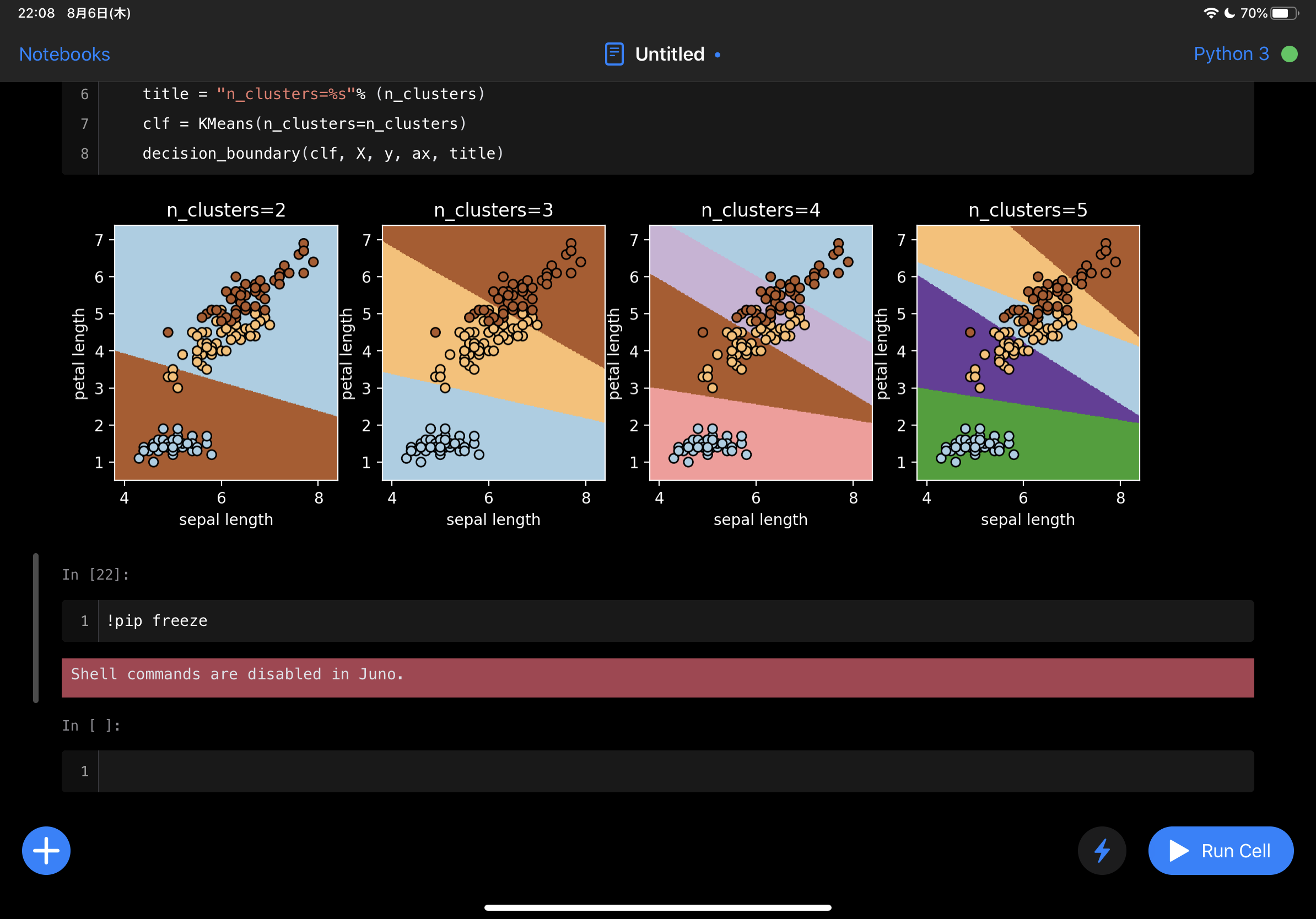
当然グラフも描けて、分類もできています。
iOS/iPadOS 13からある機能のファイルで見られる好きなところのNotebookを開くことができます。
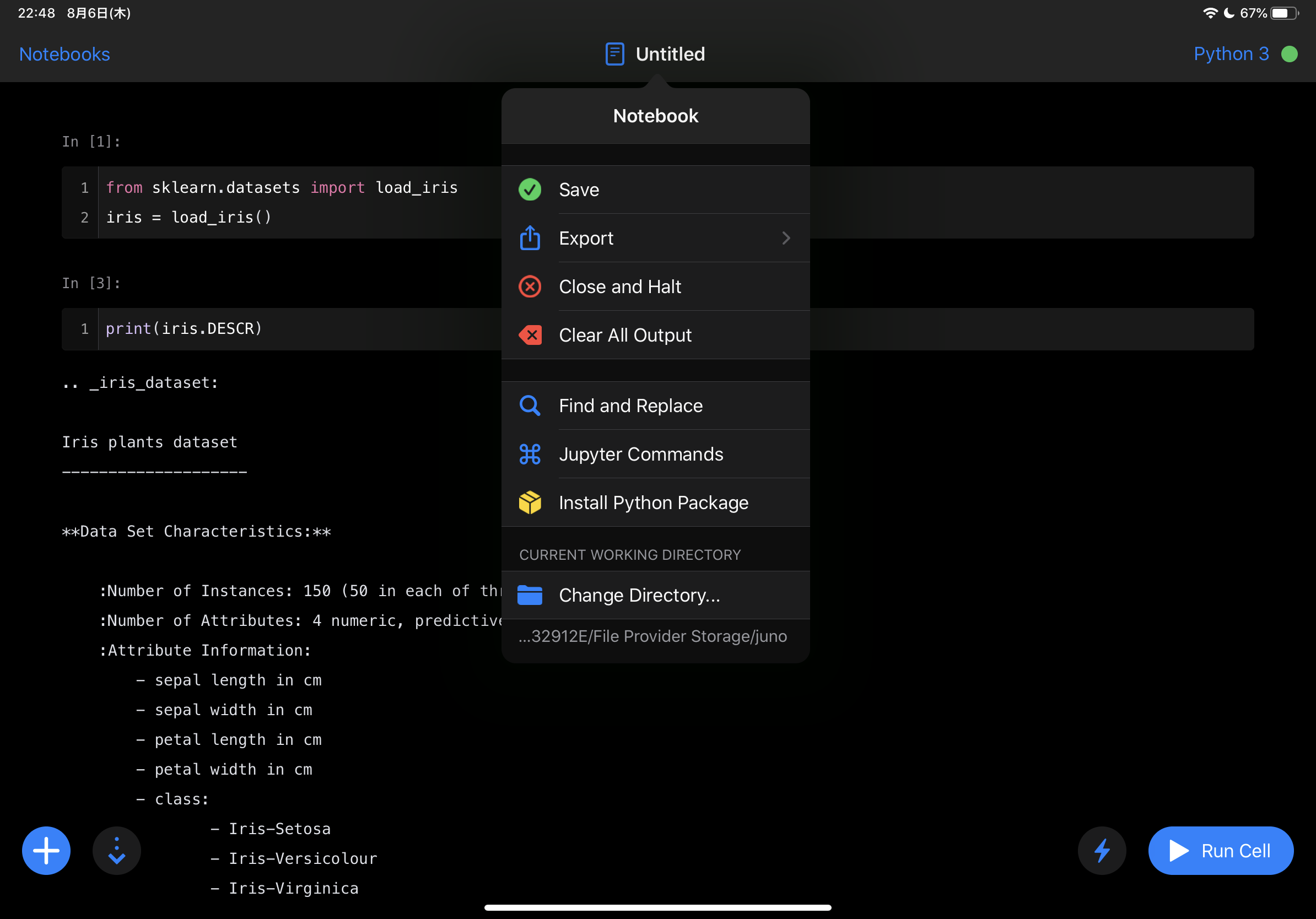
shellコマンドは実行できなくなった(?)ようです。
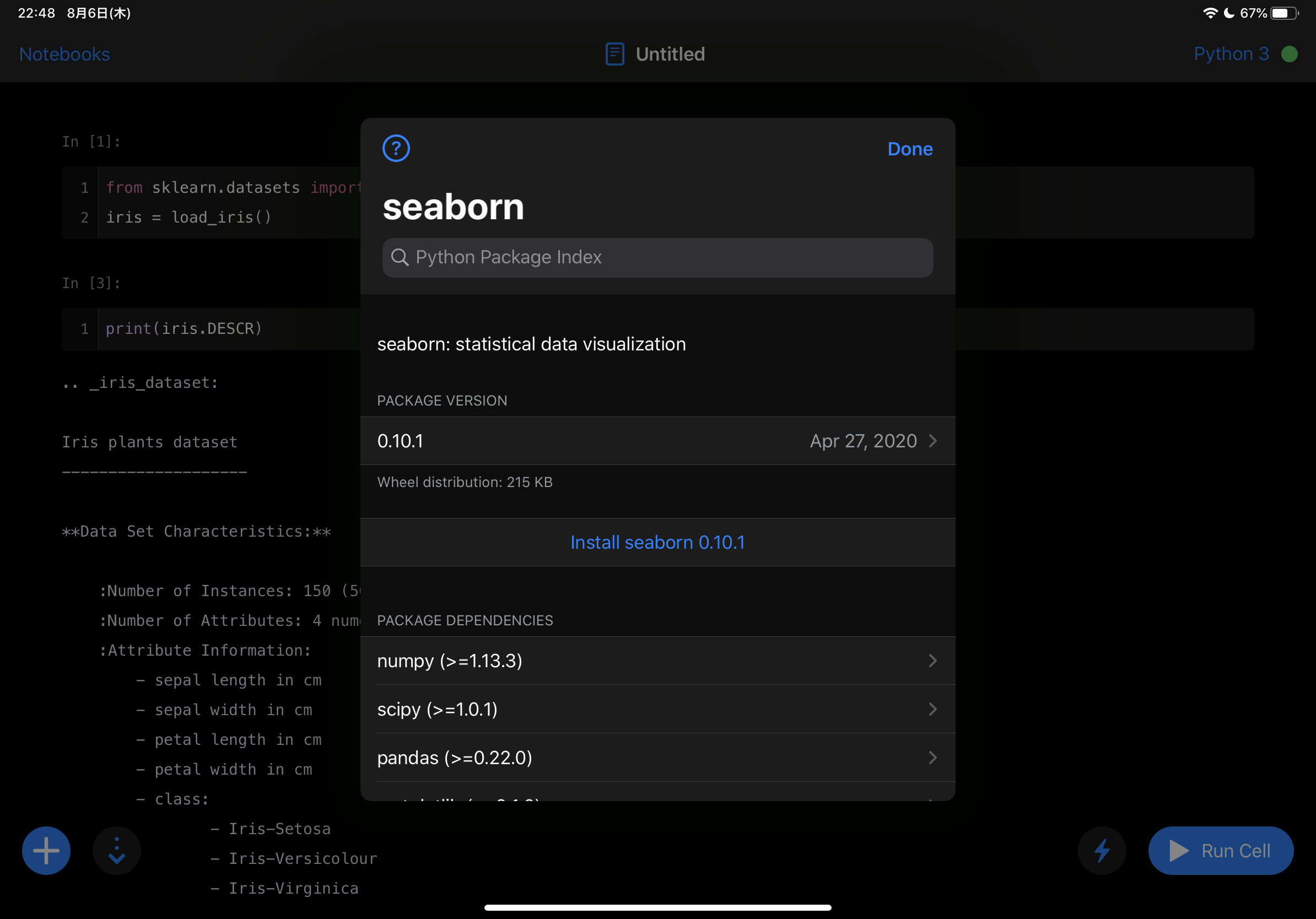
shellコマンドが使えない代わりに、ファイル名のところを押すとメニューが出てきて、Install Python Packageから不足しているパッケージのインストールが可能になっています。標準でもscikit-learnは入っていますが、seabornは標準にはありませんでした。
インストール成功し、pairplotが動作しました。
保存形式としては画像の通り、主要な物はありそう?
形式を選択すると、いつもの保存画面が表示され、AirDrop等でmac等にももちろん送ることができます。
以上が軽くJunoを触った感触です。
iPadなら持ち運びも楽なので、外で機械学習の勉強、みたいなケースで重宝するかもしれません。
- 投稿日:2020-08-11T18:50:06+09:00
引っ越すvs契約更新「今の家賃は妥当なのか?」意思決定を助ける定量データの作成
はじめに
賃貸派の方々は契約更新の時期が近づいてくる中で、こういう気持ちが生まれることはないでしょうか。
「今の家に不満はないが、更新の時にもう少し安くならないかな・・・」
「更新時に安くならなきゃ引越ししようかな」
「家賃相場より高く物件を借りているのではないか・・・?」「自分の払っている家賃が妥当かどうか確認したい」今回はここを定量的に把握する事を目標とします。
目標
分析目標を細かく分解していきます。
誰が:「賃貸契約者(住んでる人)が」
いつ:「引っ越すか、契約更新するか悩んでいるとき」
何を:「自身の払っている家賃が妥当なのか」
が分かれば
何ができる:「引っ越すか、契約更新するか、家賃交渉するかの意思決定の手助けができるようになる」このためには、あらゆる物件の家賃相場を把握する事がまず必要です。
データの準備
賃貸物件情報サービス「SUUMO」からデータをスクレイピングします。
収集時期:2020年6月
収集範囲:東京23区
データ総数:175,032件収集した特徴量は下記の通りです。(SUUMOより参照)
これらの特徴量をもとに、賃料を予測するモデルを作ります。
(賃料は、管理費も合算した金額になるよう調整しました。)特徴量の限定
自分の家賃の相場を把握するために「何階建てで」「〇〇線の△△駅から徒歩×分」など多くの情報を入力することは煩わしいですね・・・。
オッカムの剃刀の考え方に習い、少ない特徴量で「それなり」の精度の出るモデルの作成を目指します。
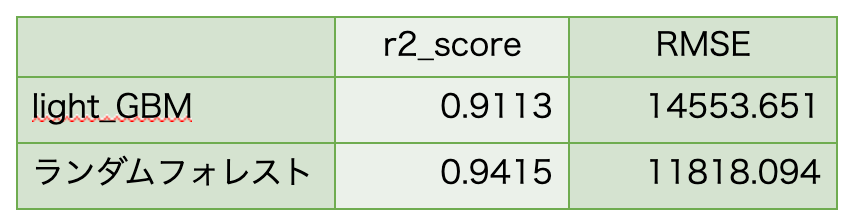
特徴量のimportanceを確認するために、ligtht_GBMを用いました。
精度もそれなりに高く、学習コストも低いので「どの特徴量を使おうかな〜」と何度も繰り返す作業にはとても重宝しました。最終的に使用する特徴量を5つに限定しました。
特徴量 例 区 新宿区 築年数 5年 物件の存する階 3階 専有面積 35㎡ 間取り 1LDK この特徴量をもとに、賃料の予想を行なっていきます。
予測精度は?
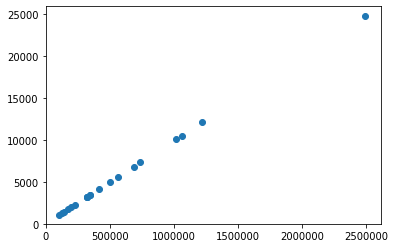
「そんな特徴量で大丈夫か?」と思いますが、
この5つの特徴量で賃料予測を行なったところ、下記のような結果となりました。
最寄駅情報によって賃料価格が大きく異なると仮定していたのですが、最寄駅よりもどの区に属しているか、が大きく関わっているようでした。ここは意外でした。
予測結果において、おおよそ±12000の誤差が確認できるとわかりました。
精度に優れたランダムフォレストを採用して、今後進めていきます。自分の家の賃料は?
特徴量をもとに、賃料を予測する学習モデルの作成は完了しました。
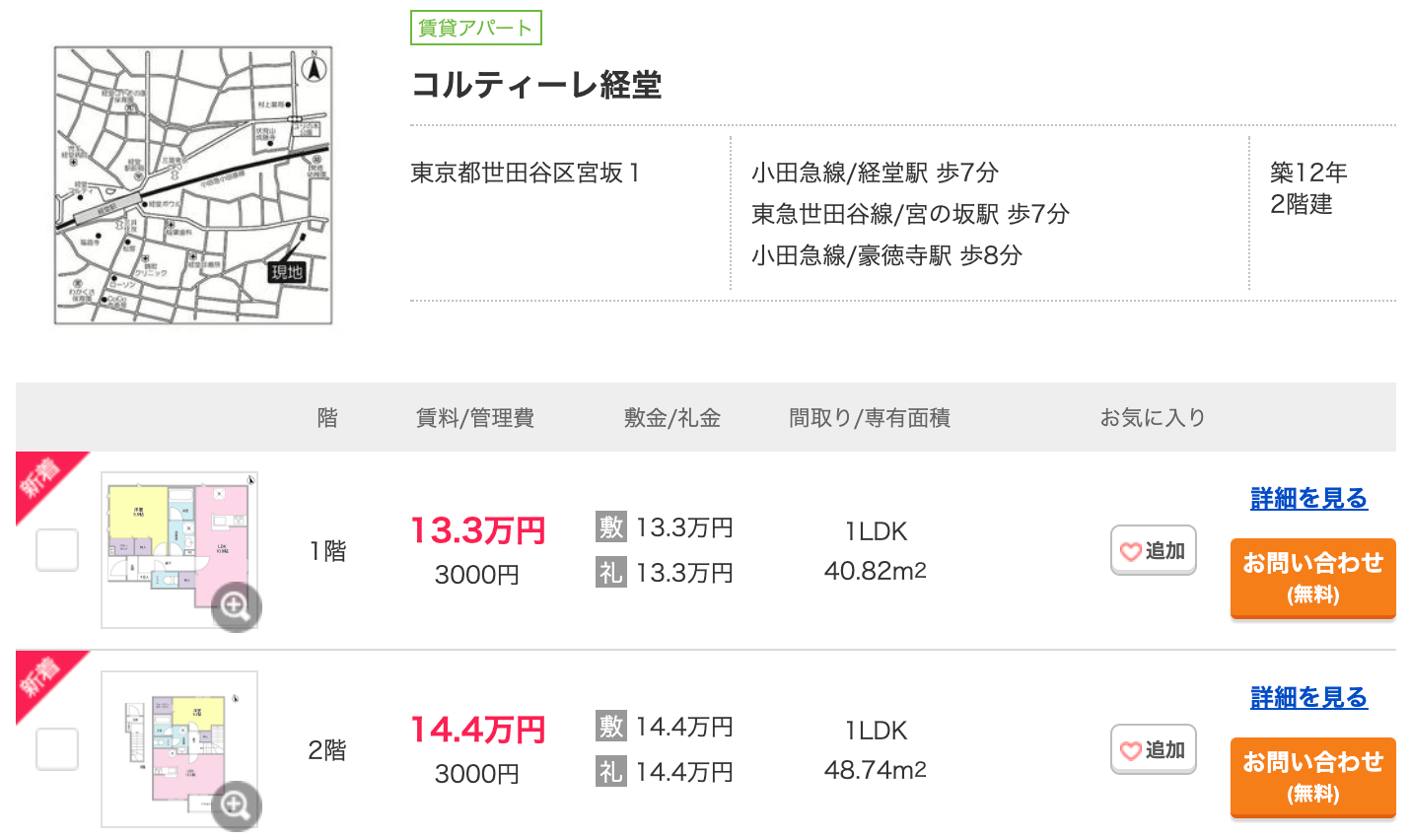
次に「今の家賃が妥当なのか」を確認するためには自宅の特徴量を新たに作成し、そこに学習モデルを食わせて予測金額(賃料)を叩き出す必要があります。サンプルとして世田谷の物件を1つ参照し、実際に住んでいる気持ちになります。(2階に住んでいるつもりで)
(SUUMOより参照)
物件の特徴を確認します。
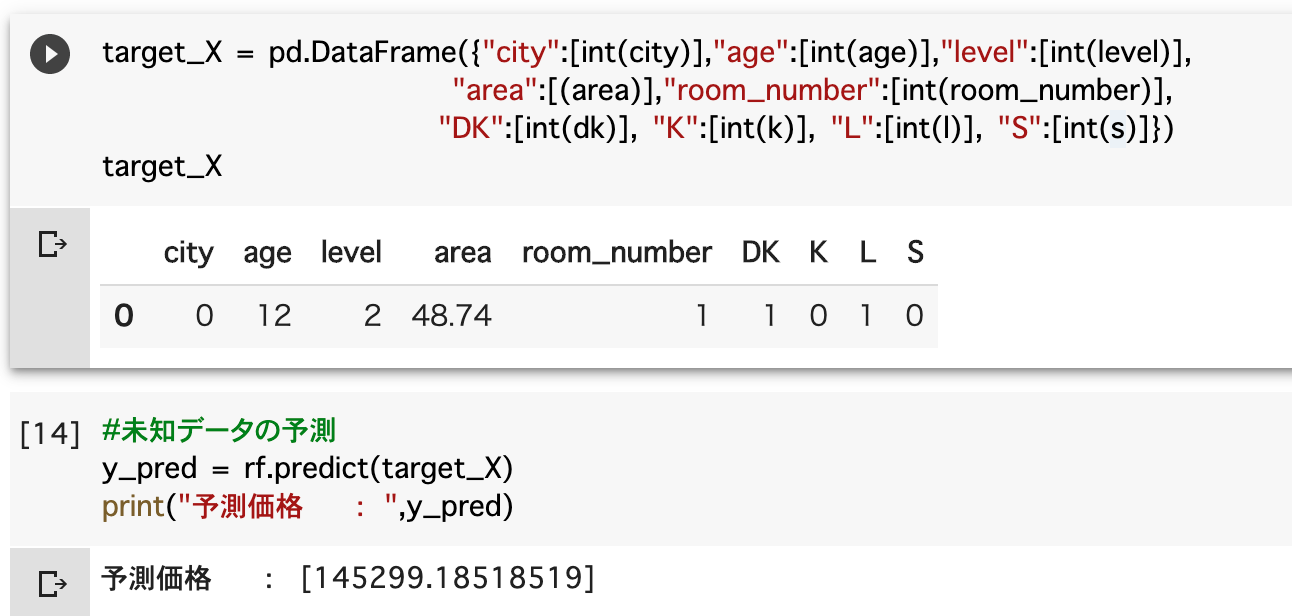
特徴量 区 世田谷区 築年数 12年 物件の存する階 2階 専有面積 48.74㎡ 間取り 1LDK 家賃(管理費込) ¥14,7000 今回はinput関数を用いて、データフレームの作成を行います。
データフレームを作成。学習データを用いて予測結果の算出。
予測結果を踏まえた考察
結果 実際の家賃(管理費込) ¥14,7000 予測結果の家賃(管理費込) ¥14,5229 実際の家賃に対して、予測結果が安い結果となりました。
引越しをしようか、契約更新をしようか、家賃交渉をしようか、
ここで家主がどう思うかは、その人それぞれですし、その他周辺環境などにもよるところがあると思います。(自分が実際に住んでいないので意見を控えときます笑)
しかしながら、意思決定においてそれなりに勘案できる情報となります。「管理費分だけ安くしてくれないか?」このくらいは交渉してみても良いかもしれませんね。
最後に
目標としていた
「引っ越すか、契約更新するか、家賃交渉するかの意思決定の手助けができるようになる」
この点には多少なりとも貢献するモデルになりました。
家賃としては数千円の違いでも、年間に換算すると何万と変わるので、さらに精度が高いモデルを作成できればより具体的な意思決定に貢献できそうですね。価格交渉の末、たとえ1000円でも家賃が安くなれば、支出が据え置きでネットフリックスとか契約できちゃいます。
侮れませんな・・・参考
今住んでいる物件の家賃を下げたい方は、下記YouTubeが参考になるとおもいますよ〜
https://www.youtube.com/watch?v=25DfwHHnKDgSUUMOのスクレイピングに関しては、下記記事がとても参考になりました。
https://qiita.com/haraso_1130/items/8ea9ba66f9d5f0fc2157
- 投稿日:2020-08-11T18:42:01+09:00
【Udemy Python3入門+応用】 62. 集合内包表記
※この記事はUdemyの
「現役シリコンバレーエンジニアが教えるPython3入門+応用+アメリカのシリコンバレー流コードスタイル」
の講座を受講した上での、自分用の授業ノートです。
講師の酒井潤さんから許可をいただいた上で公開しています。■集合内包表記
◆普通に記述した場合
s = set() for i in range(10): s.add(i) print(s)result{0, 1, 2, 3, 4, 5, 6, 7, 8, 9}◆集合内包表記で記述した場合
s = {i for i in range(10)} print(s)result{0, 1, 2, 3, 4, 5, 6, 7, 8, 9}if文を付け足すこともできる。
s = {i for i in range(10) if i % 2 == 0} print(s)result{0, 2, 4, 6, 8}
- 投稿日:2020-08-11T18:39:53+09:00
SQLAlchemyを利用して既存テーブルのメタデータを取得する
目的
SQLAlchemyを介して既存テーブルのカラム名やカラム型などのメタデータを取得する。
環境
動作環境
- Windows 10 Home
- Python 3.8.5 (Anaconda)
- Oracle Database 18c XE
必要なライブラリ
()内は検証で使用したバージョン。
- sqlalchemy (1.3.18)
- cx_oracle (7.2.3)
使用するテーブル
create table demo_tbl ( i_col integer, f_col float, c_col char(1), d_col date, PRIMARY KEY (i_col) )実装
準備
エンジンインスタンス作成
RDBに接続するための
sqlalchemy.engine.Engineインスタンスを作成する。host,port,pdb-name,user,passにはそれぞれ使用する接続情報に置き換えること。import sqlalchemy import cx_Oracle dsnStr = cx_Oracle.makedsn('host','port','pdb-name') connect_str = 'oracle://user:pass@' + dsnStr.replace('SID', 'SERVICE_NAME') engine = sqlalchemy.create_engine(connect_str, max_identifier_length=128)検査インスタンス作成
メタデータを取得するための
sqlalchemy.engine.reflection.Inspectorインスタンスを作成する。inspector = sqlalchemy.inspect(engine)検査インスタンスを用いてテーブルのメタデータにアクセスすることができる。
以下ではカラム一覧と主キーの取得方法を紹介する。それ以外の情報を取得する方法はAPIドキュメントを参照のこと。
カラム一覧
以下のように
get_columnsメソッドにテーブル名を渡すとカラム情報のリストが返却される。columns = inspector.get_columns("demo_tbl")[{'name': 'i_col', 'type': INTEGER(), 'nullable': False, 'default': None, 'autoincrement': 'auto', 'comment': None}, {'name': 'f_col', 'type': FLOAT(), 'nullable': True, 'default': None, 'autoincrement': 'auto', 'comment': None}, {'name': 'c_col', 'type': CHAR(length=1), 'nullable': True, 'default': None, 'autoincrement': 'auto', 'comment': None}, {'name': 'd_col', 'type': DATE(), 'nullable': True, 'default': None, 'autoincrement': 'auto', 'comment': None}]
typeキーで格納されている値はsqlalchemy.types.TypeEngineをベースとしたSQLAlchemyで定義された一般型のインスタンス、またはDB個別に実装されたサブクラスのインスタンスになっている。詳細はAPIドキュメントを参照のこと。# i_col(integer)の場合 type_of_col0 = columns[0]["type"] type(type_of_col0) # -> <class 'sqlalchemy.sql.sqltypes.INTEGER'> isinstance(type_of_col0, sqlalchemy.types.TypeEngine) # -> True isinstance(type_of_col0, sqlalchemy.types.Integer) # -> True isinstance(type_of_col0, sqlalchemy.types.Float) # -> False # d_col(date)の場合 type_of_col3 = columns[3]["type"] type(type_of_col3) # -> <class 'sqlalchemy.dialects.oracle.base.DATE'> isinstance(type_of_col3, sqlalchemy.types.TypeEngine) # -> True isinstance(type_of_col3, sqlalchemy.types.DateTime) # -> True isinstance(type_of_col3, sqlalchemy.types.Date) # -> False※Oracleの
date型は時刻まで格納可能なのでSQLAlchemyのクラスとしてはDateTimeとなる。主キー
以下のように
get_pk_constraintメソッドにテーブル名を渡すと主キー関連の情報がが返却される。{'constrained_columns': ['i_col'], 'name': 'sys_c007315'}※
get_primary_keysというメソッドもあるがこれは廃止予定としてdeprecatedになっている。参考
- 投稿日:2020-08-11T17:57:13+09:00
【python】dataclassでお手軽引数型チェック
はじめに
- DDDでコード書く練習していたときに、ValueObjectの型チェック方法を試行錯誤したのでその時のメモ
- Qiitaの記事練習として(初投稿)。
結論
__post_init__()で初期化時の処理がかけるので、ここで型チェックを実行- isinstanceで型チェック
self.__annotations__から期待値の型を取得するdataclasses.asdict(self)でインスタンスをdictに変換。これをisinstanceにかける。dataclassとは?
- init()を自動生成してくれる。
__init__()に引数を入れて、self.hoge=arg_hogeとかする必要ない。- ValueObjectを生成するのに適している。
普通の書き方
class Users: def __init__(self, user_name: str, user_id: int): self.user_name = user_name self.user_id = user_iddataclassでの書き方
class User: user_name: str user_id: intdataclassで書いたほうがきれいに書けますね!
dataclassでは型チェックしてくれない?
今回の本題です。
user_name: strやuser_id: intで型指定していて、型チェックしているように見えますが、実際は普通のアノテーションです。str型で指定しているのに、int型で入れられてしまいます。
コード
import dataclasses @dataclasses.dataclass(frozen=True) class User: user_name: str user_id: int c = User(user_name='ヒノヤコマ', user_id=1) print(f'{c.user_id}:{c.user_name}') c_fail = User(user_name=2, user_id='ファイアロー') print(f'{c_fail.user_id}:{c_fail.user_name}')実行結果
> python .\dataclassテスト.py 1:ヒノヤコマ ファイアロー:2上記のようになんでも入れられてしまいます。
__post_init__()で初期化時に型チェックを実行下記のようにdataclass内で、
__post_init__()関数を定義することで、初期化時の処理を書くことができます。
ここにisinstanceを使用して型チェックしましょう@dataclasses.dataclass(frozen=True) class User: user_name: str user_id: int def __post_init__(self): if not isinstance(self.user_name, str): raise Exception if not isinstance(self.user_id, int): raise Exception実行結果
> python .\dataclassテスト.py 1:ヒノヤコマ Traceback (most recent call last): File ".\dataclassテスト.py", line 17, in <module> c_fail = User(user_name=2, user_id='ファイアロー') File "<string>", line 4, in __init__ File ".\dataclassテスト.py", line 10, in __post_init__ raise Exception Exception期待通り、例外を出すことができました!
変数が多いと型チェックが大変
いままでの例では、
user_nameとuser_idの2つでしたが、たくさんあると大変です。
そのため、__post_init__で書いた型チェックを変数の数だけますようにしたいですね。というわけで書いたのが下記になります。
@dataclasses.dataclass(frozen=True) class User: user_name: str user_id: int def __post_init__(self): # 1. asdictでUserインスタンスをdict型に変換 user_dict = dataclasses.asdict(self) # 2. self.__annotations__から期待値の型を取得 # self.__annotations__には引数の名前とその指定する型がdictで入ってます。 # これから期待値の型を取得して、isinstanceで型チェックします。 for user_arg_name, user_arg_expected_type in self.__annotations__.items(): # 3. isinstance実行 # dict型に変換したUserから、アノテーションのKeyで指定して対象の変数を取り出して実行します。 if not isinstance(user_dict[user_arg_name], user_arg_expected_type): print(f'{user_arg_name} is not ok') raise Exception else: print(f'{user_arg_name} is ok')細かいことはコメントで入れました。
asdictでインスタンスが持つすべての引数(変数)を取得し、self.__annotations__で期待値の型を取得して、isinstanceをかけます。
コメントアウトや実行結果
> python .\dataclassテスト.py user_name is ok user_id is ok 1:ヒノヤコマ user_name is not ok Traceback (most recent call last): File ".\dataclassテスト.py", line 21, in <module> c_fail = User(user_name=2, user_id='ファイアロー') File "<string>", line 4, in __init__ File ".\dataclassテスト.py", line 13, in __post_init__ raise Exception Exceptionこのコードの弱点
typingなどで型を指定すると、この方法だとうまくいきません。
下記はコメントを抜いて、List[int]で引数の型を指定したもの。import dataclasses from typing import List @dataclasses.dataclass(frozen=True) class User: user_name: str user_id: int status_list: List[int] def __post_init__(self): user_dict = dataclasses.asdict(self) for user_arg_name, user_arg_expected_type in self.__annotations__.items(): if not isinstance(user_dict[user_arg_name], user_arg_expected_type): print(f'{user_arg_name} is not ok') raise Exception else: print(f'{user_arg_name} is ok') status_list=[50,51] c = User(user_name='ヒノヤコマ', user_id=1, status_list=status_list) print(f'{c.user_id}:{c.user_name}') c_fail = User(user_name=2, user_id='ファイアロー', status_list=status_list) print(f'{c_fail.user_id}:{c_fail.user_name}')以下のようにlistのところでエラーが出ています。
> python .\dataclassテスト.py user_name is ok user_id is ok Traceback (most recent call last): File ".\dataclassテスト.py", line 27, in <module> c = User(user_name='ヒノヤコマ', user_id=1, status_list=status_list) File "<string>", line 5, in __init__ File ".\dataclassテスト.py", line 19, in __post_init__ if not isinstance(user_dict[user_arg_name], user_arg_expected_type): File "C:\Users\proje\AppData\Local\Programs\Python\Python37\lib\typing.py", line 708, in __instancecheck__ return self.__subclasscheck__(type(obj)) File "C:\Users\proje\AppData\Local\Programs\Python\Python37\lib\typing.py", line 716, in __subclasscheck__ raise TypeError("Subscripted generics cannot be used with" TypeError: Subscripted generics cannot be used with class and instance checksなぜだめなのか
以下を実行するとわかりますが、List[int]は型を指定するための型なので、実際のlist型ではありません。
>>> from typing import List >>> print(type(List[dir])) <class 'typing._GenericAlias'>型チェック時に変換する必要がありそうですね。
おわりに
__post_init__を使用して型チェックするのは問題なさそうです。
ですが、型を指定する際にtypingを使用していると、for文で回してチェックするのはもう一工夫必要そうです。
mypyなどでコード内にいれるのではなく、外から確認するのもありかもしれません。
(push時に自動確認するなど、環境がしっかりしていればこちらもありですね)参考
- Python Documentation contents dataclasses --- データクラス
- 投稿日:2020-08-11T17:52:05+09:00
連番なファイルをループで読み込んで処理し、グラフ化する方法
file_1.dat,file_2.dat ...... file_100.datのような数字だけが連続で変化するdatファイルをfor文を用いて読み込んで処理し、グラフ化する方法です。
まとまった説明が見つからなかったので、備忘録として残します。何がしたいか
こんな感じのn行2列のdatファイルが数十個あり、2列目の値のみを取り出して計算し、グラフ化したいと思っています。
仮にExcelに1ファイルずつコピペして貼り付けて~となると時間も労力もかなりかかってしまいます。当初はファイル一つ一つを読み込み、処理して、x,yの値に代入して…といったプログラムだった、つまり
data_number_1,data_intensity_1 = np.loadtxt("file_1.dat",unpack = True) s1 = su(data_intensity_1) m1 = mean(data_intensity_1) x=[s1] y=[m1]これをファイルの個数分繰り返すというとんでもなく行数も分量も多いプログラムだったのですが、冒頭で述べたようにファイル名は数字の箇所だけが異なるため、Pythonでfor文を用いることで簡単に処理できるのではないか?と考えたことがプログラムを考えた動機です。
<用いた環境>
spyder(python3.7)
計算:numpy (import numpy as np)
描画:matplotrib (import matplotlib.pyplot as plt)datファイルの読み込みとfor文
datファイルの読み込みにはnp.loadtextを用います。
参考:https://deepage.net/features/numpy-loadsavetxt.html読み込みに用いたコードのサンプルは以下の通りです。
ここでは、file_1からfile_20までの連番ファイルを読み込むとします。data = [] for i in range(1,21): data.append(np.loadtxt("file_{}.dat".format(i),usecols=(1),unpack=True))以下、順を追ってコードを説明します。
for文について
for文のiの範囲を設定するとき、最大値-1の範囲が適用されるため、今回はファイルの番号である20+1として(1,21)と範囲を設定します。
ファイル名は、file_数字.datなので、formatメゾットを使用し、file_{}.dat".format(i)としてやります。
こうすることで、file_1.dat,file_2.dat......といったループ処理ができます。そしてこれらを作成したdataというリストにdata.appendで繰り返される処理の値をリストにどんどん追加していく、といった具合です。
loadtextについて
datファイルのうち、2行目の値だけを数値として扱いたいので、そのための処理を書き加えてやる必要があります。
そのままloadtextを用いて数値を読み込むと、リストには先の画像の例で言うと[0.0 72925],[1.0 70740]...[10.0 73343]といったように行ごとに入れられてしまいます。
そこでunpackを用いて転置してやり、列ごとに別の変数に入れてやります。
Trueにしてやることで転置が起こり、リストには[0.0 1.0 ... 10.0],[72925 70740 ... 73343]と列ごとに異なる変数にすることが可能です。また、1列目は使わず、2列目のみを数値として扱いたいのでusecolsによって読み取る行を指定します。
0を最初の行として読み込む行を決めているので、2列目を読み込む際には(1)としてやる必要があります。
そうすることでdataリストには[72925 70740 ... 73343]のみが入ることになります。詳しくは上記に掲載した参考サイトをご覧ください。
このようにunpackとusecolsを用いることで、各ファイルごとに2列目だけをある変数に入れてやることができました。data = [file_1の2列目,file_2の2列目,...,file_20の2列目] といった感じです。
当初のプログラムではファイル分行を打っていたのがなんとたったあれだけで済みました!行数も文字量も驚きの減少です。
もちろんファイルの量が増えれば増えるほど楽になります。処理
こうして2行目の値だけを全ファイル文取り込むことができたので、次はそれぞれについて処理をしていきます。
リストに入った値を一括処理するため、関数を定義してやり、順に処理させます。今回は横軸を各ファイルごとの値を合計した値、縦軸はその平均値としてグラフを書くとします。
関数の定義はそれぞれ次のように書くことができます。
def su(a): return np.sum(a)def mean(a): return np.mean(a)これらの処理をリストdataの個々に適用してやります。
for i in range(1,21): si = su(data[i-1]) mi = mean(data[i-1])ここで注意しなければならないのは範囲の値です。
ファイル名は1から始まっているのですが、リストの値は0から始まります。
よって関数でiの値を指定するときに(i-1)としてやる必要があります。1~20だと思っている値を0~19の範囲にしてやる必要があるということです。
グラフの作成
今回は細かい説明については省きます。matplotribで検索すれば色々説明がでてくるかと思います。
先の処理方法で計算した値をそれぞれx,yとしてやる必要があります。
そのため、ファイルを読み込む時と同様にリストを作り、処理した値を順にx,yリストに入れるようにします。x=[] y=[] for i in range(1,21): si = sum(data[i-1]) mi = mean(data[i-1]) x.append(si) y.append(mi)こうすることでx軸y軸の値を用意することができたので、後はplotを用いてグラフにすれば完成です。
全体のコードサンプル
以上を踏まえた全体のコードは以下のようになります。(値、ファイル、グラフはサンプルとして適当に用意したものであり、何の意味も持ちません)
import numpy as np import matplotlib.pyplot as plt def su(a): return np.sum(a) def mean(a): return np.mean(a) data = [] for i in range(1,21): data.append(np.loadtxt("file_{}.dat".format(i),usecols=(1),unpack=True)) x=[] y=[] for i in range(1,21): si = su(data[i-1]) mi = mean(data[i-1]) x.append(si) y.append(mi) plt.plot(x,y,ls="",marker="o"); plt.xlim(xmin=0) plt.ylim(ymin=0) plt.show()このようにループ処理を用いることで100行以上の簡略化に成功し、効率は格段にあがりました!
同じような問題で悩んでいる方の参考になれば幸いです。閲覧ありがとうございました。
- 投稿日:2020-08-11T17:03:16+09:00
python 関数でのグローバル変数の使用
関数でグローバル変数を作成または使用するにはどうすればよいですか?
ある関数でグローバル変数を作成した場合、そのグローバル変数を別の関数でどのように使用できますか?アクセスを必要とする関数のローカル変数にグローバル変数を格納する必要がありますか?
グローバル変数は、それに割り当てる各関数でglobalとして宣言することにより、他の関数で使用できます。
globvar = 0 def set_globvar_to_one(): global globvar # Needed to modify global copy of globvar globvar = 1 def print_globvar(): print(globvar) # No need for global declaration to read value of globvar set_globvar_to_one() print_globvar() # Prints 1
- 投稿日:2020-08-11T16:58:39+09:00
Google ColaboratoryのTPUでKerasを動かす
準備
%tensorflow_version 2.x import tensorflow as tf print("Tensorflow version " + tf.__version__) try: tpu = tf.distribute.cluster_resolver.TPUClusterResolver() # TPU detection print('Running on TPU ', tpu.cluster_spec().as_dict()['worker']) except ValueError: raise BaseException('ERROR: Not connected to a TPU runtime; please see the previous cell in this notebook for instructions!') tf.config.experimental_connect_to_cluster(tpu) tf.tpu.experimental.initialize_tpu_system(tpu) tpu_strategy = tf.distribute.TPUStrategy(tpu)def create_model(): #model定義 return modelwith tpu_strategy.scope(): model = create_model()学習部分
普通に学習
history = model.fit( x_train, y_train, epochs = 50, batch_size = 2048, validation_data=(x_test, y_test))
- 投稿日:2020-08-11T16:58:39+09:00
Google Colaboratory上でKeras + TPU
準備
%tensorflow_version 2.x import tensorflow as tf print("Tensorflow version " + tf.__version__) try: tpu = tf.distribute.cluster_resolver.TPUClusterResolver() # TPU detection print('Running on TPU ', tpu.cluster_spec().as_dict()['worker']) except ValueError: raise BaseException('ERROR: Not connected to a TPU runtime; please see the previous cell in this notebook for instructions!') tf.config.experimental_connect_to_cluster(tpu) tf.tpu.experimental.initialize_tpu_system(tpu) tpu_strategy = tf.distribute.TPUStrategy(tpu)def create_model(): #model定義 return modelwith tpu_strategy.scope(): model = create_model()学習部分
普通に学習
history = model.fit( x_train, y_train, epochs = 50, batch_size = 2048, validation_data=(x_test, y_test))
- 投稿日:2020-08-11T15:33:21+09:00
Docker Desktop for Windowsを使って、最新のodooを2コマンドで起動させる
docker/odoo
odooをご存じでしょうか。odooは、pythonで実装されたオープンソースのERPとして、有名ですが、実は、業務アプリケーションのノンコーディングツール(Odoo Studio)でもあります。
Odoo Studio: Build an App from Scratch with Zero Coding Experience 2019/11/13これまで、odooを試すのに、Linuxのインストールから始まり、python環境の構築、データベースとなるpostgreSQLの設定やWebサーバーの設定など、いくつかの手順が必要でした。それが、Dockerの登場により、2コマンドで、odooをインストールし、起動することができるようになりました。もちろん、odooもDocker公式イメージとして、登録されています。
準備 - Dockerのインストール
ここでは、Windows10で、Docker Desktop for Windowsを使って、最新のodooを2コマンドで起動させます。
まずは、Docker Desktop for Windowsをインストールします。
https://docs.docker.jp/docker-for-windows/toc.htmldockerでodooを起動
odooは、Docker Hubに公式イメージとして登録されていますので、2つのコマンドで起動が可能です。
コマンド1 - データベースの起動
まずは、データベースであるPostgreSQLのコンテナをバックグラウンドで実行します。
初回の起動時に、必要なモジュールのインストールが自動的に行われます。
# 項目 内容 1 コマンド docker run 2 コンテナをバックグラウンドで実行 する (-d) 3 POSTGRES_USER (-e) odoo 4 POSTGRES_PASSWORD (-e) odoo 5 POSTGRES_DB (-e) postgres 6 コンテナの名前(--name) db 7 Dockerイメージ postgres:10 doccker.db.cmddocker run -d -e POSTGRES_USER=odoo -e POSTGRES_PASSWORD=odoo -e POSTGRES_DB=postgres --name db postgres:10コマンド2 - odooの起動
先に起動したPostgreSQLのコンテナにリンクするようにして、odooのコンテナを起動します。
# 項目 内容 1 コマンド docker run 3 ホスト側に公開するコンテナのポート (-p) 8069:8069 4 コンテナの名前 (--name) odoo 5 他のコンテナへのリンク (--link) db:db 6 疑似ターミナルを割り当て する (-t) 7 Dockerイメージ odoo docker.odoo.cmddocker run -p 8069:8069 --name odoo --link db:db -t odoo結果
コマンドプロンプト.cmdMicrosoft Windows [Version 10.0.18363.959] (c) 2019 Microsoft Corporation. All rights reserved. C:\Users\dev>docker run -d -e POSTGRES_USER=odoo -e POSTGRES_PASSWORD=odoo -e POSTGRES_DB=postgres --name db postgres:10 Unable to find image 'postgres:10' locally 10: Pulling from library/postgres 75cb2ebf3b3c: Pull complete 3ca6415d2bca: Pull complete ac08e6372a7b: Pull complete b4394fce95ce: Pull complete 6edcd5da08e3: Pull complete 3380dcb7db08: Pull complete c7c147d9c90d: Pull complete 08ae47fef758: Pull complete 3d807fd80688: Pull complete 3f37a1ee389c: Pull complete cea1ff8f661e: Pull complete 064c69d8ca75: Pull complete 96c4055cb7f7: Pull complete 4477915a4c1f: Pull complete Digest: sha256:e3a02efdce3ec64cfdb76a8ff93ae14d3294e47a0203d8230c8853a3890fe340 Status: Downloaded newer image for postgres:10 05ae7b08b1800ef560a3b6dca95c4071163b3d1582ffc224d87f2e77a29ab9a9 C:\Users\dev>docker run -p 8069:8069 --name odoo --link db:db -t odoo Unable to find image 'odoo:latest' locally latest: Pulling from library/odoo bf5952930446: Already exists fa677b64d95a: Pull complete 2dd4309767ba: Pull complete 6ffbc48d26ec: Pull complete d6f4cbbe7cdf: Pull complete 338ad56bea8c: Pull complete d6c8b0326958: Pull complete 6fd5f5f6c87c: Pull complete 24ccac123804: Pull complete Digest: sha256:a0a1f89921d66751b8f94042f4bd291d4761858ef4f4262a0b27de8cf70b74d6 Status: Downloaded newer image for odoo:latest 2020-08-11 03:43:19,685 1 INFO ? odoo: Odoo version 13.0-20200629 2020-08-11 03:43:19,685 1 INFO ? odoo: Using configuration file at /etc/odoo/odoo.conf 2020-08-11 03:43:19,686 1 INFO ? odoo: addons paths: ['/usr/lib/python3/dist-packages/odoo/addons', '/var/lib/odoo/addons/13.0', '/mnt/extra-addons'] 2020-08-11 03:43:19,687 1 INFO ? odoo: database: odoo@172.17.0.2:5432 2020-08-11 03:43:19,935 1 INFO ? odoo.addons.base.models.ir_actions_report: Will use the Wkhtmltopdf binary at /usr/local/bin/wkhtmltopdf 2020-08-11 03:43:20,096 1 INFO ? odoo.service.server: HTTP service (werkzeug) running on 3dd0bcf7ca1d:8069ログイン(初回)
次のURLで、odooにログインします。
コマンドプロンプトには、疑似ターミナルとして、次のように標準出力されます。
コマンドプロンプト.cmd2020-08-11 06:12:57,654 1 INFO ? odoo.http: HTTP Configuring static files 2020-08-11 06:12:57,685 1 INFO ? odoo.http: Generating nondb routing 2020-08-11 06:12:57,709 1 INFO ? werkzeug: 172.17.0.1 - - [11/Aug/2020 06:12:57] "GET / HTTP/1.1" 303 - 1 0.013 0.036 2020-08-11 06:12:57,732 1 INFO ? werkzeug: 172.17.0.1 - - [11/Aug/2020 06:12:57] "GET /web HTTP/1.1" 303 - 2 0.005 0.010 2020-08-11 06:12:57,853 1 INFO ? werkzeug: 172.17.0.1 - - [11/Aug/2020 06:12:57] "GET /web/database/selector HTTP/1.1" 200 - 2 0.004 0.098 2020-08-11 06:12:57,895 1 INFO ? werkzeug: 172.17.0.1 - - [11/Aug/2020 06:12:57] "GET /web/static/lib/fontawesome/css/font-awesome.css HTTP/1.1" 200 - - - - 2020-08-11 06:12:57,896 1 INFO ? werkzeug: 172.17.0.1 - - [11/Aug/2020 06:12:57] "GET /web/static/lib/bootstrap/css/bootstrap.css HTTP/1.1" 200 - - - - 2020-08-11 06:12:57,904 1 INFO ? werkzeug: 172.17.0.1 - - [11/Aug/2020 06:12:57] "GET /web/static/lib/jquery/jquery.js HTTP/1.1" 200 - - - - 2020-08-11 06:12:57,964 1 INFO ? werkzeug: 172.17.0.1 - - [11/Aug/2020 06:12:57] "GET /web/static/lib/popper/popper.js HTTP/1.1" 200 - - - - 2020-08-11 06:12:57,982 1 INFO ? werkzeug: 172.17.0.1 - - [11/Aug/2020 06:12:57] "GET /web/static/lib/bootstrap/js/index.js HTTP/1.1" 200 - - - - 2020-08-11 06:12:58,005 1 INFO ? werkzeug: 172.17.0.1 - - [11/Aug/2020 06:12:58] "GET /web/static/lib/bootstrap/js/util.js HTTP/1.1" 200 - - - - 2020-08-11 06:12:58,033 1 INFO ? werkzeug: 172.17.0.1 - - [11/Aug/2020 06:12:58] "GET /web/static/lib/bootstrap/js/alert.js HTTP/1.1" 200 - - - - 2020-08-11 06:12:58,036 1 INFO ? werkzeug: 172.17.0.1 - - [11/Aug/2020 06:12:58] "GET /web/static/lib/bootstrap/js/button.js HTTP/1.1" 200 - - - - 2020-08-11 06:12:58,053 1 INFO ? werkzeug: 172.17.0.1 - - [11/Aug/2020 06:12:58] "GET /web/static/lib/bootstrap/js/carousel.js HTTP/1.1" 200 - - - - 2020-08-11 06:12:58,063 1 INFO ? werkzeug: 172.17.0.1 - - [11/Aug/2020 06:12:58] "GET /web/static/lib/bootstrap/js/collapse.js HTTP/1.1" 200 - - - - 2020-08-11 06:12:58,091 1 INFO ? werkzeug: 172.17.0.1 - - [11/Aug/2020 06:12:58] "GET /web/static/lib/bootstrap/js/dropdown.js HTTP/1.1" 200 - - - - 2020-08-11 06:12:58,094 1 INFO ? werkzeug: 172.17.0.1 - - [11/Aug/2020 06:12:58] "GET /web/static/lib/bootstrap/js/tooltip.js HTTP/1.1" 200 - - - - 2020-08-11 06:12:58,095 1 INFO ? werkzeug: 172.17.0.1 - - [11/Aug/2020 06:12:58] "GET /web/static/lib/bootstrap/js/modal.js HTTP/1.1" 200 - - - - 2020-08-11 06:12:58,112 1 INFO ? werkzeug: 172.17.0.1 - - [11/Aug/2020 06:12:58] "GET /web/static/lib/bootstrap/js/popover.js HTTP/1.1" 200 - - - - 2020-08-11 06:12:58,127 1 INFO ? werkzeug: 172.17.0.1 - - [11/Aug/2020 06:12:58] "GET /web/static/lib/bootstrap/js/scrollspy.js HTTP/1.1" 200 - - - - 2020-08-11 06:12:58,135 1 INFO ? werkzeug: 172.17.0.1 - - [11/Aug/2020 06:12:58] "GET /web/static/lib/bootstrap/js/tab.js HTTP/1.1" 200 - - - - 2020-08-11 06:12:58,152 1 INFO ? werkzeug: 172.17.0.1 - - [11/Aug/2020 06:12:58] "GET /web/static/src/img/logo2.png HTTP/1.1" 200 - - - - 2020-08-11 06:12:58,360 1 INFO ? werkzeug: 172.17.0.1 - - [11/Aug/2020 06:12:58] "GET /web/static/lib/fontawesome/fonts/fontawesome-webfont.woff2?v=4.7.0 HTTP/1.1" 200 - - - - 2020-08-11 06:12:58,399 1 INFO ? werkzeug: 172.17.0.1 - - [11/Aug/2020 06:12:58] "GET /web/static/src/img/favicon.ico HTTP/1.1" 200 - - - -おわりに
Windows 10 上で、Docker Desktop for Windows をインストールし、2つのコマンドで、最新のodooを起動できることを確認しました。
* 準備 - Docker Desktop for Windows のインストール
* コマンド1 - PostgreSQLのコンテナの起動
* コマンド2 - odooのコンテナの起動odoo は、pythonで実装されたオープンソースのERPでもありますが、Odoo Studioを使って、ノンコーディングの高速開発が可能です。
- 投稿日:2020-08-11T15:26:18+09:00
遺伝的アルゴリズムで関数の最適値を求める(その1)
進化アルゴリズムとは
進化的アルゴリズム(evolutionary algorithm, EA)とは、最適化アルゴリズムの一つで、自然界の淘汰、遺伝子組み換え、突然変異などをモデルとして組み込んだ最適化手法である。
進化的アルゴリズムのは下記の4種類に大分される。
- 遺伝的アルゴリズム
- 遺伝的プログラミング
- 進化戦略
- 進化的プログラミング今回は遺伝的アルゴリズム(genetic algorithm, GA)を扱う。
使用したライブラリ
下記のDEAPというpythonライブラリを用いる。
https://deap.readthedocs.io/en/master/コード
目的関数
下記に示すBird Bunctionの最小値を探索する。
$$f(x, y) = sin(x)e^{(1-cos(y))^2}+cos(y)e^{(1-sin(x))^2}+(x-y)^2$$
Bird Functionは多峰性の関数で、(x,y)=(4.70104, 3.15294),(-1.58214, -3.13024)に大域的最適解-106.764537、その周辺に多数の局所解を持つ。sample_GA.pyimport numpy as np def bird(x): x1 = x[0] x2 = x[1] t = np.sin(x1)*np.exp((1-np.cos(x2))**2) + np.cos(x2)*np.exp((1-np.sin(x1))**2) + (x1-x2)**2 return t, # 描画 import matplotlib.pyplot as plt from mpl_toolkits.mplot3d import Axes3D c1 = c2 = np.arange(-10, 10, 0.1) xx, yy = np.meshgrid(c1, c2) len=np.size(c1) z=np.empty((len, len)) for i in range(len): for j in range(len): xin=np.array([xx[i,j], yy[i,j]]) z[i,j], = bird(xin) fig0 = plt.figure(figsize=(12,12), dpi=80).add_subplot(111, projection='3d') fig0.plot_surface(xx, yy, z, alpha=1) plt.xlim(10, -10) plt.ylim(-10, 10)ライブラリのインポート
必要なライブラリをインポートしておく。
DEAPはrandomライブラリを使用するため、1試行ごとに結果が変化する。
これを避けるためには、random.seed(1)とすること。sample_GA.pyimport pandas as pd import random from deap import algorithms from deap import base,creator,tools # random.seed(1)初期個体群の生成
初期個体を生成する関数を作っておく。
ここでは、x, yともに-10〜10の範囲で、10×10の格子状に初期個体を生成した。sample_GA.pydef initPopulation(pcls, ind_init): x = np.linspace(-10, 10, 10) y = np.linspace(-10, 10, 10) inix, iniy = np.meshgrid(x,y) contents = np.concatenate([inix.reshape([-1,1]), iniy.reshape([-1,1])], axis=1) pcls = [ind_init(c) for c in contents] return pclstoolboxに登録
評価("evaluate")、個体群("population")、交叉("mate")、突然変異("mutate")、選択("select")等の手法をtoolboxに登録しておく。
例えば、下記の
toolbox.register("mate", tools.cxBlend, alpha=0.2)は、
tool.cxblendという関数を交叉手法として登録します、という意味(alpha=0.2はtools.cxBlendの引数)。交叉方法にブレンド交叉、突然変異にガウス変異、選択形式は10個体のトーナメントで実施する。
初期個体群を指定したい場合、"population_guess"に登録しておく。sample_GA.pycreator.create("FitnessMax", base.Fitness, weights=(-1.0,)) creator.create("Individual", np.ndarray, fitness=creator.FitnessMax) toolbox = base.Toolbox() toolbox.register("evaluate", bird) toolbox.register("population_guess", initPopulation, list, creator.Individual) toolbox.register("mate", tools.cxBlend, alpha=0.2) toolbox.register("mutate", tools.mutGaussian, mu=[0.0, 0.0], sigma=[2.0, 2.0], indpb=1) toolbox.register("select", tools.selTournament, tournsize=10)実行
ここまで設定すれば、あとは簡単なコマンドで動く。
下記の例では各世代の評価個体数、平均値、標準偏差、最大値、最小値を
logbookに記録していく。
hofとは、Holl of Fameの略で、評価が高い順に指定した数だけ保存する事ができる。algorithms.eaSimpleの引数cxpbは交叉率、mutpbは突然変異率、ngenは世代数を示す。
1世代あたりの個体数はpopで定義したものを引き継ぎ10×10=100個体となる。
初期サンプリングと世代交代時の個体数を別々に指定することも可能。sample_GA.py# 初期個体群を生成 pop = toolbox.population_guess() # 設定 hof = tools.HallOfFame(5, similar=np.array_equal) stats = tools.Statistics(lambda ind: ind.fitness.values) stats.register("avg", np.mean) stats.register("std", np.std) stats.register("min", np.min) stats.register("max", np.max) # 実行 pop, logbook= algorithms.eaSimple(pop, toolbox, cxpb=0.9, mutpb=0.1, ngen=20, stats=stats, halloffame=hof, verbose=True) # 上位3個体を表示 print("holl of fame.1 : %s" % (hof[0])) print("holl of fame.2 : %s" % (hof[1])) print("holl of fame.3 : %s" % (hof[2]))terminalgen nevals avg std min max 0 100 80.0665 99.3005 -83.8352 414.98 1 74 7.49092 62.2371 -103.255 301.724 2 89 11.499 108.449 -105.693 665.594 3 84 -60.283 53.2721 -106.71 161.645 4 87 -95.3896 32.299 -106.723 40.9594 5 75 -99.3516 28.4967 -106.758 23.0967 6 73 -104.068 15.7185 -106.764 4.33984 7 76 -103.476 18.6955 -106.764 10.0824 8 80 -101.665 22.5172 -106.764 16.8155 9 92 -102.631 20.6472 -106.764 26.921 10 77 -102.882 19.2791 -106.764 6.34586 11 90 -99.4555 30.4939 -106.764 56.3788 12 89 -100.566 27.1489 -106.764 30.2934 13 79 -100.978 25.2596 -106.764 51.745 14 78 -98.4071 32.1796 -106.764 85.5625 15 76 -105.728 10.3096 -106.764 -3.14868 16 89 -95.2292 38.3427 -106.764 80.6272 17 91 -102.44 25.6436 -106.764 96.6545 18 80 -105.432 11.2501 -106.764 4.33866 19 83 -102.271 23.504 -106.764 67.6912 20 79 -103.856 16.5553 -106.764 -3.86946 holl of fame.1 : [4.70021671 3.1529326 ] holl of fame.2 : [4.70021671 3.15293287] holl of fame.3 : [4.70021671 3.15293358]大域的最適解-106.765付近に収束した。
各個体の微分情報を使わないアルゴリズムなので、微小な誤差は出てしまう。まとめと所感
- 遺伝的アルゴリズムDEAPで関数の最適化を行った
- 乱数を使うため、試行ごとに収束するかどうかが怪しい
- 離散値で多変数の最適化向けか?
- 投稿日:2020-08-11T15:26:18+09:00
遺伝的アルゴリズムで関数を最適化する(その1)
進化アルゴリズムとは
進化的アルゴリズム(evolutionary algorithm, EA)とは、最適化アルゴリズムの一つで、自然界の淘汰、遺伝子組み換え、突然変異などをモデルとして組み込んだ最適化手法である。
進化的アルゴリズムのは下記の4種類に大分される。
- 遺伝的アルゴリズム
- 遺伝的プログラミング
- 進化戦略
- 進化的プログラミング今回は遺伝的アルゴリズム(genetic algorithm, GA)を扱う。
使用したライブラリ
下記のDEAPというpythonライブラリを用いる。
https://deap.readthedocs.io/en/master/コード
目的関数
下記に示すBird Bunctionの最小値を探索する。
$$f(x, y) = sin(x)e^{(1-cos(y))^2}+cos(y)e^{(1-sin(x))^2}+(x-y)^2$$
Bird Functionは多峰性の関数で、(x,y)=(4.70104, 3.15294),(-1.58214, -3.13024)に大域的最適解-106.764537、その周辺に多数の局所解を持つ。sample_GA.pyimport numpy as np def bird(x): x1 = x[0] x2 = x[1] t = np.sin(x1)*np.exp((1-np.cos(x2))**2) + np.cos(x2)*np.exp((1-np.sin(x1))**2) + (x1-x2)**2 return t, # 描画 import matplotlib.pyplot as plt from mpl_toolkits.mplot3d import Axes3D c1 = c2 = np.arange(-10, 10, 0.1) xx, yy = np.meshgrid(c1, c2) len=np.size(c1) z=np.empty((len, len)) for i in range(len): for j in range(len): xin=np.array([xx[i,j], yy[i,j]]) z[i,j], = bird(xin) fig0 = plt.figure(figsize=(12,12), dpi=80).add_subplot(111, projection='3d') fig0.plot_surface(xx, yy, z, alpha=1) plt.xlim(10, -10) plt.ylim(-10, 10)ライブラリのインポート
必要なライブラリをインポートしておく。
DEAPはrandomライブラリを使用するため、1試行ごとに結果が変化する。
これを避けるためには、random.seed(1)とすること。sample_GA.pyimport pandas as pd import random from deap import algorithms from deap import base,creator,tools # random.seed(1)初期個体群の生成
初期個体を生成する関数を作っておく。
ここでは、x, yともに-10〜10の範囲で、10×10の格子状に初期個体を生成した。sample_GA.pydef initPopulation(pcls, ind_init): x = np.linspace(-10, 10, 10) y = np.linspace(-10, 10, 10) inix, iniy = np.meshgrid(x,y) contents = np.concatenate([inix.reshape([-1,1]), iniy.reshape([-1,1])], axis=1) pcls = [ind_init(c) for c in contents] return pclstoolboxに登録
評価("evaluate")、個体群("population")、交叉("mate")、突然変異("mutate")、選択("select")等の手法をtoolboxに登録しておく。
例えば、下記の
toolbox.register("mate", tools.cxBlend, alpha=0.2)は、
tool.cxblendという関数を交叉手法として登録します、という意味(alpha=0.2はtools.cxBlendの引数)。交叉方法にブレンド交叉、突然変異にガウス変異、選択形式は10個体のトーナメントで実施する。
初期個体群を指定したい場合、"population_guess"に登録しておく。sample_GA.pycreator.create("FitnessMax", base.Fitness, weights=(-1.0,)) creator.create("Individual", np.ndarray, fitness=creator.FitnessMax) toolbox = base.Toolbox() toolbox.register("evaluate", bird) toolbox.register("population_guess", initPopulation, list, creator.Individual) toolbox.register("mate", tools.cxBlend, alpha=0.2) toolbox.register("mutate", tools.mutGaussian, mu=[0.0, 0.0], sigma=[2.0, 2.0], indpb=1) toolbox.register("select", tools.selTournament, tournsize=10)実行
ここまで設定すれば、あとは簡単なコマンドで動く。
下記の例では各世代の評価個体数、平均値、標準偏差、最大値、最小値を
logbookに記録していく。
hofとは、Holl of Fameの略で、評価が高い順に指定した数だけ保存する事ができる。algorithms.eaSimpleの引数cxpbは交叉率、mutpbは突然変異率、ngenは世代数を示す。
1世代あたりの個体数はpopで定義したものを引き継ぎ10×10=100個体となる。
初期サンプリングと世代交代時の個体数を別々に指定することも可能。sample_GA.py# 初期個体群を生成 pop = toolbox.population_guess() # 設定 hof = tools.HallOfFame(5, similar=np.array_equal) stats = tools.Statistics(lambda ind: ind.fitness.values) stats.register("avg", np.mean) stats.register("std", np.std) stats.register("min", np.min) stats.register("max", np.max) # 実行 pop, logbook= algorithms.eaSimple(pop, toolbox, cxpb=0.9, mutpb=0.1, ngen=20, stats=stats, halloffame=hof, verbose=True) # 上位3個体を表示 print("holl of fame.1 : %s" % (hof[0])) print("holl of fame.2 : %s" % (hof[1])) print("holl of fame.3 : %s" % (hof[2]))terminalgen nevals avg std min max 0 100 80.0665 99.3005 -83.8352 414.98 1 74 7.49092 62.2371 -103.255 301.724 2 89 11.499 108.449 -105.693 665.594 3 84 -60.283 53.2721 -106.71 161.645 4 87 -95.3896 32.299 -106.723 40.9594 5 75 -99.3516 28.4967 -106.758 23.0967 6 73 -104.068 15.7185 -106.764 4.33984 7 76 -103.476 18.6955 -106.764 10.0824 8 80 -101.665 22.5172 -106.764 16.8155 9 92 -102.631 20.6472 -106.764 26.921 10 77 -102.882 19.2791 -106.764 6.34586 11 90 -99.4555 30.4939 -106.764 56.3788 12 89 -100.566 27.1489 -106.764 30.2934 13 79 -100.978 25.2596 -106.764 51.745 14 78 -98.4071 32.1796 -106.764 85.5625 15 76 -105.728 10.3096 -106.764 -3.14868 16 89 -95.2292 38.3427 -106.764 80.6272 17 91 -102.44 25.6436 -106.764 96.6545 18 80 -105.432 11.2501 -106.764 4.33866 19 83 -102.271 23.504 -106.764 67.6912 20 79 -103.856 16.5553 -106.764 -3.86946 holl of fame.1 : [4.70021671 3.1529326 ] holl of fame.2 : [4.70021671 3.15293287] holl of fame.3 : [4.70021671 3.15293358]大域的最適解-106.765付近に収束した。
各個体の微分情報を使わないアルゴリズムなので、微小な誤差は出てしまう。まとめと所感
- 遺伝的アルゴリズムDEAPで関数の最適化を行った
- 乱数を使うため、試行ごとに収束するかどうかが怪しい
- 離散値で多変数の最適化向けか?
- 投稿日:2020-08-11T15:10:12+09:00
GCPのプリエンプティブルVMを自動で起動させる方法
GCPのプリエンプティブルVMを自動で起動させる方法
GCPにはPreemptible VMという料金が安い代わりに最長でも24時間で停止するVMがあります。
https://cloud.google.com/compute/docs/instances/preemptible?hl=japreemptの通知を受けて停止したVMは自動では起動してきません。
以下は、自動起動をシャットダウンスクリプトとCloud Functionsを利用して実現するツールについての説明です。Tool概要
- Preemptible VMが停止後に自動起動しないので、起動するようにするツールです。
- 手動で停止した場合は起動して来ないようにpreempt通知を受け取った場合だけ起動する仕組みになっています。
- 実行環境はpython3.7です。
Toolの動作
- VMにshutdown-scriptを設定します。VMの停止時に実行されて、preemptの通知を受け取っているかをチェック、TRUE or FALSEをファイルに書き込んでGCSにアップロードします。
- GCSのファイル更新をトリガーにしてCloud Functionsを実行します。
- Functionはpreemptの通知がTRUEだった場合のみVMを起動します。
設定手順:
ソースコードをダウンロード
git clone git@gitlab.com:cloud-mainte/auto-boot-preemtible-vm.git or git clone https://gitlab.com/cloud-mainte/auto-boot-preemtible-vm.gitGCSバケットの作成
- shutdown scriptを配置するバケットを作成して、shutdown scriptを配置する。
- スクリプト内の
BUCKET変数は適宜書き換える。$ gsutil mb -l ASIA-NORTHEAST1 gs://<backet name> $ gsutil cp shutdown-script.sh gs://<backet name>/ $ gsutil ls -l gs://<backet name>
- preempt通知を受け取ったかどうかの状態を保存するバケットを作成する。
$ gsutil mb -l ASIA-NORTHEAST1 gs://<backet name>対象のインスタンスにshutdown-scriptを設定
gcloud compute instances add-metadata <instance name> \ --metadata shutdown-script-url=gs://<backet name>/shutdown-script.shCloud Functionsにインスタンス起動プログラムを設定
- main.pyの
bucketは適宜書き替える。gcloud functions deploy boot_preemptible_instance --region=asia-northeast1 -- entry-point boot_instance --runtime python37 --trigger-event google.storage.object.finalize --trigger-resource gs://<backet name>動作確認
手動でインスタンスを停止してみる。
$ gcloud compute instances stop <instance name> $ gcloud compute instances list $ gsutil cat gs://<backet name>/preempted_status.out {"preempted_status":"FALSE", "instance_name":"<instance name>", "zone":"asia-northeast1-a", "project":"<project name>"}手動で停止した場合は
"preempted_status":"FALSE"となる。
ログの確認。$ gcloud logging read "resource.type=\"cloud_function\" AND resource.labels.function_name=\"boot_preemptible_instance\" AND resource.labels.region=\"asia-northeast1\"" --format="table(timestamp, textPayload)" |head -10 TIMESTAMP TEXT_PAYLOAD 2020-08-11T04:38:26.982711475Z Function execution took 1894 ms, finished with status: 'ok' 2020-08-11T04:38:26.979Z Preempted status is FALSE. : 2020-08-11T04:38:25.090798409Z Function execution startedPreemptの通知を模してみる。
$ cat preempted_status.out {"preempted_status":"TRUE", "instance_name":"<instance name>", "zone":"asia-northeast1-a", "project":"<project name>"} $ gsutil cp preempted_status.out gs://<backet name>/ $ gsutil cat gs://<backet name>/preempted_status.out {"preempted_status":"TRUE", "instance_name":"<instance name>", "zone":"asia-northeast1-a", "project":"<project name>"}ログの確認。
$ gcloud logging read "resource.type=\"cloud_function\" AND resource.labels.function_name=\"boot_preemptible_instance\" AND resource.labels.region=\"asia-northeast1\"" --format="table(timestamp, textPayload)" |head -12 TIMESTAMP TEXT_PAYLOAD 2020-08-11T05:02:51.640069088Z Function execution took 15066 ms, finished with status: 'ok' 2020-08-11T05:02:51.637Z Instance started. 2020-08-11T05:02:51.637Z Instance starting.. status = RUNNING 2020-08-11T05:02:46.246Z Instance starting.. status = STAGING 2020-08-11T05:02:40.862Z Instance starting.. status = TERMINATED 2020-08-11T05:02:39.373Z Instance status is now TERMINATED 2020-08-11T05:02:38.959Z Preempted status is TRUE. Boot up the instance. : 2020-08-11T05:02:36.577339221Z Function execution startedインスタンスが起動した。
$ gcloud compute instances list NAME ZONE MACHINE_TYPE PREEMPTIBLE INTERNAL_IP EXTERNAL_IP STATUS <instance name> asia-northeast1-a g1-small true XX.XX.XX.XX XX.XX.XX.XXX RUNNING
- 投稿日:2020-08-11T15:06:21+09:00
教科書に出てくる等高線を描いてみる(Python)
この記事は教科書に出てくる等高線を描いてみる(Fortan+gnuplot)の続編です.
はじめに
筆者は大学の工学部で習う流体力学を勉強しているが,流体力学の最初のほう(完全流体の理論)では速度ポテンシャル $\phi$ や流れ関数 $\psi$ といった物理量が現れ,それらの量を用いた代表的な流れの例がいくつか出てくる(二重わき出しなど). これらの量の2次元での等高線が教科書の図として載っているが,自分でも作図できたら理解が深まるような気がする.しかしながら,関数 $y = f(x)$を手軽に描画できるグラフソフト・サービスでは,2次元面でのある物理量の等高線を描くのは難しいことが多い.前回の記事ではFortran+gnuplotで描画してみたが,やはり人気があるプログラミング言語でやってみたいので,今回はPythonを用いて等高線を描いてみる.
事前準備: Pythonと必要なモジュールのインストール
Pythonはマルチプラットフォームなので各環境に合わせてインストールを行う.Pythonのインストール方法は色々あるので自分で調べて良さそうなものを選んで欲しい.インストールが難しそう or PCに色々インストールしたくない,という人にはGoogle Colaboratoryの利用をおすすめする.
Python本体以外に,モジュールとしてnumpyとmatplotlibが必要である.OSに元々入っているPythonなどを利用する場合は追加インストールする.
対象とする流れ場
前回の記事を同じ吹き出し・吸い込みについて対象とする. 原点からの距離を $r$ とすると, 3次元での速度ポテンシャル $\phi$ は以下のように表せる.
$$
\phi = -\frac{m}{r},\, r = \sqrt{x^2+y^2+z^2} \tag{1}
$$
ここで$m$は吹き出しの強さと呼ばれる係数である.2次元での等高線が描きたいので, $z=0$ として,
$$
\phi = -\frac{m}{r},\, r = \sqrt{x^2+y^2} \tag{2}
$$
を描画してみる.作業方針
Pythonのmatplotlibモジュールは各種の描画をできるので作業はPythonのみで解決する.
- numpyの機能で2次元の格子点(等間隔のマス目)を作成し,各格子点での $\phi (= \phi_{i,j})$の値を計算する.
- matplotlibの機能で等高線を描画する.
という流れでPythonスクリプトを作る.高度なことはしないので,オブジェクト指向などは考慮しない.
Pythonスクリプト
以下にスクリプト例を示す.
contour.py
import matplotlib import numpy as np import matplotlib.pyplot as plt xmin = -10.0 xmax = 10.0 ymin = -10.0 ymax = 10.0 delta = 0.1 x = np.arange(xmin,xmax,delta) y = np.arange(xmin,xmax,delta) m = 1.0 X, Y = np.meshgrid(x,y) r = np.sqrt(X**2 + Y**2) Z = -m/r fig, ax = plt.subplots(figsize=(6,6)) levels = np.arange(-1.0,0.0,0.2) cont = ax.contour(X,Y,Z,levels) cont.clabel(fmt='%1.1f', fontsize=12) plt.show()最初に$x$,$y$に座標値を入れ,$X$,$Y$が格子点に変換したものである.matplotのグラフ描画では2つの流儀があるが,Artistオブジェクトを使う方法を用いている.等高線に数値を入れられるのはmatplotlibの長所の一つなので入れておいた.
端末で上記スクリプトを実行するのは以下のとおりであり,等高線図が別ウィンドウで現れる.
python contour.py画像を保存したい場合はこの描画ウィンドウで保存(Save)ボタンを押して保存するか,スクリプト最後の
plot.show()を以下に変更する.plt.savefig('c2.png')まとめ
Pythonのmatplotlibの機能を用いて等高線を簡単に描画する方法を紹介した.今回はmatplotlibの設定に関して必要最低限のものしか設定していないが,matplotlibはグラフの隅々まで詳細に設定できるので,こだわれば教科書に載っている図を正しく綺麗に描画することも可能である.
- 投稿日:2020-08-11T15:05:15+09:00
Python初心者によるDjango超入門!その3 テンプレートファイルの継承機能を使ってみた
本記事について
UdemyでDjangoについて学習した結果のアウトプットページです。
前の記事の続きになります。
今回は、Djangoのテンプレートファイルの継承機能を使ってみようと思います。urls.py
今回はテンプレートファイルの継承機能の説明になるため、ulrs.pyは前回と同じです。
first\myapp\urls.pyfrom django.urls import path from . import views app_name = 'myapp' urlpatterns = [ path('', views.index, name='index'), ]views.py
views.pyも前回と同じですが、念のためコードは下記になります。
first\myapp\views.pyfrom django.shortcuts import render def index(request): context = { 'names':['鈴木','佐藤','高橋'], 'message':'こんにちは。', } return render(request, 'myapp/index.html', context)ベースとなるHTMLファイルを作る
継承元となるHTMLを作ります。
作成場所は、前回作ったindex.htmlの1個上のフォルダになります。
同じフォルダではないので注意してください。
作成出来たら以下のコードを記入します。
first\myapp\templates\base.html<html> <head> {% block title %} <title>継承元ページ</title> {% endblock %} </head> <body> <h1>Django勉強ページです</h1> <div> {% block body %} {% endblock %} </div> </body> </html>ポイントは2つ。
head部分に{% block title %}{% endblock %}を記入することと、
body部分に{% block body %}{% endblock %}と記入することです。
次は継承先となるindex.htmlを編集しましょう。継承先テンプレートファイル(index.html)の編集
前回の記事でも作りましたが、ファイルの場所は下記になります。
ファイルを開いて、以下のように記述します。
first\myapp\templates\myapp\index.html{% extends "base.html" %} {% block title %} <title>Django勉強ページ</title> {% endblock %} {% block body %} <p>{{ names.0 }}さん。{{ message }}</p> <p>{{ names.1 }}さん。{{ message }}</p> <p>{{ names.2 }}さん。{{ message }}</p> <hr> {% for name in names %} <p>{{ name }}さん。{{ message }}</p> {% endfor %} {% endblock %}コードの解説です。
まず、{% extends "base.html" %}で先ほど作成したテンプレートファイルを継承します。よくやるミスが間違った場所にbase.htmlを置いてしまいエラーになるケースです。続いて、
{% block title %}{% endblock %}の中にtitleタグを入れています。
これは、index.htmlページ独自のタイトルを入れる意味になります。
index.html以外にhtmlページを作成し、違うタイトルにしたい場合などに役に立ちます。
何も記入しないと、base.htmlにあるtitleが反映され「継承元ページ」と表示されます。続いて、前回作成したbody部を丸ごと
{% block body %}{% endblock %}で囲みます。
これもtitleと同じです。index.htmlページ独自のbodyを記述するという意味です。その外にもよくやるのがナビ部分を継承させたり、フッター部分を継承させたりします。
継承機能を利用することで、ページの一部に変更があっても全体に反映がさせやすくなります。
Djangoには色々な機能がありますが、継承機能は必ず利用する機能だと思います。動作確認
py manage.py runserverで開発用サーバーを起動し、動作確認します。
base.htmlにだけ記入した「Django勉強ページです」と記載され、
ページタイトルが「Django勉強ページ」と表示され、
body部が正常に表示できていれば成功です。
- 投稿日:2020-08-11T15:01:50+09:00
MATLABを使っている人がPythonを使うときの注意点
MATLABを使っている人がPythonを使うときの注意点
仕事で使用しているMATLABとPythonの注意点が書かれたサイトがあったので、備忘録として残しておきます。
・MATLAB に慣れた人が Python を始めるときの11の注意点
https://datachemeng.com/matlab_to_python/
Pythonを使い始めてハマったこと
1."="による代入は値のコピーではなく、参照になっている。意図しないデータ変更が行われる可能性あり
対策 値をコピーしたい場合は .copy()を使うようにする。
import numpy as np # ============================================================================= # b += 5 -> 元のaも変化する。 # ============================================================================= a = np.array([1,2,3]) print('a = ') print(a) print('\n') b = a b += 5 #元のaも変化する。 print('b = a , b += 5 を実行後') print('a = ') print(a) print('b = ') print(b) print('\n') # ============================================================================= # 'b = a , b = b + 5 -> 元のaは変化しない。 # ============================================================================= a = np.array([1,2,3]) b = a b = b + 5 #元のaは変化しない。 print('b = a ,b = b + 5 を実行語') print('a = ') print(a) print('b = ') print(b) print('\n') # ============================================================================= # 'b = a , b[0] = 100 ->元のaも変化する。 # ============================================================================= a = np.array([1,2,3]) b = a b[0] = 100 print('b = a ,b[0] = 100 を実行後') print('a = ') print(a) print('b = ') print(b) print('\n') # ============================================================================= # b = a.copy() b[0] = 100 ->元のaも変化する。 # ============================================================================= a = np.array([1,2,3]) b = a.copy() b[0] = 100 print('b = a.copy(),b[0] = 100 を実行後') print('a = ') print(a) print('b = ') print(b) print('\n')a = [1 2 3] b = a , b += 5 を実行後 a = [6 7 8] b = [6 7 8] b = a ,b = b + 5 を実行語 a = [1 2 3] b = [6 7 8] b = a ,b[0] = 100 を実行後 a = [100 2 3] b = [100 2 3] b = a.copy(),b[0] = 100 を実行後 a = [1 2 3] b = [100 2 3]2.配列の型変換が自動で行われない。(配列要素に対して演算を行う場合のみ)
対策 array作成時に整数でセットせず、少数で指定する。またはdtype = floatで型指定する。
どちらにしても型への注意は必須。
- 投稿日:2020-08-11T14:42:14+09:00
E検定取得に向けてラビットチャレンジに参加
- 投稿日:2020-08-11T13:49:53+09:00
グラフを3Dで見てみたい!そんな夢かなえます。
見てみたいよね?
三角関数、対数関数、サイクロイド…数えたらきりがないほどにこの世界ではいろんな関数や図形、グラフがあります。
そんなグラフを3Dで見てみたいですよね?で一体どうするってのさ?
今回はBlenderとExcelを使ってやっていきたいと思います。
Excelファイルに座標のデータを入れてそれを基にBlenderでオブジェクトを生成します。
では仕組みが分かったところで…
早速作ってくか
開発環境
windows 10
python 3.7
Blender 2.8
pip 20.2.1Excelでデータを作成する
まずはExcelファイルをいじいじするライブラリをインストールしましょう。
$pip install openpyxlコードはまぁざっとこんな感じですね
function_3Dgraph.pyimport openpyxl import math class funcdate(): def create_wb(self,wb_name): new_file=openpyxl.Workbook() #Excelファイルのデータを作成します。 new_file.save(wb_name+'.xlsx') #いったん保存します。 print('Excelファイルが出来ました') return new_file def write_wb(self,cell_name,cell_value,file_name): file=openpyxl.load_workbook(file_name+'.xlsx',data_only=True) #Excelファイルを読み込みます。 new_wb=file.active #シートを読み込みます。 new_wb[cell_name]=cell_value #cell_mane(例えばA1)セルに値を入れます。 def write_func(self,t,file_name): file=openpyxl.load_workbook(file_name+'.xlsx',data_only=True) new_wb=file.active print(new_wb) for i in range(1,t+1): if(i==0): i=1 x=math.cos(i*math.pi/180) #ここの式は何でもいいです。 y=math.sin(i*math.pi/180) #ここの式は何でもいいです。 new_wb['A'+str(i)]=i #各座標にデータを入れていきます。 new_wb['B'+str(i)]=x new_wb['C'+str(i)]=y print(new_wb['A'+str(i)].value,new_wb['B'+str(i)].value,new_wb['C'+str(i)].value) else: file.save(file_name+'.xlsx') print('記入出来ました') def get_func(self,file_name,t): file=openpyxl.load_workbook(file_name+'.xlsx',data_only=True) wb=file.active graph_t=wb['A'+str(t)].value #各座標にそれぞれのセルの値を入れます。 graph_x=wb['B'+str(t)].value graph_y=wb['C'+str(t)].value a=[graph_t,graph_x,graph_y] return a test=funcdate() test.create_wb('test_function') test.write_func(360,'test_function') print(test.get_func('test_function',10))ではさっそく実行してみましょう。
$python function_3Dgraph.py実行したらExcelファイルが出来上がってるはずなので、開いてみてください。
こんな感じで表に入っていたらOKです。
ちなみにグラフにするとこんな感じです。
・ x軸y軸を分けたもの
・ x軸y軸を合わせたもの
Blenderでグラフを作る
まずはBlenderをダウンロードしましょう。
Blenderをダウンロード
ダウンロードできたら起動して、pythonエディターを開きましょう。
↓ここですね
そうしたら下のコードを打ってみてください。最初に生成されているキューブを削除(xキー)しとくと見やすいですね。$import bpy $bpy.ops.mesh.primitive_uv_sphere_add(segments=32, ring_count=16, radius=1.0, calc_uvs=True, enter_editmode=False, align='WORLD', location=(0.0, 0.0, 0.0), rotation=(0.0, 0.0, 0.0))ではさっそく追記しましょう
function_3Dgraph.pyimport openpyxl import math import bpy class funcdate(): def create_wb(self,wb_name): new_file=openpyxl.Workbook() new_file.save(wb_name+'.xlsx') print('Excelファイルが出来ました') return new_file def write_wb(self,cell_name,cell_value,file_name): file=openpyxl.load_workbook(file_name+'.xlsx',data_only=True) new_wb=file.active new_wb[cell_name]=cell_value def write_func(self,t,file_name): file=openpyxl.load_workbook(file_name+'.xlsx',data_only=True) new_wb=file.active print(new_wb) for i in range(1,t+1): if(i==0): i=1 x=math.cos(i*math.pi/180) y=math.sin(i*math.pi/180) new_wb['A'+str(i)]=i new_wb['B'+str(i)]=x new_wb['C'+str(i)]=y print(new_wb['A'+str(i)].value,new_wb['B'+str(i)].value,new_wb['C'+str(i)].value) else: file.save(file_name+'.xlsx') print('記入出来ました') def get_func(self,file_name,t): file=openpyxl.load_workbook(file_name+'.xlsx',data_only=True) wb=file.active graph_t=wb['A'+str(t)].value graph_x=wb['B'+str(t)].value graph_y=wb['C'+str(t)].value a=[graph_t,graph_x,graph_y] return a test=funcdate() test.create_wb('test_function') test.write_func(360,'test_function') for i in range(1,360): bpy.ops.mesh.primitive_uv_sphere_add(segments=32, ring_count=16, radius=1.0, calc_uvs=True, enter_editmode=False, align='WORLD', location=test.get_func('test_function',i), rotation=(0.0, 0.0, 0.0))bpyライブラリはBlenderでしか使えないのでBlenderで実行します。
とその前に
Blenderでopenpyxlライブラリを使えるようにしときましょう。Blenderでopenpyxlを使うために管理者として起動してください。(権限ないと言われるので…)
先程作ったファイルを実行するためにBlenderをScriptモードにします。
これでも行けます。
そうしたらfunction_3Dgraph.pyを開いて実行してください。
グラフが出てきたら成功です!
(Blenderの1mは小さいので分かりづらいですがちゃんと振幅が1のsin波になっています。振幅を10にすると分かりやすくなります。)
とまぁこんな感じでグラフが完成しました。
ちなみにBlenderで作成したExcelファイルは C:\Program Files\Blender Foundation\Blender 2.83 に保存されます。
余談
ここでちょっと数学的な話を…
今回は媒介変数tでxとyを表してグラフを作りました。式にするとこんな感じですね。\left\{ \begin{array}{ll} x=f(t) \\ y=g(t) \end{array} \right.これで$y=f(x)$の3Dのグラフが出来ます。また$t=z$とすれば、$ax=by=cz(a,b,cは任意の定数)$と三次元の直線もできるわけです。
…ここまでは直行形式のグラフの話です。では極形式の時はどうしましょう?
たとえばこんな式があったとします。r=f(θ)この式で表されている直線を横軸(x軸)と縦軸(y軸)にそれぞれ分解します。
\left\{ \begin{array}{ll} x=f(θ) \cos(θ)\\ y=f(θ) \sin(θ) \end{array} \right.これで極形式のグラフが書けます。
実はここまでの事ならわざわざExcelファイルを使わく手もよいのです。何なら使わないほうがらkこのような式が分からないグラフの時に使います。Excelファイルに値が入ってる場合はそれを参考にしてグラフを作ればよいのです。
やや縮んでる気がしますがまぁ大丈夫でしょう。値をとるときにn倍にすれば問題ないですし…あとがき
今回はExcelを使ってBlenderで3Dグラフを作ってみました。次はこのグラフをアニメみたいに動かしてみたいですね。